基于高通量测序技术比较不同来源原料乳中的细菌特征
2021-06-04周继福王娉郭佳赵晓美陈颖
周继福,王娉,郭佳,3,赵晓美,陈颖
(1.中国检验检疫科学研究院,北京 100176)(2.南京财经大学食品科学与工程学院,江苏南京 210023)(3.天津科技大学食品科学与工程学院,天津 300457)
原料乳是乳腺直接分泌、不做加工处理且富含蛋白质、碳水化合物、脂肪酸和多种微量营养素的生鲜乳,也是唯一能为新生哺乳动物提供足量营养物质的食物[1]。研究表明,原料乳已成为病原微生物传播的重要途径,Mungai等[2]发现美国2007~2012年非巴氏杀菌乳中病原菌引起的疾病暴发率是1993~2006年的三倍。美国及欧洲多国已制定相关原料乳饮食指南[3,4],我国也制定颁布了GB 19301-2010《生乳》等一系列国家标准保证原料乳的质量安全,规定了生乳中微生物、真菌等的限量及检测标准,原料乳中微生物的检测对奶牛健康状况监测、规范挤乳操作和牛奶质量监管有积极作用。目前用于原料乳中微生物鉴定的技术和方法包括流式细胞术[5],酶联免疫吸附测定技术[6],平板培养法,基于PCR技术的(GTG)5-PCR指纹图谱[7]、结合激光诱导荧光的微芯片电泳(microchip electrophoresis coupled with laser-induced fluorescence,MCE-LIF)[8]和环介导等温扩增[9]等方法以及生化传感器[10],但这些依赖于微生物培养和仪器的特异性、稳定性。Carvalho等[11]认为实验室条件下可培养的细菌不足1%。因此亟需采用不依赖于培养的方法对原料乳中的微生物多样性进行探究。
基于高通量测序技术的宏基因组学技术可对生态位中提取的所有微生物DNA进行测序和分析,鉴定细菌种类和表征群落丰度。同时,技术的发展和成熟也使得高通量测序更多的用于乳品微生物群落分析,Dalmasso等[12]利用高通量测序技术分析奶酪中的微生物群落动态,Zhang等[13]运用高通量测序对不同产地山羊奶微生物多样性进行了研究。
本研究采用高通量测序技术分析不同地区、不同季节原料乳中微生物多样性,比较不同来源原料乳之间的细菌群落结构和相对丰度的差异,明确优势菌群和显著性差异种属,并对细菌功能基因代谢途径进行预测,为原料乳质量和安全性提供相关的微生物信息。
1 材料与方法
1.1 材料与试剂
原料乳样品为本实验室采集,分别来自北京、河北、内蒙古、天津四地,原料乳奶槽车载量约为30 t。
QIAamp®Fast DNA Stool Mini kit,德国Qiagen公司;Qubit3.0 DNA检测试剂盒,美国life公司;2×TaqMaster Mix,南京Vazyme公司;MagicPure®Size Selection DNA Beads,北京全式金生物技术(TransGen Biotech)有限公司。
1.2 仪器与设备
高速冷冻离心机,德国D-37520 Osterode;Qubit®3.0荧光计,美国Invitrogen公司;聚合酶链式反应(polymerase chain reaction,PCR)仪,美国BIO-RAD公司;凝胶成像系统,美国UVP公司;IlluminaMiSeq测序系统,美国Illumina公司。
1.3 方法
1.3.1 样本采集

表1 原料乳主要信息表Table 1 Sampling information of raw milk
23份原料乳样品收集自北京(BJ,n=11)、河北(HE,n=6)、内蒙古(NM,n=5)和天津(TJ,n=1)四地,采样时间跨夏(S,n=4)、秋(F,n=12)、冬(W,n=7)三季(表1),采样时严格按照工厂微生物检测要求进行,冷藏条件下送至实验室在-20 ℃下储存并尽快完成细菌基因组DNA提取。
1.3.2 基因组DNA提取
使用Fast DNA Stool Mini kit提取原料乳中细菌DNA,略作修改,将离心获得的沉淀中加入缓冲液(InhibitEX buffer)的体积从1000 μL减少为200 μL,以保证最终提取的DNA浓度,其余操作步骤按照试剂盒说明书进行。提取的DNA低温(-20 ℃)储存待用。
1.3.3 高通量测序
利用Qubit荧光计对基因组DNA定量以确定PCR扩增体系中模板DNA的量,扩增区域为细菌16S rRNA 基因的 “ V3-V4 ” 区,引物为 341F:5’-CCTACGGGNGGCWGCAG-3’ , 805R :5’-GACTACHVGGGTATCTAATCC-3’[14]。扩增产物经过琼脂糖凝胶电泳检测后使用核酸纯化试剂盒对DNA进行纯化回收,Illumina MiSeq测序平台用于完成高通量测序。
1.3.4 生物信息和数据统计分析
对23份原料乳样品进行测序,获得的原始图像数据经过碱基识别后形成FASTQ格式的测序序列。剔除引物、接头序列、PCR过程产生的错误序列以及低阈值序列。依据Filtered序列间的距离和相似性,采用软件Usearch(version 5.2.236)进行聚类形成操作分类单元(operational taxonomic units,OTU),聚类后的OTU常在97%的相似度下用于生物信息统计分析。Alpha多样性指数用以衡量微生物群落多样性和分布丰度。使用RDP classifier(version 2.12,数据库:http://rdp.cme.msu.edu/misc/reso urces.jsp)和Blast分类方法对OTU分类序列进行物种丰度计算,获得OTU分类序列矩阵并绘制群落结构分布柱状图。根据矩阵信息利用统计检验筛选差异物种分类单元,结果以误差线图表示。此外,为比较多样本间的差异,进行Beta多样性分析。最后,基于京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)对原料乳的细菌功能基因进行预测。统计软件包括STAMP(version 2.1.3)和Excel,当p<0.05时认为具有统计学意义。
2 结果与分析
2.1 高通量测序信息
对高通量测序数据处理后共获得1 722 199条高质量序列,质控后的序列平均长度为421.17 bp。OTU微生物物种分类结果显示,原料乳中微生物共分为33门,64纲,117目,254科,732属。
2.2 OTU聚类和分类注释
为获得样品中微生物群落结构组成信息,对高通量测序序列进行归类。韦恩图(图1)直观展示微生物共有或独特的OTU数目,北京、内蒙古、河北、天津四地的OTU数分别为6787、3856、3876、251;独有OTU数目分别为3020、981、935、52,说明四地区中,北京地区乳样中微生物群落丰度最高。四地乳样共有OTU数为70,北京和天津共有OTU数为178,北京和内蒙古共有OTU数为2368,北京和河北共有OTU数为2441,内蒙古和河北共有OTU数为1597,说明四地区中,北京和河北的乳样最相似。夏、秋、冬三季的OTU数分别为2193、5254、6132,三个季节样品共有OTU数为707,表明冬季乳样中微生物群落丰度高于夏季和秋季;夏季和秋季共有的OTU数为1096,夏季和冬季的共有OTU数为1142,秋季和冬季的共有OTU数为2780,说明秋季和冬季的乳样更相似。
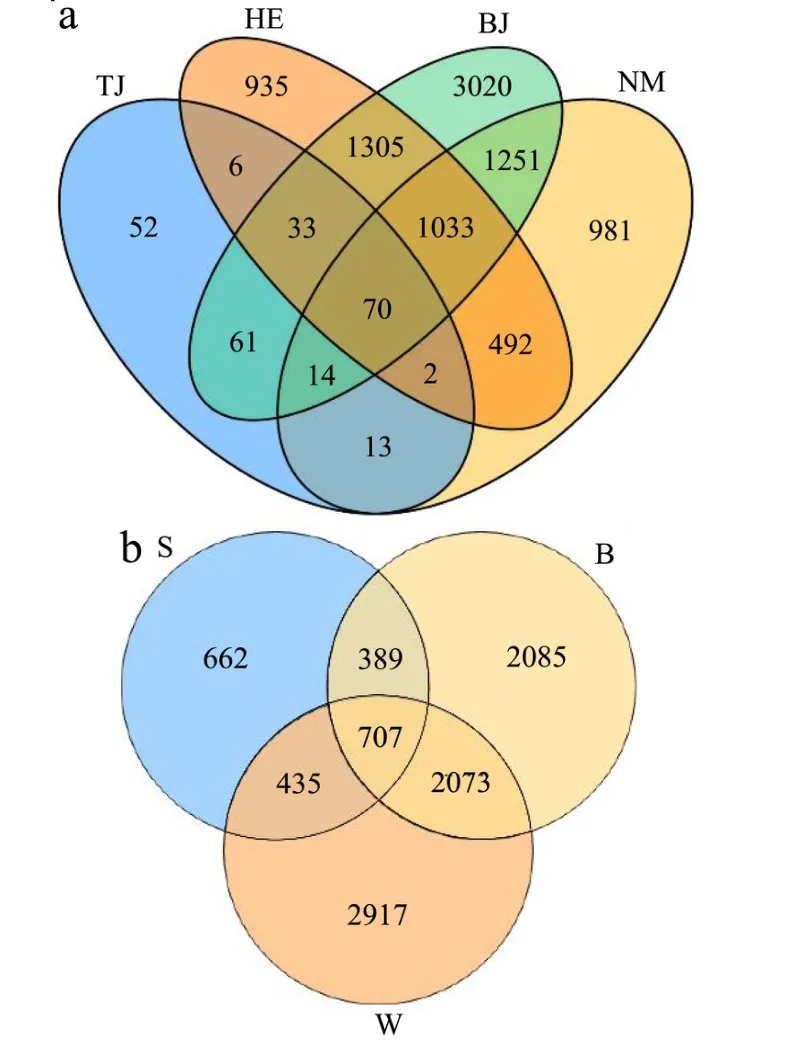
图1 不同来源原料乳中细菌OTUs分布韦恩图Fig.1 Venn diagram of bacterial OTUs distribution in raw milk from different sources
使用物种分类方法统计门(phylum)、属(genus)层次上的原料乳微生物群落组成,绘制门、属水平上的原料乳微生物群落结构分布柱状图,绘图时将门、属分类水平上相对丰度<0.50%和<5.00%的细菌群落及unclassified划分为其他菌属(others)[15],以直观反映原料乳中主要微生物数量及群落构成。
在门分类水平上的原料乳中微生物细菌菌门和相对丰度各不相同,图2显示所有乳样中均含有变形菌门(Proteobacteria),厚壁菌门(Firmicutes),拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),平均相对丰度分别为76.54%,11.17%,7.10%和4.56%。此外,所有原料乳中还含有 Candidatus Saccharibacteria,绿弯菌门(Chloroflexi),芽单胞菌门(Gemmatimonadetes),异常球菌属-栖热菌门(Deinococcus-Thermus),柔壁菌门(Tenericutes)和梭杆菌门(Fusobacteria)。Nazmul等[16]在乳腺炎乳样和健康乳样中均发现四种菌门的存在,Rozo等[17]在原料乳制成的奶酪中也得到同样的结果。此外,Sadan等[18]发现牛瘤胃中主要的菌群为厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),而瘤胃是分解饲料的主要场所,这间接说明厚壁菌门和拟杆菌门为原料乳中的常见微生物群落。
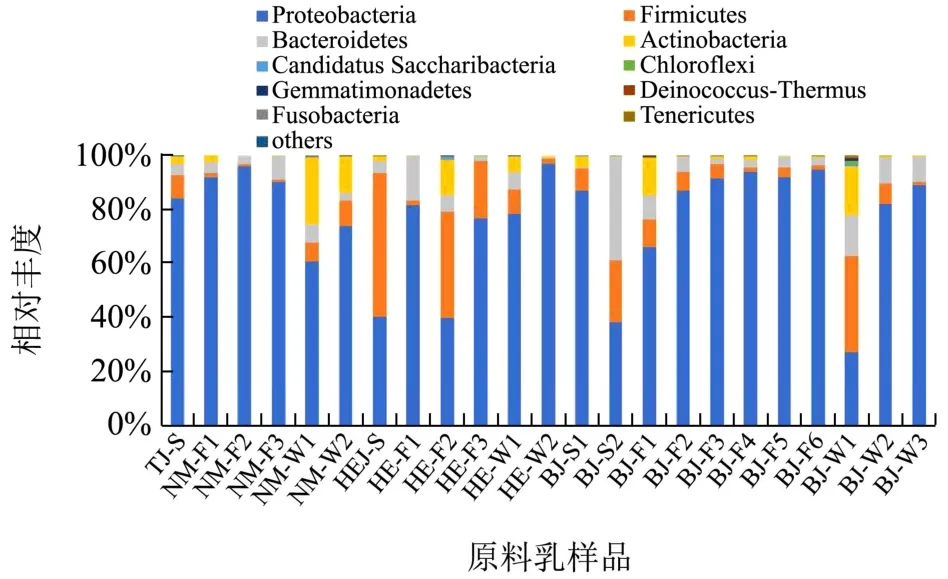
图2 基于门水平的原料乳细菌群落分布柱状图Fig.2 Barplot of bacterial community distribution in raw milk based on phylum level
从样品地区来源看,四个地区原料乳样品中细菌主要来自于变形菌门,其相对丰度在68.99%~84.08%之间,拟杆菌门和放线菌门的细菌相对较少,拟杆菌门的相对丰度在10.00%以下,放线菌门的相对丰度则在5.00%以下;河北地区原料乳样品中的厚壁菌门细菌的相对丰度比其他三个地区的均高2倍以上,达到20.99%。河北地区和内蒙古地区乳样中拟杆菌门和放线菌门细菌相对含量相似。从原料乳采样季节来看,三个季节原料乳中细菌主要为变形菌门,平均相对丰度在62.54%~83.54%之间,夏季原料乳中厚壁菌门细菌的相对丰度是秋、冬两季的2倍以上,为23.23%,三个季节的原料乳中拟杆菌门和放线菌门细菌的平均相对丰度较低,放线菌门的平均相对丰度在10.00%以下。冬季原料乳中放线菌门的相对含量高于拟杆菌门。夏季原料乳中拟杆菌门的相对丰度是秋季的2倍左右。除此之外,原料乳中相对丰度较低的CandidatusSaccharibacteria,绿弯菌门,芽单胞菌门,异常球菌属-栖热菌门,柔壁菌门和梭杆菌门在内蒙古地区的秋季乳样中均未发现。柔壁菌门在河北的冬季乳样中不存在,这表明各地区在不同季节原料乳微生物群落结构组成上存在差异。
在属分类层次上(图3),原料乳主要的菌属为假单胞菌属(Pseudomonas),不动杆菌属(Acinetobacter),黄杆菌属(Flavobacterium),水栖菌属(Enhydrobacter),乳球菌属(Lactococcus),链球菌属(Streptococcus),苯基杆菌属(Phenylobacterium),耶尔森氏菌属(Yersinia),气单胞菌属(Aeromonas),考克氏菌属(Kocuria),罗斯氏菌属(Rothia),柠檬酸杆菌属(Citrobacter),葡萄球菌属(Staphylococcus),Epilithonimonas和Rheinheimera。其中假单胞菌属(Pseudomonas),不动杆菌属(Acinetobacter),黄杆菌属(Flavobacterium)和水栖菌属(Enhydrobacter)为原料乳共有菌属,平均相对丰度分别为32.02%,28.53%,5.35%和3.81%。假单胞菌属(Pseudomonas)和不动杆菌属(Acinetobacter)的平均相对丰度之和占总微生物数的60.55%,为原料乳中两种优势菌属,该结果与已有报道类似[13,19],于国萍等[20]发现不同牧场原料乳的主要菌属中包含有不动杆菌属和乳球菌属。

图3 基于属水平的原料乳细菌群落分布柱状图Fig.3 Barplot of bacterial community distribution in raw milk based on genus level
按照不同地区分析发现,除天津地区外,其余三地区原料乳中的优势菌属均是假单胞菌属,相对丰度在30.25%~41.42%之间。不动杆菌属作为天津地区乳样中的主要菌属,相对丰度为43.62%,明显高于其余三地区原料乳中不动杆菌的丰度。除上述两种菌属外,北京地区乳样中还包含相对丰度为6.03%的黄杆菌,河北地区乳样中包含相对丰度为12.99%和6.09%的乳球菌属和链球菌属,这些在内蒙古地区的乳样中均不存在。此外,葡萄球菌属和Rheinheimera也只在天津地区乳样中存在,相对丰度分别为6.84%和5.65%。河北和内蒙古原料乳中均不存在柠檬酸杆菌属,北京、河北两地的冬季乳样中也不存在气单胞菌属。从不同季节来看,三个季节的原料乳样品中均含有假单胞菌属和不动杆菌属,除夏季乳样中的假单胞菌属为7.53%外,其余季节的两种菌属的相对丰度均在22.95%以上。此外,夏季原料乳中还存在乳球菌属和黄杆菌属,相对丰度分别为16.82%和9.89%。
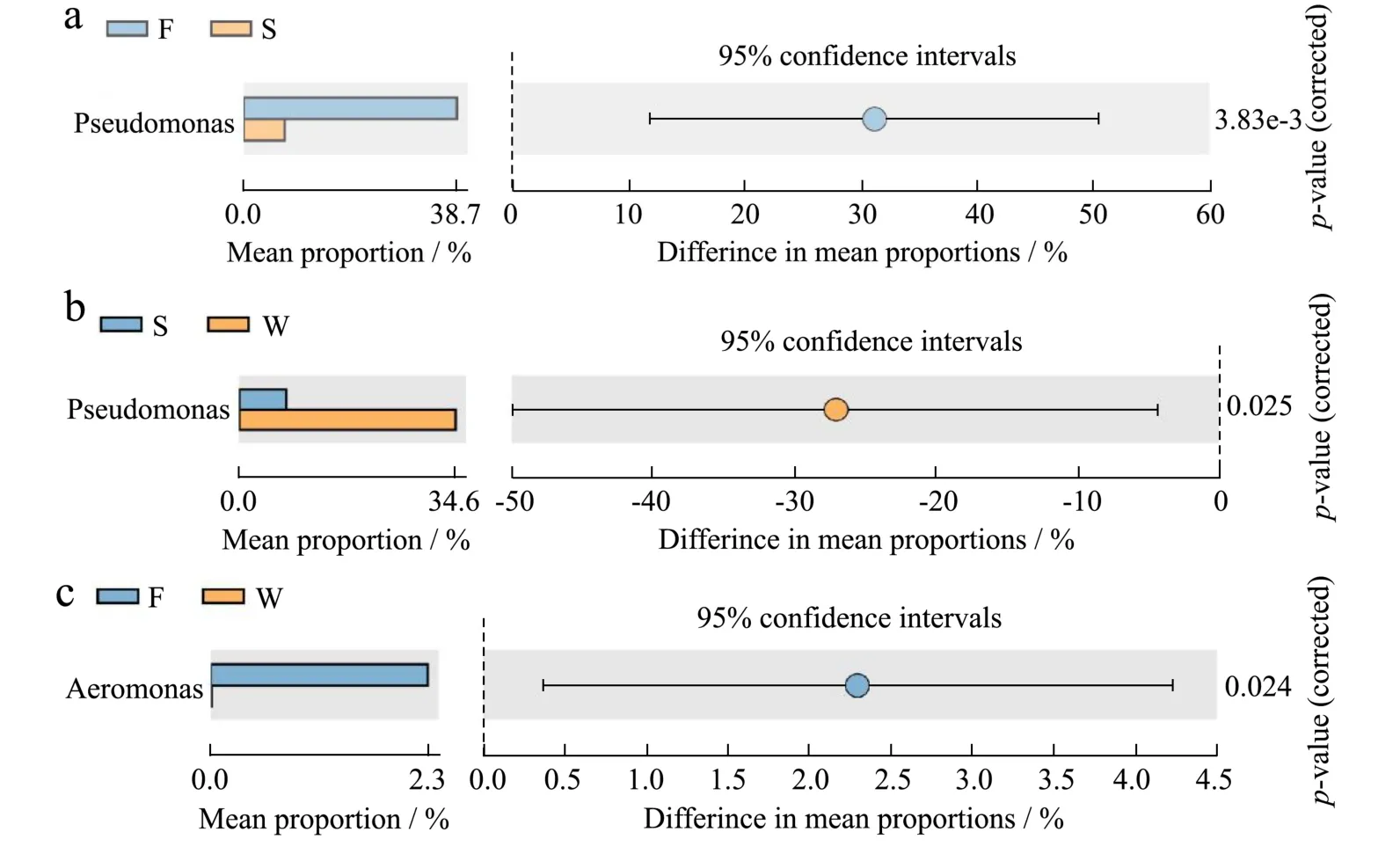
图4 不同季节原料乳中微生物丰度误差线图Fig.4 Extended error bar of microbial abundance in different seasons
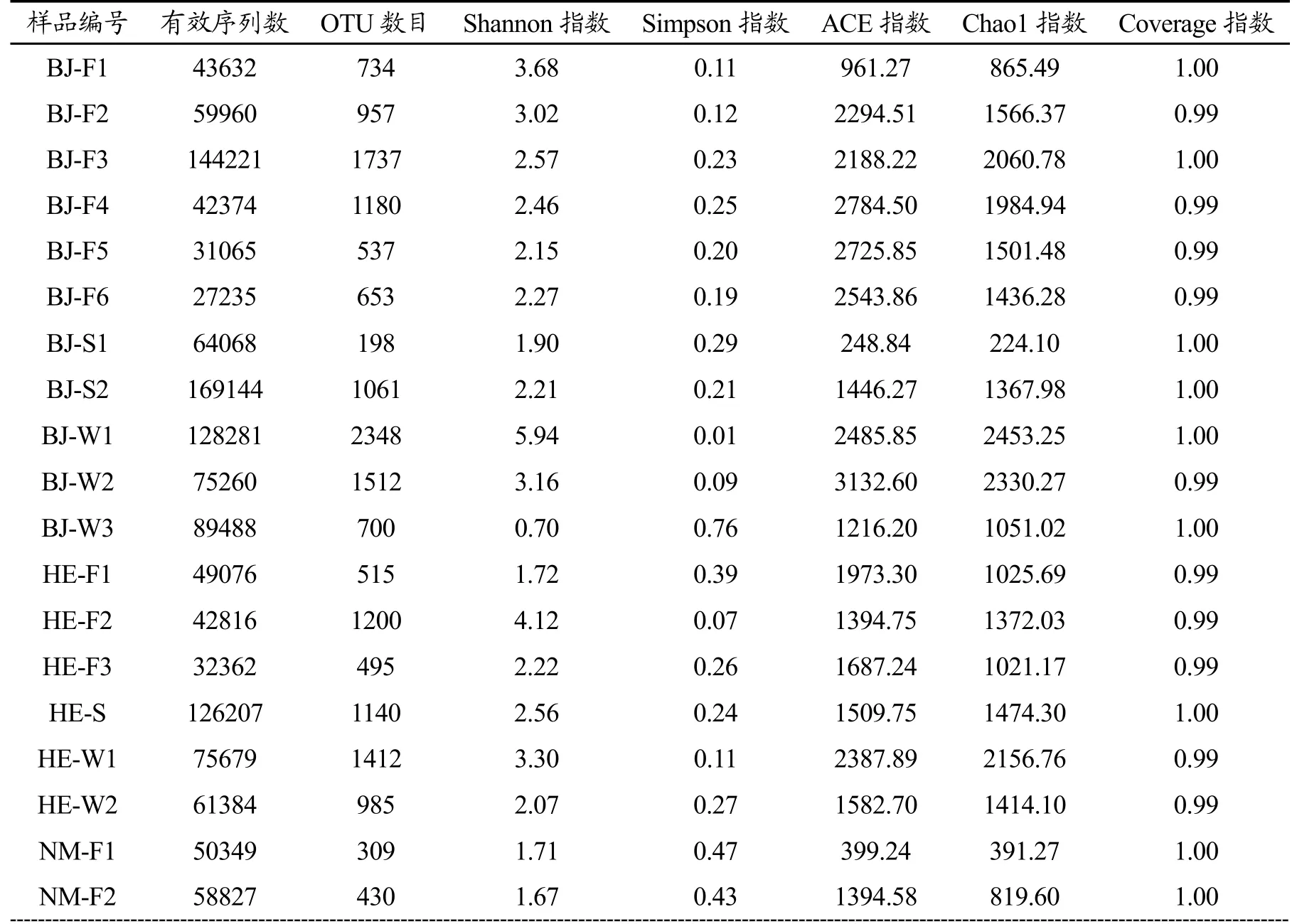
表2 原料乳样品主要测序信息及α多样性指数Table 2 Main sequencing information and α-diversity index of raw milk samples

注:BJ:北京地区;HE:河北地区;NM:内蒙古地区;TJ:天津地区;S:夏季; F:秋季;W:冬季。
有研究表明,原料乳在冷藏条件下储存一天,嗜冷菌的数量可以急剧增长到总微生物数量的90.00%以上[21,22],这或许是假单胞菌属和不动杆菌属能成为原料乳优势菌属的原因之一,实验中分析发现的主要嗜冷菌包括假单胞菌属,不动杆菌属,气单胞菌属,黄杆菌属,乳球菌属,葡萄球菌属和链球菌属[23,24]。嗜冷菌除了产生影响原料乳质量的酶以外,还可能存在潜在的致病性和抗生素耐药性[21,Danmallam等[25]在患乳腺炎奶牛的牛奶中发现葡萄球菌属细菌的存在。Decimo等[26]发现散装牛奶中分离的假单胞菌属对氨曲南展现出耐药性。
根据采样地区和采样季节对原料乳中微生物相对丰度进行显著性差异分析,不同地区微生物群落虽占比不同,但并未体现出显著性差异。而对不同季节原料乳中微生物群落相对丰度差异比较发现(图4),夏秋、夏冬季节的假单胞菌属(Pseudomonas)相对丰度存在显著性差异(p<0.05)(图4a,4b),且差异比例超过30.00%。秋冬季节的气单胞菌属(Aeromonas)相对丰度也存在显著性差异(p<0.05)(图4c)。
2.3 Alpha多样性分析
Alpha多样性既可以表示每个样品微生物群落丰度,也能表示微生物群落多样性,其衡量方法往往以指数的形式呈现(表2)。利用软件Mothur(version 1.30.1)建立的Coverage指数范围在0.99~1.00之内,表明此次高通量测序可以真实反映原料乳中的微生物群落分布信息。对不同季节原料乳群落分布丰度指数(Chao和ACE)进行统计分析,发现冬季乳样的丰度指数差异显著高于夏季原料乳(p<0.05)。BJ-S1,NM-F1和TJ-S乳样群落分布丰度指数低于其他同地区同季节样品,这说明微生物物种总数可能受牧场规模、奶牛自身状况以及季节等影响。
微生物群落分布多样性指数(Shannon和Simpson)中,Simpson指数越小代表物种多样性越高。对比不同季节、不同产地原料乳的多样性指数并未发现显著性差异。收集自冬季的两份北京地区乳样(BJ-W1和BJ-W3)的Shannon指数出现极端的大小差异,分别为5.94和0.70,这说明同一地区相同季节的原料乳微生物群落多样性可能和牧场卫生管理、奶牛自身健康、泌乳后操作及菌落总数的高低相关[27]。
2.4 Beta多样性分析

图5 基于UniFrac的原料乳中细菌主坐标图Fig.5 Principal coordinates of bacteria in raw milk based on UniFrac
Beta多样性可用于分析原料乳样本在特定的进化谱系中的微生物群落差异。使用UniFrac对Beta多样性进行评估分析,图5显示主坐标成分1(PCoA1)和主坐标成分2(PCoA2)的贡献率分别为34.00%和17.00%。图中不同地区和不同季节原料乳样品的聚类趋势较弱,这说明不同地区和不同季节乳样中的细菌群落差异不明显。
2.5 功能预测分析
基于KEGG数据库,采用PICRUSt软件(version 1.0.0)对原料乳中微生物的功能基因代谢途径进行对比预测。结果分析发现:主要的功能基因相关代谢途径为氨基酸代谢(amino acid metabolism),碳水化合物代谢(carbohydrate metabolism)和膜运输(membrane transport)(图6)。氨基酸代谢及碳水化合物代谢的丰度较高可能同原料乳中氨基酸和碳水化合物的高含量[13]及其生物功能相关,而膜运输则可能与微生物生长活动所需的酶等物质的运输相关[28]。氨基酸在原料乳的蛋白质、脂肪和乳糖生物合成中发挥重要作用,这也是氨基酸代谢活跃的一个原因。包括乳球菌和链球菌在内的微生物对乳糖的利用也促进了原料乳中碳水化合物的代谢。Zhang等[13]认为乳的组成成分和微生物群落存在某种潜在联系。吴建民等[29]发现奶牛瘤胃中的微生物通过增加氨基酸代谢对乳蛋白进行调节。有报道称,细胞信号转导(cell communication)[30]和心血管疾病(cardiovascular diseases)[31]等代谢途径曾在牛奶微生物中发现。但本研究中并未发现感觉系统(sensory system),细胞信号转导甚至心血管疾病等代谢途径。
在对代谢途径的丰度差异分析中发现了一些同人类疾病相关的代谢途径,并且在部分产地和季节样品中存在显著性差异。其中,北京-内蒙古、河北-内蒙古乳样中的微生物在神经退行性疾病(neurodegenerative diseases),循环系统(circulatory system)和癌症(cancers)的功能丰度上存在显著性差异(p<0.05),神经退行性疾病在内蒙古样品中的平均差异比例略高于北京、河北两地。夏秋、夏冬季节样品在神经系统(nervous system),内分泌系统(endocrine system)的功能丰度上也存在显著性差异(p<0.05)。这可能是原料乳中某些微生物和病原微生物有同源基因所导致的[28]。另外,夏秋、夏冬季节样品在核苷酸代谢(nucleotide metabolism),聚糖的生物合成与代谢(glycan biosynthesis and metabolism)和遗传信息处理(genetic information processing)也存在显著性差异(p<0.05)。
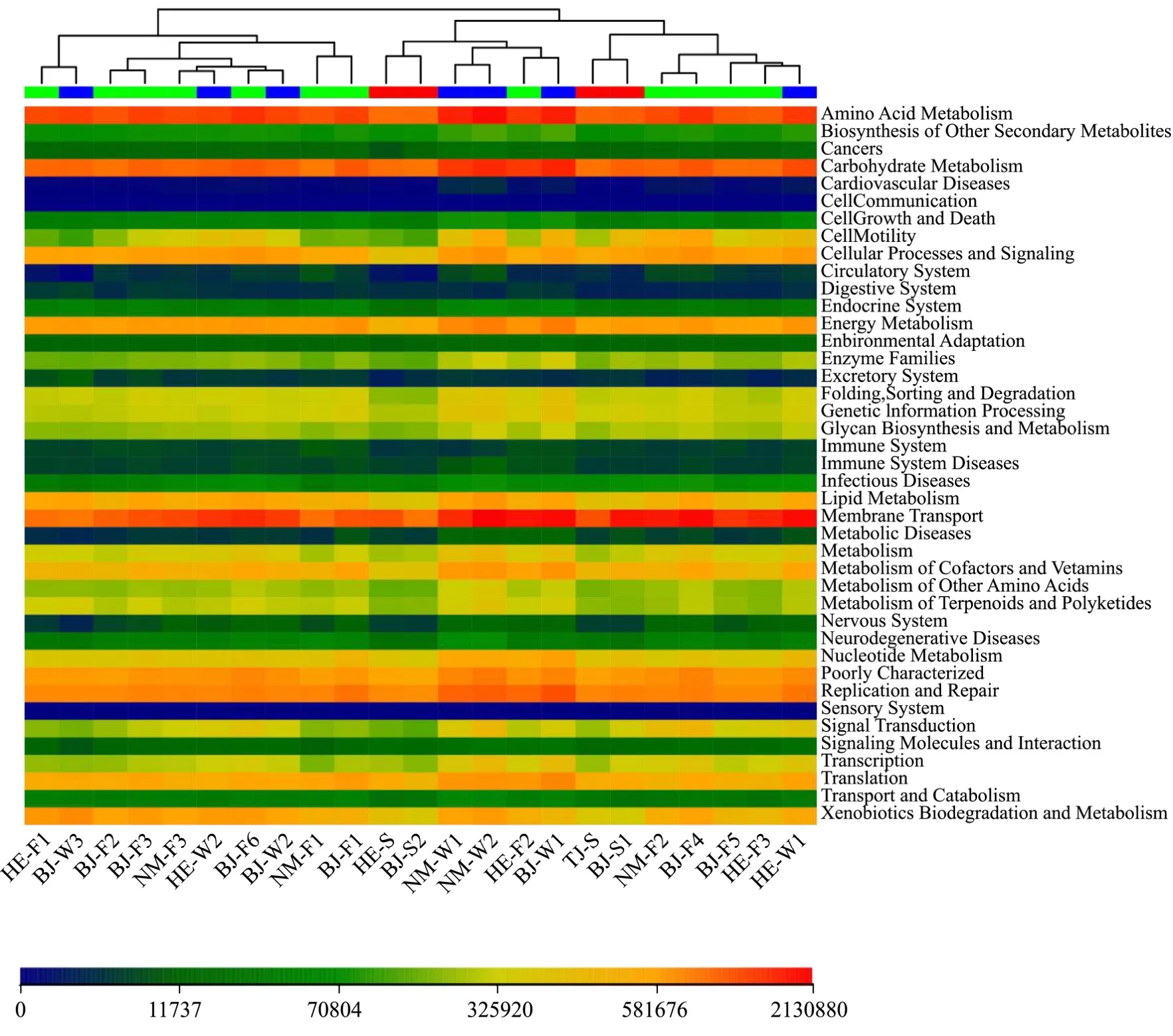
图6 不同来源原料乳中微生物功能丰度热图Fig.6 Heatmap of microbial functional abundance of raw milk from different sources
3 结论
本实验采用高通量测序技术对原料乳中微生物群落进行分析,通过探究不同采样季节、不同生产地区原料乳的优势菌群及丰度差异,并对功能基因代谢途径进行分析,以期为原料乳品质及后续加工提供相关微生物信息。结果显示,23份原料乳的优势菌门为变形菌门和厚壁菌门,优势菌属除假单胞菌属和不动杆菌属外,还包括乳球菌属和链球菌属等其他主要菌属。在门水平上,不同季节和地区原料乳菌群相对丰度比例虽然不同,但是并未体现出显著差异。在属水平上,不同季节和地区原料乳中部分微生物相对丰度存在显著性差异(p<0.05)。在功能预测分析中,氨基酸代谢、碳水化合物代谢和膜运输为原料乳细菌主要代谢途径,而研究表明这与微生物群落有关。研究并掌握原料乳中微生物组成,对于原料乳的后续生产和品质评价具有重要意义。
