平榛PYL基因家族全基因组鉴定及果实发育表达分析
2021-05-16张兴政赵豫川刘剑锋
张兴政,赵豫川,陈 玥,孙 博,刘剑锋
(吉林师范大学生命科学学院∕吉林省植物资源科学与绿色生产重点实验室,吉林四平 136000)
【研究意义】平榛(Corylus heteropylla),山毛榉目(Fagales),桦木科(Betulaceae),榛属(Corylus)植物,其繁殖方式主要通过风媒授粉,因其果实种仁富含油脂、蛋白、维生素和膳食纤维等营养物质,是一种重要经济林木[1]。榛子分布范围较广,在欧洲、亚洲和北美洲均有分布,在我国尤以东北地区为主,榛果也是东北重要特色食品之一。近年来,我国的榛子的栽培面积和产量也在不断增加[2],对东北等地方经济的发展有着重要的影响。因此,对于榛子果实发育分子机制的深入研究将有助于后续辅助榛子品种的选育,利于榛子的高质量生产。【前人研究进展】植物激素脱落酸(Abscisic acid,ABA)在植物种子成熟、休眠、生长及幼苗生长等各个生长发育过程中均发挥重要作用,也与植物的高盐、干旱、水涝等非生物胁迫响应也密切相关[3-5]。ABA对植物的调控是通过ABA信号转导途径来实现的,研究发现,PYR∕PYL∕RCAR(Pyrabactin Resistance1∕PYR1-like∕Regulatory Component of ABA Receptor),能够通过与胁迫诱导的ABA结合,从而参与植物体内ABA信号转导过程,因此被认为是一种ABA可溶性受体[6]。ABA通过结合Pyra‐bactin resistance 1-like(PYL)后与Protein Phosphatases 2C(PP2C)进一步结合形成ABA-PYL-PP2C复合体,并抑制PP2C蛋白的磷酸酶活性[7],进而与PP2C结合的Sucrose non-fermenting 1-related Protein Ki‐nase 2s(SnRK2s)被释放,调控下游效应蛋白实现ABA信号转导以参与植物发育或胁迫响应调控[8-9]。目前,在拟南芥[10-12]、水稻[13-14]、大豆[15]、葡萄[16-17]和苹果[18]等植物中均鉴定获得该基因家族成员,部分基因其生物学功能也进行了深入研究。总体上,对基因响应非生物胁迫研究较多,例如,过表达OsPYL10、Os-PYL6均能够显著提高转基因植株的耐旱性[19-20],且前者还能提高植株的耐寒性;异源表达野葡萄VyPYL9则能提高转基因拟南芥的耐旱性[21],相较而言,对植物生长发育调控领域研究较少。拟南芥中共鉴定得到14个PYLs基因家族成员,且研究发现部分基因存在功能冗余,导致部分单基因突变不影响其对ABA敏感性,但多基因成员突变则会导致ABA高度不敏感[7,10];且研究发现除了AtPYL13外,其他成员均可与ABA结合抑制蛋白磷酸酶PP2C的活性[22],但另有研究也表明,AtPYL13在ABA存在的时候可以与A类PP2C蛋白相结合以实现其生物学功能[23];在ABA处理后11个成员其表达水平产生了显著变化,AtPYL11和AtPYL13在成熟种子中特异表达,且在ABA介导的种子萌发过程中起正调控作用[11],其中,AtPYL11还可通过延迟种子萌发和通过ABA依赖或不依赖途径诱导的气孔快速关闭来响应冷害胁迫[5];外源ABA处理能够抑制拟南芥侧根生长,且敲除AtPYL8和AtPYL9后侧根生长抑制时间更长且对ABA敏感性下降[24];对烟草NtPYLs研究发现,其在不同组织中表达水平存在差异,且响应非生物胁迫表达模式也各有不同,其中NtPYL19∕20∕28∕29可能参与ABA响应及种子萌发,NtPYL6∕710∕11∕12则可能参与非生物胁迫响应[25]。研究还发现ABA信号通路也参与调控植物果实成熟,对番茄施加ABA能够通过诱导乙烯合成通路来实现促进其果实成熟[26];且番茄中的12个PYL同源基因中[27],SlPYL9在其花、种子及果实中均有表达,且过表达或抑制SlPYL9基因可影响植株对ABA的敏感性,也通过改变ABA信号通路PP2C1/2/9、SnRK2.8等基因的表达,进而影响乙烯的释放、细胞壁的形成及果实成熟[28]。
【本研究切入点】作为ABA受体,PYLs在参与植物果实发育的研究十分欠缺,因此该基因家族参与平榛果实发育的研究将对PYLs基因功能研究领域的重要补充。本研究中借助生物信息学方法从本实验测序所得的平榛(Corylus heteropylla)全基因组中筛选并鉴定得到8个ABA受体基因(ChPYR/PYL/RCAR,简称为ChPYLs),最终明确了其相应的理化、系统进化关系等,并基于课题组现有高通量转录组测序数据明确了各基因在子房及胚珠不同发育时期其表达水平,也进一步利用qRT-PCR验证了其在胚珠不同发育时期的表达情况,可初步筛选出参与榛子果实发育的候选基因,将为后续深入研究榛子果实发育提供重要理论依据。
1 材料与方法
1.1 植物材料与试剂
本研究榛子果实材料取自12年树龄的平欧杂交榛品种“达维”(吉林,四平),在胚珠发育的4个不同时期进行取样,分别为胚珠形成期(OV1),早期胚珠生长期(OV2),胚珠快速生长期(OV3)以及胚珠成熟期(OV4)[29],采集后迅速液氮冷冻;胚珠材料RNA提取选用RNAprep pure植物总RNA提取试剂盒(天根生化科技有限公司,北京);反转录试剂及荧光定量PCR试剂盒均购自天根生化科技有限公司(北京);研究中所用引物由生工生物工程(上海)股份有限公司合成。
1.2 数据下载及榛子PYL同源序列筛选及鉴定
本研究所涉及到的拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、葡萄(Vitis vinifera)和橡胶树(Hevea brasiliensis)PYL基因家族编码序列(Coding sequences,CDS)及其编码蛋白序列下载于TAIR(https:∕∕www.arabidopsis.org∕)、Phytozome 12.0(https:∕∕phytozome.jgi.doe.gov∕pz∕portal.html)及NCBI(https:∕∕www.nc‐bi.nlm.nih.gov∕)等数据库,其拟南芥基因组数据库为Arabidopsis thalianaTAIR10。平榛全基因组为本实验室首次测序完成(基因组数据上载于http:∕∕218.27.6.107∕#∕map,尚未发表)。为鉴定榛子中PYL基因家族成员,以14条拟南芥PYL蛋白序列和11条橡胶树PYL蛋白序列作为检索序列,通过BioEdit软件进行BlastP本地同源比对(E-value <e-20,其他参数设为默认参数),从平榛基因组中筛选PYLs同源序列;对筛选结果人工排除重复、相似度低或不完整短序列(无PYR_PYL_RCAR_like结构域或不完整),为确保候选基因准确和可靠性。并将其编码蛋白序列再次进行Pfam(http:∕∕pfam.xfam.org∕)以及PROSITE(https:∕∕prosite.expasy.org∕)在线工具预测;通过ProtParam在线工具(https:∕∕web.expasy.org∕protparam∕)分析蛋白分子量、理论等电点、不稳定系数等理化参数;通过WoLF PSORT在线预测工具(https:∕∕wolfpsort.hgc.jp∕)进行亚细胞定位预测。
1.3 ChPYLs基因家族系统进化分析
本研究利用MEGA 7.0软件进行基因家族的系统进化分析。提取拟南芥、水稻和橡胶树PYLs蛋白序列,并与本研究筛选的ChPYLs蛋白序列通过Clustal W程序(默认参数比对)进行多重序列比对,对比对结果进行系统进化分析,采用邻接(Neighbor-Joining,NJ)法构建基因家族系统进化树,计算模型为“No.of differences”,空位处理设置为“Pairwise deletion”,校验参数Bootstrap设置为500;对获得的系统进化树并利用Evolview在线工具(https:∕∕www.evolgenius.info∕evolview∕)进行美化和可视化。
1.4 ChPYLs基因序列及其编码蛋白比对、保守结构域分析
为研究基因对应结构,从本实验室搭建的在线平榛基因组数据库(Hazel Omic,http:∕∕218.27.6.107∕#∕map,未发表)中分别提取8条ChPYLs基因的CDS以及基因组序列,并提交到在线基因结构分析工具Gene Structure Display Server(GSDS,http:∕∕www.mybiosoftware.com∕gsds-2-0-gene-structure-display-server.html)进行可视化。
基于筛选获得的基因编码蛋白序列,通过BioEdit软件进行多序列比对(Clustal W,比对参数为默认参数);通过在线分析工具PSIPRED(http:∕∕bioinf.cs.ucl.ac.uk∕psipred∕)分析蛋白序列二级结构;利用MEME(http:∕∕meme-suite.org∕)、Pfam(http:∕∕pfam.xfam.org∕)、PROSITE,NCBI CDD(https:∕∕www.ncbi.nlm.nih.gov∕Structure∕cdd∕wrpsb.cgi)和Motif Scan(https:∕∕myhits.sib.swiss∕cgi-bin∕motif_scan)在线数据库预测分析蛋白序列中所包含的保守基序及结构域,所预测保守结构域及基序均用TBtools软件进行可视化和美化[30]。
1.5 ChPYLs基因在果实发育时期的表达分析
子房及胚珠不同发育时期高通量转录组数据源于本实验室前期研究,原始数据可下载于NCBI数据库(https:∕∕www.ncbi.nlm.nih.gov∕sra∕?term=PRJNA591492);对ChPYLs基因进行实时定量PCR验证胚珠发育的4个时期表达水平,所用引物见表1。
2 结果与分析
2.1 平榛PYL同源序列全基因组筛选、鉴定及理化性质分析
通过与拟南芥和橡胶树PYLs蛋白序列进行比对和挖掘,人工去除重复序列和短序列,在新组装的平榛全基因组中筛选获得8个PYL同源序列,基于其测序基因座位号分别命名为ChPYL1~ChPYL8。蛋白理化性质分析结果表明(表2),ChPYLs基因家族成员所编码蛋白其长度在168~208个氨基酸(aa),其相应蛋白分子量也介于18.8~22.6 kD;理论等电点(PI)其范围为5.35~7.75;对蛋白不稳定系数分析发现,其中ChPYL3,ChPYL5,ChPYL6和ChPYL8为稳定蛋白(不稳定系数小于40),其它蛋白均为不稳定蛋白;所有蛋白总平均亲水性为负值,所以均为亲水性蛋白;依据亚细胞定位预测结果显示,ChPYLs蛋白可定位于叶绿体和细胞质中。
2.2 ChPYLs基因序列比对及系统进化分析
为明确平榛ChPYLs基因的系统进化关系,本研究结合拟南芥、水稻、葡萄和橡胶树4个物种PYLs基因所编码蛋白序列进行系统进化分析,并用邻接法构建系统进化树(NJ)。系统进化分析结果显示,依据所构建进化枝,所有PYLs可分为3个组别(Group I,Group II和Group III),且各组中均包含5个物种的PYLs基因家族部分成员(图1)。Group I包括3个ChPYLs,分别为Cor0021020.1(ChPYL1)、Cor0025220.1(ChPYL2)和Cor0203430.1(ChPYL7);Group II包括3个ChPYLs,分别为Cor0031400.1(ChPYL3)、Cor0047750.1(ChPYL5)和Cor0050430.1(ChPYL6);Group III包括2个ChPYLs,分别为Cor0035760.1(Ch-PYL4)和Cor0213400.1(ChPYL8)。分析结果还表明ChPYL基因家族其进化关系与橡胶树、葡萄更近,与拟南芥和水稻较远;拟南芥、水稻和橡胶树部分成员各自组成较为独立聚集的进化枝,表明同一物种不同成员在进化过程中可能由多次复制事件多产生;平榛8条ChPYLs基因其进化分析结果则进一步显示,该基因家族在本物种中主要分为2个进化枝,表明其可能来自于2个祖先基因(图2A)。

表1 ChPYLs基因实时荧光定量PCR引物Tab.1 Primers of ChPYLs used for real-time qRT-PCR
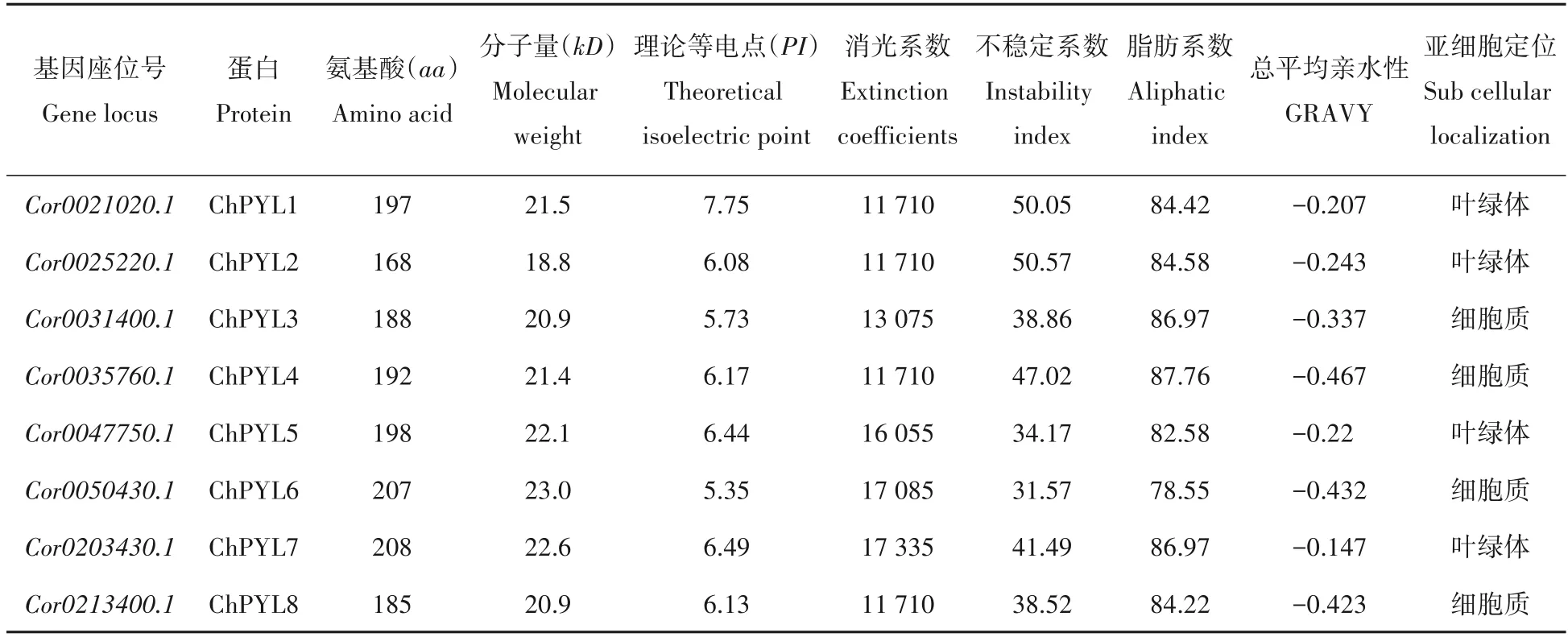
表2 平榛ChPYLs蛋白理化性质Tab.2 The physical and chemical properties analysis of ChPYLs in Corylus heterophylla
2.2 ChPYLs基因及其编码蛋白结构分析
以前人研究为基础,本研究也进一步分析了ChPYLs基因及其编码蛋白序列的结构及保守性。分析结果显示,大多ChPYLs基因不含内含子,只有ChPYL4和ChPYL8含2个内含子,ChPYL5含有1个内含子(图2A)。通过NCBI中保守结构域分析发现所有ChPYLs蛋白均含有典型的PYR_PYL_RCAR_like结构域(图2B),这与其他物种该基因家族的相关研究结果相一致;利用Pfam在线分析工具分析也发现所有蛋白均含有1个Polyketide_cyc2结构域(PF10604),该结构域是START超家族中的重要结构[31],但比较不同成员间该结构域发现,ChPYL5中该结构域长度较之其他成员更短,结合其基因结构比较结构,预示着尽管ChPYL3与其可能进化关系最近,但两者基因功能产生了差异;同时,预测结果还发现,ChPYL1、Ch‐PYL7、ChPYL3和ChPYL5还含有1个保守的Bet_v_1结构域(PF00407)(图2C),且它被认为是一个古老的保守结构域[32],该结构域包含7个β 折叠和2个α 螺旋,形成一个螺旋握结构,该结构继续折叠则形成一个中心疏水区,可与脂类、激素等疏水成分相结合[31]。结合蛋白保守模体分析发现,在该基因家族编码蛋白中所含的10种筛选的模体中,成员间所含的模体数量存在一定差异,所含的保守模体数量在4~6个,但在所有的蛋白中均含有Motif 1、Motif 2和Motif 3,同时也与PYR_PYL_RCAR_like和Polyketide_cyc2保守结构域区域相对应。
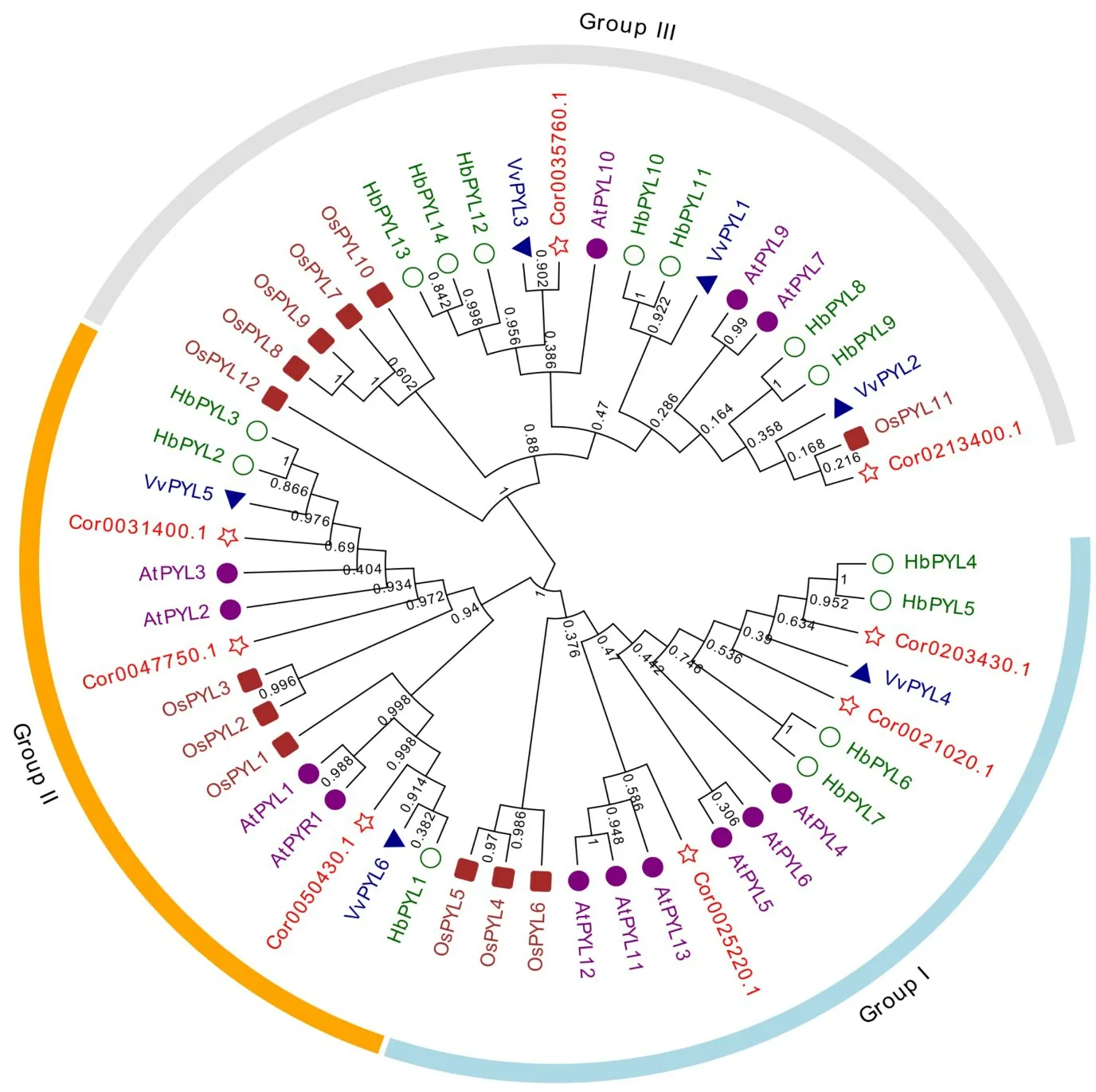
图1 ChPYLs基因家族及其他物种PYLs基因系统进化树(NJ)Fig.1 Phylogenetic analysis of ChPYLs family genes and PYLs genes in other species
2.3 基于高通量转录组测序分析ChPYLs基因表达水平
结合本实验室前期对于榛子子房及胚珠发育不同时期的高通量转录组测序数据[2,29],以8条ChPYLs基因序列为检索序列与测序数据进行比对,并获得对应基因序列及其表达量数据。结果显示,在未授粉花序(F)、子房形成(S)、子房分化(T)和子房生长(FO)4个时期基因家族成员间表达模式存在一定差异(图5A)。除未比对到对应数据的ChPYL2和ChPYL5基因外,ChPYL1、ChPYL7、ChPYL3和ChPYL6在4个时期均低表达,但ChPYL1和ChPYL7在授粉后随着子房的发育,其表达呈稍下降趋势,后2个基因则无显著差异;另外,ChPYL4和ChPYL8在4个时期均相对高表达,且后者表达量更高,同时两者在授粉后随着子房的生长发育,其表达也呈逐渐下降趋势。分析胚珠发育过程中胚珠形成(OV1)、早期生长(OV2)、快速生长(OV3)及胚珠成熟(OV4)4个时期的转录组数据发现,8个基因家族成员除ChPYL4、ChPYL6和ChPYL8外,其他基因均无表达或表达水平极低;并且ChPYL4和ChPYL6在4个时期均低表达,前者无显著差异,后者则呈现上升趋势;ChPYL8在4个时期均高表达,且在4个时期呈现双峰表达模式,即OV2和OV4时期表达量更高,且在OV2时期表达量最高,OV1时期表达量最低。
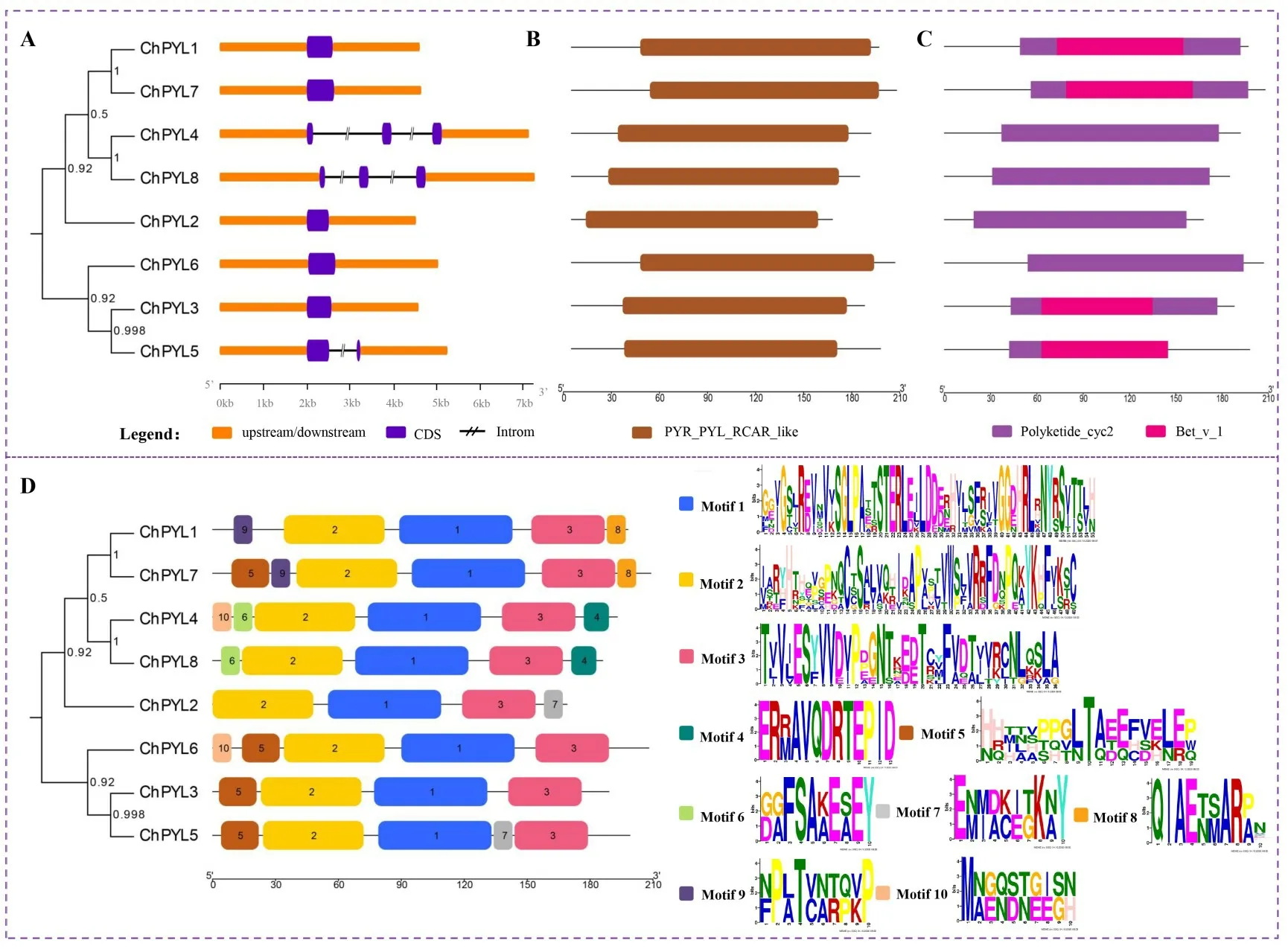
图2 ChPYLs基因及其编码蛋白结构及保守基序分析Fig.2 Analysis of gene and protein sequences structure of ChPYLs in Corylus heterophylla

图3 ChPYLs蛋白序列及其他物种部分PYLs蛋白序列比对Fig.3 Protein sequences alignment of ChPYLs and PYLs in other species

图4 ChPYLs基因在子房及胚珠发育不同时期的表达水平热图Fig.4 Expression heatmap of ChPYLs genes in different development stages of ovary and ovule
2.4 基于qRT-PCR验证ChPYLs基因在不同胚珠发育时期的表达水平
为进一步验证ChPYLs家族基因成员在胚珠发育不同时期的表达水平,笔者利用qRT-PCR对各基因进行了表达水平检测。检测结果显示,总体而言各成员间其表达模式各有差异,且该基因家族成员表达水平较低。其中,ChPYL1和ChPYL5表达呈现相似模式,OV1和OV3时期表达量更高;ChPYL2、ChPYL3和ChPYL7则在胚珠发育初期表达量较高,且前两者呈现逐渐下降的趋势;ChPYL4在OV1时期表达量最高,其他3个时期均低表达;ChPYL6则在OV3时期表达量最高;ChPYL8表达呈现双峰模式,即OV2和OV4时期表达量更高。该验证结果与转录组数据分析结果较为一致,也进一步证明转录组数据具有较高可靠性。
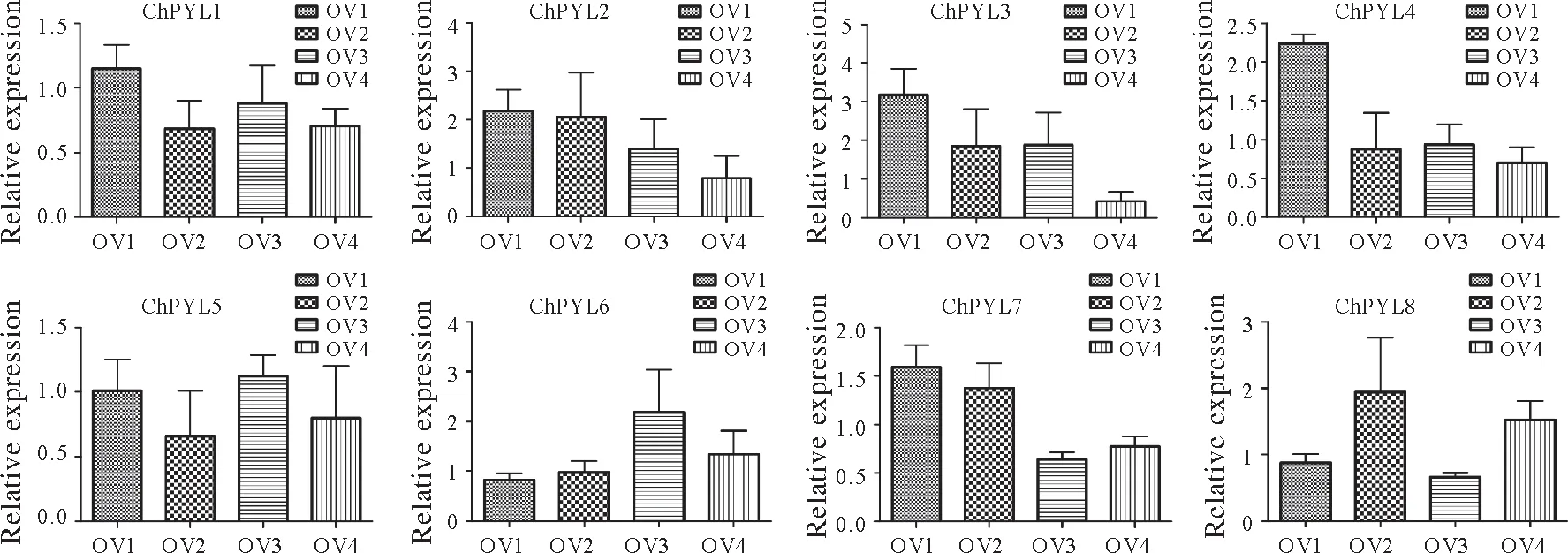
图5 胚珠发育不同时期ChPYLs表达量检测(qRT-PCR)Fig.5 Expression level analysis of ChPYLs in different development stages of ovule(qRT-PCR)
3 结论与讨论
本研究在平榛中共鉴定获得8个PYL基因家族成员,通过与拟南芥、水稻、葡萄以及橡胶树PYL基因家族进行进化分析,发现平榛PYLs与葡萄及橡胶树PYLs亲缘关系更近,这也与物种亲缘关系相一致;另外,多物种PYL基因家族进化分析发现,ChPYL4与VvPYL3亲缘关系更近(图1),且研究发现葡萄在高盐和干旱胁迫下VvPYL3表达量降低,预示着其可能作为一个负调控因子参与葡萄响应高盐和干旱胁迫;另外,研究发现与ChPYL2亲缘关系更近的AtPYL11和AtPYL12能够正调控ABA介导种子萌发[11],故在后续Ch-PYL4及ChPYL2功能研究中可依据以上预测进行侧重研究;基因结构之间的差异与各成员间进化关系密切相关,通过基因结构分析发现,平榛PYLs中除ChPYL4、ChPYL5和ChPYL8外均不含内含子,表明大部分成员均具有高度的保守性,可能在进化上较为原始;对基因编码蛋白二级结构分析发现,各成员编码蛋白保守结构域均含有α螺旋、β折叠及无规则卷曲,且数量和分布规律也与在水稻、橡胶树中的研究结果相一致;另有研究发现,PYL家族编码蛋白中CL2区域是ABA受体与PP2C互作的重要区域,且该区域中的K(赖氨酸)与I(异亮氨酸)的转变(图3)会导致对于ABA依赖性的改变,也是该区域重要的功能位点[7]。分析发现ChPYL4和ChPYL8在该位点与其他蛋白存在差异,可能影响其对ABA的依赖性。
转录组数据分析结果也显示,在子房及胚珠发育的各个时期,ChPYL4和ChPYL8均有更高的表达水平,且其他基因均表达量极低或无表达,因果实发育初期内源ABA含量极低或检测不到[33],鉴于PYL作为ABA受体可分为ABA依赖型和不依赖型,例如,在番茄的12个PYLs成员中也发现SlPYL3和SlPYL12为ABA-不依赖受体[27],故上文提及ChPYL4和ChPYL8蛋白位点的改变可能导致其对ABA依赖性更小或不依赖;ChPYL2和ChPYL5基因在子房发育时期未比对获得对应表达数据,预示着两者在该过程可能无表达且无显著差异,即不参与该过程调控;ChPYL4和ChPYL8基因在子房发育的4个时期呈下降趋势,表明它们可能作为负调控因子参与调控子房发育过程,且后者功能更显著;ChPYL8在胚珠发育的4个时期则呈现双峰表达模式,表明在胚珠中可能具有更精细的调控模式;通过荧光定量分析发现ChPYL6在胚珠发育4个时期尽管表达量较低,但发育后期表达量更高,与转录组数据较为一致,预示着该基因可能在榛子胚珠发育过程发挥正调控作用。
综上所述,基于目前PYL基因家族参与植物果实生长发育的研究相对较少、不够深入的现状,本研究以重要的经济林木植物——榛子为研究材料,也将为后续该基因家族成员参与榛子果实发育方面的研究提供重要理论基础,另外,该基因家族成员在非生物胁迫响应的生物学功能也有待深入探究。
