双通道特征提取的胶质瘤分割方法
2021-05-15丁冬冬陈世国杜进楷石平霞
丁冬冬,陈世国,杜进楷,杨 乘,石平霞
(1.贵州师范大学 物理与电子科学学院,贵州 贵阳550001;2.贵州省教育厅汽车电子技术特色重点实验室,贵州 贵阳550001;3.贵州省射电天文数据处理重点实验室,贵州 贵阳550025)
0 引 言
胶质瘤(Glioma)是一种神经系统肿瘤,不同程度的胶质瘤会产生不同的临床症状,胶质瘤的准确分割有助于疾病的诊断和治疗,有效缓解病情,延长存活时间,并且可以为后续治疗提供对比观察,因此,对胶质瘤的核磁共振图像(MRI)分割有重要的研究价值。国内外在图像分割早期应用较为广泛的有基于阈值、区域、边缘检测、小波、马尔科夫随机场等分割方法,然而传统方法进行分割耗费大量的时间和人力,且分割准确度与医生的临床经验有很大关系,易受主观因素的影响。近年来,图像分割领域多种深度学习网络结构被不断提出,主要有2D深度学习网络和3D深度学习网络两种。相比于3D深度学习网络结构,2D网络具有参数量相对较少,对电脑性能要求略低,适用范围更广的特点,因此本文针对2D深度学习网络进行改进。
文献[1]将卷积神经网络(CNN)应用在图像处理方面表现突出,然而传统CNN主要定位局部信息,易造成全局信息的丢失。文献[2]于2017年提出的全卷积神经网络(FCN)为解决语义分割提供了一种新的解决方案。2016年文献[3]提出了一种可以自动适应目标区域大小的多尺度二维卷积神经网络模型,有效解决了脑肿瘤尺度不一致的问题,网络每次利用一个图像块对图像中心像素预测,造成大量重复计算,有待优化提升。文献[4]于2018年结合模糊推理系统,建立学习规则对CNN预测肿瘤点的概率再判断,分割精确度有所提升,但为半自动化算法,分割效率不高。综上,前人所设计的2D网络未有效解决类不平衡以及形状不规则的胶质瘤更精确分割的问题。因此,设计有效的提高胶质瘤分割的2D网络有一定的研究价值。
文献[5]提出的U⁃net网络,其“编码⁃解码”结构可以进行端到端的学习,无需将图像切割为多个图像块后再进行训练,减少了大量重复计算。编码(Encode)结构对特征进行提取,用逐级下采样的方式减小特征图尺寸,增加感受野(Receptive Field)范围,捕获丰富的上下文信息。解码(Decode)过程中逐步恢复特征向量尺寸,并通过跳跃连接将编码阶段的特征与上采样得到的特征向量拼接融合,对编码过程中提取到的特征信息加以利用,提高网络的表达能力。因此,U⁃net网络在减少重复计算情况下能获得较好的分割效果。但U⁃net网络编码过程中用一条特征提取通道提取到的特征不充分,使得分割边缘清晰度不理想;解码过程中对特征向量进行拼接(Concatenate),特征融合不充分;网络中的特征向量在训练过程中会产生新的数学分布,影响网络收敛速度。针对上述存在的问题,本文提出了一种基于U⁃net网络的双通道特征提取(Dual⁃Path Feature Extraction based on U⁃net,DPFE⁃Unet)的脑部胶质瘤分割方法。
DPFE⁃Unet以U⁃net网络为基础网络结构,做了以下改进:
1)编码阶段采用双通道特征提取的方法进行特征提取,提取更加丰富的多尺度特征,更加准确地定位肿瘤边缘。
2)解码阶段,通过对来自编码阶段的特征向量和解码阶段上采样后的特征向量通过充分的跨通道融合,增加更多的非线性来增强网络的表达能力。
3)将特征向量用批归一化(Batch Normalization)处理,降低因特征向量的数学分布改变对网络收敛速度的影响,有效防止网络过拟合。
1 DPFE⁃Unet网络结构
DPFE⁃Unet的网络框图如图1所示。

图1 DPFE⁃Unet网络框图
1.1 双通道特征提取的编码结构
为了提取更加丰富的特征信息,达到提高目标区域定位精度的目的,DPFE⁃Unet在编码阶段采用两条特征提取通道进行多尺度特征提取,将双通道上对应尺度下的特征向量相加以获得更丰富的局部信息,再对结果进行批归一化和非线性激活操作,以此作为编码阶段不同尺度下的输出特征向量。
对于每条特征提取通道,输入数据首先进行两次卷积,卷积核大小为3×3,输出通道为64,并通过批归一化层和ReLU[6]非线性激活层。然后,使用最大池化层(Max Pooling)将非线性激活后的特征向量中的主要特征值保留,特征向量尺寸减半,增大感受野。为了提取更加复杂多样的特征信息,对池化后的特征向量进行两次卷积操作,卷积核大小为3×3,两次卷积的输出通道数相同,均为上一尺度特征向量通道数的两倍,最后对结果进行批归一化和非线性激活。以上“最大池化⁃卷积⁃卷积⁃批归一化⁃非线性激活”操作连续进行4次,分别得到通道数为64,128,256,512,1 024的特征向量。
最后,将双通道中对应尺度下的特征向量分别相加(Add),再对相加后的结果分别进行“卷积⁃卷积⁃批归一化⁃激活”处理,并以此作为编码阶段提取到的多尺度特征向量。
双通道特征提取的编码结构如图2所示。
1.2 跨通道特征融合的解码结构
解码过程中,对于来自编码和解码两个阶段相同尺度下的特征向量进行跨通道的特征融合,增加网络非线性表达能力,同时降低计算复杂度。
1.2.1 上采样
对某一尺度下的特征向量进行尺度恢复是通过上采样(UpSampling)操作将该特征向量尺寸加倍,与上一尺度下的特征向量尺寸相同。首先将尺度为15×15的feature4的特征通过上采样将分辨率提升1倍,恢复至30×30,同时为了减少参数量,通过3×3的卷积层将特征向量的通道数减半。同理,对每个尺度下的特征向量都进行这样的操作,最终将特征向量尺寸恢复至240×240,与输入图像分辨率相同。
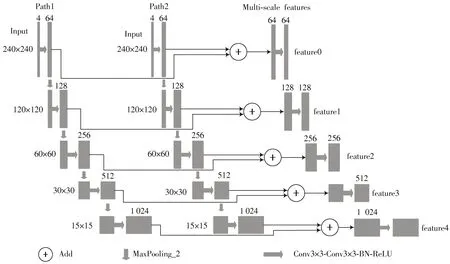
图2 双通道特征提取的编码结构
1.2.2 特征融合
为充分利用编码阶段特征向量,DPFE⁃Unet网络先对上采样后的特征向量进行批归一化和非线性激活,再将结果与编码阶段同尺度的特征向量进行拼接,最后对拼接后的结果用卷积核尺寸为1×1的卷积层[7]进行卷积,达到跨通道特征融合的目的,充分利用图像的局部信息和全局信息提高分割的精度。同时,卷积过程中将通道数减半达到减少参数量的作用。跨通道特征融合过程如图3所示。
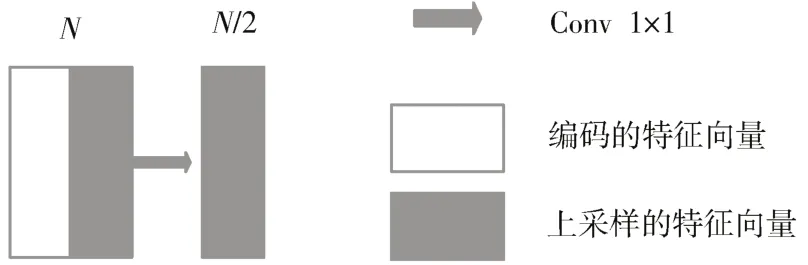
图3 跨通道融合过程
跨通道特征融合的解码结构如图4所示。
1.3 特征向量批归一化
编码阶段使用双通道特征提取的方法增加了网络复杂度,因此可能会造成过拟合现象;网络训练过程中特征向量的数学分布会发生变化,造成协方差偏移[8]。针对以上两问题,在DPFE⁃Unet网络中使用批归一化操作对编码阶段的特征向量以及各尺度下融合后的特征向量进行批归一化处理,有效防止过拟合现象的发生,加快了网络的收敛速度。同时,批归一化操作还可以有效防止梯度消失和神经元失活[9]。
批归一化公式为:
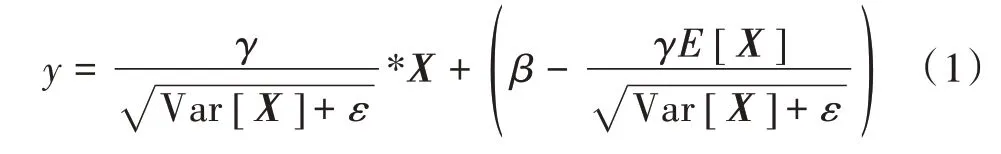
式中:X为待归一化的特征向量;E[X]为均值;Var[X]为标准差;γ,ε分别为放缩和偏移系数,均为可学习参数,ε是为了防止分母为0的较小数值。

图4 跨通道特征融合的解码结构
2 实 验
实验环境为IntelⓇCoreTMi7⁃9700K CPU@3.6 GHz,RAM大 小 为32 GB,Windows 10专业版,编程语言为Python,深度学习框架为Keras。
2.1 实验数据及预处理
实验所采用数据来源于MICCAI大赛的BRATS2015数据集[10]的高级别肿瘤(HGG)中的220例病人数据,每位病人有Flair、T1、T1c、T2四种模态MRI图像和一组专家标注真实标签(Ground Truth,GT),每种模态有155张切片,将数据集按照0.6,0.2,0.2的比例随机分为训练集、验证集和测试集。
由于肿瘤区域通常不在切片序列的两端,本实验取每种模态的第50~120张切片进行训练,进一步缓解因类不平衡造成的难以精确分割的问题。四种模态图像(Flair、T1、T1c、T2)及专家标注GT图像如图5所示。

图5 多模态数据显示图
将四种模态分别进行标准化处理,作为输入数据的四个通道进行训练[11],标准化公式为:

式中:Xi为第i个模态的图像;E(Xi)为该模态图像的均值;ε为防止分母为0的较小数值。
2.2 网络超参数设置
实验中卷积核均采用随机He分布初始化方法进行初始化[12],网络在反向传递过程中使用Adam优化器进行优化[13],为了便于解码阶段的特征拼接,将卷积函数中的valid参数设置为“same”,网络设置为自动调整学习率,初始学习率为0.000 1,批大小为10,网络评价指标为Dice系数[14]。
2.3 评价指标
Dice系数用来衡量两个向量的相似性程度,实验中用来表述模型输出的分割结果与真实标签的相似性。Dice系数定义如式(3)所示,其中,TP(True Positive)为真 阳 性,FP(False Positive)为 假 阳 性,FN(False Negative)为假阴性。

网络训练过程中选用Dice损失(Dice_loss)函数作为优化依据,Dice损失函数定义为:

2.4 实验结果对比及可视化
2.4.1 实验结果对比
将本文方法与对比文献中的3种方法进行对比,Dice系数对比如表1所示。其中,方法1中所用网络为2D U⁃net网络,方法2的DeepMedic网络在2015年的脑肿瘤分割比赛中排名前三,方法3、4所用网络均为3D网络。

表1 DPFE⁃Uet在Brats2015验证集上的Dice系数与其他方法对比
由表1中的各评价指标可以看出,本文提出的2D网络DPFE⁃Unet与方法1中所用2D U⁃net网络相比,相应的Dice系数分别提升9.7%,14.3%和7.8%;与方法2,3,4所用3D网络相比,相应的Dice系数分别至少提升4.7%,5.3%和8.8%。结果表明,基于U⁃net网络改进的DPFE⁃Unet网络在胶质瘤分割问题上与对比文献相比有更好的分割效果。
2.4.2 实验分割结果可视化
对测试集的分割结果进行可视化,如图6所示。其中,图6a)为Flair模态图像,图6b),图6c),图6d)分别为WT、TC、ET的真实标签,图6e),图6f),图6g)为本文方法对应的三个区域的分割结果。
从图6中的分割结果与真实标签对比可以看出,本文提出的方法对胶质瘤分割的结果与给定标签图像在视觉效果上基本一致,表明DPFE⁃Unet网络对胶质瘤分割较准确。

图6 分割结果
3 结 语
DPFE⁃Unet网络是在U⁃net网络结构的基础上改进的端到端的2D网络。在编码阶段,利用双通道特征提取的方法提取更加丰富的多尺度特征,改善因网络结构简单造成的特征提取不充分而无法准确分类的问题。解码阶段利用1×1的卷积层将特征信息进行充分的跨通道融合,利用局部信息和 全局信息提高边缘分割精度,同时降低网络参数,有效缓解双通道特征提取方法导致参数量增加的情况。批归一化的引入解决了特征向量的数学分布发生变化对网络收敛速度的影响。
实验结果表明,DPFE⁃Unet网络在胶质瘤分割问题上效果更好,与U⁃net网络相比分割效果有显著提升,也优于近几年的部分3D网络。因此,本文研究成果有助于提升临床医学诊断的准确率,为制定合理的治疗方案提供依据。
