胃癌组织环状RNA表达谱分析
2021-05-14杨青茹王华周松峰申红星
杨青茹,王华,周松峰,申红星
(1. 江苏大学医学院, 江苏 镇江 212013; 2. 南通大学医学院, 江苏 南通 226019)
胃癌是全球常见的恶性肿瘤,也是导致死亡的第二大主要原因[1]。胃癌的致病因素很多,如EB病毒感染、幽门螺杆菌感染等[2]。胃癌的发生不仅影响蛋白代谢异常[3],也涉及微小RNA(microRNA,miRNA)、长链非编码RNA(lncRNA)和环状RNA(circluar RNA, circRNA)等非编码RNA的异常表达。CircRNAs自2013年发现其生物学功能以来,逐渐成为研究的热点,研究显示circRNAs参与癌症等多种疾病的发生发展过程[4]。CircRNAs分子是一类不具有5′末端帽子和3′末端poly(A)尾巴,并通过共价键形成环形的非编码RNA分子,具有一定的组织、时序和疾病特异性,并发挥调节转录、翻译和表达水平等方面的生物学功能[5-6]。已有研究表明,circ-HuR异位表达在体内外均能抑制胃癌细胞的生长、侵袭和转移[7]。部分circRNAs分子含miRNAs应答元件(miRNA response element,MRE),可充当竞争性内源RNA(competing endogenous RNA,ceRNA),与miRNAs结合,发挥miRNAs海绵的作用,上调靶基因表达水平;也可以翻译成蛋白质来发挥作用[8]。CeRNA调控在癌症进展中发挥关键作用,如circRNA_LARP4通过抑制miR-424-5p和调控LATS1的表达,抑制胃癌细胞的增殖和侵袭[9];circPDSS1通过海绵miR-186-5p和调节NEK2促进胃癌进展[10];circRNA_100782通过海绵吸附miR-574-3p,抑制Rb表达,促进胃癌的增殖和转移[11]。
虽然多个报道显示circRNAs参与调节胃癌的发生发展,但是非编码RNA尤其是circRNAs和miRNAs在胃癌中的详细作用机制依然不清楚。本研究通过分析胃癌相关的多个circRNAs的测序数据,旨在揭示circRNAs-miRNAs-mRNAs网络通过生物进程、细胞组分和分子功能3个方面参与胃癌发生发展的机制。
1 材料与方法
1.1 差异表达circRNAs的获得
使用美国国家生物技术信息中心(NCBI)的GEO数据库(http://www.ncbi.nlm.nih.gov/geo/geo2r/)和在线分析工具R语言分析(http://www.dooccn.com/r/)GSE100170、GPL19978(GSE83521和GSE89143)、GSE78092和STAD(未上传)数据库中差异表达的circRNAs。
1.2 筛选circRNAs靶向的miRNAs及其验证
使用Starbase数据库(http://bioinfo.au.tsinghua.edu.cn/software-database)预测circRNAs的靶向miRNAs,根据CLIP-Data≥2的筛选条件,筛选circRNAs下游的miRNAs,并利用GEO数据库(GSE23734)作为下游miRNAs准确性的验证,其结果以韦恩图呈现(http://bioinformatics.psb.ugent.be/webtools/Venn/)。
1.3 筛选miRNAs靶向的mRNAs
使用TargetScan数据库(http://www.TargetScan.org)预测miRNAs下游的mRNAs,其筛选标准为Total context+ score≤-0.75。
1.4 GO分析、KEGG分析和ceRNA相互作用网络
基因本体论(gene ontology,GO)分析包括生物进程、细胞组分和分子功能3个方面[12]。通过GO分析和KEGG分析验证mRNAs的功能。通过Cytoscape2.0软件(http://www.cytoscape.org)分析circRNAs-miRNAs-mRNAs三者之间的相互作用关系。
1.5 靶基因相关疾病
利用比较毒物基因组学数据库(The comparative toxicogenomics database)分析靶基因导致的相关性疾病,网址为https://ctdbase.org/,筛选标准为P<0.05[13]。
2 结果
2.1 预测差异表达circRNAs的下游靶miRNAs以及验证
本文所使用的数据库以及筛选流程图如图1所示。从GEO数据库以及STAD中总共获得了83 748个circRNAs,按照Log2(fold change,FC)>1,P<0.05的筛选标准获得734个差异表达的circRNAs(表1)。将circRNAs注释后,利用韦恩网站处理,共获得59个基因(表2),其韦恩图如图2A所示。排除各种因素(没有针对性的miRNAs,并且无法同时在两个或多个数据库中找到)发现17个差异表达的circRNAs,见表3。用Starbase数据库预测17个circRNAs的下游靶miRNAs,得到 354个miRNAs。以GEO数据库中的数据集GSE23739 (共80对标本,癌组和癌旁组各占一半)作为参考,进一步验证354个miRNAs与胃癌的关系。根据P<0.05的筛选标准,从GSE23739数据库获得512个miRNAs,并与预测到的354个miRNAs作韦恩图(图2B),获得46个共同的miRNAs(表4)。由于miR-483-3p没有查询到靶基因,最终选取45个miRNAs用于下一步分析。为验证癌组和癌旁组中差异性表达的circRNAs,用R语言作为运行程序绘制热图。结果如图2所示,分别显示4个数据库GPL19978(图2C)、GSE78092(图2D)、GSE100170(图2E)和STAD(图2F)的circRNAs的差异表达。
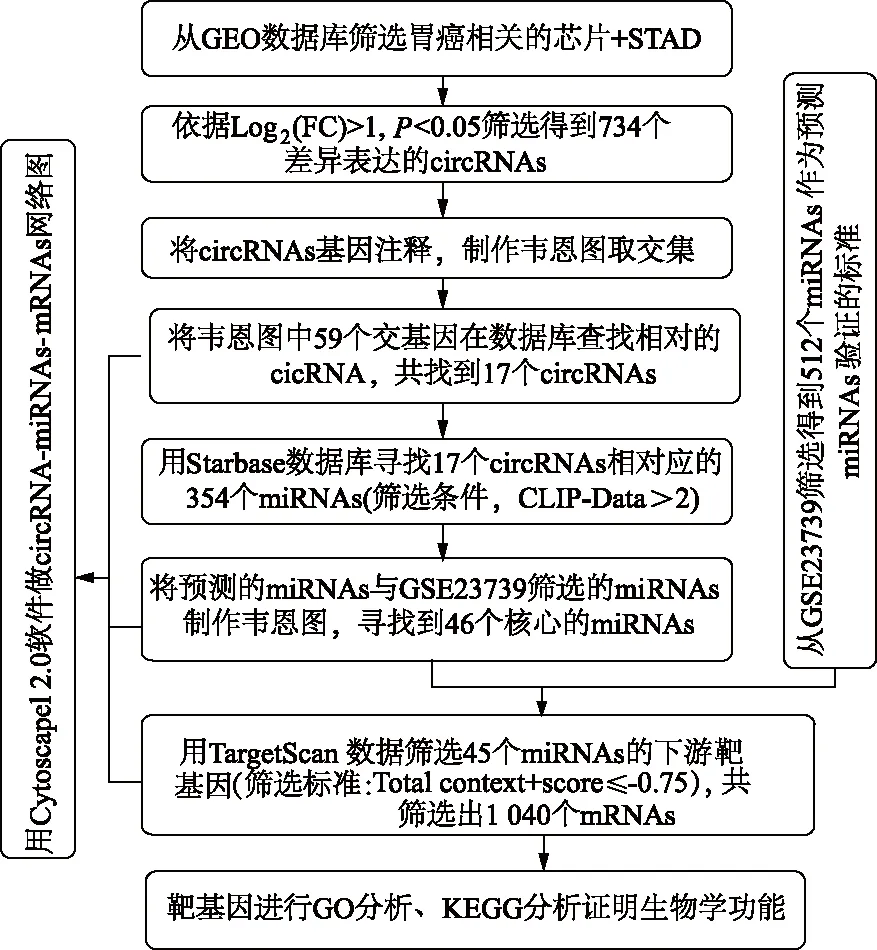
图1 差异表达circRNAs筛选技术路线图
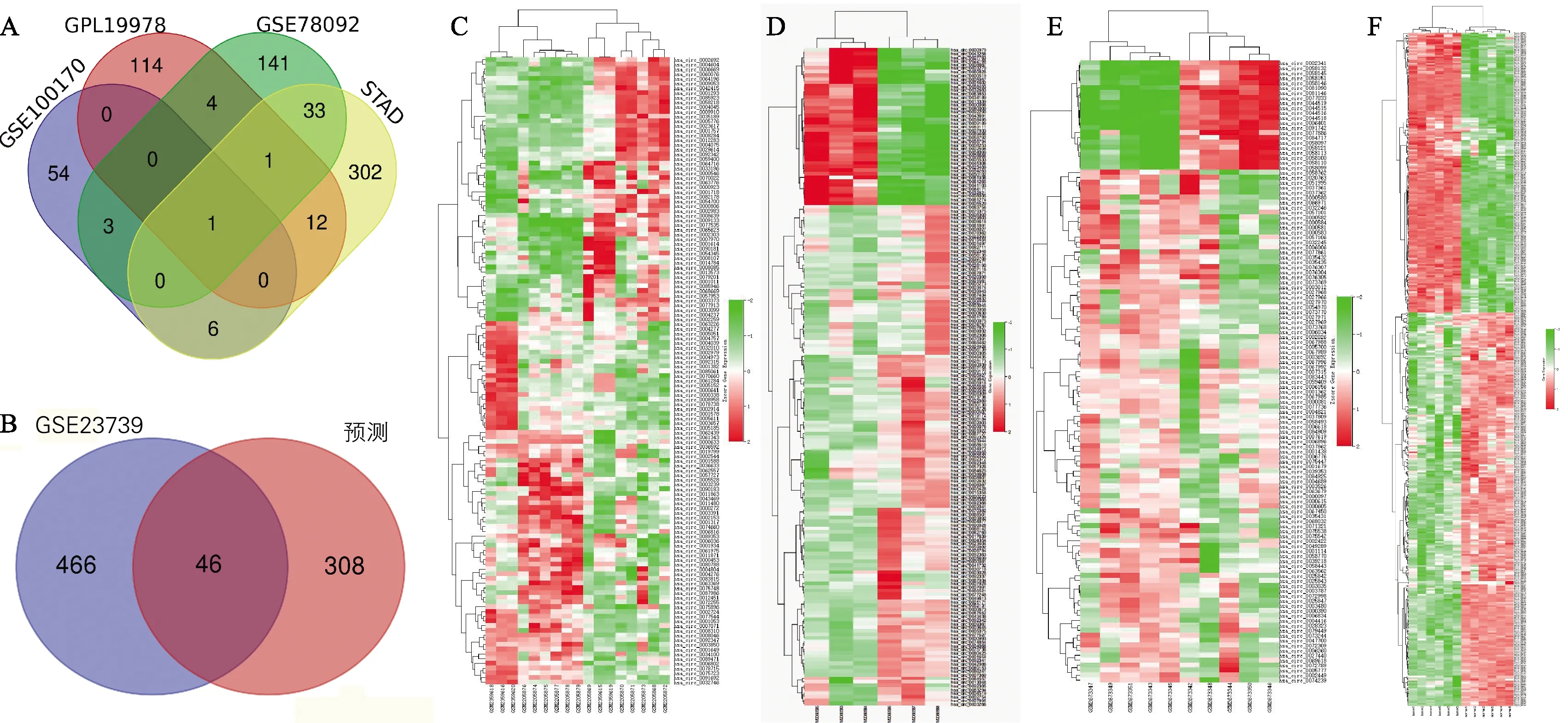
A:4个基因芯片数据(GSE78092,GSE100170,GPL19978,STAD)的韦恩图;B:GSE23739和预测数据的交集,筛选出46个miRNAs;C:GPL19978(癌组与癌旁组的比例为8 ∶9);D:GSE78092(癌组与癌旁组的比例为3 ∶3);E:GSE100170(癌组与癌旁组的比例为5 ∶5);F:STAD(癌组与癌旁组比例为6 ∶6)图2 差异表达circRNAs的筛选韦恩图以及所选数据库差异表达circRNAs的热图
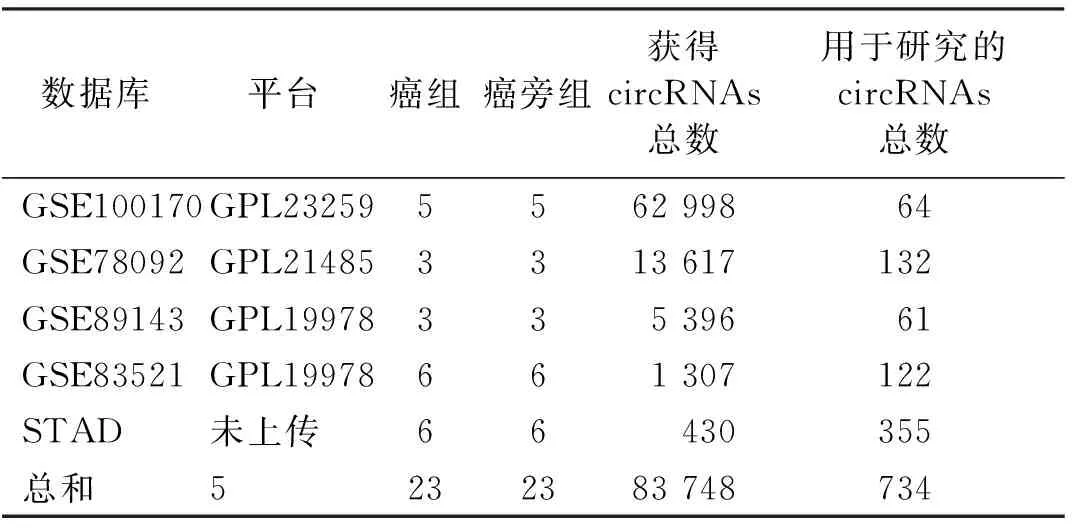
表1 有关数据源的特定信息
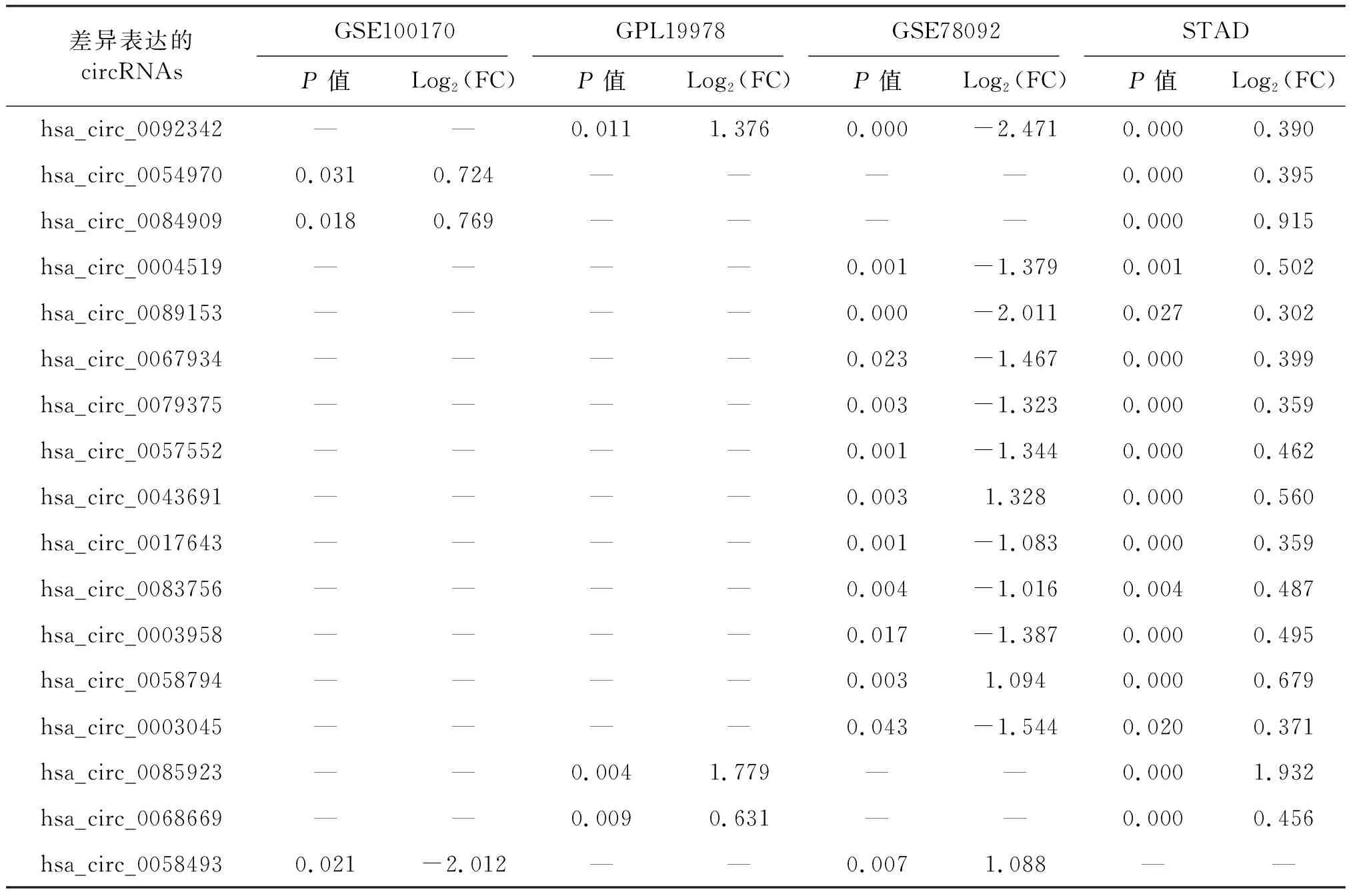
表3 差异表达的circRNAs的P值以及倍数变化的对数值

表4 CircRNAs靶向的经过筛选的下游miRNAs
2.2 靶基因的GO分析、KEGG分析以及ceRNA网络调控
利用TargetScan数据库,在Total context+score≤-0.75的筛选条件下,筛选出miRNAs下游的靶基因有1 040个。利用Cytoscape 2.0软件将本研究所分析的17个circRNAs、45个miRNAs以及1 040个靶基因进行ceRNA网络调控分析(图3A),结果表明,某些miRNAs(hsa-miR-28-5p和hsa-miR-331-3p)可以结合多个circRNAs,说明其在胃癌的发生发展中发挥核心调节作用。通过GO分析研究核心miRNAs相对应的靶基因,结果表明其主要在蛋白质泛素化,蛋白质转移、定位,病毒的转移以及tRNA的核输出等生物学进程中发挥作用(图3B)。为进一步研究circRNAs与胃癌的相关性,通过KEGG分析深入研究circRNAs相关靶基因的富集通路,由图3C可见,其富集通路的途径主要是通过囊泡转运中的SNARE相互作用(hsa04130)、氧化磷酸化(hsa00190)、MAPK信号通路(hsa04010)和N-糖基生物合成(hsa00510)来实现。而调控转录的生物过程主要是转录RNA代谢、细胞内信号传导级联和蛋白质定位。
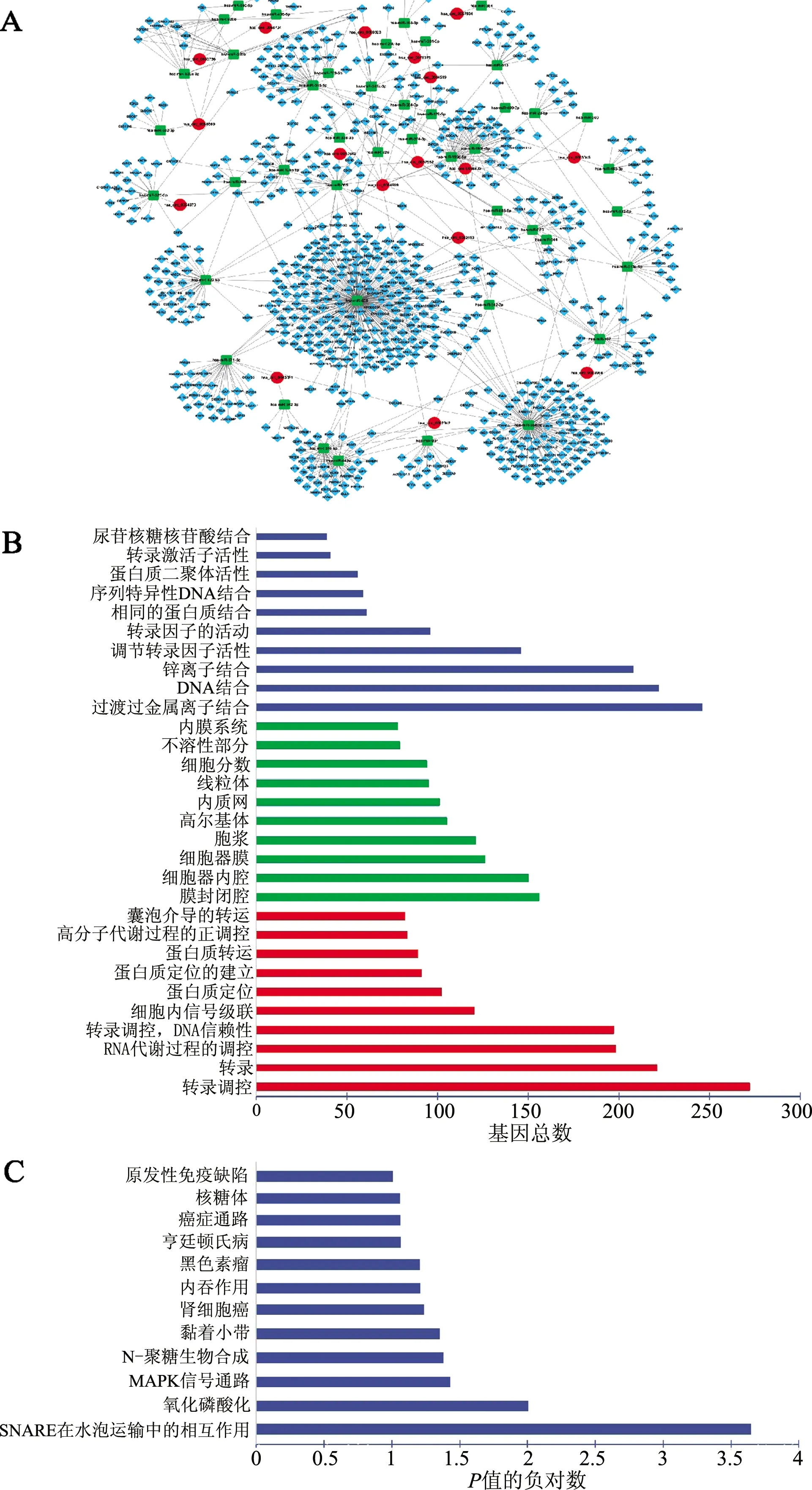
A:用Cytoscape2.0软件分析circRNAs-miRNAs-mRNAs的ceRNA关系,红色:circRNAs,绿色:miRNAs,蓝色:mRNAs; B:GO分析,红色:生物进程,绿色:细胞组分,蓝色:分子功能;C:靶基因的KEGG分析图3 靶基因的互作网络图以及靶基因GO分析和KEGG分析
由表5可见,miRNAs下游富集的核心基因相关的疾病包括肿瘤、组织学类型的肿瘤、腺癌、皮肤和结缔组织疾病、白血病和急性骨髓白血病等,而与其相关程度最大的疾病仍是肿瘤,为我们研究胃癌提供了许多参考依据。CircRNAs在肿瘤中的作用机制研究较多,其中胃癌内的ceRNA网络调控机制为本课题组的研究重点,为后续研究胃癌相关的circRNAs、miRNAs以及mRNAs奠定了基础。
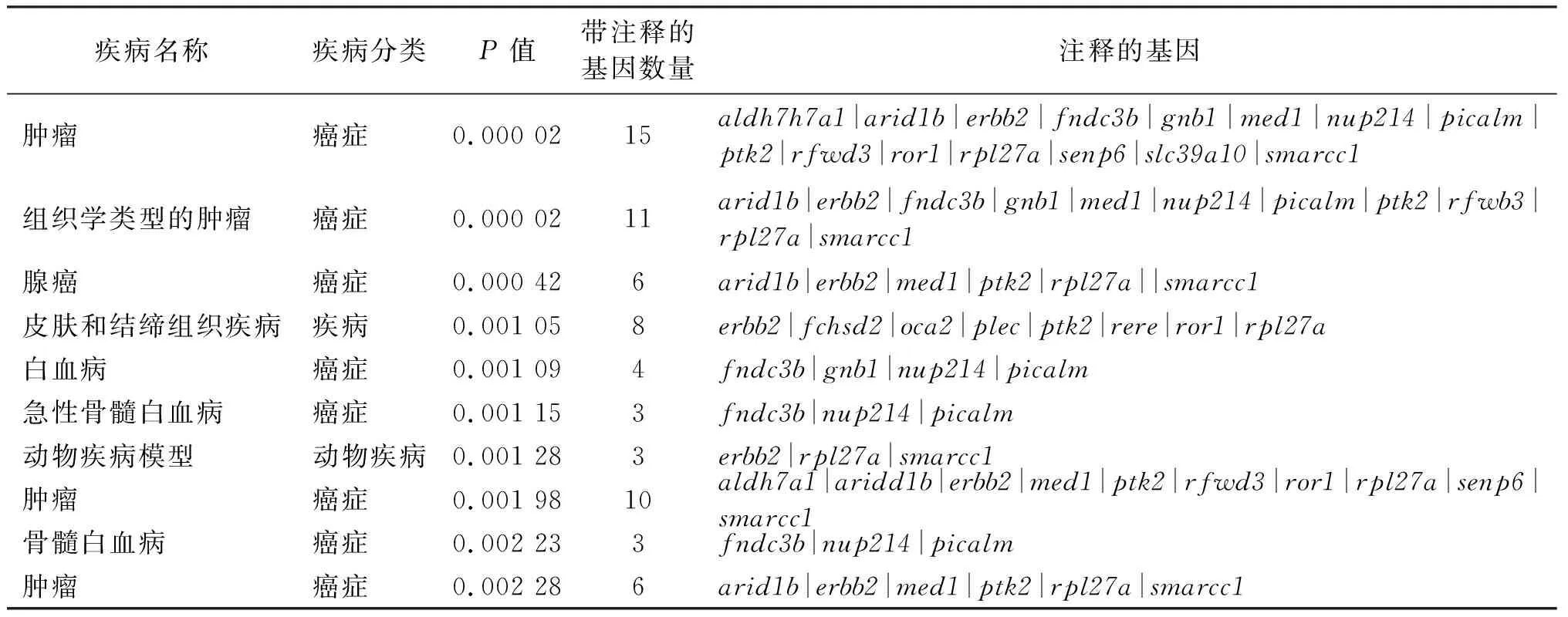
表5 核心基因相关疾病 (前10个)
3 讨论
本研究利用生物信息学方法发现胃癌差异表达的circRNAs(如hsa_circ_0092342、hsa_circ_0054970和hsa_circ_0084909)可作为胃癌诊断标志物以及发挥ceRNA调控作用,影响胃癌的发生发展。结果表明,hsa_circ_0092342可通过hsa-miR-654-3p靶向CLVS1/PGBD1发挥促进或抑制胃癌的作用;hsa_circ_0054970可通过hsa-miR-525-5p靶向TMEM65对胃癌发挥作用。KEGG分析结果表明,靶基因发挥的主要作用是转录调控、RNA代谢过程、细胞内信号级联和蛋白质定位等。此项研究的意义是发现差异circRNAs表达以及ceRNA调控网络的变化,尤其是差异表达特别明显的几个circRNAs对胃癌的诊断作用。目前为止,研究者已发现胃癌的各种生物标志物,例如脑表达X连锁4( brain-expressed X-linked 4 ,BEX4),烟酰胺N-甲基转移酶等[14-15]。多项研究已证实ceRNA调控机制,如circRNA_100269在胃癌中下调,并通过靶向miR-630抑制肿瘤细胞生长;circRNA_001569通过吸附miR-145在胃癌中促进细胞增殖;干扰circRNA_0001947表达可抑制胃癌细胞增殖并诱导其凋亡[16-18]。在胃癌组织中发现大量的miRNAs,高表达miR-125b-5p的胃癌患者生存期明显缩短,证明miR-125b-5p能促进胃癌的发生发展[19]。本研究中的核心miRNAs为miR-28-5p和miR-331-3p,其中miR-28-5p在胃癌中尚无相关报道,但在鼻咽癌中发现其影响鼻咽癌细胞的增殖、凋亡、迁移和侵袭[20];而miR-331-3p在许多癌症研究中都有报道,如circCACTIN通过吸附miR-331-3p和调节转化生长因子β受体1(TGFBR1)的表达促进胃癌进展[21],circ_0038646通过miR-331-3p/GRIK3促进结直肠癌细胞增殖和迁移[22]。
在胃癌中已发现许多circRNAs,部分存在于真核细胞的胞质中,少部分内含子来源的circRNAs存在于细胞核内。若circRNAs主要存在胞质中,则作为miRNAs海绵发挥作用,进而解除miRNAs对其靶基因的抑制作用;若定位于细胞核内,则可充当RNA结合蛋白发挥作用[23]。就目前的研究而言,多数circRNAs主要是通过ceRNA调控机制促进或者抑制胃癌的增殖和迁移,但还有很多尚未知悉的生物学功能,其在疾病诊断治疗的研究还不深入,需要进一步求证。今后应深入研究circRNAs在胃癌诊断、治疗方面的作用机制,充分了解其生物学功能,使其能更广泛地应用于研究领域。
