副干酪乳杆菌(Lactobacillusparacasei)N1115菌株定量PCR检测方法研究
2021-05-12张奕南王越张栋张娜娜朱宏刘洋王世杰
张奕南,王越,张栋,张娜娜,朱宏,刘洋,王世杰
(1.上海市质量监督检验技术研究院,上海 200233;2.石家庄君乐宝乳业有限公司,石家庄 050221)
0 引 言
副干酪乳杆菌(Lactobacillus paracasei)在细菌分类学上的位置属于厚壁菌门(Firmicutes)、芽孢杆菌纲(Bacilli)、乳杆菌目(Lactobacillales)、乳杆菌科(Lactobacillaceae)、乳杆菌属(Lactobacillus),与干酪乳杆菌(L.casei)、副干酪乳酸菌(L.paracasei)和鼠李糖乳杆菌(L.rhamnosus)在系统发育和表型上关系密切,它们一起被认为是干酪乳杆菌群,具有益生特性和食品发酵工业生产价值[1]。L.paracasei具有良好的耐酸、耐胆盐能力,并具有提高肠道上皮细胞屏障的作用。研究发现L.paracasei的多个菌株具有通过调节细胞因子的分泌,平衡炎症反应,以缓解过敏的功能[2-4]。通过代谢产物如有机酸、细菌素和苯乳酸可有效抑制致病菌和腐败菌的生长[5-6]。该菌种胞外酶产生抑制血管紧张素(ACE)的小分子肽,并且有些菌株可以高产氨基丁酸(GABA),具有调节血压的临床效果[7-9]。另外,据报道L.paracasei菌株及发酵液还具有抗肿瘤和抗氧化的生理功能[10-11]。L.paracasei的多种生理功能决定了其在不同领域的广阔应用前景。近年来在食品工业中的开发和应用研究报道较多,如:生产酸奶干酪等乳制品的优良发酵菌株开发;具有一定生理功能的经过口或黏膜途径摄入的活菌及其代谢产物的乳酸菌制剂的研制;生产GABA、L-乳酸、苯乳酸发酵菌种筛选;以及干燥、微胶囊等相关产品的加工的工艺研究[12-13]。
联合国粮食与农业组织、世界卫生组织定义益生菌(probiotics)为当摄入一定量时能对宿主产生健康益处的微生物活体[14]。L.paracasei就是一种非常具有应用前景的益生菌。乳酸菌的益生功能与特定菌株和定植肠道内活菌计量密切相关[15]。另外,从加工的角度来看,这些微生物必须适合大规模的工业生产,因此在食品的货架期结束时,其菌含量应达107CFU/g[16]。关于益生菌在食品中的使用,我国发布了一系列的法律法规,2010年卫生部发布可用于食品的菌种名单21种[17],对婴幼儿食品中规定了可用菌株的目录[18],并通过《新资源食品管理办法》(即2013年后《新食品原料安全性审查管理办法》)进行申报批准,截至2020年10月已申报批准15株。随着法规的进一步完善,食品菌种(株),包括发酵菌种的使用范围、数量和安全性要求也将更加明确和规范[19]。
L.paracasei是近年从干酪乳杆菌中划分出来的一个新种,其分类学地位一直存在争议,2008年,国际细菌分类委员会驳回了Dick等将副干酪乳酸菌归入干酪乳杆菌属的分类建议,重新将副干酪乳酸菌及其亚种,从干酪乳杆菌划分出来[20]。仅基于有限的DNA-DNA同源性比较试验,如16SrDNA序列同源性分析,完成的分类及进化分析,很难将副干酪乳杆菌和干酪乳杆菌区分开,需要与表型特征描述,如糖培养发酵试验等,相结合[21-22]。基于全基因组差异的分子生物学分析方法,实现了乳杆菌属到种水平的鉴别,并可以实现株水平的遗传多样性分析及菌株分型[23]。2019年,Stijn Wittouck等通过全基因组分析方法,对NCBI数据库中200多个乳酸杆菌属基因组数据进行种水平分类分析[24]。2020年,Piotr Jarocki等对属于L.casei,L.paracasei和L.rhamnosus3个种的30多株菌进行了属及种的特异性多聚酶链式反应(polymerase chains reaction,PCR)、多重PCR(multiplex-PCR)、实时高分辨溶解曲线分析(Real-Time HRM analysis)、PCR限制性片段长度多态性(restriction fragment length polymorphism-PCR,RFLP-PCR)、基于REP元件PCR指纹法(Repetitive extragenic palindromic elements-PCR,rep-PCR)、DNA随机扩增多态性(Random Amplified Polymorphic DNA,RAPD)、和PCR扩增片段长度多态性(Amplified Fragment Length Polymerphism,AFLP)和基质辅助激光解吸电离飞行时间质谱(Matrix-Assisted Laser Desorption/Ionization Time of Flight Mass Spectrometry,MALDI-TOF MS)等方法进行研究。研究结果显示multiplex-PCR和MALDI-TOF MS可用于种水平鉴别,而AFLP-PCR方法对于株水平鉴别分辨力更强[25]。传统的多态性比较分析方法较为随机,而且其最大的不足是这些标记都与性状没有直接的联系。单核苷酸多态性(single nucleotide polymorphism,SNP)可在干酪乳杆菌群的持家基因中稳定存在,分布密度较高,使得SNP成为理想的基因分型方法[26]。特别是单个碱基的差异可能决定一个基因功能的改变,从而影响所控制的性状。近年来,随着益生菌全基因组测序Whole Genome Sequencing(WGS)技术的发展,乳酸菌的比较基因组学研究得到了发展[27-29],其中功能相关基因及SNP位点分析技术的发展为L.paracasei种内株水平鉴别提供了研究基础。
本项目研究对象为副干酪乳杆菌N 1115(L.paracasei N 1115,简称N 1115),是从内蒙古牧区传统发酵乳制品中分离得到的一株功能性菌株。已进行了全测序,并与L.casei ATCC 334和L.rhamnosus GG进行了比较基因组分析[30]。已进行的功能性研究表明,N 1115不仅具有耐酸、耐胆盐、促进肠道细胞生长等特性,还可以刺激T细胞增殖分化,调节细胞免疫应答过程,在提高机体抗感染能力的同时降低超敏反应的发生[31-32]。而实时荧光定量聚合酶链式反应(Real-time Fluorescence Quantitative Polymerase Chain Reaction,RT-qPCR)技术,具有定量分析特异性高、灵敏度高、重复性良好、准确度高、分析通量大等优点,相关设备普及度较高[33]。故本研究在N 1115比较基因组和基于菌株功能相关的cSNP位点分析基础之上,建立了CDS 247开放阅读框架中株特异性SNP位点探针的RT-qPCR分析方法。并建立了定量检测饮料、菌粉中的N 1115副干酪乳杆菌方法。该方法的建立为益生菌株水平鉴别提供了应用方法借鉴,并为保障相关新食品原料、新资源食品、以及保健食品的申请、生产及质量安全监管的顺利进行提供了理论支持。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种
本研究试验菌株包括15株副干酪乳杆菌(包括分离株N 1115和标准菌株),5株干酪乳杆菌、13株其他类乳杆菌、1株嗜热链球菌、3株乳酸乳球菌及16株双歧杆菌的标准菌株或工业生产用菌株,菌株编号及标准菌株保藏号详见表1。表1菌株购买获赠后均由上海市质量监督检验技术研究院自行保存。
1.1.2 试剂及培养基
溶菌酶,北京索莱宝科技有限公司;蛋白酶K溶液(10 mg/m L),溶菌酶溶液(20 mg/m L),十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB),Na2EDTA,氯仿,异戊醇,无水乙醇,NaCl,中国医药集团;Tris饱和酚,北京索莱宝科技有限公司;TE buffer(p H 8.0),生工生物工程(上海)股份有限公司;Premix TaqTM(TaKaRa TaqTMVersion 2.0),Premix Ex TaqTM(Probe qPCR),ROX plus宝生物工程(大连)有限公司;细菌基因组DNA提取试剂盒(DP302),天根生化科技(北京)有限公司;MRS培养基,北京陆桥技术股份有限公司;所有分子生物学用水均为18 MΩ纯水。
CTAB裂解液(p H 8.0):称取4.00 g CTAB,16.38 g NaCl,2.42 g Tris,1.50 g Na2EDTA,用适量水溶解后,调节p H,定容至200 mL,高压灭菌,存放于4℃备用。
CTAB-II裂解液(pH 8.0):称取1.00 g CTAB,16.38 g NaCl,2.42 g Tris,1.50 g Na2EDTA,用适量水溶解后,调节p H,定容至200 mL,高压灭菌,存放于4℃备用。

表1 试验菌株及编号

(续表1)
1.2 仪器与设备
高速台式离心机(Eppendorf 5417R),德国Eppendorf公司;微量移液器(2μL,10μL,100μL,1 000μL),法国吉尔森公司;微量分光光度计(DeNovix DS-11),美国DeNovix公司;分析天平(Mettle toledo AL204(感量0.0001 g)),梅特勒-托利多(常州)称重设备系统有限公司;厌氧培养系统(ANOXOMAT MARK II),荷兰Mart Microbiology B.V;电热恒温培养箱,美国Shellab公司;ESCO LA2-4A1生物安全柜,新加坡艺思高公司;荧光定量PCR仪(ABI 7500),赛默飞世尔科技(中国)有限公司。
1.3 实验方法
1.3.1 SNP分析
下载GenBank数据库中多个副干酪乳杆菌菌株基 因 组,包 括ATCC_334、BD-II、CAUH 35、EG、FAM 18149、HD1.7、HDS-01、IIA、JCM_8130、KL1、L9、LC2W、LC355、LOCK919、Lpc10、TK1501、TMW、Zhang全基因组序列,以N 1115菌株作为参考菌株,通过变异检测软件SAMtools进行SNP Calling,并对原始结果进行过滤,得到SNP位点分析结果。对差异位点,用R语言转化矩阵,做hclust聚类分析,得到基于SNP位点的聚类分析图。相关分析由上海昂朴生物技术有限公司完成。
1.3.2 糖代谢相关序列在线分析
使用在线RAST[34-36]进行N 1115全基因组序列注释,得到N 1115代谢预测途径。通过NCBI在线Blast对假基因进行分析,用http://www.pathogenomics.sfu.ca/islandviewer/进行N 1115染色体组序列的基因岛分析[37]。
1.3.3 RT-qPCR引物的设计与合成
结合现有N 1115菌株基因组注释信息,寻找位于开发阅读框架中的特异性位点。特异位点前后共约200 bp片段,使用Primer Express 3.0进行引物探针设计,选择探针包含特异性位点的探针引物组。通过引物特异性位点个数及位置,并结合Tm值、引物末端稳定性、GC含量、二级结构等参数,得到30套理论可行的引物及探针,并优选其中的5套引物及探针进行合成。最终经过试验验证,得到一组引物探针组,位于开放阅读框架CDS 247,具体序列见表2。引物由上海英俊生物技术服务有限公司合成。
1.4 DNA模板的提取
裂解液提取法过程如下:样液1 mL,加至2 mL的微量离心管中,12 000 g离心10 min后,加入40μL 20 mg/mL的溶菌酶,37℃孵育30 min后,加入56℃预热的700μL CTAB裂解液(或CTABII裂解液)和10μL蛋白酶K,振荡混匀,置于56℃水浴中处理2 h(或水浴过夜),期间每隔30 min混匀一次;取出冷却至室温后按照核酸提取步骤抽提核酸[38]。试剂盒提取DNA模板过程按照试剂盒说明书进行。煮沸法提取DNA过程为:取样液1 mL加至2 mL的微量离心管中,100℃煮沸5 min后,10 000 g离心10 min后取上清。

表2 N1115扩增引物探针名称、序列及基因来源
1.5 RT-qPCR方法
1.5.1 反应体系
按照Premix Ex TaqTM(Probe qPCR)说明书,配置反应液,包含:Mix反应缓冲液7.5μL,上下游引物终浓度为400μmol/L,探针终浓度为400μmol/L,DNA模板1.0μL,去离子水(DW)补充至15μL。
1.5.2 反应过程
将混合物置于荧光PCR仪中实时监控扩增反应,反应程序为:第一阶段95℃2 min;第二阶段95℃5 sec,64℃35 sec 40个循环;于64℃35 sec处收集荧光信号,荧光通道为FAM/none。
1.6 RT-qPCR检测方法验证
1.6.1 扩增引物的特异性及传代稳定性测试
对目标序列进行在线比对分析方法的特异性,并分别提取15株副干酪乳杆菌菌株(种内株间)、31株近缘及生产常见乳酸菌(种间)、以及10株其他生产常见益生菌基因组DNA为模板。采用所设计的引物对上述各种DNA分别进行扩增,确定检测方法对N 1115菌株的特异性。以无菌水作为阴性对照。N 1115连续传代3至8代得到的菌液提取DNA,调节模板浓度一致,分析Ct值差异性。没有显著差异性,表明特异性序列传代稳定性好。
1.6.2 N 1115染色体DNA RT-qPCR方法的检出限、精确度及线性检测
对N 1115提取的染色体DNA溶液进行稀释,核酸质量浓度依次为50、5、0.5、0.05、0.005、0.0005、0.00005 ng/μL,进行RT-qPCR扩增。每个质量浓度设置10个平行,计算Ct值的标准偏差(Standard Deviation,SD)和相对标准偏差(relative standard deviation,RSD)。确定方法检出限、精确度及线性。以无菌水作为阴性对照样品。
1.6.3 N 1115菌悬液定量检测方法的确度、线性、及定量检出限
对N 1115菌悬液的6个浓度梯度进行DNA提取和扩增,每个菌浓度10个平行,计算Ct值的SD和RSD。确定方法的检出限、精确度及线性。同时参考菌液的常规菌落计数结果,得到方法的准确度。
1.6.4 N 1115菌体定量检测方法的专属性验证
专属性验证包括在干扰菌专属性和基质专属性,具体试验如下。
(1)干扰菌专属性验证。
在高浓度干扰菌两歧双歧杆菌存在的情况下,制备N 1115菌的6个浓度梯度按照CTAB裂解方法提取DNA后,以提取液原液为模板进行RT-qPCR扩增,做3个平行。考察所建方法的准确度、线性及检出限。
(2)基质专属性验证。
制备N 1115菌液,以企业提供的3种菌粉溶液为稀释液进行稀释。稀释6个菌浓度梯度,按照CTAB裂解方法提取DNA后。以DNA提取液原液为模板,进行RT-qPCR扩增。以传统培养计数方法进行菌液的活菌计数为参考,比较3种菌粉中N 1115菌株计数结果与传统培养计数结果的差异。
1.7 模拟样品检测
利用建立好的RT-qPCR技术对模拟样品进行检测,模拟样品的制作:N 1115培养至109CFU/m L,以不含N 1115的菌粉和饮料作为基质,进行梯度稀释,最终将稀释液进行核酸提取,以该核酸为模板扩增。每个浓度进行3个平行。
1.8 数据与图像处理方法
实验使用的仪器自带软件7500 System Software v1.3.2在扩增过程中自动进行分析并得出Ct值。
1.9 N 1115菌粉实际样品检测过程
企业提供N 1115菌粉及其产品不同批次共2份,按建立的RT-qPCR方法对样品进行检测。每个样品4个平行。取标准菌株N 1115菌液109CFU/mL进行10倍系列稀释5个浓度。检测样品根据标签明示值,称取0.1g(A)样品至10 mL(B)的无菌稀释液,并用无菌稀释液进行10倍稀释,使菌粉样本浓度在105~109CFU/mL之间,记录稀释倍数(C)。标准菌悬液和样品溶液在同样的条件下使用CTAB-II法提取核酸。以标准菌浓度对数值(lg Co)为x轴,Ct值为y轴作图,得到标准线性拟合曲线及公式。样品Ct平均值,带入线性拟合公式得到菌浓度对数值x。样品中菌浓度按照以下公式计算:
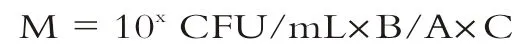
式中:M为样品中菌量(CFU/g);A为检测样品称取质量(g);B为稀释液体积(mL);C为样品菌液稀释倍数。
2 结果与分析
2.1 基因组特异性序列分析
2.1.1 L.paracasei菌株SNPs位点及聚类分析
比较公共资源可获得的20株L.paracasei基因组序列,得到9219个N 1115特异性SNPs位点。SNP位点聚类分析结果显示,分析的20株菌L.paracasei由聚类成3簇,包括菌株L9、IIA、LC355、TMW、ATCC334、EG、X 8700.2、HD1.7、HDS.01、LOCK919、Lpc10、Zhang和TK1501(第1簇);菌株FAM 18149、CAUH 35、JCM 8130、KL1、LC2W、BD.II(第2簇);菌株N 1115(第3簇),如图1。SNPs聚类分析显示N 1115与其他株相似度较低。据报道,SNP分析在一定程度上可以表现L.paracasei种内的进化过程,但由于这种突变有可能是种间重组造成的,也可能造成进化上分析的误差,所以并不能完全反映菌株的进化关系。
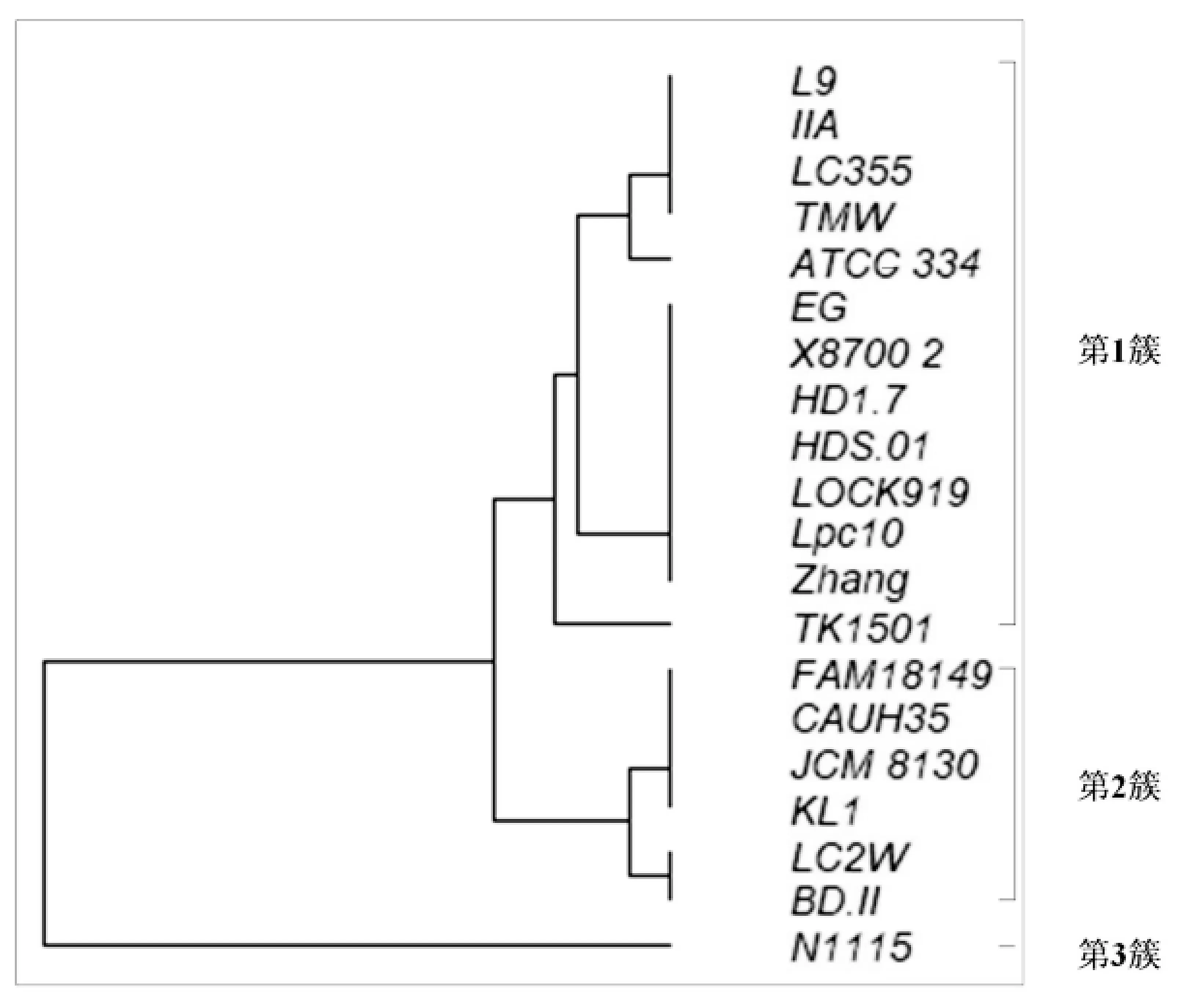
图1 公共资源获得的20株L.paracasei中SNP位点预测及聚类分析
2.1.2 代谢功能性相关基因及SNP位点选择
经过对N 1115全基因组注释,对N 1115株代谢途径进行了预测如图2。注释基因占全基因组23%。其中糖代谢、蛋白代谢以及氨基酸及其同系物代谢分别对应功能基因个数163、131和131,分别占注释基因总数(935)的17.43%、14.01%和14.01%。乳酸菌可利用的碳源种类与环境或在肠道中的生态位适应密切相关[27]。生态位碳源代谢能力对于乳酸菌益生功能的发挥及相关生产能力的开发非常关键。相关的研究报道指出,基因水平转移事件在细菌基因组引入多种功能,从而增强细菌在自然环境中的竞争力。编码糖转运蛋白和碳水化合物水解的基因是水平转移获得的大部分菌株水平特异性基因的代表[39]。另外,昱宿主作用相关的胞外定位蛋白也与生态位适应性有关[28],但没有相关研究显示其对应基因差异具有菌株水平的特异性。
关于L.paracasei泛基因组相关研究指出,糖盒在L.paracasei基因组中的分布及其多变,特别是在糖岛A和B中,泛基因组显示共有74个糖盒,但各个菌株含有糖利用盒的数量差异很大。只有15个糖盒属于核心基因组,且位于糖岛A和B之外。L.paracasei的进化复杂,并不总是与生态位适应有关,预测可以利用的糖源为果糖、蔗糖、肌醇、葡萄糖酸盐、氨基半乳糖、山梨醇、纤维二糖、核糖、甘露糖、甘露醇、山梨糖、木糖、半乳糖苷、β-葡萄糖苷、α-葡萄糖苷、葡萄糖苷、透明质酸盐、半乳糖醇、抗坏血酸和某些未知糖源[29]。除了乳酸菌常规的乳糖和半乳糖途径、糖原代谢途径、以及甘露糖、D-核糖、脱氧核糖等单糖代谢途径外,N 1115还预测出较为独特的糖代谢途径,包括几丁质(Chitin)/N-乙酰葡萄糖氨(NAG)代谢途径、D-葡萄糖酸和酮葡萄糖酸盐单糖代谢途径。

图2 N1115代谢途径预测
N 1115株预测得到的Chitin/NAG代谢途径,含有5个代谢相关基因,包括2个NAG核心基因nag A和nag B。预测结果显示,该途径没有NAG激酶,也没有NAG磷酸转运系统(phosphotransferase system,PTS)。推测可能与唾液酸的利用有关,即该途径与NAG-P的NAG利用途径进行了合并。而经过进一步比对,CDS 247与N-乙酰氨基葡萄糖的PTS转运系统的subunit IIB序列十分相似,但仅有417 nt碱基,翻译产物为139 aa,而典型的具有糖转运活性的nag E基因约为1,947 nt,产物包括ABC三个亚单位,大小约为648 aa。分析该CDS 247可能为序列上被删减的没有功能的假基因。细菌基因组中的假基因是在不断发生的突变过程中不断产生的,并且会随着突变的进一步积累而退化,最终被去除。与真核生物进化尺度上的假基因保守性截然不同,细菌假基因的保留时间似乎非常短,即使与非常接近的细菌相比,属于同一种细菌的菌株之间也很少有相同的假基因[40]。本次实验,仅进行了序列比对,并未进行进一步的功能性研究。但CDS 247在多株L.paracasei中都有发现,说明该基因对于L.paracasei来说遗传较为稳定,位于该基因的SNP位点,适合用于种内菌株鉴别。经分析该基因并不位于基因岛(Genomic island),说明该序列水平转移获得可能性较低,遗传上也更为稳定。
2.2 引物特异性及传代稳定性验证
2.2.1 引物特异性验证及传代稳定性
使用设计的引物对15株L.paracasei种内株进行扩增(图3)。扩增结果显示,除N 1115外的其他株扩增均为阴性,N 1115株扩增为阳性,对应两平行样品Ct值分别为19.0297和20.2145。41株其他种乳杆菌和食品及保健品常见益生菌扩增结果均为阴性,空白对照均未发生扩增。这表明本项目所设计引物对N 1115菌株检测具有很好的特异性(表3)。
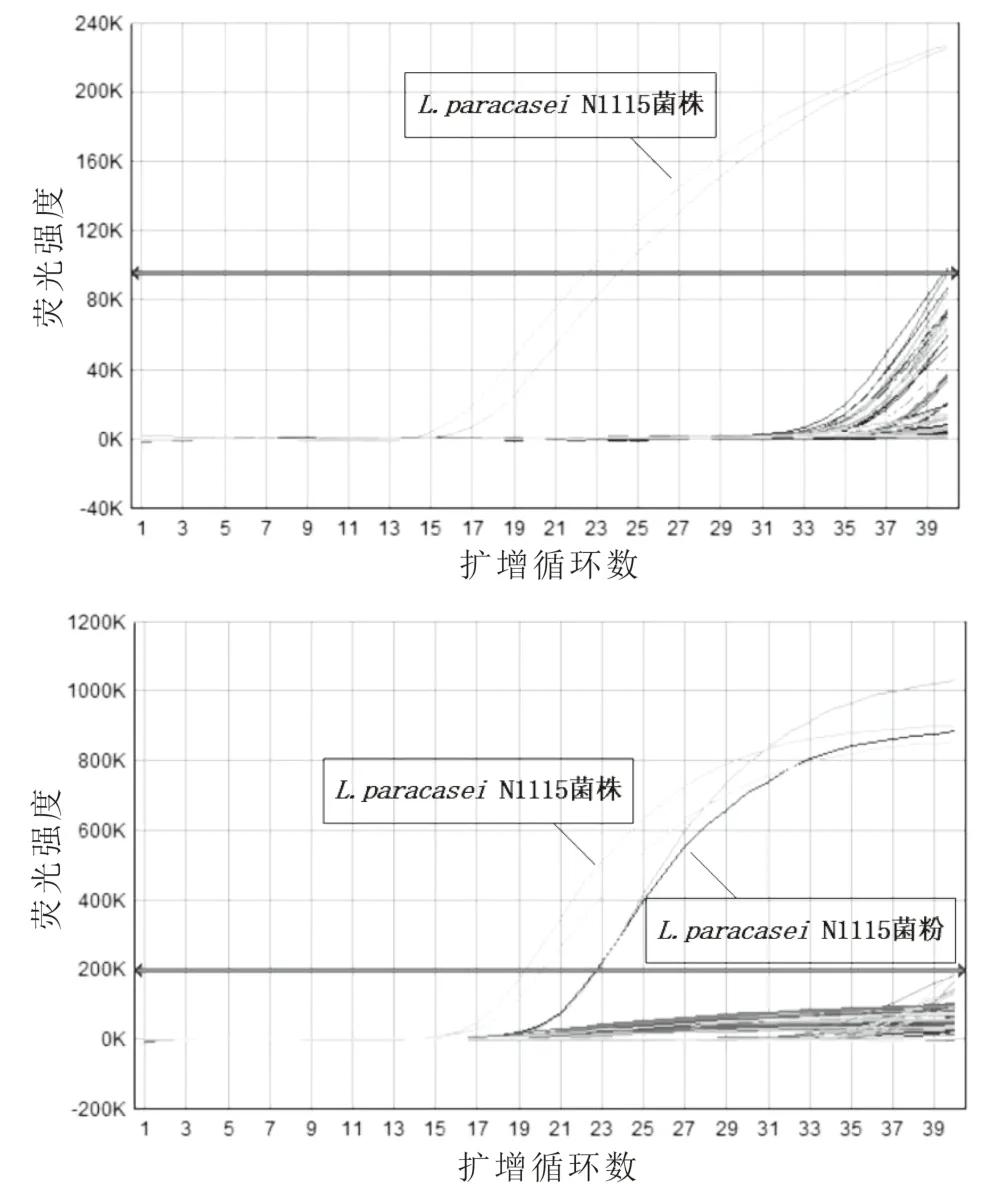
图3 N1115DNA扩增引物特异性
N 1115菌株传代3到8次,特异性检测Ct值相对标准偏差为2.89%,方法对于传代多次的检测精确度高,特异性扩增的传代稳定性好,可用于有限代数菌株的检测(表4)。
2.3 N 1115菌液定量检测方法的检出限、线性检测及精确度
用9.9×108CFU/mL菌液提取的DNA溶液用无菌水进行10倍系列梯度稀释,稀释至对应菌浓度为99 CFU/mL。每个浓度3个平行。扩增结果显示对应菌浓度990 CFU/mL及以上时,RT-qPCR为扩增阳性。以菌浓度对数值(Log[菌浓度])为x轴,Ct值为y轴作图,做线性拟合得到公式y=-3.6487x+46.716,R2=0.9993,线性拟合度好。这种情况下,定量检出限可达到990 CFU/mL,见图4。

表3 特异性检测结果
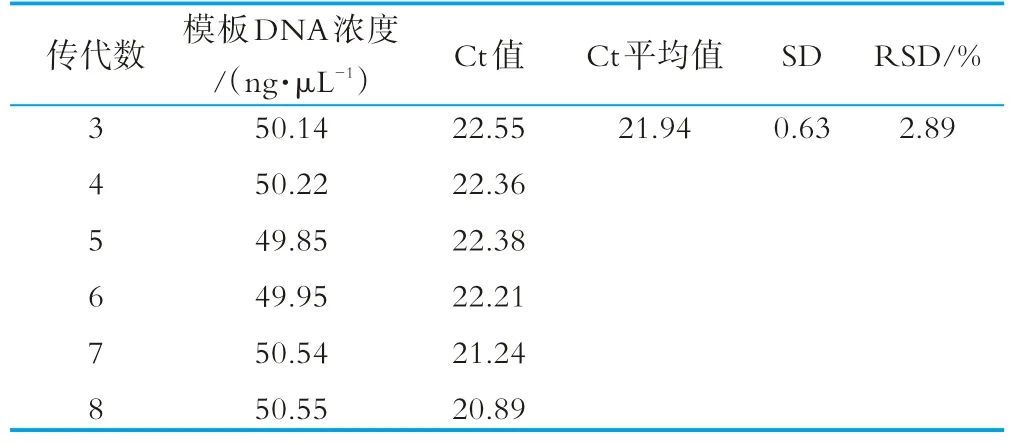
表4 特异性扩增的传代稳定性验证
同时,用无菌生理盐水对厌氧培养得到的菌液做10倍系列稀释,得到6个浓度梯度的菌液。取菌液1 mL,采用CTAB法进行DNA提取。提取后DNA为模板进行RT-qPCR扩增。每个菌浓度10个平行,同时对菌液进行培养计数。扩增结果显示,菌浓度大于等于9.9×103CFU/mL时,每个菌浓度的10个检测结果均为阳性。Ct值的SD介于0.29~0.66之间,RSD介于1.03%~3.02%之间,均在可接受范围内。证明建立的RT-qPCR方法的DNA浓度定量精确度较好。以菌浓度对数值(Log[菌浓度])为x轴,Ct值为y轴作图,做线性拟合得到公式y=-3.9332x+51.006,R2=0.9984,线性拟合度好。定量检出限可达到9.9×103CFU/m L,见图4。
在B.bifidum为4.8×107CFU/mL时,N 1115菌株的10倍系列稀释液采用CTAB裂解液提取DNA后,进行扩增。每个梯度3个平行。各浓度Ct值的SD介于0.33~0.57之间,RSD介于1.06%~3.05%之间,与未加干扰菌的情况相似。扩增结果显示:对应菌浓度2.9×104CFU/mL及以上时,RT-qPCR扩增为阳性。以菌浓度对数值(Log[菌浓度])为x轴,Ct值为y轴作图,做线性拟合得到公式y=-4.0903x+52.961,R2=0.983,线性拟合度好。这种情况下,定量检出限可达到2.9×104CFU/mL,见图4。

图4 N 1115菌液梯度稀释检测结果及线性拟合
通过比较梯度浓度N 115菌株DNA,梯度浓度N 1115菌液提取DNA和添加高浓度(至107CFU/mL)干扰菌的梯度浓度N 1115菌液提取的DNA,分别为模板扩增得到的线性拟合曲线可以看出,3组曲线线性拟合度较好(R2>0.98)。菌液的拟合曲线位于DNA直接为模板的拟合曲线之上,说明菌液DNA的提取效率不能达到100%,而且菌液的拟合曲线的斜率绝对值相较于DNA直接为模板的拟合曲线也有增加,这说明随着菌浓度的减少,DNA提取效率呈逐渐降低的趋势。提示qPCR方法并不能实现样品中N 1115菌的绝对定量,需要对照已知菌浓度的标准品进行换算。
2.3.1 针对菌粉基质的提取方法优化
提取方法的稳定性对于菌的定量检测的准确度也非常重要,故通过考察提取DNA质量和浓度相对偏差对DNA提取方法进行了选择优化。选择较低浓度菌液(约105CFU/mL)用4种方法提取DNA优化结果显示,使用CTAB-II裂解液的方法菌粉DNA产物质量好(A260/A280约为2)、准确度在4种方法中最好(4次提取得到的DNA浓度RSD最低),试剂盒在菌浓度为105CFU/mL时提取回收率较低,数据未列出,见表5。
以菌浓度对数值(Log[菌浓度])为x轴,分别以CTAB-II裂解液方法提取的DNA和试剂盒提取DNA为模板进行扩增的Ct值为y轴作图,线性拟合分别得到公式y=-3.68x+48.478,R2=0.9994和y=-3.9588x+45.899,R2=0.9872,线性拟合度较好,但曲线斜率为-3.9588,绝对值超了出RT-qPCR理论斜率值。一般来说,试剂盒提取DNA纯度较高,不易产生反应体系抑制现象。故有可能随着菌浓度的降低,试剂盒提取得率降低,导致DNA浓度偏低,反映到扩增效果上为斜率与理论值不符。故在菌浓度>104CFU/mL时,CTAB-II和试剂盒法两种提取方法得到的模板DNA,定量分析结果准确。菌浓度<104CFU/mL时,改良CTAB法提取得到的模板DNA准确度更高。
2.3.2 模拟样本检测
使用CTAB-II裂解法提取模拟菌粉DNA,并进行RT-qPCR扩增结果(图5)显示:对应菌浓度2.0×104CFU/mL及以上时,RT-qPCR扩增为阳性。以菌浓度对数值(Log[菌浓度])为x轴,Ct值为y轴作图,各菌粉基质中梯度加入N 1115菌株线性拟合得到公式为:L45植物乳杆菌菌粉基质中y=-3.505x+48.107,R2=0.9981;植物乳杆菌菌粉基质中y=-2.9183x+42.496,R2=0.9838;嗜酸乳杆菌菌粉基质中y=-3.4358x+46.185,R2=0.9994;3种菌粉线性拟合度好,定量检出都可达到2.0×104CFU/mL,且扩增效率较稳定。
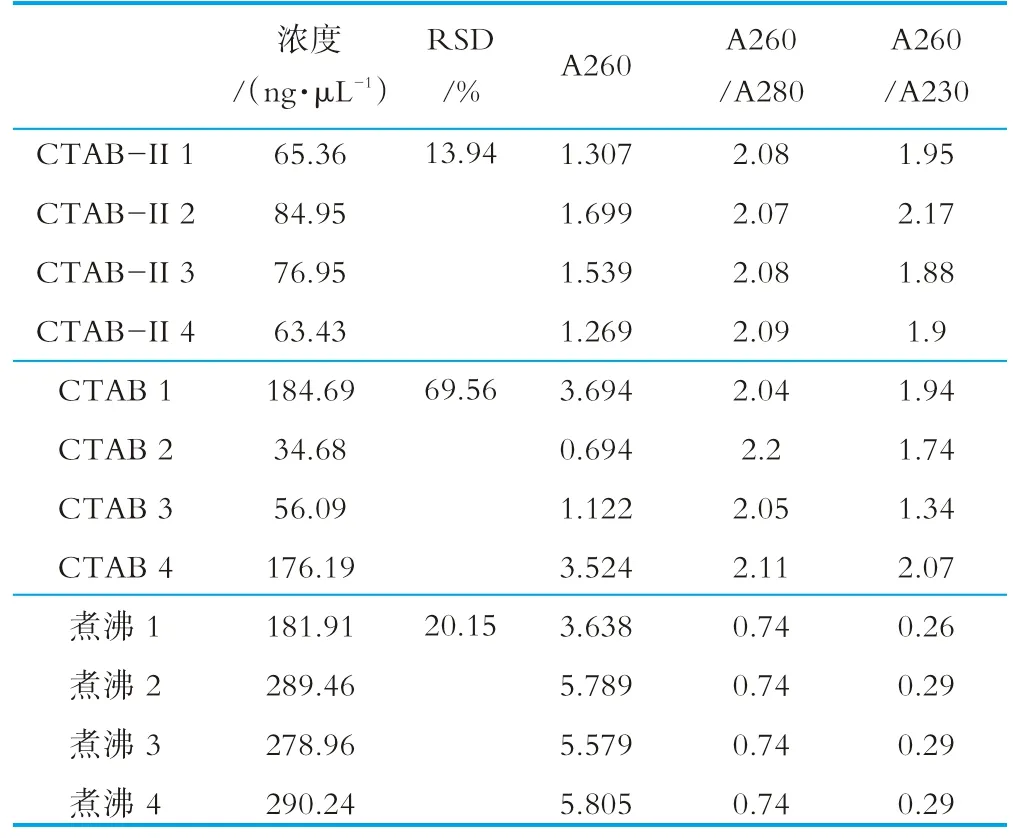
表5 DNA提取方法比较

图5 CTAB-II裂解法提取各种菌粉中DNA的N1115特异基因扩增Ct值及线性拟合
2.3.3 N 1115菌粉实际样本检测结果
标准曲线拟合方程为:y=-3.68x+48.478,R2=0.9994。菌粉1称取0.1 g至10 mL,稀释100倍后,检测4平行Ct值分别为23.25,23.91,23.40,23.02平均为23.40,带入标准曲线拟合方程得到x=6.81,故菌粉1中N 1115菌量M=106.81CFU/m L×10 m L/0.1 g×100=6.46×1010CFU/g;菌粉2称取0.1 g至10 mL稀释10倍后,检测4平行Ct值分别为21.72,22.72,22.30,22.20平均为22.24,带入标准曲线拟合方程得到x=7.13,故菌粉2中N 1115菌量M=107.13CFU/mL×10 mL/0.1 g×10=1.35×1010CFU/g。计算结果与产品明示值相符,建立的方法对含有大量低聚糖及辅料的菌粉样品适用。
3 结 论
每种益生菌的独特的益生功能与一定数量的特定菌株密切相关。方便快速的益生菌株水平特异性鉴别和定量检测方法对于益生功能评价,新食品原料的申请,新资源食品研制和申报,以及后续产品的质量安全控制不可或缺。针对株功能性的位点的特异性PCR目前仍是方法开发的首选方法。SNPs位点的分析可以在一定程度上体现株间的进化关系,是寻找株水平特异性的一个切入点。结合对菌株的特殊功能的不同需求,通过生物信息学手段,对功能性基因、水平转移基因、假基因等多种进化来源中SNPs位点进性筛选,结合目的菌株益生功能、生境、进化特点,找到较为稳定的突变位点,可以有效地简化益生菌株水平的特异性位点分析过程,从而建立较为可靠的菌株检测方法。本研究针对糖代谢功能基因上的SNPs位点分析,建立RT-qPCR检测方法,实现了菌粉中N 1115株水平的定量检测,方法特异性、精确度和稳定性较好。随着对益生功能基因水平研究的不断深入,与菌株独特表型相关的功能基因、转录表达调控等多特异性位点的分析和筛选,将有助于菌株功能、活性、定植能力等多角度的量化评价方法的开发,进一步促进益生菌产业的健康发展。
