高级别卵巢浆液性囊腺癌差异基因的生物信息挖掘
2021-05-08车莹莹白米雪卢坤傅琳
车莹莹 白米雪 卢坤 傅琳
[摘要]目的利用生物信息學的方法筛选高级别卵巢浆液性囊腺癌(HGSC)的差异表达基因(DEGs),并从基因水平挖掘这些DEGs在HGSC中发挥的潜在作用。方法从GEO数据库中下载GSE10971、GSE14001、GSE18521、GSE27651、GSE12470数据集,运用R软件和Bioconductor安装包筛选HGSC组织中与正常组织相比上调的DEGs和下调的DEGs,对这些基因分别进行基因本体(GO)富集分析和KEGG通路分析,蛋白质相互作用(PPI)网络分析及预后生存分析,并运用网络分析插件(CytoHubba)筛选关键基因,最后通过Kaplan-Meier plotter数据库分析筛选出的关键基因的表达与HGSC病人生存预后的关系。结果从GEO数据库中筛选出134个DEGs,其中94个上调DEGs作为细胞质的组成成分,与蛋白质二聚活性有关,参与细胞内代谢过程的调控和细胞周期的调控;40个下调DEGs主要以细胞外基质成分居多,并且大多具有poly(A)聚合活性,参与肿瘤信号通路的调控。筛选出的6个上调关键基因BUB1B、CENPF、BIRC5、UBE2C、ASPM、TOP2A与病人预后有显著相关性(r=0.87~1.55,P<0.05)。结论筛选出的DEGs参与了HGSC发生发展的分子功能,其中的关键上调基因BUB1B、CENPF、BIRC5、UBE2C、ASPM、TOP2A可能对HGSC的临床治疗及预后判断具有潜在的指导价值。
[关键词]卵巢肿瘤;囊腺癌,浆液;计算生物学;基因本体;蛋白质相互作用图;预后
[中图分类号]R737.31[文献标志码]A[文章编号]2096-5532(2021)01-0019-06
[ABSTRACT]ObjectiveTo screen out the differentially expressed genes (DEGs) in high-grade ovarian serous cystadenocarcinoma (HGSC) using the bioinformatics method, and to investigate the potential role of these DEGs in HGSC at the gene level. MethodsGSE10971, GSE14001, GSE18521, GSE27651, and GSE12470 datasets were downloaded from gene expression database (GEO), and R software and Bioconductor installation package were used to screen out the upregulated and downregulated DEGs in HGSC tissue compared with normal tissue. These genes were analyzed by Gene Ontology (GO) enrichment analysis, kyoto Encyclopedia of Genes and Genomes (KEGG) pathway analysis, protein-protein interaction (PPI) network analysis, and prognosis survival analysis, and the network analysis plug-in (CytoHubba) was used to screen out hub genes. Finally, the Kaplan-Meier plotter database was used to analyze the association of the expression of hub genes with the survival and prognosis of HGSC patients. ResultsA total of 134 DEGs were screened out in GEO database, among which 94 upregulated DEGs were cytoplasmic components associated with protein dimerization activity and were involved in the regulation of intracellular metabolism and cell cycle, and 40 downregulated DEGs were mainly the components of extracellular matrix, most of which had poly (A) polymerization activity and were involved in the regulation of tumor signaling pathways. Six upregulated hub genes, i.e., BUB1B, CENPF, BIRC5, UBE2C, ASPM, and TOP2A, were significantly correlated with the prognosis of patients (r=0.87-1.55,P<0.05). ConclusionThe DEGs screened out are involved in the molecular functions of the development and progression of HGSC, and the upregulated hub genes, i.e., BUB1B, CENPF, BIRC5, UBE2C, ASPM, and TOP2A, may have a potential value in guiding clinical treatment and prognostic evaluation of HGSC.
[KEY WORDS]ovarian neoplasms; cystadenocarcinoma, serous; computational biology; gene ontology; protein interaction maps; prognosis
卵巢浆液性囊腺癌(OV)是比良性浆液性囊腺瘤和交界性浆液性囊腺瘤(SBT)严重的一种卵巢上皮性癌亚型[1]。根据美国KURMAN教授提出的卵巢癌“二元模型”理论,可以将OV分为两种类型:Ⅰ型的低级别浆液性囊腺癌(LGSC)和Ⅱ型的高级别浆液性囊腺癌(HGSC)[2]。目前认为,HGSC发病起源于输卵管,与LGSC在分子学和组织学水平上存在明显差异[3-4]。相较于LGSC,HGSC具有发病年龄较晚(55~65岁)、发病率高、生存率低、对化疗药物敏感性高且易复发等特点,因此对HGSC预后判断和治疗策略的深入研究也显得尤为迫切。本研究运用生物信息学的方法,从GEO(Gene Expression Omnibus)数据库获取OV基因芯片数据,从中挖掘HGSC的差异表达基因(DEGs),进行基因本体(GO)富集分析和KEGG信号通路分析,构建蛋白质相互作用(PPI)网络,筛选出关键基因,并分析关键基因表达与HGSC预后的关系,从而为HGSC的靶向治疗提供一定的理论依据。
1资料与方法
1.1数据来源
从GEO数据库(http://www.ncbi.nlm.nih.gov/GEO/)中检索并下载的OV相关数据集有5个(GSE10971、GSE14001、GSE18521、GSE27651、GSE12470)[5-6],其中前4个数据集对应的检测平台为GPL570,而最后1个数据集对应的检测平台为GPL887。在每个GSE数据集中,只选择HGSC样本以及与之匹配的正常样本数据。其中GSE10971数据集中包含肿瘤样本13个和正常样本12个,GSE14001数据集中包含肿瘤样本10个和正常样本3个,GSE18521数据集中包含肿瘤样本53个和正常样本10个,GSE27651数据集中包含肿瘤样本22个和正常样本6个, GSE12470数据集中包含肿瘤样本35个和正常样本10个[7-11]。利用GEO2R (http://www.ncbi.nlm.nih.gov/geo/geo2r)分析工具进行在线分析,将结果汇总在Excel表格中,去除没有基因名称或基因探针以及同一个基因对应多个基因探针的数据。
1.2DEGs的筛选
使用R 3.6.2软件(https://www.r-pro-ject.org/)中的edgeR包对数据进行标准化处理,之后对数据进行筛选。筛选标准如下:P<0.01,差异倍数logFC≥1或≤-1[12]。然后再对筛选出的DEGs进行火山图的可视化分析。
1.3上调基因和下调基因的筛选
将上一步筛选的DEGs数据,按照logFC>1为上调基因的标准、logFC<-1为下调基因的标准,进行再次筛选。然后,将5个数据集中的上调基因或下调基因全部导入Bioinformatics & Evolutionary Genomics(http://bioinformatics.psb.ugent.be/webtools/Venn/)在线数据库中,以寻找5个数据集中上调基因或着下调基因的交集[13]。
1.4GO和KEGG富集分析
利用DAVID 6.8(Database for Annotation,Visualization and Integrated Discovery, https://david.ncifcrf.gov/)数据库分析基因组规模数据集的生物信息,并进行基因和蛋白质的功能信息的可视化[14]。GO分析用于分析大量注释基因的生物学过程、分子功能及细胞组成[15]。KEGG(Kyoto Encyclopedia of Genes and Genomes)分析是从分子水平上了解基因和蛋白质所参与的信号通路和生物学功能。GO和KEGG富集分析均以P<0.05为差异有统计学意义。
1.5PPI网络的构建及关键基因的筛选
将全部的DEGs导入String数据库(http://string-db.org)[16]中进行分析,以置信度≥0.4为PPI显著。将分析结果导入Cytoscape 3.7.2软件中进行可视化分析[17]。应用Cytoscape软件中的cytoHubba插件从PPI网络中筛选关键基因,选择度定位≥12的DEGs作为关键基因。
1.6关键基因的生存预后分析
通过在线生存分析工具Kaplan-Meier plotter(http://kmp lot.com/analysis/),根据上述筛选条件,按照关键基因排名从上至下进行生存预后分析,评估每个关键基因在OV中的预后意义[18]。根据基因的表达中值,将病人样本分为两组(高表达组和低表达组)进行分析,参数设置为默认,以P<0.05为差异有统计学意义。
2结果
2.1从5个数据集中筛选出的DEGs
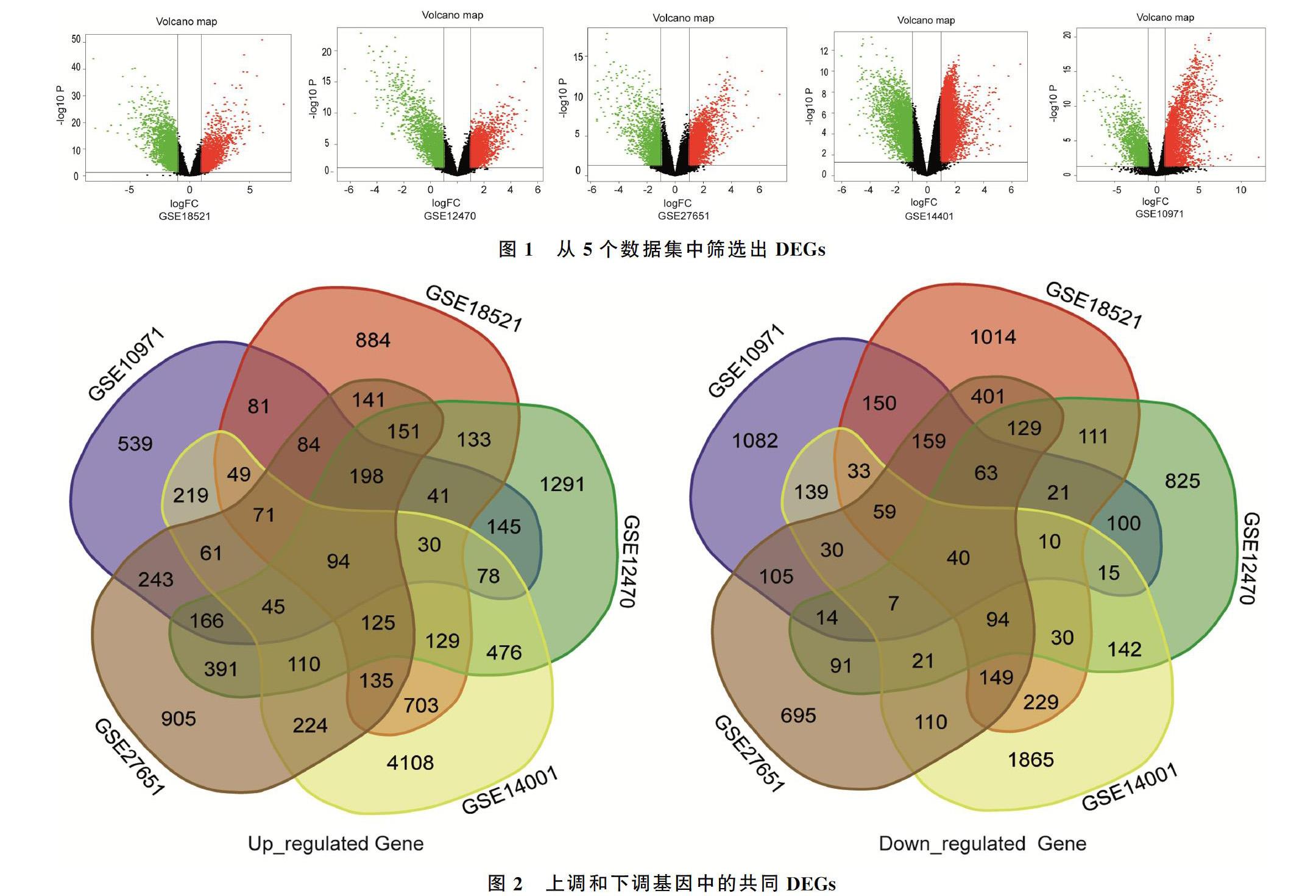
本文从GSE18521数据集中筛选出了6 669个DEGs(共有45 118个基因),从GSE12470数据集中筛选出了6 068個DEGs(共有18 819个基因),从GSE27651数据集中筛选出了6 593个DEGs(共有45 118个基因),以及从GSE14001数据集中共筛选出了12 408 个DEGs(共有45 118个基因),从GSE10971数据集中筛选出了5 612个DEGs(共有45 118个基因),其结果通过火山图直观展示,红色代表高表达基因,绿色代表低表达基因,黑色表示表达水平差异并不显著的基因(图1)。
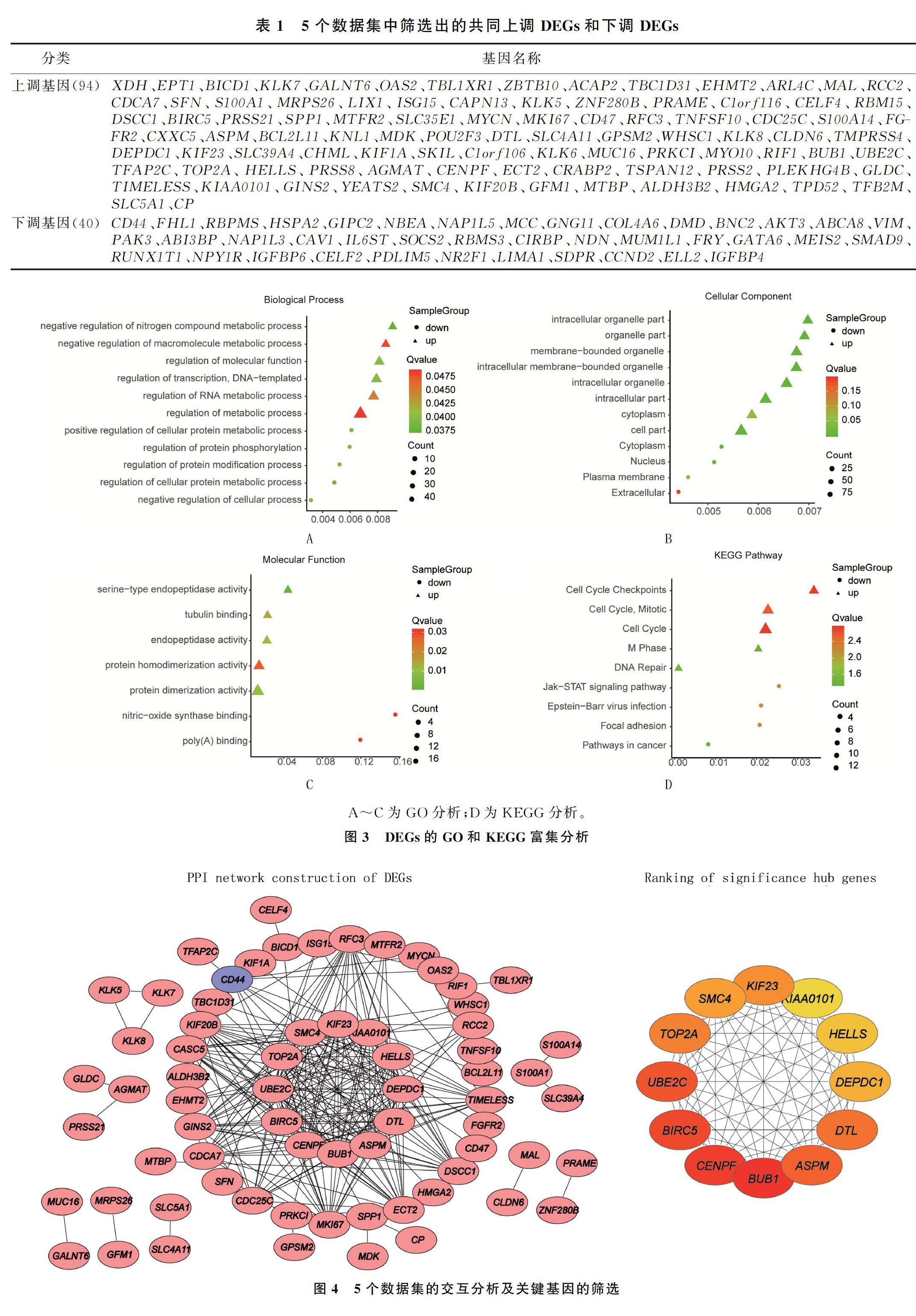
进一步对5个独立数据集进行交集分析,找出5个数据集的共同DEGs,其中表达上调基因94个(logFC>1,P<0.05),下调基因为40个(logFC<-1,P<0.05)(图2),具体的基因名称见表1。
2.2DEGs的GO和KEGG富集分析
在生物过程上,上调DEGs大多参与RNA代谢过程和其他代谢过程的调节,RNA转录和DNA模板的调控以及分子功能调节,大分子代谢过程和氮化合物代谢过程的调节;而下调DEGs大多参与细胞过程、细胞蛋白质代谢过程、蛋白质修饰过程和蛋白质磷酸化过程的调控(图3A)。在细胞成分上,上调DEGs属于细胞内细胞器成分、膜结合细胞器成分、细胞内膜结合细胞器成分、细胞质成分抑或属于细胞外成分;而下调DEGs分布于细胞核、细胞外基质、细胞质和质膜上(图3B)。在分子功能上,上调DEGs一般具有丝氨酸型内肽酶活性、蛋白质二聚活性、内肽酶活性、微管蛋白结合和蛋白质均聚活性等;而下调DEGs一般具有polyA结合功能和一氧化氮合酶结合功能(图3C)。在KEGG信号通路上,上调DEGs多数参与细胞周期及细胞周期中的有丝分裂过程,细胞周期检验点过程,DNA修复和M期信号途径;而下调DEGs多参与STAT信号通路、黏附斑粘连途径、Epstein-Barr病毒感染和肿瘤信号途径等(图3D)。
2.35个数据集的交互分析及关键基因的筛选
为了从系统角度发现和分析相关DEGs之间的相互作用,通过String在线数据库分析得到5个数据集的134个DEGs之间的PPI交互网络(图4A)。在PPI网络中,存在一些基因能夠与其他基因发生强的相互作用,而往往这些基因还处于PPI网络中的关键位置,因此被称为关键基因,它们也被认为是疾病发生的潜在驱动因子[19]。为找出导致HGSC发生的关键基因,我们使用Cytoscape软件插件过滤出69个DEGs,再根据排名筛选出前12个关键基因,颜色由红至黄,红色越深表示关键基因在PPI中具有的作用越大(图4B)。
2.4关键基因的生存预后分析
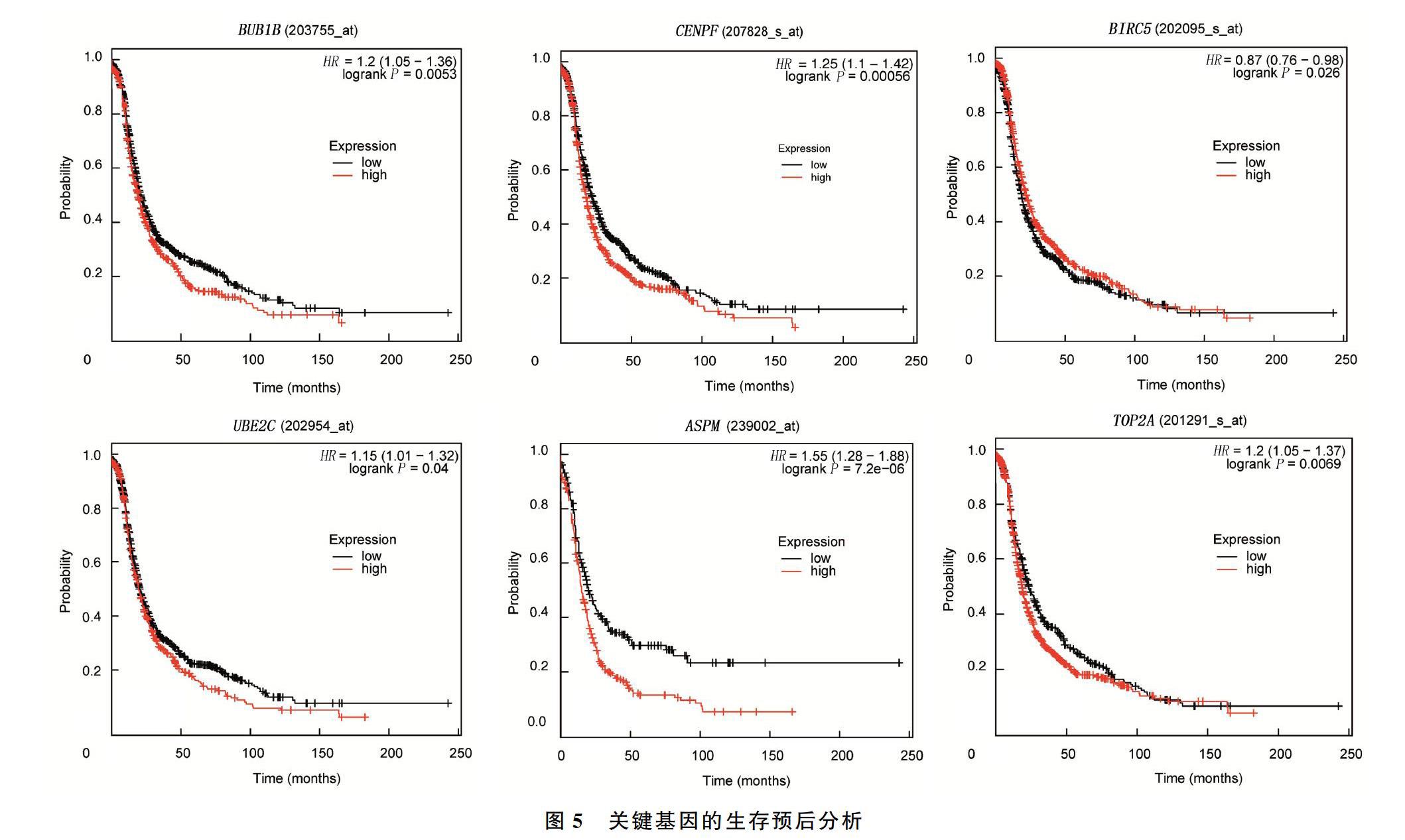
通过Kaplan Meier-plotter网站对筛选出的12个关键基因进行生存预后分析,其中6个基因对HGSC预后有显著影响,分别为BUB1B(r=1.20,P<0.05)、CENPF(r=1.25,P<0.05)、BIRC5(r=0.87,P<0.05)、UBE2C(r=1.15,P<0.05)、ASPM(r=1.55,P<0.05)、TOP2A(r=1.20,P<0.05)(图5)。这些上调基因的高表达会显著降低HGSC病人的生存率。
3讨论
目前认为,LGSC由卵巢上皮性包涵体(OEI)至良性囊腺瘤再至SBT连续发展而来,而HGSC由输卵管远端发展而来,即使二者在起源上有相似之处,但目前普遍认为,两种疾病在临床上具有不同的病理特征,这意味着寻找能鉴别LGSC和HGSC的肿瘤标志物极为重要[20]。
有研究表明,50%的HGSC与DNA修复缺陷有关[21]。根据GO和KEGG富集分析,本研究显示上调DEGs参与DNA模板的调控和DNA修复,这可以作为寻找HGSC靶基因的依据。之后通过生物信息学分析找到6个与预后显著相关的基因,这6个基因在HGSC中都表现为表达上调。有研究结果表明,BUB1B基因的GLEBS结构域对治疗胶质母细胞瘤有重要作用,并且PTTG3P-FOXM1-BUB1B信号轴上调成为肺腺瘤的治疗靶点[22-23];CENPF基因相关级联信号轴的失调促进前列腺癌的转移[24];BIRC5基因的高表达对淋巴瘤的细胞活力具有重要作用,使用相关药物降低BIRC5在淋巴瘤中的表达具有潜在靶向治疗作用[25];在高风险的乳癌病人中,UBE2C基因高表达者具有不良预后[26];ASPM基因可作为肝细胞癌血管侵袭、早期复发和预后不良的新型标记物[27];在早期乳癌病人中检测到TOP2A基因表达异常[28]。目前研究发现BUB1B基因在高级别肿瘤疾病中的表达较高,并与长期预后有关[29],这与本研究生物信息学分析的结果一致。虽然上述基因在卵巢癌中的研究甚少,但是根据它们在其他肿瘤中的研究,我们猜测这些基因处于肿瘤信号通路的某个关键节点上,影响机体的正常生理功能,从而引起肿瘤的发生。
综上所述,本研究通过对5个数据集进行生物信息学分析,挖掘出了与HGSC有关的DEGs共134个,其中与HGSC预后显著相关的基因6个,这6个基因可能对HGSC的临床治疗及预后判断具有潜在的指导价值,并为后续的实验研究提供新的思路。但是,对于本研究筛选出的这些基因是否能够有效鉴别LGSC和HGSC,还需要在今后的研究中进一步探讨。
[参考文献]
[1]MCCLUGGAGE W G. Morphological subtypes of ovarian carcinoma: a review with emphasis on new developments and pathogenesis[J]. Pathology, 2011,43(5):420-432.
[2]SHIH I M, KURMAN R J. Ovarian tumorigenesis: a proposed model based on morphological and molecular genetic analysis[J]. Am J Pathol, 2004,164(5):1511-1518.
[3]KURMAN R J. Origin and molecular pathogenesis of ovarian high-grade serous carcinoma[J]. Ann Oncol: Off J Eur Soc Med Oncol, 2013,24 Suppl 10:x16-x21.
[4]MEDEIROS F, MUTO M G, LEE Y, et al. The tubal fimbria
24青島大学学报(医学版)57卷
is a preferred site for early adenocarcinoma in women with familial ovarian cancer syndrome[J]. Am J Surg Pathol, 2006,30(2):230-236.
[5]BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: archive for functional genomics data sets: update[J]. Nucleic Acids Research, 2012,41(D1):D991-D995.
[6]EDGAR R, DOMRACHEV M, LASH A E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository[J]. Nucleic Acids Res, 2002,30(1):207-210.
[7]LI J, YUE H R, YU H L, et al. Development and validation of SIRT3-related nomogram predictive of overall survival in patients with serous ovarian cancer[J]. J Ovarian Res, 2019,12(1):47.
[8]LOU W Y, DING B S, ZHONG G S, et al. Dysregulation of pseudogene/lncRNA-hsa-miR-363-3p-SPOCK2 pathway fuels stage progression of ovarian cancer[J]. Aging, 2019,11(23):11416-11439.
[9]TUNG C S, MOK S C, TSANG Y T M, et al. PAX2 expression in low malignant potential ovarian tumors and low-grade ovarian serous carcinomas[J]. Mod Pathol: Off J U S Can Acad Pathol Inc, 2009,22(9):1243-1250.
[10]MOK S C, BONOME T, VATHIPADIEKAL V, et al. A gene signature predictive for outcome in advanced ovarian can-cer identifies a survival factor: microfibril-associated glycoprotein 2[J]. Cancer Cell, 2009,16(6):521-532.
[11]KING E R, TUNG C S, TSANG Y T M, et al. The anterior gradient homolog 3 (AGR3) gene is associated with differen-tiation and survival in ovarian cancer[J]. Am J Surg Pathol, 2011,35(6):904-912.
[12]OXNARD G R, LO P C, NISHINO M, et al. Natural history and molecular characteristics of lung cancers harboring EGFR exon 20 insertions[J]. Journal of Thoracic Oncology, 2013,8(2):179-184.
[13]MICHOEL T, MAERE S, BONNET E, et al. Validating module network learning algorithms using simulated data[J]. BMC Bioinform, 2007,8 Suppl 2:S5.
[14]DENNIS G, SHERMAN B T, HOSACK D A, et al. DAVID: database for annotation, visualization, and integrated discove-ry[J]. Genome Biol, 2003,4(5):P3.
[15]CONSORTIUM G O. The Gene Ontology (GO) project in 2006[J]. Nucleic Acids Research, 2006,34(90001):D322-D326.
[16]SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: quality-controlled protein-protein association networks,made broadly accessible[J]. Nucleic Acids Res, 2017:45(1):362-368.
[17]SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003,13(11):2498-2504.
[18]SZSZ A M, LNCZKY A, NAGY , et al. Cross-validation of survival associated biomarkers in gastric cancer using transcriptomic data of 1,065 patients[J]. Oncotarget, 2016,7(31):49322-49333.
[19]XIAO Y B, FENG M, RAN H Y, et al. Identification of key differentially expressed genes associated with non-small cell lung cancer by bioinformatics analyses[J]. Mol Med Rep, 2018,17(5):6379-6386.
[20]LI J, FADARE O, XIANG L, et al. Ovarian serous carcinoma: recent concepts on its origin and carcinogenesis[J]. J Hematol Oncol, 2012,5:8.
[21]HILL S J, DECKER B, ROBERTS E A, et al. Prediction of DNA repair inhibitor response in short-term patient-derived ovarian cancer organoids[J]. Cancer Discov, 2018,8(11):1404-1421.
[22]DING Y, HUBERT C G, HERMAN J, et al. Cancer-specific requirement for BUB1B/BUBR1 in human brain tumor isolates and genetically transformed cells[J]. Cancer Discov, 2013,3(2):198-211.
[23]SHIH J H, CHEN H Y, LIN S C, et al. Integrative analyses of noncoding RNAs reveal the potential mechanisms augmenting tumor malignancy in lung adenocarcinoma[J]. Nucleic Acids Res, 2020,48(3):1175-1191.
[24]LIN S C, KAO C Y, LEE H J, et al. Dysregulation of miRNAs-COUP-TFII-FOXM1-CENPF axis contributes to the metastasis of prostate cancer[J]. Nature Communications, 2016,7:11418.
[25]PISE-MASISON C A, RADONOVICH M F, DOHONEY K M, et al. Gene expression profiling of ATL patients: compilation of disease-related genes and evidence for TCF4 involvement in BIRC5 gene expression and cell viability[J]. Blood, 2009,113(17):4016-4026.
[26]PSYRRI A, KALOGERAS K T, KRONENWETT R, et al. Prognostic significance of UBE2C mRNA expression in high-risk early breast cancer. A Hellenic Cooperative Oncology Group (HeCOG) Study[J]. Annals of Oncology, 2012,23(6):1422-1427.
[27]LIN S Y, PAN H W, LIU S H, et al. ASPM is a novel mar-ker for vascular invasion, early recurrence, and poor prognosis of hepatocellular carcinoma[J]. Clin Cancer Res: Off J Am Assoc Cancer Res, 2008,14(15):4814-4820.
[28]TUBBS R, BARLOW W E, BUDD G T, et al. Outcome of patients with early-stage breast cancer treated with doxorubicin-based adjuvant chemotherapy as a function of HER2 and TOP2A status[J]. J Clin Oncol: Off J Am Soc Clin Oncol, 2009,27(24):3881-3886.
[29]MUKHERJEE A, JOSEPH C, CRAZE M, et al. The role of BUB and CDC proteins in low-grade breast cancers[J]. Lancet Lond Engl, 2015,385 Suppl 1:S72.
(本文編辑 马伟平)
