基于16S rRNA高通量测序技术的生态学实验教学
2021-04-27张乃方
何 磊,张乃方,程 磊,陈 欣
(浙江大学a.国家级生物实验教学示范中心;b.生命科学学院,杭州 310058)
0 引言
土壤微生物在陆地生态系统有机质的分解、物质循环及土壤结构的保持等方面具有其重要的作用[1-6]。温度变化会影响土壤微生物活性,温度升高促进了微生物呼吸等代谢过程,提高了微生物的活性;另一方面,土壤微生物活性也会对气候变化产生正反馈作用[7-9],微生物活性的提高会促进热量的产生,加剧土壤温度的升高,一些不易分解的碳在升温的作用下成为较易分解的碳,从而促进微生物对有机碳的分解[10]。因此,了解微生物群落的多样性对于气候变化的响应具有十分重要的意义。
土壤微生物群落具有多样性高且大部分不能单独纯培养的特点[11-12],近年来,科研工作者越来越多地使用分子生物学方法进行微生物多样性的研究[13-15]。微生物rRNA具有很高的保守性,能够反映微生物之间的亲缘关系;同时,也具有一定的变异性,通过核酸序列特征能够鉴别不同的微生物种类。原核微生物的rRNA按照沉降系数可分为5S、16S和23S rRNA,由于16S rRNA的长短适中并同时含有保守区和变异区,因此是很好的生物标志物。由于传统一代测序的数据通量过小,无法测出环境样品中庞大数量的微生物,目前,与16S rRNA扩增结合的高通量测序手段成为了研究微生物多样性的重要方法之一。
本文从微生物生态学科研中提炼出一个生态学科研训练实验——土壤微生物多样性的分子生物学测定,该实验先提取土壤中微生物的基因组DNA,利用微生物16S基因特定区域的通用引物对16S rRNA进行扩增,再对PCR产物进行纯化和质量测定,最后用Illumina测序仪进行高通量二代测序。该实验用于多年的实验教学实践,大大激发了学生探索科学的欲望,提高学生综合实验能力,为学生日后进行相关科学研究夯实基础。
1 实验材料与仪器设备
1.1 实验材料
MP FastDNA SPIN Kit for DNA试剂盒(MO BIO laboratories,Inc.,Carlsbad,CA,USA),Bioflux 切胶回收试剂盒(Bioer Technology Co.,Ltd,Hangzhou,CHN),15 mL离心管,2 mL离心管,枪头,TAE电泳缓冲液,10 ×Taq 酶buffer,2.5 mmol/L dNTPs,Taq 酶。
1.2 仪器设备
天平,离心机(Eppendorf,Centrifuge 5424),PCR仪(Eppendorf,nexus SX1),移液器,电泳仪(BIORAD,Power Pac Basic),微量紫外-可见光分光光度计(Nanodrop,ND LITE Spectrophotometer,Thermo Fisher Scientific Inc.,Wilmington,DE,USA),切胶仪(上海培清,JS-BLUE 350E)。
2 实验内容与步骤
实验土壤取于升温实验基地,实验基地包括两个处理,一个是对照处理;另一个是土壤升温处理,升温处理的样地使用红外线加热器来为土壤增温,悬挂高度约为1.5 m,对照处理样地在同样的位置悬挂一个假的加热器。据温度测定结果,空气日均温度增加了1.27℃,而土壤日均温度增加了1.71℃。分别在每个处理点内随机选取3个采样点,使用土壤柱状取样器收集0~10 cm的表层土以及10~20 cm的底层土,将土壤装入密封袋后低温保存待用。
2.1 稳定同位素探针(SIP)实验
为了研究升温对微生物群落以及分解作用的影响,设计了一个室内培养实验。首先,将不同处理的土壤分成若干个20 g干重的小份,然后统一调整其湿度至60%,放入165 mL的广口瓶中。之后,将不同处理中的其中一瓶作为对照,其余广口瓶中加入碳十三标记的小麦(Trticum aestivum,13C含量>97%,IsoLife BV,Wageningen,The Netherlands)植物材料并混合均匀,植物材料和土壤的质量比为1∶150。将培养瓶遮光并在25℃的温度下培养9个星期。培养实验结束后取土壤进行DNA提取和后续13C-DNA的分离及测序。
2.2 土壤中DNA的提取与测定
(1)称取500 mg土壤,加入MP Fast DNA SPIN Kit for DNA 试剂盒(MO BIO laboratories,Inc.,Carlsbad,CA,USA)中的Lysing Matrix E Tube,再加入978 μL Sodium Phosphate Buffer和122 μL MT Buffer,混匀40 s。
(2)设置离心机转速为14 000 g,离心10 min,将上清液转移到新的干净的2 mL离心管,加入250 μL PPS并用枪头吹打均匀,用手上下摇10次。
“互联网+”的应用让人们的整个生活发生了很大的改变,包括人们的出行方式,支付方式,都在逐步地走向便捷,当然也包括我们的学习方式,从以前的书籍到现在的电子版,从以前的当面授课到现在的视频直播,都在发生着悄悄地改变,而且这种改变对我们来说是有益的,也是值得提倡的[3]。下面就介绍现在几种常见的教学方法。
(3)设置离心机转速为14 000 g,离心5 min,小心吸取上清液至干净的15 mL离心管中,加入1 mL Binding Matrix Suspension(用之前要将其摇晃重悬),手工反转2 min,然后将管子静置3 min。
(4)小心去除500 μL上清液,避免碰到沉淀。将剩下的液体重悬,将其中的600 μL转移到SPIN Filter中,以14 000 g的转速离心1 min,将Catch Tube倒空,再将剩下的混合物加到SPIN Filter中,以同样的转速离心1 min。
(5)添加500 μL SEWS-M 到SPIN Filter并把其中的混合物用枪头轻柔地重悬,以14 000 g的转速离心1 min,倒出流过液并把SPIN Filter放回Catch Tube。以14 000 g的转速再离心2 min,清干SPIN Filter中剩余的SEWS-M。
(6)移动SPIN Filter到新的Catch Tube上,打开盖子,室温风干5 min。
(7)加入40 μL无菌水轻轻重悬,用枪头轻轻搅动或用手指轻弹。
(8)以14 000 g的转速离心1 min,DNA被洗脱到Catch Tube中。
(9)取1.5 μL DNA 溶液用微量紫外-可见光分光光度计测定DNA的浓度与质量,当OD260/OD280在1.7~2.0之间(纯度较好的DNA 该比值在1.8附近),可以进行之后的步骤。
2.3 16S rRNA扩增与检测
(2)PCR扩增。PCR程序为:94℃ 5 min,30个循环×(94 ℃ 30 s,55℃ 30 s,72℃ 1 min),72℃ 10 min。
(3)取1 μL PCR产物进行琼脂糖电泳以验证DNA序列是否扩增成功。称取2 g琼脂糖,与100 mL TAE缓冲液混合,加热后再混入染料,使其凝固制成凝胶,之后将DNA样品与Loading buffer以1∶5的比例混合,加入点样孔,进行电泳。在目标区域看到明显的条带说明成功进行PCR扩增(见图1)。
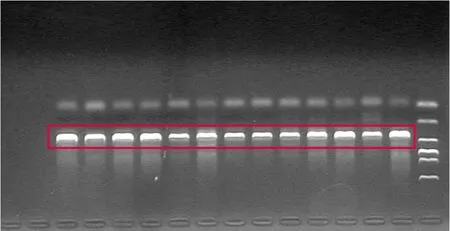
图1 成功扩增DNA片段的电泳图示例(红色方框内为符合目标片段长度的DNA)
2.4 Illumina MiSeq测序
使用干净的刀片,对符合目标序列长度的PCR产物进行切胶分离,并且使用切胶回收试剂盒(Bioer Technology Co.,Ltd,Hangzhou,CHN)溶出DNA。经微量紫外-可见光分光光度计验证其浓度与纯度后,将样品混合,使用Illumina测序仪上机测序。
2.5 454焦磷酸测序
对于SIP实验分离出的13C-DNA以及不含标记的DNA组分,使用针对16S序列V4-V8区的F515(5′-TCGTGCCAGCMGCCGCGG-3′)和 R1391(5′-AGACGGGCGGTGTGTRCA-3′)进行16S 扩增。其中F和R引物上都带有8位碱基的barcode和测序需要的linker,每个样品设有3个技术重复。100 μL的PCR体系包括AccuPrime Tag DNA聚合酶(Invitrogen,Carlsband,CA)3 个单位,10 × PCR buffer 10 μL,50 mmol/L MgSO410 μL,2.5 mmol/L dNTP(Invitrogen)8 μL,BSA(New England BioLabs,Ipswich,MA)0.5 μL,F 和R 引物各0.2 μmol/L,以及模板DNA5 ng,其余加无菌去离子水。PCR程序为:94℃ 2 min,25个循环×(94 ℃30 s,56 ℃30 s,68 ℃ 45 s),68 ℃10 min。PCR产物通过切胶回收和浓度纯度测定之后进行454焦磷酸测序。
2.6 数据处理与分析
(1)本实验中主要利用QIIME软件对数据进行后续处理:首先对数据进行质量控制、序列拼接以及去除嵌合体,之后以97%的相似度进行OTU聚类,再将OTU信息与Greengenes rDNA数据库进行比对,注释各OTU的分类单元,得到OTU分布表格。
(2)利用QIIME中自带的数据处理语句或者R软件中vegan软件包的程序对OTU分布信息进行分析,并比较不同样品间多样性及微生物分布的差异。QIIME 的具体使用方法见网址:http://qiime.org/tutorials/tutorial.html。
3 结果与讨论
3.1 升温对于下层土土壤微生物分类以及功能上分布的影响
根据下层土土壤微生物的16S rRNA序列测序结果表明,下层土中相对丰度较高的门有酸杆菌门(Acidobacteria),放线菌门(Actinobacteria),绿弯菌门(Chloroflexi),芽单胞菌门(Gemmatimonadetes),浮霉菌门(Planctomycetes),变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia),升温没有显著影响微生物的群落分布(图2(a)),在较低的尺度的门、纲等尺度上,升温也没有对微生物群落分布造成显著的影响(图2(b))。测序结果的DCA分析结果表明升温处理的样品点并没有与对照处理分开(图2(d))。这些结果都说明升温并不会影响土壤微生物群落的整体组成。
本文使用GeoChip比较升温处理和对照之间微生物功能基因丰度的差异。共检测了49 857个基因,其中85%属于细菌,10%属于真菌。与16S测序结果不同,DCA分析结果显示,对于功能基因的分布,升温处理样品点和对照样品点都是分开聚集,这说明升温处理极大地改变了土壤微生物功能性基因的分布。升温处理显著改变了16%总的功能性基因的相对丰度,并改变了15.6%的细菌功能性基因的分布以及21%的真菌功能性基因的分布;同时升温处理显著提升了20.4%的碳循环相关功能基因的丰度。实验结果表明,升温处理虽然没有显著改变微生物群体分类方面的结构,却改变了微生物群体的功能结构。这与之前类似的研究结果是一致的,环境变量会大幅度地改变微生物的功能结构,却没有明显改变微生物的分类结构[17]。
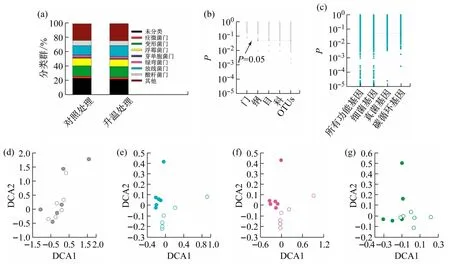
图2 升温对于下层土土壤微生物分类以及功能上分布的影响。(a)相对丰度较高的门;(b)温度对于微生物不同分类水平分布效应的P值;(c)温度对所有基因以及细菌、真菌和碳循环功能基因效应的P值。(b)图和(c)图中虚线以下代表升温效应在统计上显著(P<0.05);(d)~(g)微生物群体的DCA 排序分析,包括:基于16S rRNA测序结果的细菌分类(d),基于GeoChip的微生物功能基因(e)、细菌基因(f)和真菌基因(g)。实心圈代表升温处理,空心圈代表对照
3.2 升温和对照处理下微生物群落在分类和功能上的结构
SIP实验分离出的13C-DNA和12C-DNA的16S序列扩增和测序结果表明,利用13C的微生物群体和不利用13C的微生物群体有很大的差异(图3(a),(b))。SIP实验分离出的DNA的GeoChip测定结果表明,无论是对于13C-DNA还是12C-DNA,升温处理下的功能基因分布都与对照处理下差异很大,而可利用13C的微生物功能基因又与利用12C的微生物功能基因分布差异很大(图3(c)~(e))。结果说明,升温对于碳分解相关功能基因分布的影响十分显著。

图3 SIP实验中升温和对照处理下微生物群落在分类和功能上的结构。(a)占优势的门的相对丰度;(b)~(e)总的微生物群体以及可利用13C的微生物群体在分类和功能上的结构分布。其中:(b)基于16S测序的细菌群体;(c)GeoChip测的全部功能基因;(d)细菌的功能性基因;(e)真菌的功能性基因。图中实心符号代表升温处理,空心符号代表对照处理
4 结语
本实验将16S rRNA高通量测序技术与同位素探针技术应用于生态学本科实验教学中。通过本实验,一方面学生切身体验到如何用试剂盒进行土壤DNA的提取,琼脂糖电泳扩增与检测,Illumina MiSeq测序以及测序结果的分析等一系列过程,大大激发了学生探索土壤微生物多样性奥秘的兴趣;另一方面,学生学习了如何利用统计分析软件对OTU分布信息进行分析,并比较不同样品间多样性及微生物分布的差异,开拓了学生在生态学统计及应用方面的思路。实践证明,该实验提高了学生综合性实验能力,包括实验操作、数据分析处理、结果探索以及多种实验手段的融合,为学生的进一步科学研究奠定了基础。
