结合卷积神经网络与图卷积网络的乳腺癌病理图像分类研究
2021-04-22汪琳琳施俊韩振奇刘立庄
汪琳琳 施俊 韩振奇 刘立庄
0 引言
乳腺癌是女性的高发疾病之一,其发病率和死亡率均占女性恶性肿瘤的首位[1]。乳腺癌的精确诊断对后续治疗具有重要意义。组织病理学诊断被认为是肿瘤诊断的“金标准”[2]。组织中的细胞结构和空间分布存在着潜在的关系[3],发生病变的组织在形态、细胞空间分布等方面与正常组织存在明显的区别[4]。组织结构之间的形态变化、相邻关系和空间分布等因素对于疾病诊断也具有重要作用[5]。在临床诊断中,病理医生通过观察细胞的形态和分布进行诊断[6]费时、费力,并且诊断结果容易受到病理医生经验和知识水平等主观因素影响。使用计算机辅助诊断(computer-aided diagnosis,CAD)对病理图像进行分析诊断,能够为医生提供更加客观、可靠的诊断结果[6-7]。
以卷积神经网络(convolution neural network,CNN)为代表的深度学习方法广泛应用于病理图像分析[8-10]。CNN通过层次化的深层结构来学习特征,具有强大的抽象特征学习和表达能力[9]。由于卷积核具有局部感知和权重共享的特点,CNN能较好地表达局部的特征[8]。目前基于CNN的病理图像分类方法大多侧重于局部特征的表达[11-15],然后组合局部特征得到图像的全局特征。但是CNN没有足够的上下文感知能力,也无法有效捕捉到组织细胞间的空间关系。
图论(graph theory)被广泛用于表示拓扑结构,利用图论对病理图像建模,可以准确捕捉细胞组织间的空间关系[3,5]。图卷积网络(graph convolution network,GCN)是一种对图结构进行卷积运算的有效方法[16],在多种图任务中取得良好性能[17]。GCN主要分为基于空域和基于频域(谱)的方法[17-18]。前者通过定义聚合函数聚合自身节点与邻节点信息,代表算法有GraphSAGE[19]、EdgeConv[20]等;后者基于图谱理论在谱空间定义卷积,代表算法有ChebNet[21]、GCN[16]等。
近年来,学者们已经开始探索GCN在病理图像上的应用。例如,Zhou等[22]提出一种基于GCN的细胞图网络对结直肠癌分级;Wang等[23]提出一种基于GCN的弱监督方法对前列腺组织切片分级。上述两个工作都是先分割出病理图像的细胞核并提取细胞核的外观特征,然后构建以细胞核为节点、细胞核间的空间关系为边的细胞图[3],最后对细胞图进行图像级分类。这种方法比较精细,能较好地模拟组织微环境中的复杂结构,但是处理时比较复杂,并且使用大量细胞核为节点,构建的细胞图规模较大,需要很大的计算开销。
在全切片图像(whole slide image,WSI)上,常利用图像子块(patch)构建图。Li等[24]提出一种具有注意学习机制的GCN,对肺癌和脑癌WSI进行生存分析;Adnan等[25]提出一种GCN结合多示例学习(multiple instance learning,MIL)的方法对肺癌亚型进行分类。上述两个工作都从WSI中采样具有代表性的图像子块,然后将子块表示为节点,再根据子块节点之间的空间关系生成边,构建WSI上的图结构,最后通过GCN对图结构进行处理。这种基于子块构图的方法能较好地缩小图的规模,平衡好效率和计算开销,但如何选择子块作为节点需要一定的先验知识,图像子块的数目和位置都会对结果产生一定影响。已有研究[22-25]表明通过GCN的方法可以有效利用病理图像中细胞组织间的空间关系,为分类提供有意义的空间结构特征,但算法性能仍有进一步提升的空间。
针对CNN具有良好的局部特征表达能力但空间感知能力不足,而图结构能较好地弥补这种缺憾的特点,本文提出一种结合CNN与GCN的深度神经网络框架,应用于乳腺病理图像分类。将病理图像上不重叠的子块特征表示为节点,根据子块特征的距离生成边,简单有效地实现图网络构建。
1 方法
1.1 总体流程
本文提出一种结合CNN与GCN网络的CNN-GCN-fusion融合框架,其总体流程如图1所示,主要包含3个部分:用于提取特征的CNN模块、用于捕捉空间结构关系的GCN模块、特征融合分类模块。在该框架下,可以使用多种GCN算法,本文采用文献[16]中的谱卷积(为与广义GCN区别,记为sGCN)算法。具体步骤如下。

图1 CNN-GCN-fusion框架总体流程Figure 1 Overview of the CNN-GCN-fusion framwork
(1) 使用CNN对乳腺病理图像进行特征提取及下采样,得到一组具有抽象语义的特征图。
(2) 将特征图上同一个像素位置的特征向量表示为一个节点,通过K最近邻(k-nearest neighbor,KNN)算法[26]寻找最邻近的其他节点,在这些节点之间形成边,将特征图表示为图。
(3) 通过sGCN对构建的图进一步特征映射,得到图上的空间结构特征。
(4) 将CNN得到的特征图进行全局池化,与sGCN得到的空间结构特征融合。
(5) 将融合后的特征通过分类器分类,对整个网络进行训练及反馈调参,得到CNN-sGCN-fusion模型。
1.2 CNN特征学习
CNN网络通常由输入层、卷积层、池化层、全连接层组成[8]。输入一般为RGB图像,然后通过卷积核提取局部特征,由池化层进行下采样缩小特征图的尺寸,并由激活函数增强网络的非线性表达能力。经过多层的卷积和池化操作后,输入图像由低层特征到高层特征逐步学习,得到一组由多个特征映射叠加成的特征图,在全连接层组合局部特征得到高表达能力的全局特征。CNN网络经常使用预训练模型进行训练。与从头开始训练的模型相比,预训练网络能更快地提取通用特征,并且在一定程度上减少过拟合,增强泛化能力[27]。
ResNet通过跳跃连接的方式克服了CNN网络随着网络层数加深带来的梯度消失问题[28]。本文选择了代表性的ResNet18网络进行后续网络的构建。ResNet18网络的结构如图2所示,共18层,由4个具有不同通道数的残差块(residual block)组成。随着通道数的加深及下采样,逐渐从低层特征中提取出高级特征。本文在预训练ResNet18的基础上进行微调:保留4个残差块,去掉特定的分类任务部分(即平均池化层和全连接层)。将病理图像输入微调的网络结构中,得到一组由512个通道特征堆叠成的特征图。
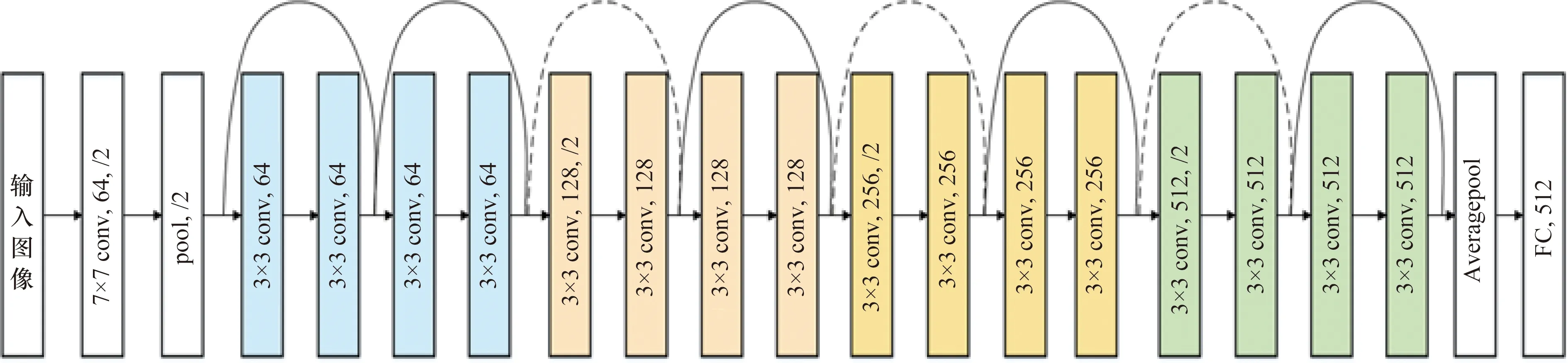
图2 ResNet18网络结构Figure 2 Network structure of ResNet18
1.3 GCN特征学习
1.3.1 GCN原理
不同于CNN在二维矩阵等规则的欧氏空间中进行卷积运算,GCN将卷积运算推广到了具有图结构的非欧氏数据。GCN将图结构作为输入,通过对图中每个节点的邻节点进行图卷积运算得到新的节点表示,然后对所有节点进行池化,能够得到整个图的表示。
空域GCN通过聚合函数从邻节点聚合特征来更新当前节点的特征,聚合函数可以有多种形式,如平均聚合,最大池化聚合,LSTM聚合等[19]。频域GCN(sGCN)基于图谱理论,利用图傅里叶变换,先将空域上的节点特征和卷积核转换到频域,然后在频域中相乘,再通过傅里叶反变换转换回空域。sGCN对频域的卷积核进行一阶切比雪夫近似,简化了计算复杂度[16]。
将一个包含N个节点的无向图定义为G=(V,E),其中vi∈V表示节点,ei,j=(i,j)∈E表示两个节点之间的边。两两节点之间的关系用一个邻接矩阵A∈RN×N表示,如果两个节点之间存在边连接,则Aij>0。假定每个节点包含D维特征,将这些特征表示为一个N×D维的矩阵X,则X∈RN×D。
谱图卷积可以定义为式(1):
xGg=F-1[F(x)·F(g)]
(1)
式中:x表示节点特征;Gg表示频域卷积核;F()表示傅里叶变换;F-1()表示傅里叶逆变换;·表示点乘。
拉普拉斯矩阵L可进行谱分解:
L=UΛU-1
(2)
式中:U是特征向量组成的正交矩阵;Λ是特征值对角矩阵。将U作为图傅里叶变换的基函数,式(1)可表示为式(3):
xGg=U(UTx·UTg)
(3)
令gθ=diag(UTg),式(3)等价为式(4):
xGg=UgθUTx
(4)
为简化计算,使用一阶切比雪夫多项式来近似表示卷积核Gg:

(5)

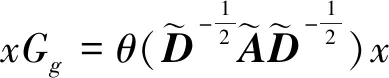
(6)
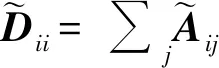
一个多层的sGCN最终表示为式(7):

(7)
式中:H(l)∈RN×dl表示第l层的节点特征;H(l+1)∈RN×dl+1表示第l+1层更新的节点特征。输入层的特征为H(0)=X。W(l)∈Rdl×dl+1是每一层中的可训练权重,表示激活函数σ(),本文采用ReLU函数。
因此,只要知道输入特征X与邻接矩阵A,就可以计算出更新的节点特征。实际上,谱图卷积运算是将每个节点的特征与其邻节点的特征加权后传播到下一层中。随着层数的加深,每个节点能聚合到更远邻节点的特征,感受野越大。但堆叠多个层会使得反向传播过于平滑,导致梯度消失,sGCN一般不超过4层[29]。
1.3.2 图构建
经过多层卷积后的多通道特征图包含了高级的语义特征,多通道特征图可以看作是输入图像的高维特征表示;特征图上一个像素位置的特征向量可以表示为输入图像对应子块的特征。因此,在特征图上构图并将不同像素位置的特征向量当作节点,实际上是对输入图像对应位置的不重叠子块之间构图,可以充分捕捉子块之间的空间关系。
本研究将ResNet得到的特征图表示为X,X上每个像素位置定义为一个节点Xi,其沿着通道方向的512维特征向量为该节点的初始特征,即Xi∈RN×512。其中N为节点个数,512为节点特征维数。边定义为两个节点之间的潜在相互作用,假设距离越小的节点越容易产生相互作用。如果两个节点之间的距离在一定范围内,则在这两个节点之间生成一条边。本文采用式(8)中定义的欧氏距离,首先计算所有节点两两之间的相关性dis(Xi,Xj),得到距离矩阵Dis∈RN×N,然后根据KNN算法对Dis从小到大排序,选出每个节点距离其最近的K个节点作为邻节点,并在这些节点之间生成边,由此得到图的结构。

(8)
邻接矩阵A∈RN×N定义为式(9):

(9)
将特征图X与邻接矩阵A根据式(7)进行谱图卷积运算,得到新的节点表示。本研究设置了3层图卷积层,对每个节点的特征再次表达,得到具有空间结构信息的特征表示。
1.4 分类
为了更好地利用乳腺病理图像的信息,本研究将全局特征与空间结构特征相融合。首先将ResNet的特征图进行全局平均池化,得到512维特征向量,代表了输入图像的全局特征。经过3层sGCN的节点特征仍然保持着N个节点之间的连接,为了对整个图进行分类,本文对N个节点特征进行全局平均池化,通过一个全局节点特征来代表整个图的节点信息。然后将这个全局节点的特征拉伸成64维的特征向量,与512维的全局特征拼接融合,得到576维特征向量。再将融合的特征通过2个全连接层进一步映射,通过softmax分类器计算每一类的分类概率,最后利用交叉熵损失(cross-entropy loss)函数训练整个网络。
在训练过程中,通过反向传播进行参数的更新。在网络前向传播时,对CNN模块进行ImageNet预训练参数初始化,GCN模块随机初始化,然后在迭代训练中根据损失梯度下降方向反向调整各层参数,直至损失收敛。在本文的网络框架中,由于直接对ResNet得到的特征图进行图卷积运算,再通过分类器进行分类,整个过程是端到端的,因此在反馈调参时,同时对CNN模块、GCN模块、分类模块的参数进行了更新。随着参数更新,CNN的特征值得到调整,同时图的结构也得到微调和优化。
2 实验和结果
2.1 实验数据与预处理
为了验证所提出方法的有效性,本研究在2个公开数据集上进行了实验,分别为2015生物成像挑战赛(简称2015挑战赛)公开的乳腺组织数据集[12]和Databiox公开的乳腺组织数据集[30]。
2015挑战赛数据集由249张训练图像和20张测试图像组成,包含4类乳腺病理图像:正常组织、良性病变、原位癌和浸润癌,每一类数据是均衡的。图像由HE染色,具有高分辨率(2 048×1 536像素)。所有图像在相同的采集条件下数字化,尺寸为0.42 μm×0.42 μm,放大200倍。这些图像由两位有经验的病理学专家进行标记,并丢弃有歧义的图像。数据集可在https://rdm.inesctec.pt/dataset/nis-2017-003上公开获得,每类图像的形态如图3所示。
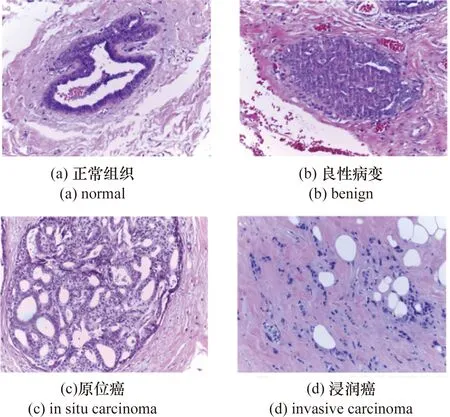
图3 2015挑战赛数据集的4种乳腺癌类型Figure 3 Four types of breast cancer in Bioimaging Challenge 2015 Breast Histology Dataset
Databiox数据集共由922张图像组成,通过对124例浸润性导管癌(invasive ductal carcinomas,IDC)患者的乳腺肿瘤组织采用不同放大倍数(4×,10×,20×,40×)得到,根据分化程度分为3个级别。本研究选择40×放大数据进行实验,并将图像裁剪成2 048×1 536像素以去除周围非组织区域。数据集在http://databiox.com上公开获得,40×放大下的图像形态如图4所示。

图4 40×浸润性导管癌分级 Figure 4 Grading invasive ductal carcinomas in 40×
与自然图像相比,病理图像更难获得,数据集样本量较少,容易使网络过拟合。病理图像分类问题是旋转不变的,病理医生可以从不同的方向进行诊断,而不会影响诊断结果[12]。旋转和镜像在不降低数据集质量的情况下可以增加数据集的大小,让数据集尽可能地多样化,使得训练的模型具有更强的泛化能力。本研究对2个数据集的数据均进行了增强,通过旋转不同角度(90°,180°,270°)和水平翻转来扩充数据。然后减去RGB 3个通道的平均值,并除以标准差,将RGB通道的值归一化到-1~1之间[31]。
2.2 实验设计
本文所提出的CNN-sGCN-fusion算法与以下4个算法进行对比。
(1) ResNet20:在ResNet18的4个残差块后增加3层卷积,共包含20层卷积,将该算法作为基准对比。
(2) CNN-GraphSAGE:GraphSAGE[19]是一种通用的归纳式空域GCN算法,可通过平均聚合函数聚合邻节点特征。平均聚合函数求取当前节点与邻节点的均值来更新当前节点的特征。由GraphSAGE提取图上的空间特征分类,不进行特征的融合。
(3) CNN-EdgeConv:EdgeConv[20]是一种通过对称聚合邻节点特征的空域GCN算法。聚合函数通过拼接当前节点与邻节点的差值来更新当前节点的特征。由EdgeConv提取图上的空间特征分类,不进行特征的融合。
(4) CNN-sGCN:由sGCN[16]提取图上的空间特征分类,不进行特征的融合。
对所有算法使用相同的数据,进行5折交叉验证,最后取5次实验的平均值,并计算方差。由于2个数据集都是多分类问题,使用分类准确率(Accuracy)、精确率(Precision)、召回率(Recall)、F1分数作为评价指标。
2.3 实现细节
本文提出的CNN-sGCN-fusion算法通过预训练ResNet18获得特征图,4个残差块输出通道分别为64、128、256、512。邻节点数从2、4、6、8、10、12中选择,最终确定为8。设置的3个图卷积层,输出通道分别为256、128、64。2个全连接层的隐藏节点数设为576、64,为了避免过拟合,设置全连接隐藏层的droupout为0.5。本研究采用pytorch框架来实现模型,实验进行100个epoch,设置初始学习率为0.001,每30个epoch减小为原来的0.1。将输入图像大小调整为1 024×1 024像素,批大小设为4,模型采用随机梯度下降SGD进行反向传播。为了进行公平对比,对其他对比算法设置相同的网络层数,并且超参数设置尽可能相同。
2.4 实验结果
2015挑战赛数据集在不同算法中的分类结果如表1所示。可以发现结合了GCN的算法(CNN-GraphSAGE、CNN-EdgeConv、CNN-sGCN)相比于ResNet20在各个指标上均有提高,这表明这种构图方法是有效的,利用图的拓扑结构将病理图像中的空间结构关系考虑进来更有利于分类。CNN-GraphSAGE与CNN-EdgeConv的性能相差不大,基于谱图卷积的CNN-sGCN算法性能略高于两种基于空域的GCN算法。因此在CNN-sGCN的基础上再融合病理图像的全局特征。本文提出的算法CNN-sGCN-fusion在准确率、精确率、召回率、F1分数上均获得最高的结果,分别为93.53%±1.80%、93.88%±1.78%、93.69%±1.70%、93.63%±1.83%,相比于ResNet20分别提高3.32%、3.04%、2.89%、3.10%。该结果表明将病理图像的全局特征信息与空间结构特征相结合可进一步提升分类性能。
此外,本文还分析了在CNN-sGCN-fusion算法下不同邻节点个数K对分类结果的影响。2015挑战赛数据集中不同邻节点个数下的分类结果如表2所示。可以发现,随着邻节点数的增长,分类准确率呈先上升后下降的趋势,当邻节点数(包括自身节点)为8时,达到最高的准确率。当邻节点较少时,病理图像的空间关系还不能很好地表达,当邻节点过多时,造成冗余或将相关性不是特别大的节点特征聚合过来,导致准确率下降。

表1 2015挑战赛数据集在不同算法中的分类结果(单位:%)Table 1 Classification results of different algorithms in Bioimaging Challenge 2015 Breast Histology Dataset (unit:%)

表2 2015挑战赛数据集在CNN-sGCN-fusion算法下不同邻节点数的分类结果(单位:%) Table 2 Classification results of the different number of neighbor nodes in Bioimaging Challenge 2015 Breast Histology Dataset under CNN-sGCN-fusion algorithm (unit:%)
在Databiox数据集上,不同算法的分类结果如表3所示。虽然不同级别的IDC在颜色、细胞形态中具有很大的相似性,增加了分级的难度,但仍然可以发现结合GCN的算法在分类性能上高于ResNet20,谱图卷积算法略高于其他两种空域图卷积模型。其中结合了全局特征和空间结构特征的CNN-sGCN-fusion算法具有最高的分类性能,在准确率、精确率、召回率、F1分数上分别为78.47%±5.33%、79.07%±5.28%、79.00%±4.60%、78.69%±5.17%,相比于ResNet20分别提高了2.19%、1.88%、2.15%、2.29%。

表3 Databiox数据集在不同算法中分类结果(单位:%)Table 3 Classification results of different algorithms in Databiox dataset (unit:%)
Databiox数据集在CNN-sGCN-fusion算法下由不同邻节点个数K得到的分类结果如表4所示。随着邻节点数的增长,分类准确率呈现先增长后下降的趋势,在邻节点数为8时,达到最高的准确率。

表4 Databiox数据集在CNN-sGCN-fusion算法下不同邻节点数的分类结果(单位:%)Table 4 Cassification results of the different number of neighbor nodes in Databiox dataset under CNN-sGCN-fusion algorithm (unit:%)
3 讨论
针对CNN无法很好表示高分辨率乳腺病理图像组织细胞间的空间关系问题,本文提出一种结合CNN与GCN的病理图像分类框架,通过图的拓扑结构来表示图像子块间的关系,从而有效提取病理图像中隐含的空间结构信息。在对比实验中,通过替换不同的GCN算法,发现所有结合了GCN的算法在性能上都有所提升。其中基于谱图卷积的sGCN算法略高于两种基于空域的GCN算法,这是由于上述两种空域图卷积的聚合函数采用了较为简单的聚合方式。这在一定程度上会缩小节点之间的差异,使得经过多层图卷积后,不同节点的特征趋于同质化。而谱图卷积通过拉普拉斯算子进行图傅里叶变换,根据分解的特征值计算,低特征值对应的特征向量变化比较平滑,高特征值对应的特征向量变化比较剧烈,在一定程度上保持甚至放大了节点之间的差异。
相比于细胞图方法,本文方法避免了细胞分割及细胞核特征提取等一系列复杂的操作,利用特征图上的像素特征向量代表原始病理图像的子块,在一定程度上简化了图。相比于通过提取子块的图构建方法,本文方法不需要先验知识,并且充分利用所有子块位置的特征,避免了采样不足或选取的位置不具代表性而造成的问题。本文提出的通过特征图构图的方法相对简单,并且在分类准确率上比传统CNN方法有所提高。对于细胞组织之间存在较强空间相关性的病理图像来说,具有一定的研究意义和临床应用价值。
下一步工作将考虑在特征图上加入注意力机制,选择更有代表意义的位置作为节点,并对邻节点分配不同的权重;同时考虑对图结构进行分层池化,逐步减小图的规模,降低图结构信息的损失。
4 结论
本文提出一种结合CNN与GCN的病理图像分类框架,应用于乳腺病理图像辅助诊断。通过图的构建,获得病理图像内部细胞组织间的空间分布关系,为分类提供有意义的特征。并进一步将空间结构特征与全局特征融合,使特征表达更加丰富。在两个公开乳腺癌数据集上进行了实验,算法分别获得93.53%±1.80%和78.47%±5.33%的分类准确率,优于同类算法,证明了其有效性。实验表明,通过图卷积网络将病理图像的空间结构特征与全局特征融合,有利于分类结果的提升,具有一定的研究意义和临床应用价值。
