八肋游仆虫NMD途径因子的生物信息学分析及分子鉴定
2021-04-21周宝春王软林柴宝峰
周宝春,王软林,柴宝峰
(山西大学 生物技术研究所 化学生物学与分子工程教育部重点实验室 ,山西 太原 030006)
0 引言
无义介导的mRNA降解(nonsense-mediated mRNA decay,NMD)途径是一种mRNA质量监控机制,保障细胞内基因的精准表达。在真核生物的细胞中,它的功能是用来识别和降解含有提前终止密码子(premature termination codons,PTCs)的异常转录本,从而避免存在潜在毒性的蛋白截短体产生[1]。这种监控机制不仅在高等真核生物中普遍存在,而且在原生生物的三大超群——陷摄虫群(Ex-cavata)、多貌生物群(Diaphoretickes)和单鞭毛生物群(Amorphea)中均有发现[2-4]。
在不同进化水平的生物中,NMD途径具有一定的进化特征,从高等真核生物到低等原生生物,NMD途径相关蛋白因子的种类、数量、结构和功能均存在一定的差异[3]。例如在识别含PTC转录本的方式上,进化程度相对较高的哺乳动物是以依赖于外显子连接复合体(exon junction complex,EJC)的方式,在停滞的核糖体上通过招募SMG1(Suppress with morphogenetic effect on genitalia-1)、UPF1(upframeshift 1)和 eRF(eukaryotic release factor)因子形成SURF复合体,随后再招募结合UPF2和UPF3b因子,通过这两个因子再与外显子连接复合体结合[1]。在低等真核生物酿酒酵母中会招募形成eRF-UPF1-UPF2-UPF3 复合体[1,5]。而在进化程度相对较低的原生动物中,识别PTC的分子机制更为复杂。嗜热四膜虫(Tetrahymena thermophila)是以一种不依赖于外显子连接复合体的方式,招募形成UPF1-UPF2-UPF3复合体[6]。贾第虫(Giardia lamblia)可能是以一种依赖于不均一核小核糖核蛋白1(HRP1)的方式并招募形成SURF复合体[7-8]。
八肋游仆虫(Euplotes octocarinatus)是一类单细胞原生生物,细胞中有一个“C”形营养大核和一个球形生殖小核。据统计,大核中大约含有三万条高拷贝片段化的基因大小染色体(gene size chromosome)[9],即一条染色体中含有一个基因。而且,由于游仆虫大核基因阅读框中存在无义密码子编码氨基酸现象和翻译移码现象[10],使得该类生物的NMD途径机制更具特殊性。为此,在前期对八肋游仆虫NMD途径初步研究的基础上[11-12],利用生物信息学方法对八肋游仆虫NMD途径因子进行分析,以期对原生动物NMD途径的深入研究提供支持,具体包括NMD相关蛋白因子的同源鉴定和结构功能域注释、核心蛋白因子eUPF1与eUPF2的三级结构预测、分子对接和相互作用的关键氨基酸分析。最后利用定点突变和Pull-down实验验证eUPF2与eUPF1之间相互作用的关键结构域与预测结果的一致性。
1 材料与方法
1.1 材料
八肋游仆虫(69号)为本实验室培养,菌株E.coliDH5α、E.coliBL21为本实验室保存,质粒pGEX-6P-1、pET-28a分别用于重组载体的构建和体外基因原核表达。
1.2 主要实验试剂
EasyTaq®DNA Polymerase、EasyPure®Quick Gel Extraction Kit试剂盒、TransStart®FastPfu FlyDNA Polymerase、EasyScript®One-Step gDNA Removal and cDNA Synthesis SuperMix、High Pure dNTPs、His抗体及GST单克隆抗体购自Transgene公司;限制性核酸内切酶、T4 DNA连接酶购自Thermo Scientific公司;Plasmid Mini KitI购自OMEGA公司;寡核苷酸引物合成和重组载体测序由华大基因有限公司完成;PMSF蛋白酶抑制剂购自于BBI Life Sciences公司;Eastep®Super Total RNA Extraction Kit试剂盒购自Promega公司;Tris购自Solarbio公司;配制LB培养基所用酵母粉、蛋白胨购自Oxoid公司;氯化钠、琼脂粉购自Sangon公司。
1.3 生物信息学分析
为了鉴定八肋游仆虫中NMD途径相关蛋白因子,首先从 UniProt数据库(https://www.uniprot.org)中下载人类NMD途径相关蛋白因子的氨基酸序列,然后在游仆虫大核基因组数据库(http://ciliates.ihb.ac.cn/database/home/#eo)[9]中 作 tblastn 搜索。再将得到的序列,在NCBI非冗余蛋白数据库中作blastx序列比对,进一步对获取的基因序列作验证。四膜虫NMD相关蛋白因子基因序列下载自TGD(http://ciliate.org/index.php/home/welcome);酿酒酵母(Saccharomyces cerevisiae)NMD相关蛋白因子基因序列下载自SGD(https://www.yeastgenome.org/);蓝氏贾第虫NMD相关蛋白因子基因序列 下 载 自 GiardiaDB(https://giardiadb.org/giardiadb/);拟南芥(Arabidopsis thaliana)NMD相关蛋白因子基因序列下载自TAIR(https://www.arabidopsis.org/)。
蛋白多序列比对和系统发育树采用Mega 7.0.14 软件[13],采用 Jalview2.10.5 软件[14]进行可视化。其他生物中的同源蛋白序列均下载自Uni-Prot数据库。eUPF1蛋白质三级结构模型采用Swiss-model(https://www.swissmodel.expasy.org/)[15]进行在线预测,eUPF2 蛋白质三级结构模型通过Zhang Lab I-TASSER Protein Structure&Function Predictions(https://zhanglab.ccmb.med.umich.edu/I-TASSER/)进行在线预测[16]。采用 InterPro Protein sequence analysis(http://www.ebi.ac.uk/interpro/search/sequence-search)对蛋白质功能结构域注释;利用ZDOCK SERVER(http://zdock.umassmed.edu/)进行蛋白分子对接,对接结果采用PDBePISA(http://www.ebi.ac.uk/msd-srv/prot_int/cgi-bin/piserver)[17]进行 相互作用 关键氨基 酸位点的寻找,采用Discovery Studio 4.5 Visualizer软件分析相互作用蛋白因子间的疏水作用和氢键。
1.4 重组表达质粒的构建与突变
以八肋游仆虫cDNA为模板,采用PCR法,用引物BF781/BF782和BF783/BF784分别扩增获取eUPF1 CH结构域和eUPF2 U1BD结构域基因,然后用相应的限制酶分别对目的基因片段和载体进行双酶切(Table 1),最后用T4 DNA连接酶,将目的基因分别克隆到pET-28a和pGEX-6P-1质粒上,得到重组表达质粒pET-28a-eUPF1-CH和pGEX-6P-1-eUPF2-U1BD(WT);利用定点突变技术,设计引物DT901/DT902,利用PCR法将eUPF2的αA-helix结构对应的DNA片段进行缺失突变,得到重组表达质粒pGEX-6P-1-eUPF2-U1BD(MT)。
1.5 蛋白纯化与Pull-down分析
分别将构建好的pET-28a-eUPF1-CH、pGEX-6P-1-eUPF2-U1BD(WT)和pGEX-6P-1-eUPF2-U1BD(MT)重组表达质粒转化到E.coliBL21感受态细胞中,置于37℃恒温培养箱中,过夜培养。第二天,将阳性单克隆菌落接种到LB液体培养基中。在37℃,180 r/min的条件下,培养5 h后,加入IPTG诱导剂,16℃低温诱导过夜,使融合蛋白大量表达。离心收菌,加入PMSF蛋白酶抑制剂,冰浴超声破碎细胞,4℃离心收集带标签(GST/His)融合蛋白上清液,将收集的两种蛋白上清液混合,混合均匀后分成两等份,一份缓慢注入平衡过的含Glutathion-Sepharose 4B柱材料的柱子中,另一份缓慢注入平衡过的含Ni-NAT柱材料的柱子中,冰浴过夜结合。用洗涤缓冲液除去杂蛋白,随后用洗脱缓冲液洗脱目的蛋白。洗脱的目的蛋白经低温浓缩后制备样品。质量分数15% SDS-PAGE电泳后,对互作蛋白分离,Western blot湿转法转至NC膜上,封闭液中封闭1 h,PBST溶液充分洗膜后,用质量分数5%脱脂牛奶分别稀释Anti-GST/Anti-His一抗(1∶1000稀释)和荧光二抗(1∶1000稀释)。再将膜置于蛋白标签相对应的一抗稀释液中,4℃过夜孵育。然后用PBST溶液充分洗膜,再将膜置于荧光二抗稀释液中孵育2 h,最后用PBST溶液清洗膜上残留二抗,利用Odyssey红外激光成像系统成像。
2 实验结果
2.1 八肋游仆虫NMD途径相关蛋白因子的鉴定
陷摄虫超群类生物处在真核生物进化谱系最早的分支,进化程度最低[2,4],对于研究 NMD 途径的起源与进化十分重要。而蓝氏贾第虫刚好处于这一分支,所含NMD因子在种类上比较少,不存在外显子连接复合体组分蛋白因子。在多貌生物超群中,八肋游仆虫和嗜热四膜虫均属于囊泡虫超门(Alveolatas)中的纤毛虫类。拟南芥则属于陆生高等植物,其NMD核心蛋白因子种类都在两种及以上,也含有EJC组分蛋白因子。真菌和动物界均划定在单鞭毛超群中,酿酒酵母的NMD相关蛋白因子种类也比较少,而人类的因子种类最为丰富。对于mRNA降解相关蛋白因子,无论物种进化程度高低,在各种生物细胞中均存在。从八肋游仆虫基因组数据库中,通过同源比对鉴定到11种NMD相关蛋白因子基因(图1),包括UPF1(GenID:Contig 9114)、UPF2(GenID:Contig 14745)、MAGO(Gen-ID:Contig 11747)、Y14(GenID:Contig 15189)、UAP56(GenID:Contig 12743)、eIF4AIII(GenID:Contig 8403)、POP2(GenID:Contig 5432)、Exosome(GenID:Contig 25198)、XRN1(GenID:Contig 15461)、DCP2(GenID:Contig 21304)和PABP(Gen-ID:Contig 7968)。
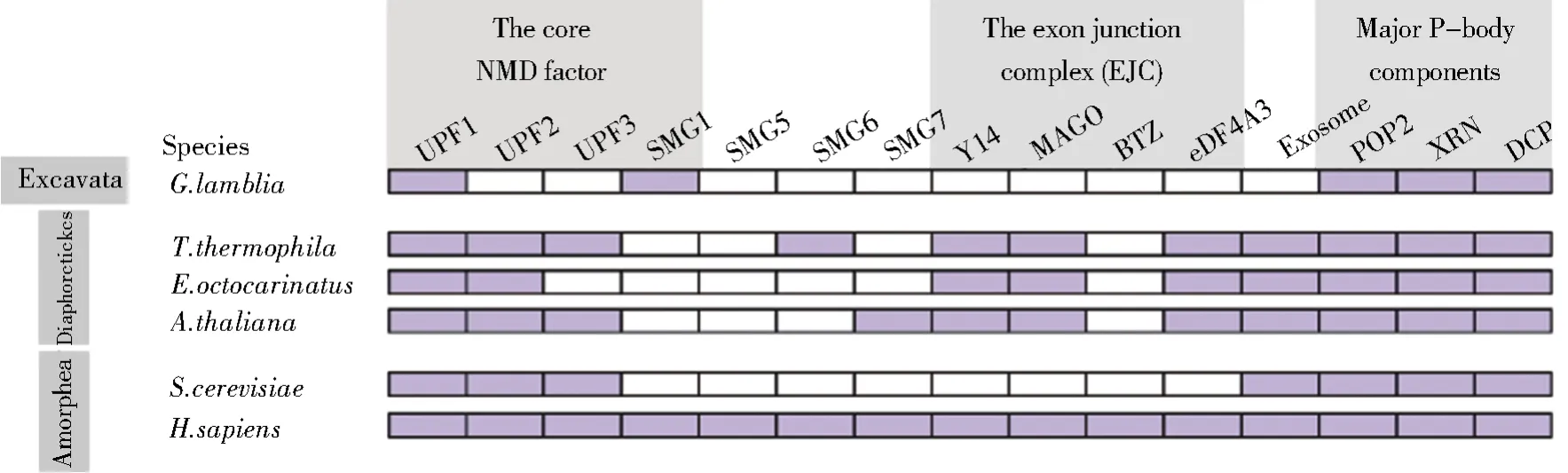
图1 八肋游仆虫与其他生物的NMD途径相关同源蛋白因子比较Fig.1 Comparison of NMD pathway-related homologous protein factors betweenE.octocarinatusand other organisms

表1 本研究所用到的寡核苷酸引物Table 1 List of oligonucleotide primers used in this work
用人类SMG1蛋白氨基酸序列在Euplotes octocarinatusGenome Database中作tblastn搜索时,得到一条属于PIKK(Phosphatidylinositol 3-kinase-reated protein kinase)蛋白激酶家族的序列,PIKK蛋白激酶家族由SMG1、ATM、ATR、PRKDC和mTOR成员组成,它们在基因准确表达的监控上起到重要作用[18],功能结构域注释和系统发育树结果(图2)显示,其与 mTOR(mammalian target of rapamycin)聚为一支,所以认为mTORL蛋白(GenID:Contig 9214)可能是八肋游仆虫的PIKK家族成员。对mTORL的功能结构域预测,发现在其N端没有人类 mTOR 的 HEAT(huntingtin,elongation factor 3,PP2A and TOR1)重复结构域,而是未知功能的结构域DUF3385,但拥有FAT、FRB、PIKK和FATC这些PIKK家族蛋白保守结构域。
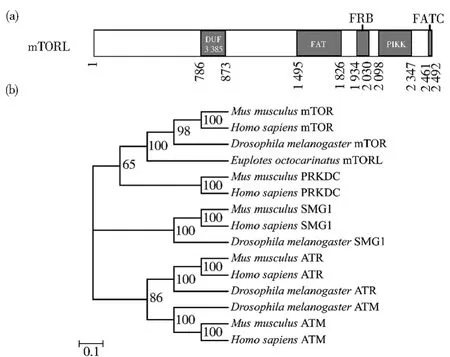
图2 八肋游仆虫mTORL蛋白功能结构域注释(A)及PIKK家族蛋白系统发育树(B)Fig.2 Schematic representation of mTORL ofE.octocarinatus(A)and phylogenetic tree of PIKK family proteins(B)
对鉴定到的这些NMD相关蛋白因子进行结构域注释(图3)。八肋游仆虫UPF1与人类UPF1的相似度为40%,与嗜热四膜虫UPF1a的相似度为43%,与嗜热四膜虫UPF1b的相似度为23%。S/T模体(motifs)是UPF1上蛋白激酶SMG1的磷酸化修饰位点,在八肋游仆虫、嗜热四膜虫以及酵母UPF1蛋白中均没有人类UPF1蛋白C末端的S/T模体,并且在这三种生物细胞中也都没有找到SMG1蛋白因子。八肋游仆虫UPF2具有3个MIF4G结构域,与人类UPF2蛋白相似度为18%,与嗜热四膜虫UPF2蛋白相似度为17%;鉴定到的八肋游仆虫的四种外显子连接复合体组分蛋白因子eIF4AIII、MAGO、Y14和UAP56中,前三种是EJC核心蛋白组分,其与人类相应的因子相比,相似度分别为65%、67%、41%和59%;与嗜热四膜虫相比,相似度分别为67%、68%、36%和45%。加工体(processing body,P-body)是真核生物细胞中mRNA降解的场所[19-20],从八肋游仆虫基因组中分别鉴定到的P-body组分蛋白有XRN1、POP2和DCP2。这三种蛋白与人类同源蛋白相似度分别为24%、38%和19%。外切体(exosome)在mRNA去腺苷酸化后会沿着5′→3′的方向降解mRNA[21],八肋游仆虫外切体蛋白与人类外切体9相似度为22%。PABP(poly A binding protein)蛋白与Poly(A)尾巴的结合对于维持mRNA的稳定性以及无义mRNA的识别至关重要[22],八肋游仆虫PABP蛋白与人类PABP蛋白的相似度为23%。

图3 八肋游仆虫NMD相关蛋白因子功能结构域示意图Fig.3 Schematic representation of functional domain of NMD-related protein factors inE.octocarinatus
2.2 eUPF1与eUPF2蛋白三级结构预测与蛋白分子对接
eUPF1和eUPF2是NMD途径核心蛋白因子,eUPF1-CH结构域与eUPF2-U1BD结构域之间的相互作用对于NMD途径的启动至关重要。UPF1-CH结构域多序列比对结果表明该结构域在进化上比较保守(图4A),CH结构域是一段富含Cys和His残基的结构域。其中,在eUPF1(71-166 aa),即对应人类hUPF1的123-213 aa的这段比对区域中,Cys-His占16.67%;对UPF2-U1BD结构域的多序列比对,及其二级结构与人类hUPF2-U1BD结构域二级结构比对的结果(图4B)表明,eUPF2-U1BD-αA在结构和序列上相对保守。前期通过体外Pull-down和酵母双杂交实验验证了eUPF1-CH与eUPF2-U1BD之间的相互作用关系,并阐明了UPF2对NMD途径启动的重要性[11-12],但这二者之间如何进行相互作用仍不清楚。

图4 八肋游仆虫UPF1-CH和UPF2-U1BD结构域的多序列比对Fig.4 Sequences alignment of UPF1-CH and UPF2-U1BD domains ofE.octocarinatuswith that of other species.(A)Multiple sequence alignment analysis of amino acid sequences of UPF1-CH protein.In UPF1(71-166 aa)of E.octocarinatus,using small triangle mark at the conservative sequence of Cys and His residues.(B)Multiple sequence alignment analysis of amino acid sequences of UPF2-U1BD protein.The secondary structures of UPF2-U1BD ofE.octocarinatusand human were marked,respectively
蛋白质分子的空间结构决定其功能,采用同源建模预测eUPF1蛋白的三级结构(图5A)。以人类UPF1-UPF2复合体的晶体结构(PDB ID 2wjv.1.A)为模板建模,模型质量QMEAN得分为-1.81,可信度GMQE得分为0.66,与模板的相似度为47.91%。对eUPF2蛋白三级结构进行预测(图5B),模型质量评估系数C-score得分为-0.48,两两结构相似度系数TM-score得分为(0.65±0.13),两两结构间的距离偏差RMSD得分为(8.4±4.5)Å。
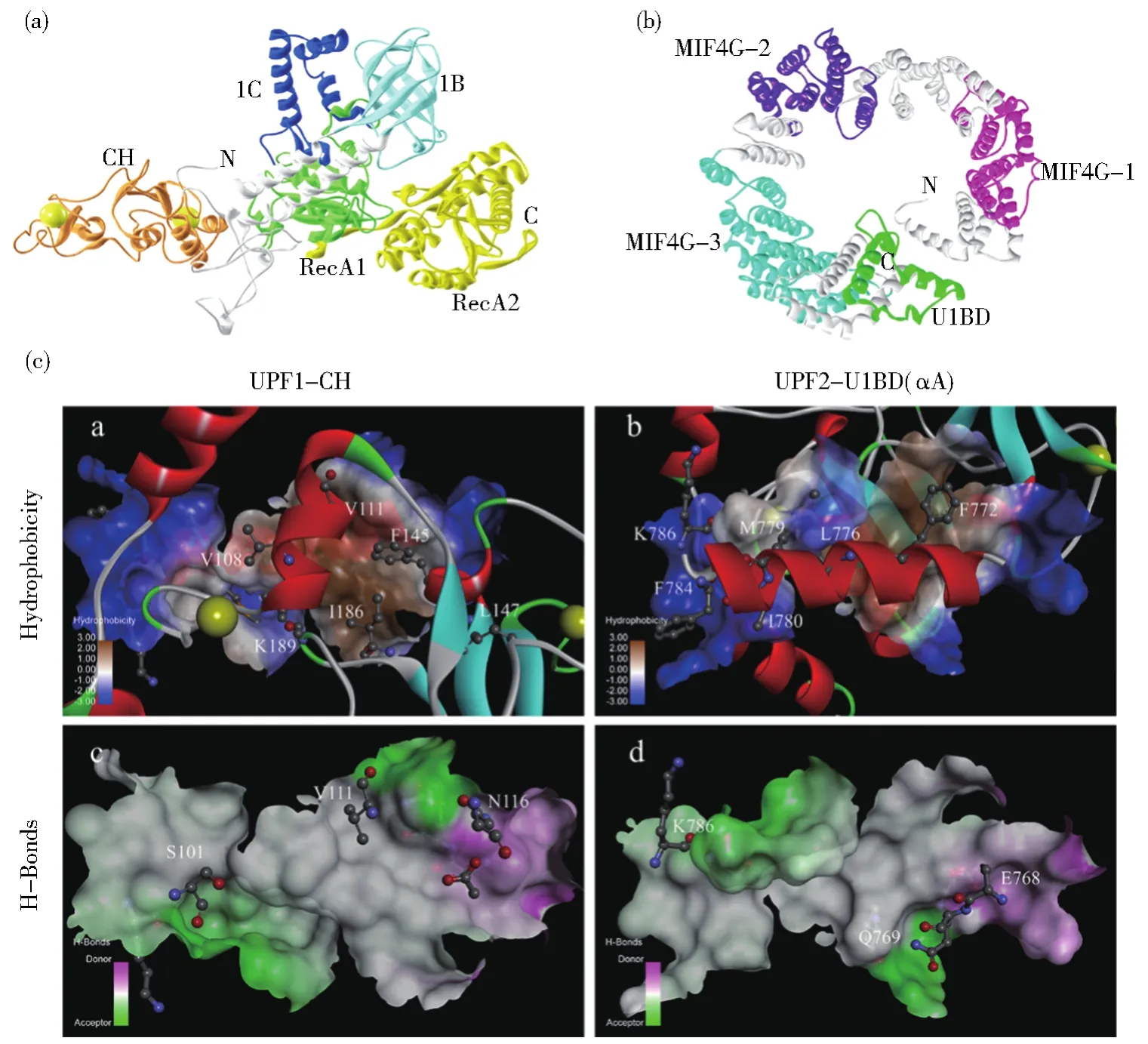
图5 八肋游仆虫UPF1与UPF2蛋白三级结构预测及其相互作用关键氨基酸分析Fig.5 Prediction of tertiary structure of eUPF1 and eUPF2 proteins and analysis of key amino acids for their interaction
为了进一步研究eUPF1-CH与eUPF2-U1BD(αA-helix,767-786 aa)之间相互作用的关系,将二者进行蛋白分子对接,以hUPF1与hUP2相互作用的关键结构和氨基酸为参照,从对接结果中挑选合理的对接模型。对挑选好的蛋白复合体模型,采用PDBePISA进行在线分析。分析结果显示,它们之间的相互作用面积为793.6 Å2,在这种对接方式下的自由能ΔiG的值为-13.1。
已有研究证实hUPF1-CH上与hUPF2-U1BD相互作用的关键氨基酸残基有V157、V161、F192、L193、I233、R236[23]。根据多序列比对结果(图4A)和PISA结果,我们推测eUPF1-CH上与eUPF2-U1BD相互作用的关键氨基酸残基是V108,V111,F145,L147,I186 和 K189,其中eUPF1-CH 残基V111、F145和 I186,与hUPF1-CH 残基 V161、F192和I233在多序列比对上相一致,eUPF1-CH残基V108和L147分别对应hUPF1-CH残基V157的+1位氨基酸和L193的+2位氨基酸,而hUPF1-CH上氨基酸残基R236对应eUPF1-CH上残基K189,二者均为带正电荷的氨基酸,来维系蛋白质的空间结构。
在hUPF2-U1BD中的αA-helix结构(767-786 aa)中,与hUPF1-CH结构域相互作用的关键氨基酸有 F1113、I1114、L1117、M1120、M1121 和 L1125[23]。而在对接模型中,eUPF2-U1BD通过PISA分析、多序列比对(图4B)、三级结构预测(图5B),找到两种因子相互作用的关键的氨基酸是F772、L776、M779、I780、F784 和 K786。其中F772、L776、M779与hUPF2中氨基酸残基F1113、L1117和M1120在序列比对上相一致。
对接模型经过Discovery Studio 4.5 Visualizer软件进行分析(图5C)后,发现eUPF2-U1BD中的αA-helix结构与eUPF1-CH相互作用面具有较强的疏水作用(图5.Ca,Cb)。此外,这两个相互作用的结构域还靠氢键来维持(图5.Cc,Cd),形成氢键的氨基酸残基分别是N116与E768、S101与K786、V111与Q769。
2.3 eUPF2-U1BD空间结构中的αA-helix对于eUPF1与eUPF2的相互作用起到关键作用
已有研究证实,在hUPF2-U1BD的N端部分(1108-1128 aa)会形成一个长而微弯的α-螺旋,C末端部分会折叠形成β发夹结构(1167-1189 aa)[23]。这两个结构分别独立地结合在hUPF1 CH结构域的两个相对的面上。在eUPF2蛋白中,通过多序列比对以及结构预测发现,eUPF2-U1BD空间结构存在三个α-螺旋(图4B和图5B),但只有αA与hUPF2-U1BD在结构和序列上与之相对应,即αA-helix结构(767-786 aa)。那么αA-helix是否也是eUPF1与eUPF2之间相互作用的关键结构呢?为了验证这一问题,我们利用定点突变技术,将这段αA-helix对应基因进行缺失突变(图6A)。然后利用原核表达系统进行表达,以野生型为对照。采用GST Pull down和His Pull down实验来分别验证野生型和突变型eUPF2-U1BD与eUPF1-CH之间的相互作用。研究结果(图6B)发现,eUPF2-U1BD突变型丧失了其与eUPF1-CH相互作用的能力,说明αA-helix对于eUPF2-U1BD与eUPF1-CH之间的相互作用至关重要。
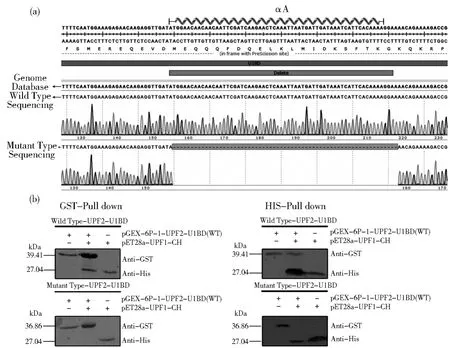
图6 αA-helix结构在eUPF2-U1BD与eUPF1-CH互作过程中关键作用Fig.6 Important role of αA-helix for the interaction between eUPF2-U1BD and eUPF1-CH domain
3 讨论
纵观NMD通路的研究历程,在以人类为代表的高等真核生物和以酿酒酵母为代表的低等真核生物上取得了丰硕的研究成果,并提出了多种合理的模型来解释含有PTC的转录本如何被识别和降解。人类NMD途径(图7a)监控mRNA的质量基于首轮的翻译过程,当核糖体滑动停滞到PTC处时,首先招募形成SURF复合体,随后UPF2作为桥梁连接起了与EJC结合的UPF3b因子,从而提出了依赖于外显子连接复合体的经典模型[1]。值得注意的是,PTC需处于最后一个外显子连接处上游50-55 nt时,mRNA才能被NMD识别和降解,这也被称为“50 nt边界规则”[24]。随着人类NMD因子的复合晶体结构逐渐被解析[23,25-27],各个因子间的相互作用又充分得到证实。而对于酵母的NMD途径(图7b)有学者提出了“伪3’UTR模型”[5],认为PTC的出现导致了3’UTR的加长,阻碍eRF3与PABP的正常结合,触发了NMD途径。
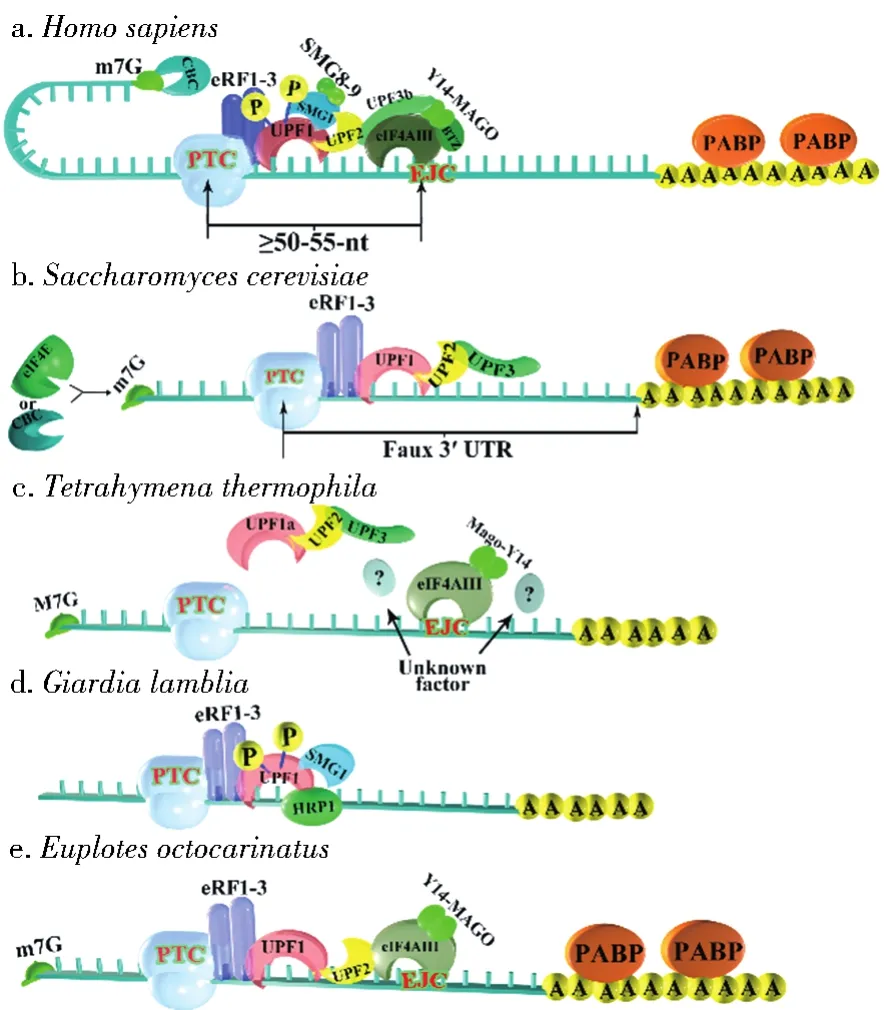
图7 不同物种识别含PTC转录本的机制比较Fig.7 Schematic diagram showing how different organisms′NMD factors are involved in PTC-containing transcript degradation
原生动物在起源与进化上处于特殊地位,通过对原生动物的NMD通路研究,可以更好地了解NMD通路的起源与进化,但关于原生动物的NMD通路的研究报道却十分稀少。嗜热四膜虫NMD通路的研究(图7c)[6]证实,在敲除了EJC组分核心因子MAGO的基因后,细胞中含PTC的转录本不会因此上调,且通过GST-Pull down实验也证实了EJC与UPF3并未有相互作用关系,进而证实了四膜虫的NMD途径不依赖于EJC。通过基因敲除等技术手段,证实了UPF蛋白作为NMD途径的核心因子,初步推断了嗜热四膜虫的NMD模型,但是该模型仍然不系统、不完整。
本课题组之前曾对蓝氏贾第虫NMD通路(图7d)进行过研究,基于酵母双杂交和Pull-down实验,发现停滞在PTC处的核糖体上也会形成SURF复合体,随后UPF1又会与HRP1相互作用,对无义mRNA识别和锚定,激活了NMD途径,同时也证实了XRN1和Ski7P作为降解含PTC转录本的下游降解因子[7-8]。此外,课题组前期也对八肋游仆虫的NMD通路进行过研究[11-12],发现八肋游仆虫NMD通路存在UPF1、UPF2、MAGO和Y14因子,基于酵母双杂交和Pull-down实验,证实了UPF1的CH结构域与UPF2的U1BD结构域之间存在相互作用关系。在此基础上,本研究结合人类同源蛋白晶体结构的研究[23],即通过hUPF2 U1BD结构域上保守的α-螺旋和β发夹结构分别独立地与hUPF1 CH结构域中两个相对的面结合。利用Pull-down实验证实了八肋游仆虫eUPF2可能通过其保守的αA-helix结构域参与eUPF2与eUPF1之间的相互作用。此外,课题组之前也证实了Mago与Y14之间的相互作用关系。本研究在此基础上又找到一个EJC核心因子eIF4A3,那么它是否与Mago和Y14存在相互作用关系仍有待于实验证明。另外本课题组之前证实了UPF2的第三个MIF4G结构域与Y14之间的相互作用关系,初步提出了eRF1-eRF3-UPF1-UPF2-EJC的模型(图7e)。然而八肋游仆虫的NMD通路与人类相比仍然不完善,不系统。在通路的下游,无义mRNA又会被哪些因子降解呢?为此,基于同源序列搜索,从八肋游仆虫大核基因组数据库中共鉴定出11种NMD相关因子,其中可能直接参与mRNA降解的因子有脱帽酶DCP2、去腺苷酸化酶POP2、外切酶XRN1和外切体exosome,那么这些因子是否也具有类似人类同源蛋白的功能[1],仍有待于后续的实验验证。
在八肋游仆虫基因组数据库中并未找到SMG1,在其他真核生物分类谱系中也能找到SMG1缺失的物种(如拟南芥,酿酒酵母)[2]。在人类NMD通路中,SMG1可以磷酸化修饰hUPF1的S/TQ模体,改变蛋白质分子的空间构象,调节UPF1的解旋酶活性[25]。磷酸化UPF1执行完功能后会被去磷酸化酶PP2A(serine/threonine protein phosphatase 2A)去磷酸化[1],然而在游仆虫大核基因组数据库中也没有找到PP2A。那么在SMG1缺失的物种中,UPF1是否需要被磷酸化激活或被其他激酶(如酪氨酸蛋白激酶)激活仍然是NMD机制悬而未解的重要问题。
