探讨DNA 条形码对摇蚊属物种界定的有效性
2021-04-10余海军
余海军,王 茜
(天津农学院水产学院/天津市水生生态及养殖重点实验室,天津 300384)
摇蚊属(Chironomus)隶属于双翅目摇蚊亚科(Diptera:Chironomidae),该属由德国昆虫学家Mei⁃gen 于1803 年建立,迄今为止全世界已记录种类达303 种;摇蚊属的幼虫可以生存于各种水体(尚未污染至严重污染的水体),其中宽叶摇蚊亚属(Lobochi⁃ronomus)在缺乏营养的水体中较为丰富,并且能耐受酸性环境,另一些摇蚊亚属喜欢生长在盐分较高的环境中,对重金属污染具有耐受性[1]。摇蚊属部分种类雄成虫能引起人类过敏性疾病;裸露在外和正在产卵的摇蚊属种类可以成为新鲜水体中鱼类(鳟鱼)的主要食物来源;在日常生活中,某些垂钓者所用诱饵也为摇蚊属幼虫[2]。
摇蚊属是摇蚊科最古老的属之一,由于种种历史原因,该属的研究在世界上一直存在很大的争议,属内物种的分类地位至今没有获得广泛的认可。依据中国摇蚊名录中统计,中国已有该属种类共23种,其中只有13种具有明确记录,另包括3种可疑种和7种只进行鉴定而没有正式发表的[3]。因此,摇蚊属的物种界定一直存在争议[3]。随着DNA 条形码的提出,其通过将基因组的标准部分提取一个或几个相对较短的基因序列,用于识别物种[4,5];虽然使用DNA序列识别生物并不新颖[6],但DNA 条形码能快速且可靠地识别所有生命形式的物种层次,包括动物、植物、真菌、微生物;其在论证物种群落组成、食物链和种内遗传变异方面也有所研究[7-9];同时也扮演着生物安全和淡水生态监测中一个不可缺少的角色[10-13]。Lin 等[14]使用DNA 条形码对长跗摇蚊属(Tanytarsus)121 个形态种进行物种鉴定有效性分析,结果表明DNA 条形码明确区分出94.6%的先前通过形态学研究分类的长跗摇蚊属物种;Song 等[15]使用DNA 条形码对多足摇蚊属(Polypedilum)161 个形态种进行物种鉴定有效性分析,结果表明现已有研究中多足摇蚊属94.4%的种类能有效被DNA 条形码区分。
本研究基于编码细胞色素C 氧化酶甲基I(COI)基因对摇蚊属157 种的749 条DNA 条形码进行分析研究,并利用相关软件对摇蚊属类群进行了遗传距离和系统发育树的分析,目的是探索DNA 条形码在摇蚊属物种界定方面的有效性,同时为完善摇蚊科DNA 条形码数据库提供参考,以填补当前国内该属的研究空白。
1 材料与方法
1.1 分类抽取和收集信息
研究中695 条DNA 条形码数据为BOID 和Gen⁃bank 数据库提供,54 条为收集样本,采集于新疆、宁夏、海南、贵州、广西、天津、浙江等地;采集方式包括灯诱、马氏网、D 型网、扫网;将采集后的幼虫样本保存于95%乙醇中,成虫样本保存于85%乙醇中,并存于4 ℃冰箱,用于后续的形态学和分子学研究。
1.2 DNA 提取和PCR 扩增
本研究通过Qiagen DNA Blood and Tissue kit 试剂盒,采用摇蚊成虫的头、胸部和幼虫的腹部提取基因组DNA。提取后成虫的头、胸部保存到与翅、足和触角一致的玻片标本上,用Euparal 树胶封片,凭证标本均存于天津农学院水产遗传育种实验室。
使用通用引物LCO1490和HCO2198扩增COI条形码[16]。PCR 扩增反应体系为25 μL,包含12.5 μL 2×Es Taq MasterMix,上下游引物各0.625 μL,dd H2O 8.75 μL,DNA模板2.5 μL。通过Master Cycler Gradient进行扩增,扩增程序为98 ℃预变性10 s,94 ℃5 min,35个循环(94 ℃1 min,51 ℃1 min,72 ℃1 min),最后72 ℃延伸5 min;10 ℃保存。PCR 扩增产物经1.5%琼脂糖凝胶电泳检测后,送北京六合华大基因科技股份有限公司进行纯化测序。
1.3 序列分析和对比
各个基因片段都采用双向测序,根据测序公司返回的测序结果,使用软件SeqMan 7.1.0.44 观察峰图质量编辑序列[17]。将编辑好的FASTA 文件导入MEGA7.0 后选择标准密码子进行后续密码子比对。
用MEGA7.0 软件分析对比后,对各样本碱基的组成、简约信息位点、变异位点(Variable sites)、保守位点、自裔位点、平均碱基含量、遗传距离和碱基转换与颠换比值进行统计。选用Kimura-2-parameter(K2P)分子进化模型,节点处置信度为1 000,其他设置均为默认值,建立邻接树。
利用ABGD 软件分析mOTU 的数量[18]:将编辑好的FASTA 文件在线提交到ABGD 网站(http://ww⁃wabi.snv.jussieu.fr/public/abgd/),选择K2P 模型,种内差异先验值P 为0.001~0.100,相对gap 宽度值X 为1.0,其余参数设置为默认值[15]。本研究的54 条DNA 序列及其相关信息均已上传BOLD(http://www.boldsystems.org)数据库。
2 结果与分析
2.1 序列特征
研究共获得摇蚊属749 条DNA 条形码,其中实验 室 获 得54 条DNA 条 形 码,695 条 来 自BOID 和Genbank 数据库,所有序列的长度均为658 bp,各碱基的平均含量分别为A=26.56%,T=37.67%,C=18.82%,G=16.92%,序列存在碱基的偏倚性,C+G的含量显著低于A+T 的含量,特别是第三核酸位点A+T 的含量为85.15%。通过比对序列得出,信息位点有244个,占37.08%;变异位点有261 个,占39.67%。大部分的变异位点都集中在第三核酸位点,其比例为78.16%,表明在第三核酸位点上的碱基进化速率快。
2.2 遗传距离
在DNA 条形码研究中,种内与种间遗传距离的差异是一项非常重要的指标,当种间遗传距离大于种内遗传距离,就会形成一个明显的空白区[15,19]。
实验室共获得54 条DNA 条形码,形态鉴定约为12 种。结合BOID 和Genbank 数据库已下载的数据,分析统计共获得形态种约50 种。在MEGA 7.0软件中,对所有形态种进行分组,即60 个组。种内遗传距离为0~7.21%,平均值为1.10%;种间遗传距离为0.2%~19.4%,平均值为14.25%;种间平均遗传距离是种内平均遗传距离的12.95 倍,因此本研究的749 条形码数据存在明显的空白区。
Hebert 等[5]通过对无脊椎动物及脊椎动物11门13 320 个物种进行了DNA 条形码的研究,并提出了种间平均遗传距离大于种内平均遗传距离的10 倍作为区分物种的标准;随着DNA 条形码的深入研究,不同领域的学者对该标准进行了验证,在摇蚊科不同类群中也出现了相应的研究,详细结果见表1。
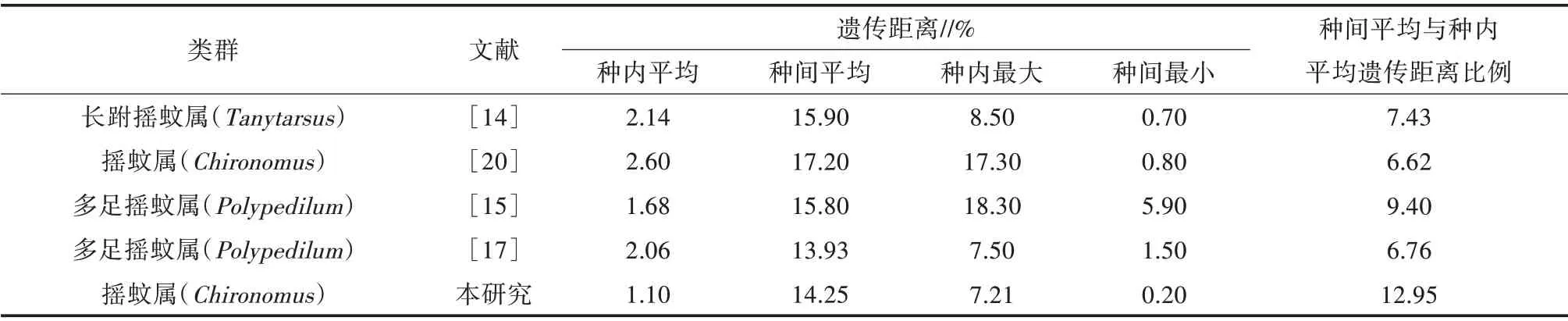
表1 摇蚊科不同类群遗传距离比较
2.3 系统发育树结果
本研究采用DNA 条形码分析中最常用的邻接法,基于54 条DNA 条形码明显分离出13 个分类操作单元,代表摇蚊亚科中12 个已命名的物种,结果显示DNA 条形码的鉴别结果与形态学鉴定结果一致(图1)。
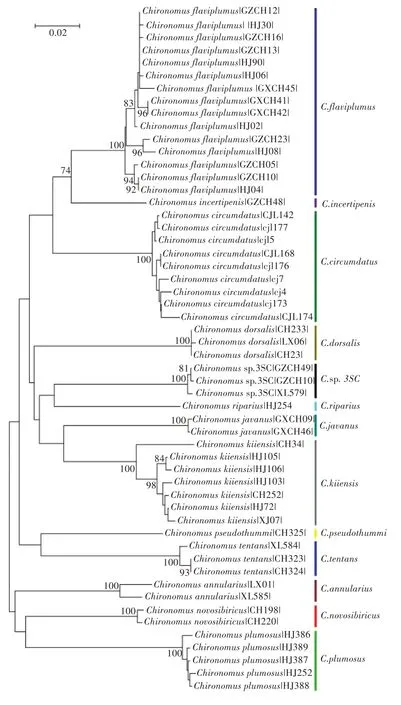
图1 54 条DNA 条形码邻接树
2.4 ABGD 评估结果
ABGD 在线软件通过比较种内和种间的遗传距离,当种间最小遗传距离大于种内最大遗传距离时则出现空白区,DNA 条形码空白区的存在很容易将物种分开(图2)。
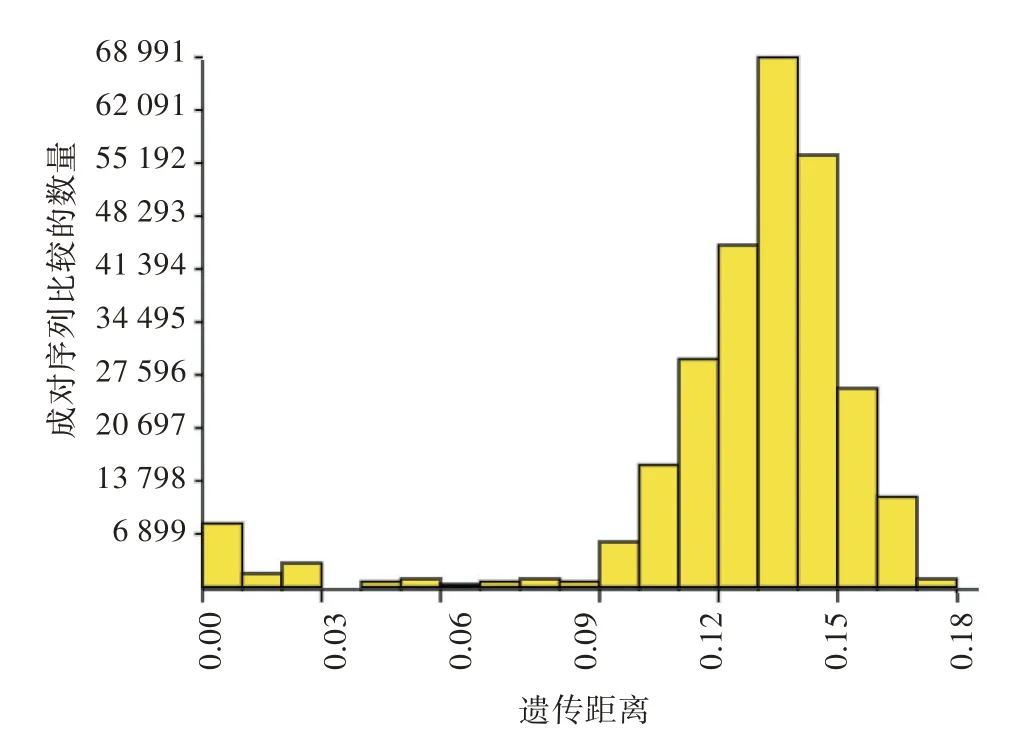
图2 749条DNA条形码基于K2P模型得到的遗传距离分类
基于ABGD 在线软件评估摇蚊亚科OTUs 的数量,即通过设定不同的阈值P,得到不同的OTUs 数量;随着P 增大,OTUs 的数量减少(图3)。当P 为0.002 637 时,749 条DNA 条形码被分为104 OTUs;P 为0.026 827 时,被分为61 OTUs。即OTUs 的置信区间可为61~104,ABGD 所评估的OTUs 的数量略大于形态评估的种类。当P 为0.037 276 时,被分为58 OTUs,最为接近邻接树法评估的分子操作单元数量,因此ABGD 方法评估749 条摇蚊亚科DNA条形码的遗传距离阈值为3%~4%。
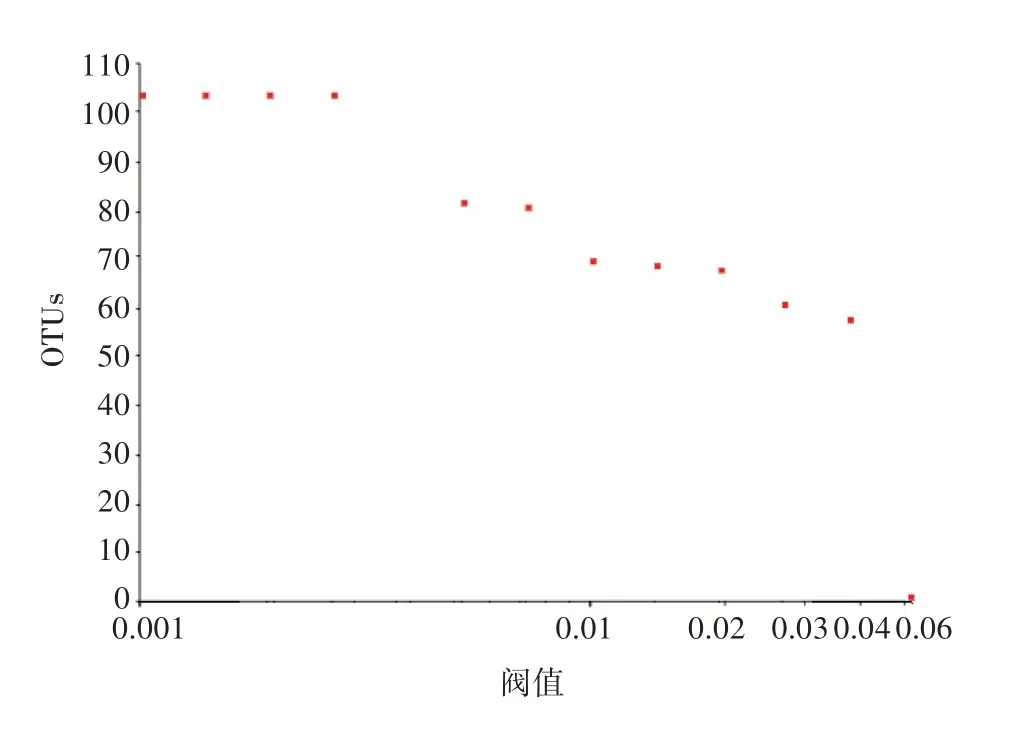
图3 ABGD 在线评估OTUs的数量随P 的变化
3 讨论
目前为止,DNA 条形码技术存在5 种分析方法,即遗传距离的分析、遗传相似度的分析、系统发育树的分析、序列特征的分析和统计分类法的分析[21],而在摇蚊DNA 条形码研究中,主要是基于遗传距离和系统发育树的分析[22-28]。
基于遗传距离的分析,即通过计算DNA 条形码序列的遗传距离,从而实现物种的区分,主要分析方法包括DNA 条形码间隙和遗传距离阈值分析[21]。DNA 条形码间隙的一项非常重要指标是种内与种间遗传距离的差异,当种间遗传距离大于种内遗传距离,就会形成一个明显的空白区。本研究中种内平均遗传为距离为1.10%,种间平均遗传距离为14.25%,后者大约是前者的12.95 倍,形成了空白区。遗传距离阈值是指物种在能形成空白区的前提下,使用物种种间遗传距离与参比的遗传距离阈值进行对比,当种间遗传距离大于参比的阈值,则可以将物种区分为不同物种。阈值法在DNA 条形码的物种鉴定中起着不可替代的作用,然而随着DNA 条形码的研究不断出现,有学者发现不同物种DNA 条形码的阈值各不相同,没有一个统一的标准阈值[29]。BOLD 数据库最初将遗传距离1%作为生物物种的划分阈值,由于其阈值宽泛,在一定程度上造成了物种的鉴别过度,导致同种异名的出现,为了不使物种混乱,近年来BOLD 系统默认鉴别阈值为3%[30];Meier 等[31]在对449 个双翅目昆虫物种的研究中,发现在物种划分时,其遗传距离大于3%;He⁃bert 等[5]以3%作为阈值对200 个鳞翅目昆虫物种进行鉴别;蜉蝣目[32-34]、襀翅目和毛翅目[35,36]的物种以2%的阈值就能将物种区分开。本试验研究的物种隶属于双翅目,通过使用ABGD 方法评估得到的遗传距离阈值为3%~4%,与Meier 等[31]的研究相符,且与摇蚊亚科中其他属研究的阈值差异不大,Song 等[15]区分多足摇蚊属的遗传距离阈值为5%~8%,Lin 等[14]区分长跗摇蚊属的遗传距离阈值为4%~5%。本研究是分析摇蚊属物种的DNA 条形码,其属于摇蚊亚科中比较大的属级之一,属内物种的丰富度极高,但由于时间有限,加之中国地大物博,从而不能完全覆盖已记录所有物种和一些隐种;为此还需要更深入的研究,才能达到对中国地区摇蚊属遗传距离的准确界定。
系统发育树能直观地反映物种之间的亲缘关系,本研究采用的是邻接树法,其基于遗传距离和最小进化原理,通过自举检验法进行聚类,并通过统计给定树分支的可信度,得出最优树[37]。在邻接树中,基于54 条来自实验室的DNA 条形码明显分离出12 个物种,与形态学鉴定结果一致,匹配率达92.3%;通过结合BOID 和Genbank 数据库中的695条DNA 条形码,分离出50 个形态种,其中发现本试验数据Chironomus pseudothummi 与参比数据Chi⁃ronomus sp.TE11 聚为一支(图4),由于数据库中参比数据Chironomus sp.TE11 不能提供证据标本与本研究比较,将基于本研究的数据将参比数据Chi⁃ronomus sp.TE11 修 改 为Chironomus pseudothummi,并将相关信息上传至BOLD 数据库。
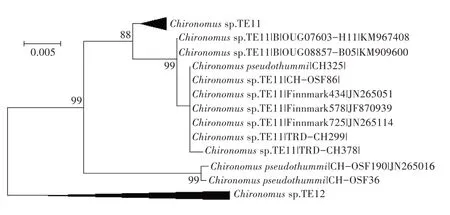
图4 Chironomus pseudothummi 类群邻接树
4 结论
本研究通过采集中国地区的摇蚊属昆虫12 种54 个样本,结合BOID 和Genbank 数据库中的相关数据,共计749 条DNA 条形码,运用ABDG 法分析该数据集遗传距离,结果显示种内与种间存在一个明显的空白区;基于邻接树结果得出数据集分离出50 个物种,与传统形态学鉴定一致,匹配率高达92.3%,并修正数据库中Chironomus sp.TE11 为Chironomus pseudothummi,证明DNA 条形码能有效鉴别摇蚊属昆虫。本研究的数据均已上传中国DNA 条形码数据库,在丰富中国摇蚊科数据库的同时,也为探索DNA 条形码在摇蚊昆虫中鉴别的有效性提供了基础数据。
