常用基因序列在摇蚊科昆虫系统发育研究中的应用进展
2016-12-14闫春财赵广君戈昕宇王新华
闫春财,郭 琴,赵广君,戈昕宇,王新华,刘 婷
(1.天津师范大学a.生命科学学院,b.天津市动植物抗性重点实验室,天津300387;2.南开大学生命科学学院,天津300071)
常用基因序列在摇蚊科昆虫系统发育研究中的应用进展
闫春财1,郭 琴1,赵广君1,戈昕宇1,王新华2,刘 婷1
(1.天津师范大学a.生命科学学院,b.天津市动植物抗性重点实验室,天津300387;2.南开大学生命科学学院,天津300071)
摇蚊科隶属于双翅目长角亚目,现行分为11亚科.本文整理了常用核基因和线粒体基因在摇蚊科系统发育研究中的应用情况:摇蚊科中的9亚科(不含智利摇蚊亚科和乌桑巴摇蚊亚科),除前寡角摇蚊亚科外,其余均为单系;直突摇蚊亚科与摇蚊亚科的亲缘关系较近,寡脉摇蚊亚科与长足摇蚊亚科为姊妹群.在摇蚊亚科中,摇蚊族(Chironomini)、长跗摇蚊族(Tanytarsini)都是单系群.常用核基因和线粒体基因在摇蚊科昆虫系统发育研究中,18SrDNA和28SrDNA适合研究从界到科所有高级阶元的系统发育关系,对于部分类群可以进行属间分析;CAD基因能较好地解决从亚科到属之间各分类阶元的系统发育关系;ITS和EF-1ɑ可以解决种或属间的系统发育关系;COⅠ和COⅡ适合研究属、种及种以下阶元的系统发育关系,但不能很好地解决较高的阶元如族间、亚科以及科的系统发育关系;16SrDNA适用于种、属阶元水平的系统学研究,但不适用于种内;Cytb适合研究种内到种间甚至科间的系统发育关系.
摇蚊科;基因;分子标记;分子系统学;系统发育
摇蚊科(Chironomidae)隶属于昆虫纲双翅目长角亚目(Diptera:Nematocera),现行分类系统中该科分为11亚科,世界范围内约有2万种,已记述种类超过5 000种.摇蚊科广布于全球各大生物地理区域,即使在高山寒冷地带,如喜马拉雅山海拔5 600 m的地区仍有分布,某些种类在-16℃下仍能够保持活跃状
态[1].摇蚊体型微小至中型,成虫口器退化,幼虫生活在各类型水体中,是分布最广、种类最多、生物量最大的淡水底栖动物类群之一,同时也是重要的水体环境指示生物.在摇蚊的系统发育研究中,以往多采用形态学方法对种、属进行鉴定,但对于形态相近的物种,如摇蚊属(Chironomus)的某些种,则需借助细胞遗传学方法,如通过染色体数目与形态、带型、组型、性染色体位置及染色体减数分裂行为模式等特征来鉴别昆虫种类[2].虽然该方法在种属鉴定上很有帮助,但在研究摇蚊科的系统发生关系上仍有不足,如无法通过染色体带型及组型来确定外群,从而无法通过外群建立系统发育树.而分子生物学方法能够很好地解决这一问题,由于DNA序列中富含生物学信息,因此成为研究生物系统发育和进化规律的重要工具[3-4].
摇蚊科的系统发育研究始于1914年Goetghebuer[5]对摇蚊亚科(Chironominae)主要类群间系统发育关系的探讨.1966年Brundin[6]首次报道了摇蚊种间水平的系统发育关系.此后,许多学者基于形态学研究了摇蚊科的多个类群[7-8].同传统方法相比,基于分子的系统学研究能够解决一些单纯依靠形态特征无法解决的某些类群的分类地位,并能为分类学中的种类鉴定、亲缘种及单系类群的识别提供简便快捷的方法.目前常用的主要有核基因序列和线粒体基因序列,两者在昆虫系统发育的不同分类阶元中各具优势.
本文总结了常用核基因和线粒体基因在摇蚊科昆虫系统发育研究中的应用情况,整理了摇蚊科主要类群的演化和系统发育关系,归纳出几种常用核基因(18S rDNA、28S rDNA、ITS、EF-1ɑ、CAD、组蛋白H3基因)和线粒体基因(COⅠ、COⅡ、Cytb、16S rDNA)在摇蚊科昆虫各分类阶元中的适用范围和应用情况.
1 常用核糖体基因在摇蚊科昆虫分子系统学上的应用
1.1 18S rDNA
18S rDNA是真核生物染色体上编码核糖体小亚基RNA的基因,在核糖体合成蛋白质的过程中具有重要作用.由于其核酸序列和二级结构具有高度保守性,因此被认为比较适用于高级阶元系统发育的研究,但其不同区域上的进化速率不同,因此也适用于不同的分类层[8].如Cranston等[9]用18SrDNA、28SrDNA、CAD联合COⅠ基因序列片段对冈瓦纳大陆寡脉摇蚊亚科(Podonominae)(共15属)中的13个属进行了系统发育分析,采用贝叶斯法和最大似然法(ML)构建的发育树表明该亚科为单系,是长足摇蚊亚科(Tanypodinae)的姊妹群;如果把长足摇蚊属(Trichotanypus)考虑在内,寡脉摇蚊族(Podonomini)为单系;拟绿摇蚊属(Parochlus)为单系,是 Podonomus属的姐妹群;Podochlus属为单系,是拟绿摇蚊属和Podonomus属的姐妹群;Afrochlus属与Archaeochlus属互为姊妹群. Cranston等[10]又利用文献[9]中的基因片段对摇蚊科的9个亚科(不包括智利摇蚊亚科(Chilenomyiinae)和乌桑巴摇蚊亚科(Usambaromyiinae))的系统发育关系进行了分析,发现除前寡角摇蚊亚科(Prodiamesinae)外,其余亚科均为单系;似蠓摇蚊亚科(Buchonomyiinae)是所有亚科的姊妹群;南部的阿福罗摇蚊亚科(Aphroteniinae)是除了似蠓摇蚊亚科以外所有亚科的姊妹群;在寡角摇蚊亚科(Diamesinae)中,Heptagiini、Harrisonini与寡角摇蚊族(Diamesini)互为姐妹群;在长足摇蚊亚科(Tanypodinae)中,五脉摇蚊族(Pentaneurini)单独形成一支;在直突摇蚊亚科(Orthocladiinae)中,直突摇蚊族(Orthocladiini)和中足摇蚊族(Metriocnemini)(不包括棒脉摇蚊属(Corynoneura)和布摇蚊属(Brillia))的分类地位也得到了支持.Martin等[11]利用2个线粒体基因和一个核基因18S rDNA对摇蚊亚科的系统发育关系进行了研究,结果证实基于不同的分子标记研究能够解决不同阶元的系统发育关系;拓扑学结果显示,该亚科主要分成两大支,二叉摇蚊属(Dicrotendipes)为这两大支的姐妹群:一支由球附器摇蚊属(Kiefferulus)、阿克西摇蚊属(Axarus)、光摇蚊属(Xestochironomus)、裸突摇蚊属(Goeldichironomus)、李摇蚊属(Lipiniella)及哈摇蚊属复合体(Harnischia complex)构成,属间有较近的亲缘关系;另一支由摇蚊属(Chironomus)(包括Camptochironomus和Lobochironomus 2个亚属在内)、弱摇蚊属(Baeotendipes)、恩菲摇蚊属(Einfeldia)等聚在一起,亲缘关系较近.
1.2 28S rDNA
28S rDNA是真核生物染色体上编码核糖体大亚基RNA的基因,在进化过程中比较保守,但变异性高于18SrDNA,其序列上含有D1~D12个高变区.28SrDNA同18S rDNA一样适合从界到科所有高级阶元的系统发育研究,对于部分类群可以进行属间分析[12].如Allegrucci等[13]利用28S rDNA的D1和D3~D5基因片段对南极的摇蚊科(Chironomidae)15个类群之间的系统学关系进行分析,最大简约树(MP)显示,直突摇蚊亚科(Orthocladiinae)、摇蚊亚科(Chironominae)、滨海摇蚊亚科(Telmatogetoninae) 和寡脉摇蚊亚科(Podonominae)各分别聚为一支,这4个亚科很容易区
分,其中直突摇蚊亚科与摇蚊亚科的亲缘关系较近.王茜等[14]利用28S rDNA的D1部分基因序列研究直突摇蚊亚科属级阶元的系统发育情况,基于距离临近法(NJ)和MP法建立的系统发育树如图1所示.2种发育树均表明,滨海摇蚊属位于系统发育树的基部,心突摇蚊属和真开氏摇蚊属互为姐妹群,流环足摇蚊属和刀突摇蚊属互为姐妹群.Huang等[15]基于28S rDNA的D1~D3、组蛋白H3的2个核基因序列以及COⅠ、Cytb 2个线粒体基因序列对海摇蚊属(Pontomyia)4个已知种和1个未知种的系统发育关系进行分析,结果表明,海摇蚊属为单系;海生摇蚊(Pontomyia oceana)和Pontomyia pacifica形成一大支,有较近的亲缘关系;形态上难以区分的2个种Pontomyia natans和Pontomyia cottoni聚在一起,支持率较高,无论是传统方法还是基于分子生物学的方法都难以区分,因此将二者视为1种,Pontomyia cottoni被认为是Pontomyia natans的次异名.
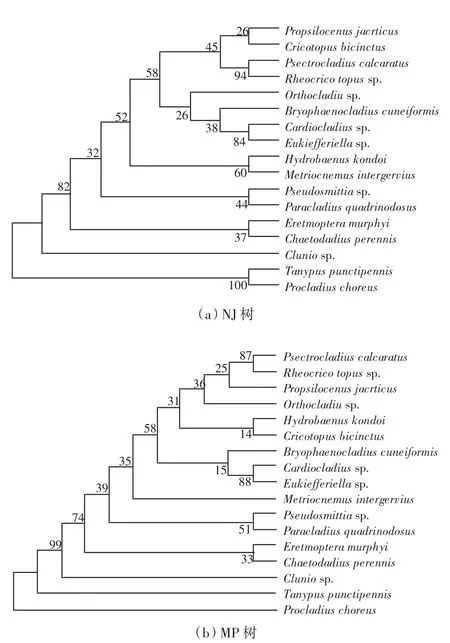
图1 基于28S rDNA D1序列构建的17种摇蚊系统发育关系NJ和MP树(引自文献[14])Fig.1 NJ and MP trees of 17 species of Chironomidae based on D1 region of 28S rDNA D1 sequence(cited from document[14])
1.3 ITS
由18S、5.8S和28S rDNA构成的转录单元中有2个可变区,即核糖体内转录间隔区ITS1和ITS2,ITS1位于18S和5.8S rDNA之间,ITS2位于5.8S rDNA和28S rDNA之间.由于ITS为非编码区,承受的选择压力较小,相对变化较大,并且能够提供详尽的系统学分析所需要的可遗传性状,因此可以解决种和属间的系统发育关系[16].如宗祖胜等[17]对我国南方某城市4座自来水厂沉淀池池壁上的摇蚊进行采集,将样品进行ITS1序列的PCR扩增和PCR-RFLP分析,采用最大简约法构建系统进化树,结果表明,摇蚊的ITS1序列在种内相当保守,在种间表现出一定的差异性,因此摇蚊ITS1序列可以作为鉴别摇蚊物种的理想分子标记之一.Sharma[18]基于核基因ITS1和ITS2序列对摇蚊的7个种(Thienemanniella xena、Xylotopus par、Tribelos fuscicorne、Robackia demeijerei、Xylotopus jucundum、Polypendilum aviceps和 Chironomus tentans)作了详细分析,结合从基因库中获得的15个物种序列,研究显示这些物种具有很高的种间变异,在欧洲分布的物种比在北美分布的物种更容易聚群;在进化树中较高的置信度和较短的聚类分枝能够表明可能存在的各种聚群及快速分化的物种.Asari等[19]利用核糖体内转录间隔区ITS2对日本水摇蚊属(Hydrobaenus)中的3个种,即Hydrobaenus kondoi、Hydrobaenus biwaquartus和Hydrobaenus kisosecundus进行区分,基于ITS2的分子生物学方法很容易把Hydrobaenus biwaquartus与其他2个物种加以区分,这与形态学研究的结果一致;但不支持通过传统方法(例如阳茎刺突、抱器端节的长度、触角比、第九背板的形状等)判定的Hydrobaenus kondoi、Hydrobaenus biwaquartus为同物异名的说法,可见ITS2对于水摇蚊属(Hydrobaenus)中相近物种的区分能够提供更加有用的信息.
ITS基因虽然可以用于摇蚊科物种的鉴别及系统发育分析,但由于该序列中A或T有较多的重复,从而会干扰测序,而且长度变化较大也可能会使序列的对齐产生误差.
2 常用编码蛋白的核基因在摇蚊科昆虫分子系统学上的应用
2.1 CAD
CAD是由3种参与从头合成嘧啶核苷酸途径的酶共同构成的融合蛋白,3种酶分别为氨甲酰磷酸合成酶(CPS)、天冬氨酸转氨甲酰酶(ATC)以及二氢乳清酸酶(DHO),其中CPS的基因片段长度约4 kb,含
遗传信息最多[20-21].Silva等[22]利用CAD的部分序列结合形态学特征对拉布长足摇蚊属(Labrundinia)(长足摇蚊亚科)中22个种的系统发育进行了验证,与单独利用CAD基因序列或者基于形态特征数据所做贝叶斯分析的结果基本相同,即拉布长足摇蚊属为单系,有较高支持率;分子结合形态数据产生的贝叶斯树结果显示,Labrundinia mayaca+L.paravirescens+L.jasoni+ L.paulae与L.semicurvata的亲缘关系最近,仅L. mayaca+L.paravirescens 2个分枝的支持率较高,这些结果不同于仅用CAD基因序列分析得到的结论;此外,L.amandae+L.unicolor互为 L.reniformis+L. parareniformis的姐妹群.Andersen等[23]对一种来自克罗地亚980 m深处的雌性Troglocladius hajdi进行研究,发现其部分形态特征类似于直突摇蚊族(Orthocladiini)和中足摇蚊族(Metriocnemini)中的一些类群,但仅凭传统方法很难将其归为任何一属.基于分子标记COⅠ、18S rDNA、28S rDNA、CADⅠ及CADⅣ分析该物种在直突摇蚊亚科(Orthocladiinae)中的系统发育关系,采用贝叶斯法构建系统树,结果表明,Troglocladius hajdi没有与直突摇蚊族中的环足摇蚊属(Cricotopus)聚在一起,而与中足摇蚊族(Metriocnemini)中的特维摇蚊属(Tvetenia)、心突摇蚊属(Cardiocladius)、真开氏摇蚊属(Eukiefferiella)聚在一起形成一大支,表明Troglocladius hajdi与这3个属间有较近的亲缘关系.
2.2 H3和EF-1ɑ
H3是一个编码与染色体结构形成有关的碱性蛋白质的基因,其序列高度保守,可用于研究高级分类阶元的系统发育;同时,其序列还存在进化速率较快的间隔区,适于分析亲缘关系较近的物种间的系统发育.因此H3可用于不同分类阶元层次上的分子系统学研究.EF-1a是单拷贝基因,在蛋白质翻译过中具有重要作用,其第一、二密码子高度保守,被看作有潜力研究高级分类阶元系统发育的基因,而第三密码子替换常常达到饱和,因此也可用其分析低阶元的系统发育关系[24-26].
为了研究海摇蚊属(Pontomyia)的系统进化,Huang等[27]测定其28S rDNA D1~D3、H3两个核基因序列以及COⅠ、Cytb两个线粒体基因序列,以Chironomus tepperi、C.thummi和Tanytarsus allicis为外群,采用最大简约法、最大似然法和贝叶斯法建立系统发育树,结果均支持海摇蚊属(Pontomyia)为单系.Ekrem等[28]分析了2个核基因(CAD、EF-1ɑ)和3个线粒体基因(COⅠ、COⅡ、16S rDNA)在摇蚊亚科(Chironominae)小突摇蚊属(Micropsectra)、泉摇蚊属(Krenopsectra)、拟长跗摇蚊属(Paratanytarsus)和Parapsectra属系统发育关系中的效用,结果发现,在一些属中COⅠ的系统学信号最弱,而CAD包含的信号最强;拟长跗摇蚊属是单系群,支持率较低;另外,在一些物种中用高度可变的EF-1ɑ序列首次发现了双翅目长角亚目的旁系同源基因拷贝.
3 常用线粒体基因在摇蚊科昆虫分子系统学上的应用
线粒体DNA在昆虫系统发育研究中应用广泛,其中,细胞色素C氧化酶亚基Ⅰ(COⅠ)和亚基Ⅱ(COⅡ)的序列既保守又有足够变异,是较好的分子标记. 3.1 COⅠ
在摇蚊科的昆虫系统学研究中,Krosch等[29]通过线粒体基因COⅠ和2个核基因28S rDNA、CAD对直突摇蚊亚科(Orthocladiinae)的系统学关系进行了分析,结果发现,拟开氏摇蚊属(Parakiefferiella)单独形成一支且较早地分离出来;真开氏摇蚊属(Eukiefferiella)与心突摇蚊属(Cardiocladius)、特维摇蚊属(Tvetenia)聚在一起,说明3者有较近的亲缘关系;Botryocladius(包括南美和澳大利亚种群)形成一个单系群;Anzacladius limnophyes与Ferringtonia patagonica形成姐妹群;南美的Echinocladius sp.与Naonella forsythia、Tonnoirocladius commensalis形成姐妹群.姜丽等[30]测定了摇蚊亚科(Chironominae)23个物种的线粒体COⅠ基因的部分序列,以环足摇蚊属(Cricotopus)作为外群,采用邻接法和最大简约法构建系统发育树,结果表明,多足摇蚊属复合体(Polypendilum complex)中的斑摇蚊属(Stictochironomus)、明摇蚊属(Phaenopsectra)、倒毛摇蚊属(Microtendipes)和内摇蚊属(Endochironomus)聚在一支,亲缘关系较近;狭摇蚊属(Stenochironomus)单独为一支;哈摇蚊属群(Harnischia complex)仅有部分属聚在一起;长跗摇蚊属群(Tanytarsus complex)中大部分属的亲缘关系也较近.
COⅠ在物种界定和识别方面具有良好的应用性,被视为DNA条形码[31-32],已证实在许多动物类群中运用该基因能够成功鉴定到种级水平.如Silva等[33]利用条形码技术结合形态学特征对巴西棒脉摇蚊属(Corynoneura)中的14种86个样本进行了物种鉴定;Amora等[34]利用条形码技术对形态相似的采自日本、韩国和巴西的摇蚊属(Chironomus)类群进行了物种鉴定.此外,DNA条形码技术提高了摇蚊在水体生态系统评估及环境监测方面的应用[35-36].如Ekrem等[37]利用DNA条码技术调查了挪威中部自然保护区内雌性摇
蚊在摇蚊生物多样性方面所起的作用;Brodin等[38]评估了COⅠ基因在提高物种鉴定准确率以及在环境监测中的有效性,如运用DNA条形码技术使波罗的海的摇蚊物种鉴定成功率提高了80%.很多研究集中在相对较小的类群以及形态分类较清晰的类群中.如Meier等[39]用二代测序技术分析了1 015个热带地区分布的摇蚊(490个幼虫和525个成虫)样本;Lin等[40]使用不同的分析工具探究了长跗摇蚊属(Tanytarsus)的2 790个DNA条形码在物种鉴定上的意义.
3.2 COⅡ
Ekrem等[41]基于线粒体基因COⅡ分析了摇蚊科16属属级阶元的系统发育关系,采用最大简约法和贝叶斯法构建系统发育树,贝叶斯分析与形态学的研究结果一致,除Corynocera ambigua和Tanytarsus gracilentus互为姊妹群外,其余各属均为单系群,且得到很高的支持率;系统发育树支持长跗摇蚊亚族(Tanytarsina)和刷毛摇蚊亚族(Zavreliina)这2个亚族分类地位的划分.Stur等[42]用COⅡ结合形态学特征对小突摇蚊属(Micropsectra)的部分物种进行了系统发育分析,最大简约法和贝叶斯法分析均表明,Micropsectra pallidula、M.calcifontis、M.atrofasciata、M.sofiae和 M. schrankelae组成一个单系群,M.contracta和M.insignilobus、M.sofiae和M.schrankelae分别互为姐妹群.M.appendica和M.logani的雄性成虫在形态上与M.atrofasciata很相似,但基于线粒体基因COⅡ的结果显示并非同一物种.王茜等[43]利用部分COⅡ基因序列对直突摇蚊亚科(Orthocladiinae)的24属30种摇蚊进行了分子系统学研究,以Chironomus samoensis为外群,采用邻接法和最小进化法构建的分子系统树显示,拟开氏摇蚊属(Parakiefferiella)和克莱施密摇蚊属(Krenosmittia)、伪直突摇蚊属(Pseudorthocladius)和陆直突摇蚊属(Georthocladius)各自互为姐妹群,这与形态学分析结果一致;毛突摇蚊属(Chaetocladius)和直突摇蚊属(Orthocladius)、拟环足摇蚊属(Paracricotopus)和施密摇蚊属(Smittia)、真开氏摇蚊属(Eukiefferiella)和肛脊摇蚊属(Mesosmittia)各自互为姐妹群,并得到较高支持率.
3.3 16S rDNA和Cytb
16S rDNA基因序列一级结构比较保守,而二级结构又表现出螺旋差异性,较适用于种、属水平的系统学研究[44].细胞色素b基因(Cytb)已被证明在不同门类的不同分类阶元中均含有亲缘信号,其基因片段含有缓慢和快速进化的密码子位点、更保守区以及多个可变区或结构域,由于其进化速度适中,已被广泛用于多样性和系统发育的研究[45-47].
如Cranston等[48]基于线粒体16S rDNA基因的IV和V域,以蠓科(Ceratopogonidae)和蚋科(Simuliidae)种类作为外群,对澳大利亚地区的摇蚊科昆虫作了系统发育分析.结果显示:相对于分布在非洲的Archaeochlus和Afrochlus单系群而言,澳大利亚分布的Archaeochlus为单系,这与形态学分析一致;非洲分布的Archaeochlus是Afrochlus的姐妹群.Papoucheva等[49]测定了丝摇蚊属(Sergentia)7个种的COⅠ和Cytb基因片段,其中5个是贝加尔湖特有种,用邻接法和最大简约法建树. 2种方法的结果均支持丝摇蚊属(Sergentia)的单系性及贝加尔湖种的特有性;NJ树显示,同S.baueri相比,Sergentia electa的亲缘关系更接近贝加尔类群;S.nebulosa和S.assimilis为姊妹群,节点支持率为99%,S.baicalensis和S.flavodentata为姊妹群,但支持率仅为58%,S.affinis的地位没有得到解决;MP树中,S.baueri的亲缘关系更接近贝加尔类群,S.affinis的分类地位也得到了解决.Carew等[50]利用2个线粒体基因COⅠ、Cytb和1个核基因CAD对来自澳大利亚南部的前突摇蚊属(Procladius)类群中用传统方法不易区分的物种进行区分,前突摇蚊属(Procladius)是检测水体环境和污染状况的优良指示生物之一.基于线粒体基因的NJ树表明,研究中采集到的前突摇蚊属样品至少包含5个类群,且支持率大于99%;而基于CAD的NJ树显示有6个类群,Procladius sp.没有与Procladius paludicola聚在一起.
4 总结与展望
系统发育学主要研究物种形成及物种间的进化关系.近年来,随着分子生物技术的快速发展,尤其是PCR技术的广泛应用,越来越多的学者把核基因及线粒体基因应用在昆虫系统发育研究中.由于单一基因片段所提供的系统学信息非常有限,据其构建的进化树可信度低,因此许多研究都把核基因组序列和线粒体基因组序列结合起来开展联合基因分析,便于更客观、真实、科学地反映物种间的系统发育关系[51].
摇蚊科昆虫种类丰富,个体众多,通过系统发育学研究,可以了解该科各分类群的进化关系,解决一些有争议物种的界定以及相关物种的生物地理分布.本文总结了常用核基因和线粒体基因在摇蚊科昆虫系统发育研究中的国内外研究概况,大致观点为:摇蚊科中的9亚科(不含智利摇蚊亚科(Chilenomyiinae)和乌桑巴摇蚊亚科(Usambaromyiinae)),除前寡角摇蚊亚科(Prodiamesinae)外,其余亚科均为单系;寡脉摇
蚊亚科(Podonominae)与长足摇蚊亚科(Tanypodinae)为姊妹群,直突摇蚊亚科(Orthocladiinae)与摇蚊亚科(Chironominae)的亲缘关系较近.在摇蚊亚科(Chironomidae)中,摇蚊族(Chironomini)和长跗摇蚊族(Tanytarsini)都是单系群,而在摇蚊族(Chironomini)中,摇蚊属(Chironomus)、隐摇蚊属(Cryptochironomus)、内摇蚊属(Endochironomus)和长跗摇蚊属(Tanytarsus)均为单系;球附器摇蚊属(Kiefferulus)、阿克西摇蚊属(Axarus)、光摇蚊属(Xestochironomus)、裸突摇蚊属(Goeldichironomus)、李摇蚊属(Lipiniella)及哈摇蚊属复合体(Harnischia complex)有较近的亲缘关系,此外摇蚊属(Chironomus)(包括Camptochironomus和Lobochironomus这2个亚属在内)、弱摇蚊属(Baeotendipes)、恩菲摇蚊属(Einfeldia)等聚在一起,亲缘关系较近,弱摇蚊属(Baeotendipes)与摇蚊属(Chironomus)有较近的亲缘关系;在长跗摇蚊族(Tanytarsin)中,拟长跗摇蚊属(Paratanytarsus)、枝长跗摇蚊属(Cladotanytarsus)是单系群.在直突摇蚊亚科(Orthocladiinae)中,直突摇蚊族(Orthocladiini)和中足摇蚊族(Metriocnemini)(不包括棒脉摇蚊属(Corynoneura)和布摇蚊属(Brillia))的分类地位也得到了支持;拟开氏摇蚊属(Parakiefferiella)和克莱施密摇蚊属(Krenosmittia)、伪直突摇蚊属(Pseudorthocladius)和陆直突摇蚊属(Georthocladius)、心突摇蚊属(Cardiocladius)和真开氏摇蚊属(Eukiefferiella)各自互为姊妹群,Belgica antarctica和Eretmoptera murphyi的分类地位与传统形态学的结果一致.在长足摇蚊亚科(Tanypodinae)中,大粗腹摇蚊族(Macropelopiini)、五脉摇蚊族(Pentaneurini)分别单独形成一支,拉布长足摇蚊属(Labrundinia)、大粗腹摇蚊属(Macropelopia)和扎长足摇蚊属(Zavrelimyia)均为单系.在寡角摇蚊亚科(Diamesinae)中,Heptagiini与寡角摇蚊族(Diamesini)互为姐妹群.对现有研究进行总结可以看出,摇蚊亚科(Chironomidae)、直突摇蚊亚科(Orthocladiinae)、长足摇蚊亚科(Tanypodinae)等的研究较多,而智利摇蚊亚科(Chilenomyiinae)、乌桑巴摇蚊亚科(Usambaromyiinae)、阿福罗摇蚊亚科(Aphroteniinae)等的研究则相对薄弱,将会是后续研究的重点.
在摇蚊科的系统发育关系研究中,18S rDNA和28S rDNA适合从界到科的所有高级阶元,对于部分类群可以进行属间分析;CAD基因能较好地解决从亚科到属之间各分类阶元的系统发育关系,ITS和EF-1ɑ可以解决种或属间的系统发育关系;COⅠ、COⅡ适合研究属、种及种下阶元的系统发育关系,但不能很好地解决较高的阶元,如族间、亚科以及科的系统发育关系;16S rDNA适用于种、属阶元水平的系统学研究,但不适用于种内;Cytb适合研究种内到种间甚至科间的系统发育关系.使用单个基因研究摇蚊科系统发育的研究较少,更多的是多个基因的联合使用(核基因的联合、线粒体基因的联合或者核基因结合线粒体基因).对于同一类群,采用不同基因序列可能会得到不同的系统发育关系结果,出现基因树冲突等问题,而系统发育基因组学的建立对于解决基因树的冲突能够提供有力帮助.
[1]ARMITAGE P,CRANSTON P S,PINDER L C V,et al.The Chironomidae:the Biology and Ecology of Non-biting Midges[M].London:Chapman&Hall,1995.
[2]李志勤,闫洪波,李成德.昆虫分类的主要技术手段[J].河北林果研究,2006,21(4):398-403. LI Z Q,YAN H B,LI C D.The main techniques of insect taxonomy[J]. Hebei Journal of Forestry and Orchard Research,2006,21(4):398-403(in Chinese).
[3]GURYEV V,MAKAREVITCH I,BLINOV A,et al.Phylogeny of the genus Chironomus(Diptera)inferred from DNA sequences of mitochondrial cytochrome b and cytochrome oxidaseⅠ[J].Mol Phylogenet Evol,2001,19(1):9-21.
[4]时号,张婧,蒋国芳.线粒体基因和核基因在蝶类分子系统学中的研究进展[J].广西科学,2006,13(2):156-160. SHI H,ZHANG J,JIANG G F.A progress in the molecular systematics of butterflies based on mitochondrial gene and nuclear gene[J]. Guangxi Sciences,2006,13(2):156-160(in Chinese).
[5] GDETGHEBUER M.Recherches sur les chironomides de Belgique[J]. Mém Acad Rog Belg,1914,3(7):1-48.
[6] BRUNDIN L Z.Transantarctic relationships and their significance,evidenced by chirononind midges with a monograph of the rubfamilies of Podonominae and Aphrotenilnae and the austral Heptagyiae[J].Kunylica Svenska Vetenskadenicens Handlinggar,1966,11:1-472.
[7] DONATO M,SIRI A.A new species of Metriocnemus van der Wulp(Diptera:Chironomidae)with a tentative phylogeny of the genus[J]. Neotropical Entomology,2010,39(1):50-60.
[8] FU Y,SAETHER O,WANG X H.Thienemanniella kieffer from east Asia,with a systematic review of the genus(Diptera:Chironomidae:Orthocladiinae)[J].Zootaxa,2010,2431:1-42.
[9]CRANSTON P S,HARDY N B,MORSE G E,et al.When molecules and morphology concur:the‘Gondwanan’midges(Diptera:Chironomidae)[J].Systematic Entomology,2010,35(4):636-648.
[10]CRANSTON P S,HARDY N B,MORSE G E.A dated molecular phylogenyfortheChironomidae(Diptera)[J].Systematic Entomology,2012,37(1):172-188.
[11]MARTIN J,BLINOV A,ALIEVA E,et al.A Molecular Phylogenetic Investigation of the Genera Closely Related to Chironomus meigen(Diptera:Chironomidae)[M].Columbus:The Caddis Press,2007.
[12]DIETRICH C H,RAKITOV R A,HOLMES J L,et al.Phylogeny of the major lineages of Membracoidea(Insecta:Hemiptera:Cicadomor-
pha)based on 28S rDNA sequences[J].Molecular Phylogenetics and Evolution,2001,18(2):293-305.
[13]ALLEGRUCCI G,CARCHINI G,TODISCO V,et al.A molecular phylogeny of antarctic chironomidae and its implications for biogeographical history[J].Polar Biology,2006,29(4):320-326.
[14]王茜,王新华.直突摇蚊亚科部分属28S rDNA D1序列的分子系统发育关系(双翅目,摇蚊科)[J].动物分类学报,2011,36(1):93-98. WANG Q,WANG X H.Molecular phylogenetic analysis on some genera of the subfamily Orthocladinae(Diptera,Chironomidae)based on D1 variable region of 28S ribosomal DNA[J].Acta Zootaxonomica Sinica,2011,36(1):93-98(in Chinese).
[15]HUANG D W,CRANSTON P S,CHENG L.A complete species phylogeny of the marine midge Pontomyia(Diptera:Chironomidae)reveals a cosmopolitan species and a new synonym[J].Invertebrate Systematics,2014,28(3):277-286.
[16]MEI H,LUO W,LIU G X,et al.Phylogeny of Oedogoniales(Chlorophyceae,Chlorophyta)inferred from 18S rDNA sequences with emphasis on the relationships in the genus Oedogonium based on ITS-2 sequences[J].Plant Systematics and Evolution,2007,265(3):179-191.
[17]宗祖胜,张金松,刘丽君,等.自来水厂中摇蚊rDNA系统学分析[J].中国给水排水,2005,21(3):1-4. ZONG Z S,ZHANG J S,LIU L J,et al.Analysis on molecular systematics of chironomus rDNA in water treatment plants[J].China Water& Waste Water,2005,21(3):1-4(in Chinese).
[18]SHARMA M.Molecular Identification of Chironomid Species Based on ITS1 and ITS2 Regions of rDNA[D].Dayton:Wright State University,2007.
[19]ASARI H,KASUYAS T K.Identilication of closely related Hydrobaenus species(Diptera:Chironomidae)using the second internal transcribed spacer(ITS2)region of ribosomal DNA[J].Aquatic Insects,2004,26(3):207-213.
[20]王诗迪,赵喆,张春田.常用核基因序列在双翅目昆虫系统学中的研究进展[J].环境昆虫学报,2012,34(1):220-228. WANG S D,ZHAO Z,ZHANG C T.Advances in systematics of Diptera based on nuclear genes[J].Journal of Environmental Entomology,2012,34(1):220-228(in Chinese).
[21]MOULTON J K,WIEGMANN B M.Evolution and phylogenetic utility of CAD(rudimentary)among Mesozoic-aged Eremoneuran Diptera(Insecta)[J].Molecular Phylogenetics and Evolution,2004,31(1):363-378.
[22]SILVA F L,EKREM T R,FONSECA-GESSNER A A.Out of south America:phylogeny of non-biting midges in the genus Labrundinia suggests multiple dispersal events to central and north America[J].Zoologica Scripta,2015,44(1):59-71.
[23]ANDERSEN T,BARANOV V,HAGENLUND L K,et al.Blind flight?A new Troglobiotic orthoclad(Diptera,Chironomidae)from the Lukina Jama-Trojama cave in Croatia[J].PLoSOne,2016,11(4):1-15.
[24]刘殿锋,蒋国芳.核基因序列在昆虫分子系统学上的应用[J].动物分类学报,2005,30(3):484-492. LIU D F,JIANG G F.Application in molecular systematics of insect based on nuclear genes[J].Acta Zootaxonomica Sinica,2005,30(3):484-492(in Chinese).
[25]成新跃,周红章,张广学.分子生物学技术在昆虫系统学研究中的应用[J].动物分类学报,2000,25(2):121-130. CHENG X Y,ZHOU H Z,ZHANG G X.Application in systematics of insect based on molecular biology techniques[J].Acta Zootaxonomica Sinica,2000,25(2):121-130(in Chinese).
[26]MORSE G E,NORMARK B B.A molecular phylogenetic study of armoured scale insects(Hemiptera:Diaspididae)[J].Systematic Entomology,2006,31(2):338-349.
[27]HUANG D,CHENG L.The flightless marine midge Pontomyia(Diptera:Chironomidae):ecology,distribution,and molecular phylogeny[J].Zoological Journal of the Linnean Society,2011,162(2):443-456
[28]EKREM T,WILLASSEN E,STUR E.Phylogenetic utility of five genes for dipteran phylogeny:a test case in the Chironomidae leads to generic synonymies[J].Molecular Phylogenetics and Evolution,2011,57(2):561-571
[29]KROSCH M,BAKER A M,MATHER P B,et al.Systematics and biogeography of the Gondwanan Orthocladiinae(Diptera:Chironomidae)[J].Molecular Phylogenetics and Evolution,2011,59(2):458-468.
[30]姜丽,闫娇,王婷婷,等.基于COⅠ基因的摇蚊亚科部分属的系统发育分析(双翅目,摇蚊科)[J].天津师范大学学报:自然科学版,2015,35(3):7-11. JIANG L,YAN J,WANG T T,et al.A molecular phylogeny for some genera of the subfamily Chironominae(Diptera:Chironomidae)based on cytochrome oxidase I gene[J].Journal of Tianjin Normal University:NaturalScienceEdition,2015,35(3):7-11(In Chinese).
[31]EKREM T,WILLASSEN E,STUR E.A comprehensive DNA sequence library is essential for identification with DNA barcodes[J]. Molecular Phylogenetics and Evolution,2007,43(2):530-542.
[32]SINCLAIR C S,GRESENS S E.Discrimination of Cricotopus species(Diptera:Chironomidae)by DNA barcoding[J].Bulletin of Entomological Research,2008,98(6):555-563.
[33]SILVA F L,WIEDENBRUG S.Integrating DNA barcodes and morphology for species delimitation in the Corynoneura group(Diptera:Chironomidae:Orthocladiinae)[J].Bulletin of Entomological Research,2014,104(1):65-78.
[34]AMORA G,HAMADA N,FUSARI L M,et al.An asiatic chironomid in Brazil:morphology,DNA barcode and bionomics[J].Zookeys,2015,514:129-144.
[35]CAREW M E,PETTIGROVE V J,METZELING L,et al.Environmental monitoring using next generation sequencing:rapid identification of macroinvertebrate bioindicator species[J].Frontiers in Zoology,2013,10(45):1-15.
[36]CRANSTON P S,ANGY C,HEYZER A,et al.The nuisance midges(Diptera:Chironomidae)of Singapore′s Pandan and Bedok Reservoirs [J].Raffles Bulletin of Zoology,2013,61(2):779-793.
[37]EKREM T,STUR E,HEBERT P.Females do count:documenting Chironomidae(Diptera)species diversity using DNA barcoding[J].Organisms Diversity&Evolution,2010,10(5):397-408.
[38]BRODIN Y,EJDUNG G,STRANDBERG J,et al.Improving environmental and biodiversity monitoring in the Baltic Sea using DNA barcoding of Chironomidae(Diptera)[J].Molecular Ecology Resources,
2013,13(6):996-1004.
[39]MEIER R,WONG W,SRIVATHSAN A,et al.$1 DNA barcodes for reconstructing complex phenomes and finding rare species in specimen-rich samples[J].Cladistics,2016,32(1):100-110.
[40]LIN X L,STUR E,EKREM T.Exploring genetic divergence in a species-rich insect genus using 2 790 DNA barcodes[J].Plos one,2015,10(9):1-24.
[41]EKREM T,WILLASSEN E.Exploring Tanytarsini relationships(Diptera:Chironomidae)using mitochondrial COⅡgene sequences[J]. International Journal of Systematic and Evolutionary Microbiology,2004,35(3):263-276.
[42]STUR E,EKREM T.A revision of West Palaearctic species of the Micropsectra atrofasciata species group(Diptera:Chironomidae)[J].Zoological Journal of the Linnean Society,2006,146(42):165-225.
[43]王茜,王新华.直突摇蚊亚科部分属基于线粒体COⅡ基因部分序列的系统发育关系(双翅目,摇蚊科)[J].四川动物,2011,30(5):711-716. WANG Q,WANG X H.Phylogenetic relationship of some genera of the subfamily Orthocladiinae(Diptera:Chironomidae)based on partial sequences of mitochondrial COⅡgene[J].Sichuan Journal of Zoology,2011,30(5):711-716(in Chinese).
[44]HARRISON J S.Evolution,biogeography,and the utility of mitochondrial 16s and COⅠ genes in phylogenetic analysis of the crab genus Aus-tinixa(Decapoda:Pinnotheridae)[J].Molecular Phylogenetics and Evolution,2004,30(3):743-754.
[45]CANTATORE M,ROBERTI G,PESOLE A,et al.Evolutionary analysis of cytochrome b sequences in some Perciformes:evidence for a slower rate of evolution than in mammals[J].Molecular and Evolution,1994,39:589-597.
[46]IRWIN D M,KOCHER T D,WILSON A C.Evolution of the cytochrome b gene of mammals[J].Molecular and Evolution 1991,32(2):128-144.
[47]KUMAZAWA Y,NISHIDA M.Molecular phylogeny of Osteoglossoids:a new model for Gondwanian origin and plate tectonic transportation of the Asian Arowana[J].Molecular Biology and Evolution,2000,17(12):1869-1878.
[48]CRANSTON P S,EDWARD D,COOK L G.New status,species,distribution records and phylogeny for Australian mandibulate Chironomidae(Diptera)[J].Australian Journal of Entomology,2002,41(4):357-366.
[49]PAPOUCHEVA E,PROVIZ V,LAMBKIN C,et al.Phylogeny of the endemic Baikalian Sergentia(Chironomidae,Diptera)[J].Molecular Phylogenetics and Evolution,2003,29(1):120-125.
[50]CAREW M E,MARSHALL S E,HOFFMANN A A.A combination of molecular and morphological approaches resolves species in the taxonomically difficult genus Procladius Skuse(Diptera:Chironomidae)despite high intra-specific morphological variation[J].Bull Entomol Res,2011,101(5):505-519.
[51]邹新慧,颂葛.基因树冲突与系统发育基因组学研究[J].植物分类学报,2008,46(6):795-807. ZHOU X H,SONG G.Conflicting gene trees and phylogenomics[J]. Journal of Systematics and Evolution,2008,46(6):795-807(in Chinese).
(责任编校 纪翠荣)
Application progress of common genes in the phylogenetic studies of Chironomidae
YAN Chuncai1,GUO Qin1,ZHAO Guangjun1,GE Xinyu1,WANG Xinhua2,LIU Ting1
(1a.College of Life Sciences,1b.Tianjin Key Laboratory of Animal and Plant Resistance,Tianjin Normal University,Tianjin 300387,China;2.College of Life Sciences,Nankai University,Tianjin 300071,China)
Chironomidae belongs to Nematocera in Diptera and includes 11 subfamilies.The paper summarized the applications of common nuclear genes and mitochondria genes which were adopted in the phylogenetic studies of Chironomidae:In Chironomidae(excluding Chilenomyiinae and Usambaromyiinae),all sampled subfamilies were confirmed to be the monophyly,with the exception of Prodiamesinae;the monogeneric Podonominae was supported as sister to Tanypodinae;the subfamily Orthocladiinae and Chironominae were sister groups.In the subfamily of Chironominae,Chironomini and Tanytarsini were monophyletic groups.The results also showed that:18S rDNA and 28S rDNA genes can accurately infer the phylogenetic relationships of the levels from kindom to family,and also can be used for some groups among genus.CAD gene can infer the phylogenetic relationships of the levels from subfamily to genus.ITS and EF-1ɑ genes are more suitable for the levels of species or genus.COⅠand COⅡare applicable for the levels of genus,species and subspecies,but can not infer the phylogenetic relationships of the higher levels of tribe,subfamily and family.16S rDNA can infer the phylogenetic relationships of the levels of genus and species,but can not be used in intraspecific.Cytb is applicable for the levels of intraor inter-species,even for the level of family.
Chironomidae;genes;molecular marker;molecular systematics;phylogeny
Q951
A
1671-1114(2016)06-0054-08
2016-03-25
国家自然科学基金资助项目(31101653,31272284,31672324);天津市自然科学基金资助项目(14JCQNJC14600);天津市高等学校科技发展基金资助项目(20090608);天津师范大学引进人才基金资助项目(5RL104);天津市优秀青年教师资助计划资助项目(2013).
闫春财(1979—),男,副教授,主要从事昆虫分子系统学方面的研究.
