基于改进生成式对抗网络的编码DNA 分子识别
2021-04-10随学杰王慧锋颜秉勇
随学杰, 王慧锋, 颜秉勇
(华东理工大学 1. 信息科学与工程学院;2. 化学与分子工程学院,上海 200237)
作为最具前景的第3 代DNA 测序技术,纳米通道单分子检测技术引起了广大研究者的兴趣[1-4]。该技术的基本原理是在嵌有纳米孔的磷脂双分子层两侧施加电压,电解液中的离子在电场力驱动下穿过纳米孔,产生微弱的开孔电流,当有DNA、RNA、氨基酸、金属离子等分子穿过纳米孔时,由于分子在纳米孔道的占位改变了离子流量,将产生pA 级的阻断电流信号[5-6]。通过对阻断信号进行分析,可得到分子在溶液中的浓度、分子与孔的相互作用、分子类型、碱基序列等信息。传统的数据分析方法是依据阻断信号的时间和电流散点图分布实现分子类别的识别,然而,由于低信噪比、DNA 链折叠和缠绕、分子类别间信号重叠等原因,导致传统方法对分子的识别率较低[7]。为提高纳米孔道信号的识别率,可通过改进实验条件和仪器设备精度等方法提高纳米孔测量的电流和时间分辨率,但这些方法通常会增加实验的复杂度和难度,或达到难以突破的物理极限[8]。为提高纳米孔道单分子检测中对单个分子阻断信号的识别精度,发展高效且智能的纳米孔道数据分析识别方法具有重大意义[9-11]。
在机器学习和深度学习领域,样本不平衡是指在分类学习算法中,不同类别样本的数据量相差悬殊,导致以总体准确率为目标的分类任务中过多地关注多数类,从而使少数类样本的分类性能下降,模型准确性较差[12-14]。由于纳米孔道对不同类型单分子信号的捕获率存在着巨大差异,因此基于深度学习模型对纳米孔道单分子信号进行分类训练时存在数据集不平衡的问题,进而影响模型对单个分子识别的准确率。Sui 等[15]针对单级阻断事件提出了基于HMM-AdaBoost 的分类模型,对阻断信号有重叠的AA3和GA3分子进行了识别,Aerolysin 纳米孔道对AA3分子的捕获率大于对GA3分子的捕获率,导致训练集中两种分子的数量比约为3∶1。Karolis 等[16]提出了基于卷积神经网络的QuipuNet 模型,对编码为“000”~“111”的8 种DNA 分子和有无蛋白质绑定进行分类,训练集中编码为“011”的分子与编码为“100”的分子数量比为17∶1 等。从分类结果可以观察到,少数类样本的分类准确率均低于其他类别。多数基于机器学习和深度学习的分类任务研究表明,解决样本不均衡问题,增加训练样本数据量,可显著提高模型的分类性能[17]。
生成式对抗网络是一种基于对抗策略的生成式模型,可生成与训练样本分布相同的仿真样本,在深度学习、图像领域和序列数据等领域都受到广泛关注[18-19]。本文通过改进深度卷积生成式对抗网络(Deep Convolutional Generative Adversarial Networks,DCGAN)模型,研究类别不平衡的纳米孔道单分子数据集,并实现数据集的扩充与分类。首先对数据进行预处理,并将所有分子的阻断事件处理为相同长度;然后使用改进DCGAN 模型对少数类样本数据集进行扩充,生成相应分子的仿真阻断信号;最后应用QuipuNet 卷积神经网络对扩充前后的数据集进行分类。本文方法可显著改善纳米孔道对分子捕获率不同所带来的数据集不平衡问题,不仅可提高单个分子阻断事件的识别准确率,同时对深度学习等人工智能算法应用于纳米孔道研究提供了新的数据处理方法。总流程图如图1 所示。
1 数据集描述
1.1 数据来源
数据来源于文献[18]的多通道蛋白质检测实验。由32 个纳米通道产生的58178个阻断事件被存储至HDF5 文件中,包含“000”~“111”8种编码DNA分子的阻断信息,每个分子被记录了纳米通道编号、编码信息、有无蛋白质绑定、表征阻断信号的电流序列等信息。
1.2 数据预处理
采用阈值滤波[16]去除由于DNA 片段不完整、分子与孔道的非特异性相互作用而产生的异常阻断信号,对数据集进行归一化处理,从而消除纳米孔道作用时间差异所引起的阻断信号变化。由于卷积神经网络输入信号维度固定,为使阻断信号长度相同,采用如下处理方法:长度大于700 的阻断信号,保留前700 个数据点;取样本集中所有阻断信号开孔电流的前50 个数据点,计算其均值为0.009 5,对长度小于700 的信号,用均值为0、标准差为0.0095的高斯白噪声补齐至长度为700。
2 结果与讨论
2.1 基于改进DCGAN 的数据集平衡
Goodfellow 等[20]提出的生成式对抗网络是一种基于博弈理论扩充数据集的方法,最初用于图像的生成。生成式对抗网络由生成器(Generator,G)和判别器(Discriminator,D)两部分组成,其中生成器接收随机噪声用于学习和捕捉真实数据集的分布,并生成与之相似的新数据集;判别器的功能是一个二分类器,判断数据是来自真实数据集还是由生成器生成的伪数据集。训练过程中,生成器的目标是尽量生成与真实数据相似的伪数据,使判别器难以区分;而判别器的目标是尽量辨别出真实数据和伪数据,最终达到纳什平衡,即判别器无法判断数据是否来自生成器。该模型绕过了求解似然函数的困难,可直接生成样本,从而拟合训练数据的分布。
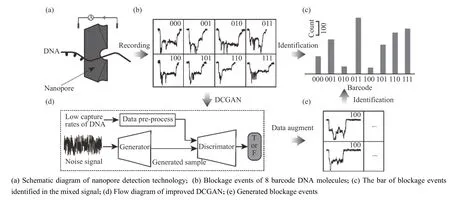
图1总流程图Fig.1General flow diagram
DCGAN 对原始生成式对抗网络的改进[21]:(1)采用带步长的卷积代替D 中的池化层;(2)在D 和G中加入批量标准化(Batch Normalization, BN)层,加快模型的收敛速度;(3)去除全连接层;(4)在G 中,除最后一层激活函数使用tanh,其余层的激活函数为ReLU;(5)在D 中,均使用LeakReLU 激活函数,该激活函数可保证导数总是不为零,能减少静默神经元的出现。
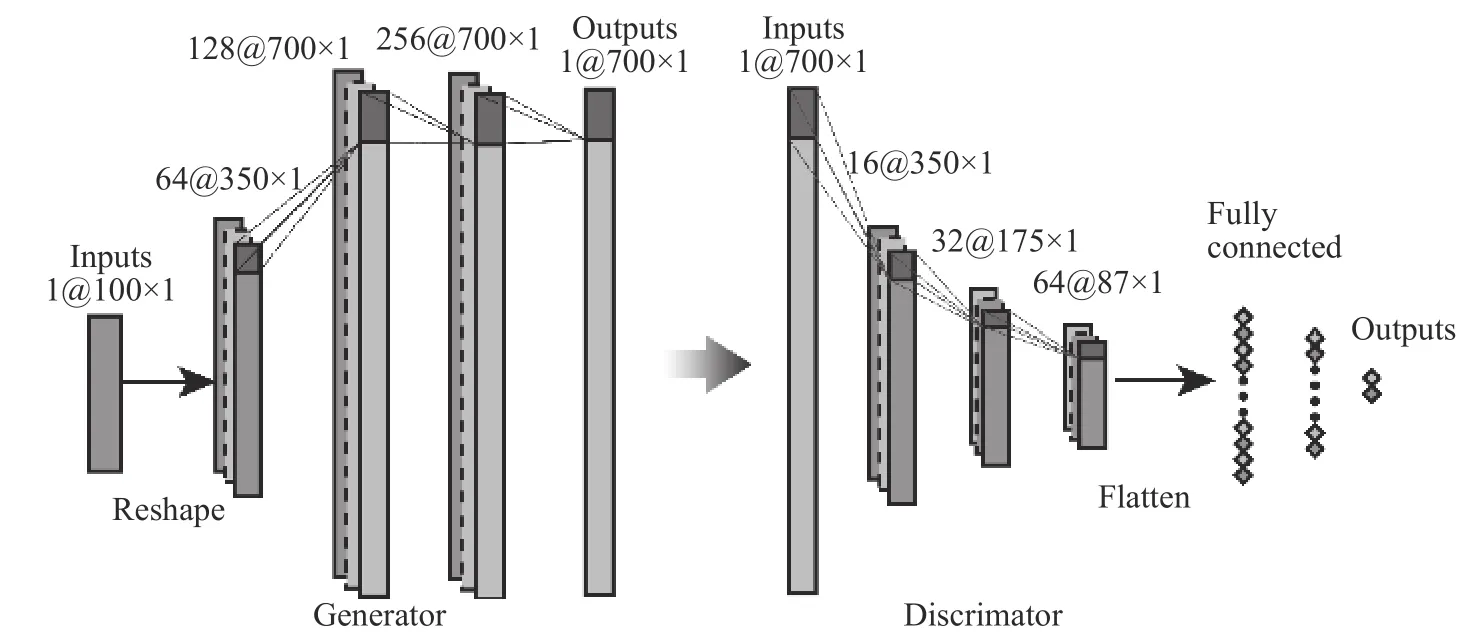
图2生成器和判别器的网络结构图Fig.2Network structure of generator and discriminator
本文以DCGAN 为基本框架,设计了适合纳米孔道单分子数据分析的网络模型结构(如图2 所示),包含生成器和判别器两部分。G 的输入为服从正态分布、长度为100 的特征向量,由4 层卷积神经网络构成。其中,卷积神经网络的基本结构为Conv1d+BN+ReLu,最后一层采用tanh 激活函数,卷积核大小为3、3、3。D 的输入为真实样本 x 和G生成的伪样本G( z ),主要由3 层卷积神经网络构成,特征图大小按16、32、64 逐级递增,相应的卷积核为7、5、3,使用LeakRelu 激活函数,最后经过一个全连接层,由sigmod 函数判断当前样本为真实样本(标签为1),或为伪样本(标签为0)。tanh、ReLU 和LeakReLU 激活函数表达式如式(1)~式(3)所示。
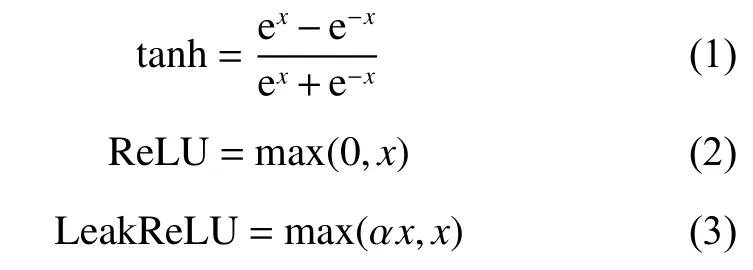
训练过程中,batch_size 设为64,初始学习率learning_rate=0.000 1, 采用Adam 优化算法训练600次。因编码DNA 分子的标签已知,本文在目标函数中引入标签信息,构成条件约束,解决深度卷积对抗网络训练太过自由的问题。目标函数用V(D,G)表示,如下式所示。
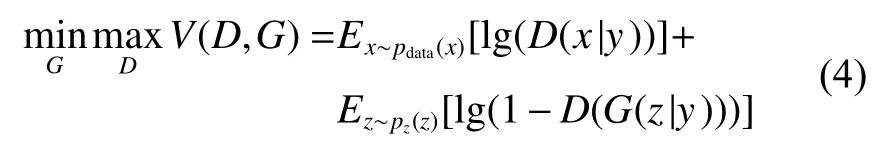
其中:x 为真实样本,z 为随机噪声,E 表示数据分布,D(x|y)为判别器D 判断真实样本是否真实的概率,D(G(z))为判别器D 判断生成器G 生成的伪样本是否真实的概率。为防止判别器过于强大而造成训练不平衡,使用标签平滑,即当判断为正样本时,用0.95 代替1。以编码为“100”的分子为例,图3 示出了模型在不同的训练次数生成的阻断事件。
2.2 编码DNA 分子的识别
原始数据集中,编码为“011”的分子在实验中的捕获率最高,对应的阻断事件最多;而编码为“100”和“010”的分子捕获率较低,仅占“011”数据集的8%和23%,造成数据集的类别不平衡。将各阻断事件预处理后,使用上述改进DCGAN,对除“011”编码之外的7种编码DNA 分子的阻断事件进行扩充。分别将原始数据集与平衡后的编码DNA 载体数据集作为QuipuNet 的输入,其中,两次实验的测试集大小相同。为验证该网络生成数据集的有效性,采用混淆矩阵以及平均准确率作为评价标准。混淆矩阵是以预测标签为横坐标、真实标签为纵坐标的交叉表,用以直观展示各类别分类情况。平均准确率的计算公式如下:
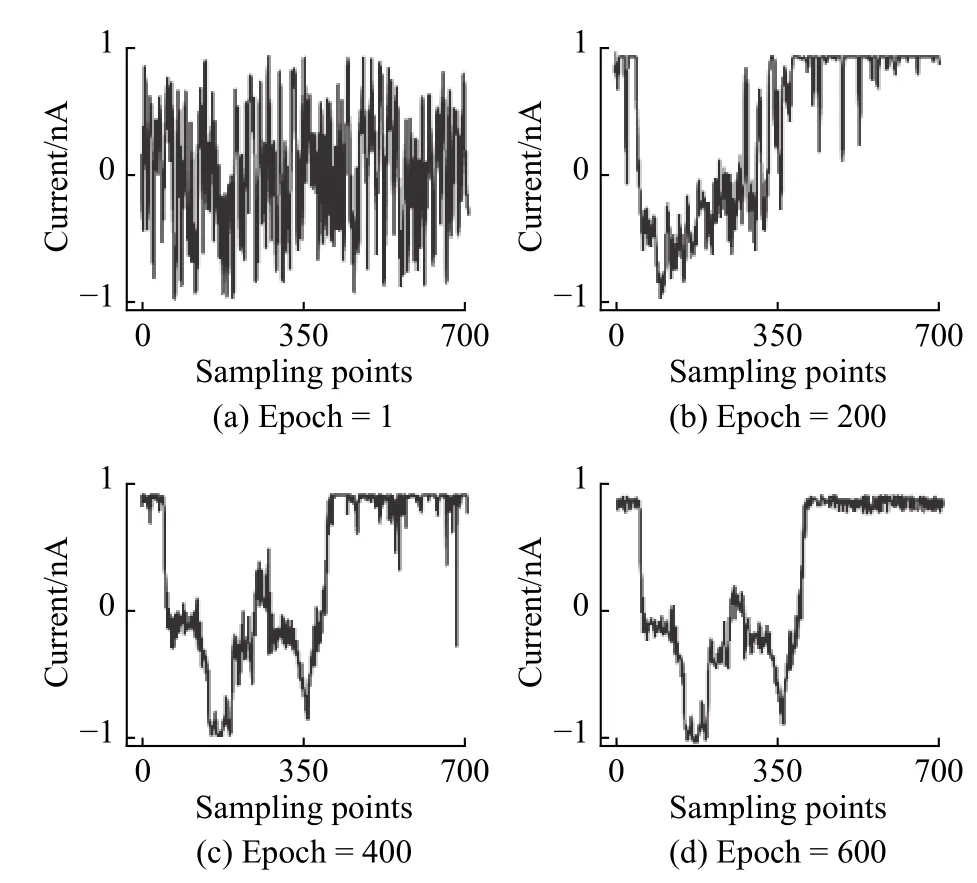
图3编码为“100”的DNA 分子的阻断事件生成过程Fig.3Generation process for blockage event of barcode ‘100’
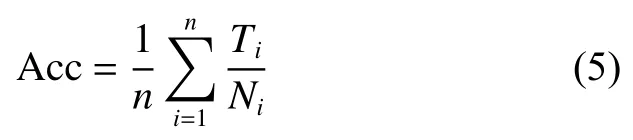
其中:n 为类别数;Ni为第i 类别中样本的数量;Ti为第i 类别中预测正确的样本数。QuipuNet 包含8层卷积网络,基本结构为Conv1d+BN+ReLU,卷积核大小为{7,7,5,5,3,3},特征图大小分别为{64,64,128,128,256,256},最后一层为softmax 分类器,进行8 分类。训练时,batch_size 设为32,使用Adam 优化算法,初始学习率为0.001,损失函数选择交叉熵损失函数,表达式如下:
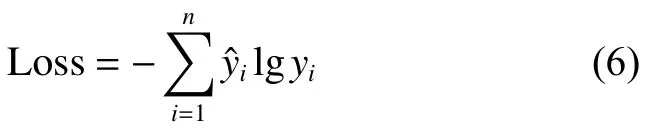
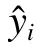
将测试集送入训练好的QuipuNet 模型,得到的混淆矩阵如图5 所示。混淆矩阵的横坐标为预测标签,纵坐标为真实标签,对角线即为各类别的分类准确率,颜色深浅代表数值的大小,混淆矩阵右边为对应的各类别训练数据的规模。可见,使用改进DCGAN 对数据集进行平衡后,少数类别的分类准确率具有明显的提升。平衡前后编码为“100”极少数类样本的分类准确率由0.82 提升至0.96,编码为“010”的分子的准确率由0.89 提升至0.93,由于二者在原始数据集中本身所占比例不同,识别准确率存在一定差异。此外,“110”和“001”等分子的识别准确率也有了一定幅度的提升。可见,在对数据集进行平衡后,少数类分子信号的识别准确率获得了显著提升,均达到0.93 及以上,总体平均准确率由0.92 提升至0.96,说明改进DCGAN 可用于生成阻断事件以平衡数据集,从而提高混合分子中捕获率低的分子的识别准确率。
2.3 不同数据集平衡方法分类结果
目前常用的数据集扩充方法有重采样法和添加高斯噪声等[22-23],为进一步验证改进DCGAN 模型在纳米孔道数据分析中的有效性,将两种传统扩充方法与改进DCGAN 模型进行对比。重采样法对少数样本进行有放回抽样m 次,数据集的数量与图5(b)中各类别训练数据集大小相等;噪声法在抽样得到的阻断电流信号上添加白噪声。分别将两种数据集送入分类模型,经不同扩充数据集训练后的QuipuNet模型对测试集的平均识别准确率如表1 所示。可知,用本文方法得到的平衡数据集训练QuipuNet 后,测试集的分类准确率较高。重采样法使数据集中含有较多重复样本,尤其是数据倾斜较为严重的编码为“100”分子的阻断事件,使得模型产生过拟合,降低准确率。而噪声法在一定程度上增加了数据的多样性,但所加随机噪声的大小不易控制。对比结果表明,两种传统的数据集扩充方法不适用于纳米孔道数据集的扩充,将大数据分析方法应用到纳米通道数据分析领域时,本文方法能在一定程度上解决纳米通道对分子捕获率不同而造成的数据集不平衡问题,提高分类准确率。
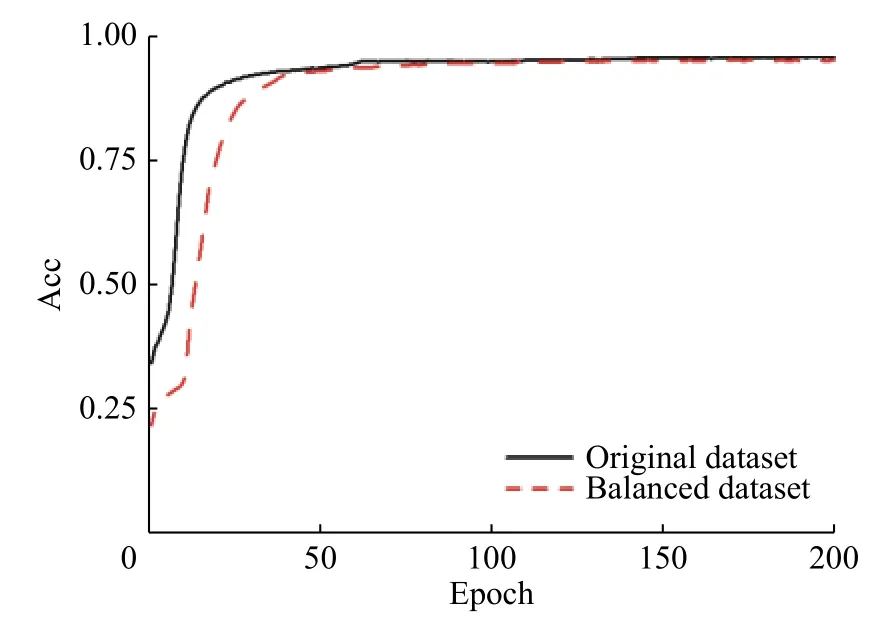
图4训练准确率变化图Fig.4Diagram of training accuracy
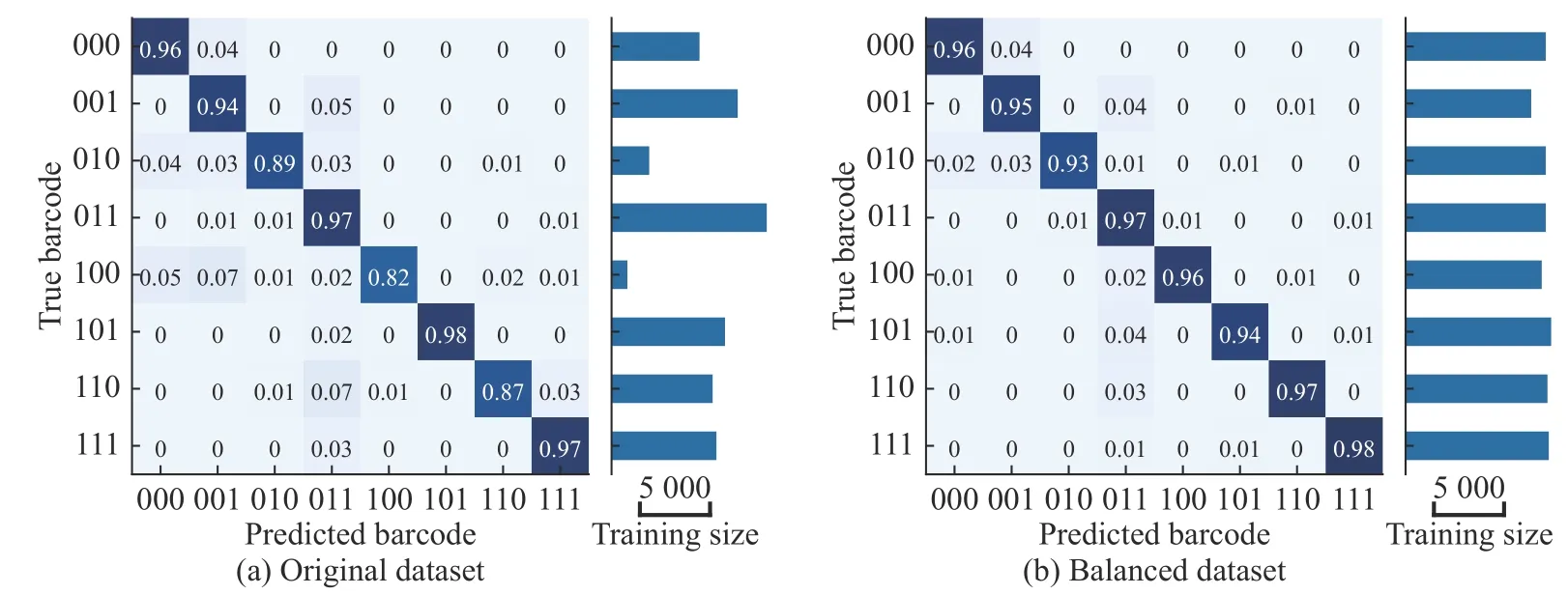
图5模型在不同训练集下的测试数据的混淆矩阵Fig.5Confusion matrix of test data for model trained with different datasets

表1不同数据扩充方法的分类结果Table1Classification results using different data augmentation methods
3 结 论
纳米孔道单分子阻断信号存在重叠或阻断台阶不明显的问题,使用传统散点图法较难进行区分,同时,由于纳米孔对分子的捕获率不同,致使不同分子阻断事件数量不平衡,进而影响模型的准确率。基于编码DNA 分子的阻断事件,本文将改进DCGAN用于少数类单分子信号样本的扩充,避免类间不平衡问题。通过与原始数据集以及由重采样法和噪声法扩充的数据集相比,本文方法显著提高了模型训练后对单分子的识别准确率。此外,本文的研究也展示了生成式对抗网络扩充训练数据的方法在纳米孔道单分子数据分析算法研究中的重要意义和作用,并可进一步用于基于纳米孔道的复杂DNA 测序数据或基于多测量方法的纳米孔道数据分析研究中。
