不同乳酸菌组合对苜蓿青贮细菌群落结构的影响
2021-04-08王丽学陈龙宾余新越刘景喜霍文娟
王丽学, 韩 静, 陈龙宾, 余新越, 刘景喜, 马 毅*, 霍文娟
(1. 天津市农业科学院信息研究所, 天津 300192; 2. 天津市农业动物繁育与健康养殖重点实验室, 天津300384; 3. 天津市农业科学院畜牧兽医研究所, 天津 300381; 4. 天津师范大学地理与环境科学学院, 天津 300387;5. 天津市农业科学院农产品保鲜与加工技术研究所, 天津 300381)
紫花苜蓿(Medicagosativa)是豆科苜蓿属多年生草本植物,为世界上广泛种植的优质蛋白类牧草,被誉为牧草之王[1]。为避免雨季苜蓿收贮过程中的损失,通常制作青贮以减少养分流失,便于长期保存[2]。优质苜蓿青贮具有柔软多汁、适口性好、消化率高等优点,但因苜蓿水溶性碳水化合物低且缓冲能值高,在青贮过程中较难形成低pH值状态,极易发生梭菌等有害菌发酵,进而降低其青贮品质[3]。因此,在苜蓿青贮过程中通常需要添加青贮添加剂[2,4]。青贮添加剂按其作用效果可分为发酵促进剂、发酵抑制剂和营养性添加剂[5]。乳酸菌属于发酵促进剂[3],可促进乳酸发酵、抑制杂菌生长、减少养分损失同时提升青贮发酵品质[6]。目前已发现的乳酸菌主要包括细菌界5门43属[7],其中青贮添加剂常用的有乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、乳球菌属(Lactococcus)、肠球菌属(Enterococcus)等[8-12],因乳酸菌特征不同,在实际应用中常搭配使用。
青贮是一个复杂的微生物菌群如乳酸菌、腐败菌、酵母菌、霉菌、芽孢杆菌等的活动过程[13],微生物群落组成可直接影响青贮品质[14],甚至进一步影响反刍动物瘤胃微生物群系[15-16]。研究表明,青贮微生物群落与青贮物料自身特征[17-20]、外源添加物质[21-22]或添加剂[23-24]、青贮方式[25]及青贮物料所生长的土壤环境[26]等多种因素有关。不同微生物添加剂对苜蓿青贮细菌群落结构影响不同,有研究表明,接种植物乳杆菌(Lactobacillusplantarum)的苜蓿青贮由植物乳杆菌主导整个发酵过程[24,27];接种产纤维素酶的短小芽孢杆菌(Bacilluspumilus)后,苜蓿青贮肠球菌属(Enterococcus)丰度增加,而当短小芽孢杆菌与植物乳杆菌混合应用时,苜蓿青贮肠球菌属丰度降低而乳杆菌属(Lactobacillus)和肠杆菌属(Enterobacter)丰度增加[27];大肠杆菌(EscherichiacoliO157:H7)与植物乳杆菌或布氏乳杆菌(Lactobacillusbuchneri)的组合处理降低了苜蓿青贮Shannon多样性指数,且大肠杆菌与布氏乳杆菌的组合还降低了厚壁菌门(Firmicutes)丰度但增加了变形菌门(Proteobacteria)丰度[28]。由此可见,不同微生物添加剂对苜蓿青贮细菌群落结构的影响存在互作效应,揭示这种互作效应对于苜蓿青贮添加剂开发具有重要意义。
鉴于此,本研究在对5种乳酸菌对苜蓿青贮营养和发酵品质影响[29]研究的基础上,运用高通量测序技术分析了6种不同乳酸菌组合苜蓿青贮细菌群落结构差异,揭示其对苜蓿青贮营养和发酵品质影响的作用机理,为后续相关乳酸菌在苜蓿青贮中的进一步应用提供参考。
1 材料与方法
1.1 试验材料
试验用苜蓿为丹农种子公司提供的‘北极熊’品种,种植于天津市农业资源与环境研究所的滨海盐碱地绿化土壤改良科研试验基地。
试验用乳酸菌:2种植物乳杆菌(Lactobacillusplantarum),菌种保藏号分别为ACCC11016(记为L1)和CICC20765(记为L2);乳酸片球菌(Pediococcusacidilactici,记为P1),菌种保藏号为CGMCC 1.4,戊糖片球菌(Pediococcuspentosaceus,记为P2),菌种保藏号为CICC 22737;凝结芽孢杆菌(Bacilluscoagulans,记为B),菌种保藏号为ACCC10229。其中植物乳杆菌CICC20765和戊糖片球菌购于中国工业微生物菌种保藏管理中心,其他菌种均由天津市农业科学院畜牧兽医研究所保藏。
1.2 试验设计
试验于2017年5月开展,设置6个菌种组合处理,分别为L1P1,L1P2,L2P1,L2P2,P1B,P2B。将菌种L1,L2,P1,P2,B分别活化后培养于MRS液体培养基中,稀释或浓缩至菌落总数1.0×109CFU·mL-1;刈割后的苜蓿晾晒24 h,其干物质含量为(29.03±0.79)%,切割成2~5 cm的小段,分别按1.5 mL·kg-1的剂量添加等比例混合的乳酸菌组合,以等量蒸馏水替代乳酸菌组合作为对照组(CK),为确保添加剂与苜蓿均匀混合,将添加物稀释4倍后均匀喷洒在切割好的苜蓿上,装入特制聚乙烯塑料袋(25 cm×35 cm),每袋300 g,每个处理3个重复,用真空封口机同时进行抽气和封口。室温避光保存45 d后,在每个样品的非表层部分用经高温灭菌的镊子按照五点采样法采集苜蓿青贮样品20 g左右,先液氮速冻,再存于-80℃冰箱中,用于细菌群落结构测定与分析。
1.3 细菌群落结构测定与分析
苜蓿青贮样品细菌群落结构测定由上海派森诺生物科技股份有限公司完成。采用Illumina MiSeq平台选用长度约为280 bp的细菌16S rRNA基因的高度可变的V3-V4区进行测序分析,其PCR扩增引物为338F(5′-3′)ACTCCTACGGGAGGCAGCA,806R(5′-3′)GGACTACHVGGGTWTCTAAT。PCR反应体系(25 μL):Q5高保真DNA聚合酶0.25 μL,5×反应缓冲液5 μL,5×高GC缓冲液5 μL,10 mMdNTP0.5 μL,模板DNA1 μL,正向引物(10 μM)和反向引物(10 μM)各1 μL,ddH2O 11.25 μL。PCR程序:98℃30 s;98℃15 s,50℃30 s,25~27个循环;72℃ 30 s,72℃5 min,于4℃保存。PCR扩增结果进行2%琼脂糖凝胶电泳,切取目的片段然后用Axygen凝胶回收试剂盒回收目的片段。利用Quant-iT PicoGreen dsDNA Assay Kit对PCR产物在Microplate reader (BioTek,FLx800)上进行定量,然后按照每个样品所需的数据量进行混样。利用Illumina公司的TruSeq Nano DNA LT Library Prep Kit构建文库。在Agilent Bioanalyzer机器上用Agilent High Sensitivity DNA Kit对文库做2100质检,利用Quant-iT PicoGreen dsDNA Assay Kit在Promega QuantiFluor上对文库进行定量,合格的文库在MiSeq机器上利用MiSeq Reagent Kit V3(600cycles)进行2×300 bp的双端测序。
1.4 数据统计与分析
运用R软件对测序数据进行分析。首先对所获序列预处理,评估有效序列质量,获得可用于后续分析的高质量序列;然后对高质量序列按照97%的序列相似度划分可操作分类单元(Operational Taxonomic Unit,OTU);再将丰度值高于0.001%的OTU代表序列与细菌16S rRNA基因数据库的模板序列进行比对,即可获取每个OTU所对应的分类学信息。
细菌群落多样性选择Chao1,ACE,Shannon和Simpson4个指数进行分析。Chao1丰富度指数通过计算群落中只检测到1次和2次的OTU数(即“Singleton”和“Doubleton”),估计群落中实际存在的物种数;ACE丰富度指数默认将序列量10以下的OTU都计算在内,从而估计群落中实际存在的物种数;多样性指数Shannon和Simpson综合考虑了群落的丰富度和均匀度,前者对群落丰富度和稀有OTU更敏感,后者对均匀度和群落中的优势OTU更敏感。
文中数据均以“均值±标准差”表示。通过IBM SPSS statistics 20.0对不同处理组数据进行多重比较,通过SigmaPlot12.0进行制图。
2 结果与分析
2.1 不同乳酸菌组合处理对苜蓿青贮细菌群落结构的影响
高通量测序结果显示,21个苜蓿青贮样品获得46 551~63 375个有效序列(合计1 194 775个),物种累计曲线最终趋于平缓,说明样本量足以反映群落的丰富度,且所有的OTU均可归属至已知分类单元(门、纲、目、科、属)。
由表1可知,CK,L1P1,L1P2,L2P1,L2P2,P1B和P2B的OTU总数分别为347,442,449,468,502,383,460个,其中P2B处理与CK的共有OTU数占比最高(58.26%),其次是P1B(48.56%),然后依次是L1P2(39.20%),L2P2(38.05%),L2P1(29.35%),L1P1(20.81%),说明含凝结芽孢杆菌的处理与对照组菌群相似性较高。由表2可知,对OTU进行划分及分类地位鉴定,在门、纲、目、科及种水平下,OTU数量均表现为L1P2处理显著高于CK(P<0.05),其他处理组间差异均不显著,而在属水平OTU数量各处理组间差异均不显著;对各分类水平的细菌类群数量进行统计,门、目、科、属水平细菌类群数量处理组间差异均不显著,在纲水平上细菌类群数量表现为L1P2和L2P1处理显著高于P2B处理(P<0.05),其他处理组间差异均不显著,在种水平上细菌类群数量表现为CK显著高于L2P2处理(P<0.05)。
2.1.1门水平细菌组间差异 由苜蓿青贮在门水平相对丰度的组间差异(表3)可知,各处理均以厚壁菌门(Firmicates)相对丰度最高,为69.3%~83.1%,然后是蓝细菌门(Cyanobacteria)和变形菌门(Proteobacteria),分别为3.9%~26.4%和0.4%~14.6%,而放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、软壁菌门(Tenericutes)、异常球菌-栖热菌门(Deinococcus-Thermus)、绿弯菌门(Chloroflexi)、梭杆菌门(Fusobacteria)、疣微菌门(Verrucomicrobia)相对丰度均小于2%(数据未在表3中体现)。其中,厚壁菌门相对丰度在组间差异均不显著;蓝细菌门表现为L1P1处理显著高于CK和P2B处理(P<0.05);而变形菌门表现为P2B处理和CK显著高于除P1B外的其他处理(P<0.05)。综合而言,各处理的优势菌门均为厚壁菌门,乳酸菌处理组的次优势菌门为蓝细菌门。
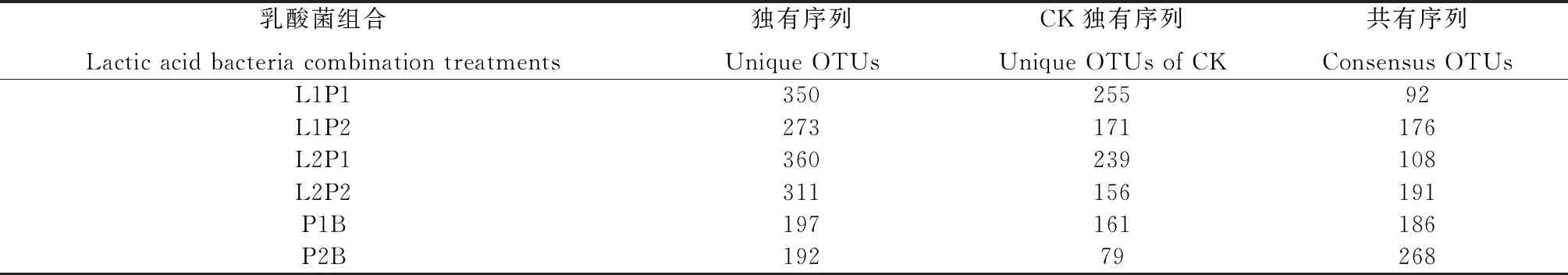
表1 乳酸菌组合处理苜蓿青贮细菌序列(OTU)与对照的对比关系
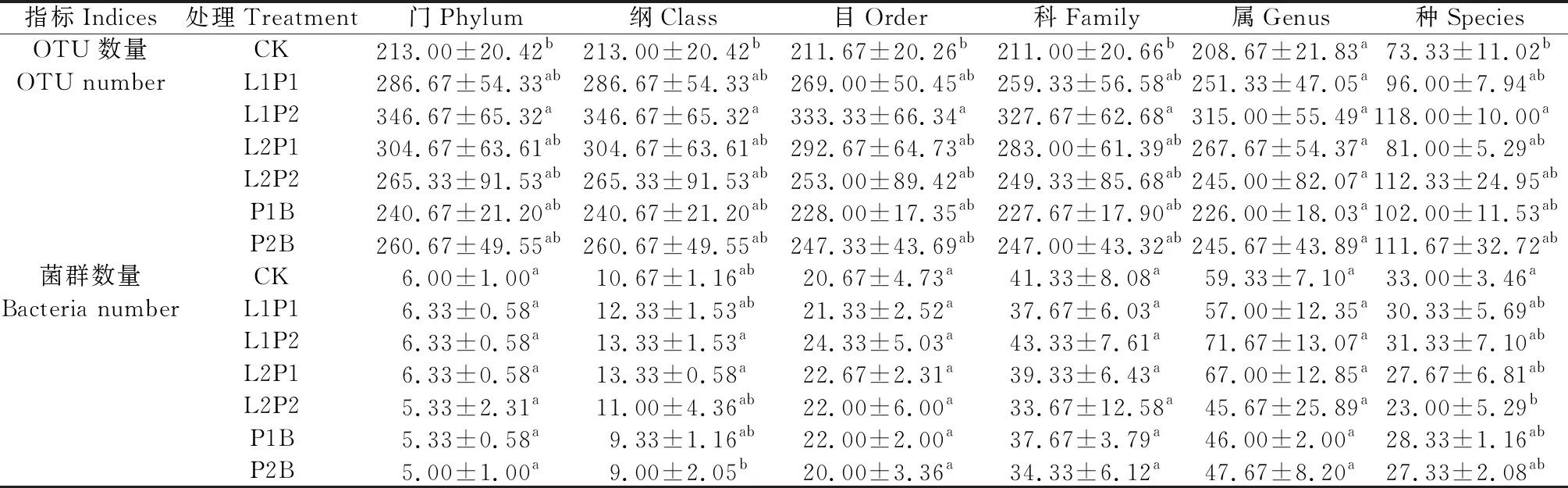
表2 苜蓿青贮样品细菌序列(OTU)划分及细菌类群分类地位统计
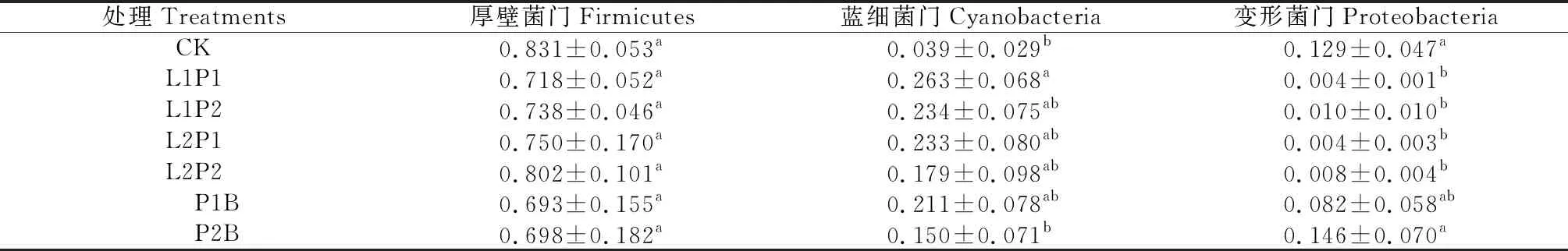
表3 苜蓿青贮细菌群落门水平相对丰度的组间差异
2.1.2属水平细菌组间差异 分析苜蓿青贮组间相对丰度有显著差异的目/科/属共有28个,其中相对丰度大于1%的属详见表4。由表4可知,乳杆菌属(Lactobacillus)在L1P1,L1P2,L2P1,L2P2处理显著高于CK及P1B和P2B处理(P<0.05),其他组间差异均不显著;片球菌属(Pediococcus)在P2B处理最高,然后依次是P1B和CK,三者差异不显著,均显著高于L1P1和L2P1处理(P<0.05),P2B显著高于L1P2(P<0.05),P2B和P1B均显著高于L2P2(P<0.05);肠球菌属(Enterococcus)在P1B处理显著高于除CK和P2B以外的其他处理(P<0.05);乳球菌属(Lactococcus)、芽孢杆菌属(Bacillus)和鞘氨醇单胞菌属(Sphingomonas)相对丰度均表现为CK显著高于其他各处理(P<0.05);棒球菌属(Corynebacterium)相对丰度则表现为P1B处理显著高于其他处理(P<0.05);野豌豆属(Viciafaba)表现为CK显著低于除P1B和P2B外的其他处理(P<0.05)。
由此可见,CK的优势菌群为片球菌属,相对丰度为43.5%,次优势菌群为乳杆菌属(23.9%);P1B和P2B处理优势菌群为片球菌数,相对丰度分别为46.9%,49.7%,次优势菌群是乳杆菌属(20.2%,15.7%)和野豌豆属(10.1%,14.6%);L1P1,L1P2,L2P1,L2P2处理的优势菌则为乳杆菌属,相对丰度分别为49.2%,43.8%,55.2%,54.2%,次优势菌群是片球菌属(21.5%,28.1%,16.8%,24.8%)和野豌豆属(24.9%,23.1%,22.9%,17.5%)。CK,L1P1,L1P2,L2P1,L2P2,P1B,P2B处理优势乳酸菌群(乳杆菌属+片球菌属)的相对丰度分别为67.4%,70.7%,71.9%,72.0%,79.0%,67.1%,65.4%。
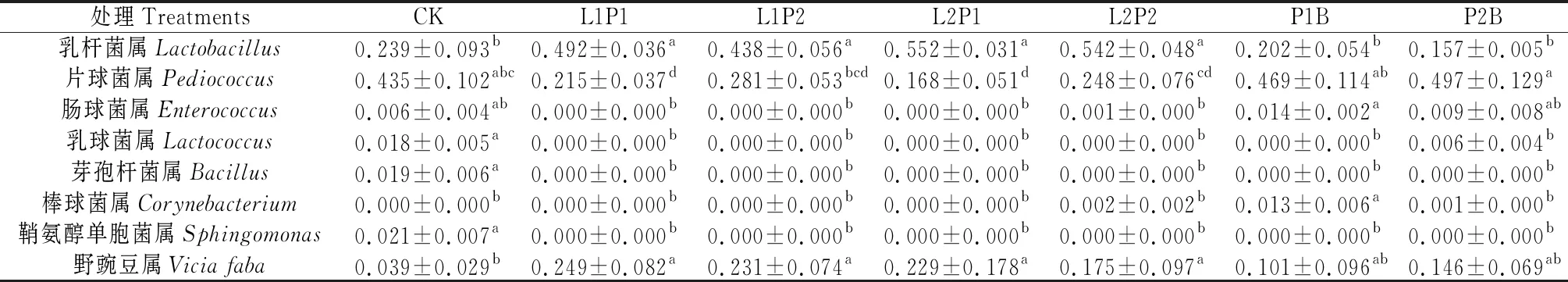
表4 苜蓿青贮细菌群落属水平相对丰度的组间差异
2.2 苜蓿青贮样品细菌群落多样性
Chao1指数和ACE指数可体现群落丰富度,Shannon指数和Simpson指数兼顾群落丰富度和均匀度。由表5可知,苜蓿青贮样品的细菌群落Chao1指数和ACE指数在添加剂处理均较CK有所增加,其中Chao1指数在L1P2处理显著高于CK(P<0.05),ACE指数在L1P2处理显著高于P1B处理和CK(P<0.05);Simpson和Shannon指数在添加剂处理均较CK有所下降但差异未达显著水平。

表5 苜蓿青贮样品细菌多样性指数
对不同处理苜蓿青贮样品的细菌丰度和细菌群落组成进行基于Unifrac距离的NMDS非度量多维尺度分析,结果表明,除L2P2处理外其他各组内差异均相对较小,其中CK独立分为一组,P2B和P1B处理分为一组,距离CK较近,L1P1,L1P2,L2P1组距离较近,可分为一组(图1)。
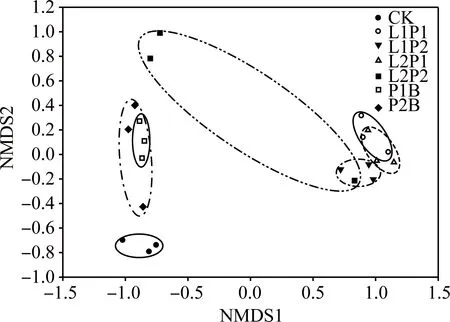
图1 不同苜蓿青贮样品微生物丰度的NMDS分析图
2.3 苜蓿青贮细菌群落结构及多样性与其营养品质的关系
2.3.1细菌群落结构 由表6可知,厚壁菌门和蓝细菌门的相对丰度与苜蓿青贮营养和发酵指标之间的相关关系均未达显著水平,但变形菌门的相对丰度与苜蓿青贮粗蛋白、非纤维性碳水化合物及乳酸含量极显著负相关(P<0.01),而与中性洗涤纤维、酸性洗涤纤维、灰分、pH值和乙酸含量极显著正相关(P<0.01),说明变形菌门丰度的增加容易造成养分损失,降低乳酸含量增加乙酸含量,不利于纤维分解和pH值的降低,对发酵品质提升不利。
2.3.2细菌群落多样性 由表7可知,Chao1和ACE指数与苜蓿青贮非结构性碳水化合物含量极显著负相关(P<0.01),说明苜蓿细菌群落丰富度的增加会导致非结构性碳水化合物的降低;Simpson指数与各营养指标之间的相关关系均未达到显著水平;Shannon多样性指数兼顾细菌群落丰富度和均匀度,对稀有菌群敏感,与粗蛋白含量极显著负相关(P<0.01),与乳酸含量显著负相关(P<0.05),与中性洗涤纤维和乙酸含量显著正相关(P<0.05),说明苜蓿青贮杂菌数量增加引起的Shannon多样性指数的提高,会导致其蛋白损失,乙酸含量增加,且不利于纤维可消化性的提高及乳酸的积累。
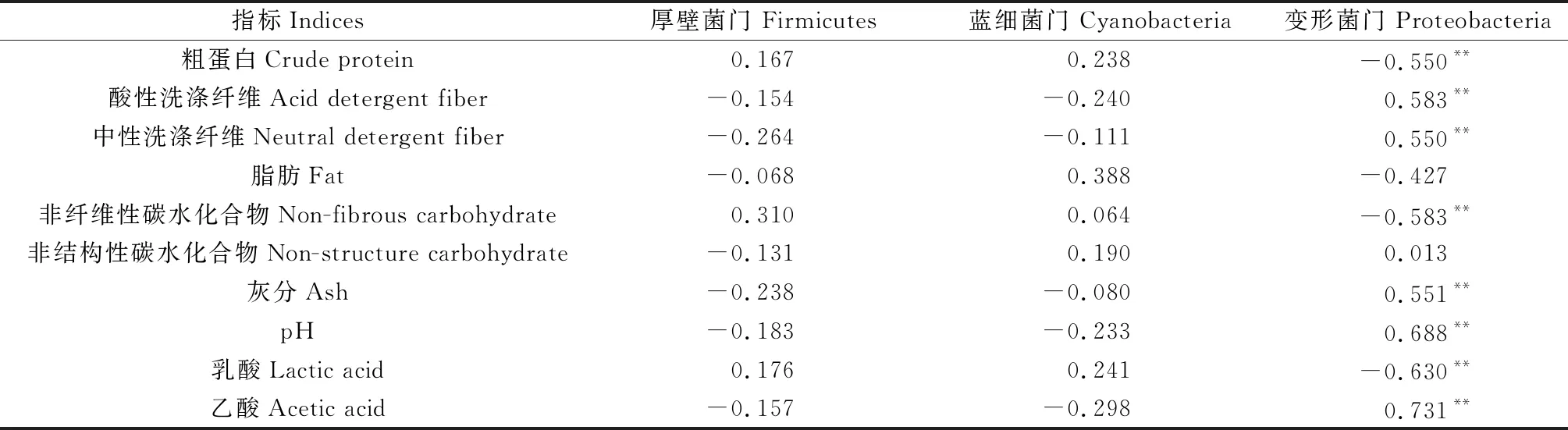
表6 苜蓿青贮样品细菌群落结构与营养品质的关系
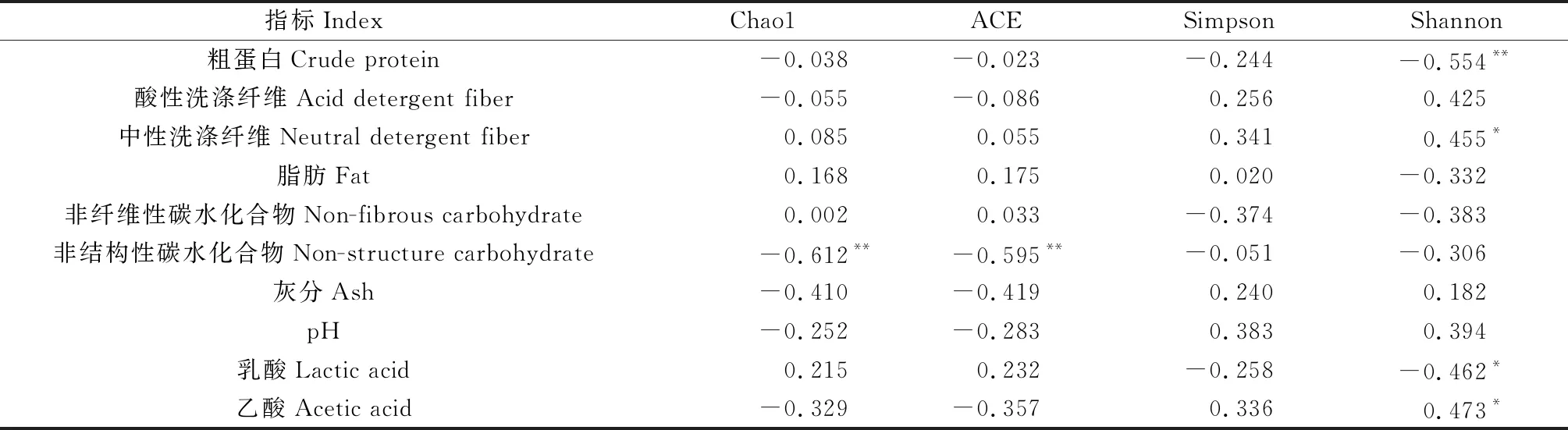
表7 苜蓿青贮样品细菌群落多样性与营养品质的关系
3 讨论
发酵过程即微生物群落的演替过程,经过最初的有氧发酵和后续的厌氧发酵阶段,不良菌群减少,乳酸菌开始主导发酵过程产生大量乳酸[30]。据报道,青贮饲料中参与乳酸发酵的菌群大多数属于厚壁菌门[28],其与变形菌门和蓝细菌门共同构成了鲜苜蓿的主要附生细菌群落[18,27],发酵后厚壁菌门逐渐占优势[13,28]。本研究结果与之相似,各苜蓿青贮中细菌群落也是以厚壁菌门(Firmicates)为主,其次是蓝细菌门(Cyanobacteria),经鉴定主要为Viciafaba属,推断其应该是来自于苜蓿青贮的植物样本部分,第三大类群为变形菌门(Proteobacteria),仅在对照组和P2B处理相对丰度大于10%。变形菌门部分菌属可通过脱氨反应生成氨,减缓pH值的降低[23],同时可与乳酸菌竞争可利用的水溶性碳水化合物[17],故其相对丰度与苜蓿青贮非纤维性碳水化合物含量极显著负相关而与pH值极显著正相关(表6),这与闫晶等[31]研究中认为变形菌门与pH成正相关的结果相似。
不同添加剂种类会导致青贮微生物群落结构的差异[30]。乳酸菌是青贮发酵成功的关键微生物[32],主要通过增加群落乳酸菌丰度促进乳酸发酵而实现抑制有害菌繁殖、保存青贮物料的目的[29]。一般而言,在不应用青贮添加剂的情况下,青贮优势菌群主要受植物附生菌影响,而附生菌因气候、地理位置、管理措施等多方面因素而存在较大的异质性[18,27]。Ni等[33]研究报道肠球菌科(Enterococcaceae)和肠杆菌科(Enterobacteriaceae)是鲜苜蓿的主要附生菌群;马召稳等[13]研究报道含水量65%的苜蓿青贮微生物群落在属水平上主要为海洋杆菌属(Oceanobacillus)、乳杆菌属(Lactobacilli)、芽孢杆菌属(Bacilli)、枝芽孢杆菌属(Virgibacillus)、乳酸球菌属(Lactococcus)等;Agarussi等[17]研究报道萎蔫的苜蓿青贮中植物乳杆菌(L.plantarum)占优势,同时也鉴定出乳酸片球菌(P.acidilactici)和戊糖片球菌(P.pentosaceus)等其他乳酸菌。本研究中,对照组未添加任何外源添加剂的情况下,其优势菌为片球菌属(Pediococcus)和乳杆菌属(Lactobacillus),说明苜蓿自身附着的细菌中有二者的存在,且推断在发酵过程中片球菌的作用大于乳杆菌,故青贮后片球菌属的相对丰度显著高于乳杆菌属。据报道,在应用乳酸菌剂后,苜蓿青贮优势菌群基本上是添加剂中使用的乳酸菌,如植物乳杆菌、乳酸片球菌、屎肠球菌(E.faecium)等[17,24]。本研究所用的乳酸菌组合中,含凝结芽孢杆菌处理的优势乳酸菌为片球菌属,其次是乳杆菌属,即在片球菌与凝结芽孢杆菌共同添加的情况下,片球菌属具有一定的优势,但芽孢杆菌属相对丰度甚至低于对照组(表4),推测芽孢杆菌属包括凝结芽孢杆菌的繁殖在苜蓿青贮过程中被抑制,可能其不宜应用于苜蓿青贮中;含植物乳杆菌处理的优势乳酸菌均为乳杆菌属,其次为片球菌属,说明在植物乳杆菌和片球菌共同添加的情况下,植物乳杆菌的优势明显。
外源添加剂处理可影响细菌群落多样性,且不同的多样性指数变化趋势不同[27]。有研究表明,苜蓿经γ射线+外源菌剂处理后青贮微生物Chao1和Shannon指数降低,而仅添加外源菌剂处理与对照差异不显著[18];接种植物乳杆菌或布氏乳杆菌可增加苜蓿青贮优势菌属的丰度从而降低细菌群落多样性[28]。本研究中,苜蓿青贮菌群丰富度均较对照有所提高,菌群丰富度的增加可能导致更多青贮发酵底物的消耗,表现在营养成分上就是非结构性碳水化合物的降低,故菌群丰富度指数与苜蓿青贮非结构性碳水化合物含量极显著负相关(表7);对优势度敏感的菌群Simpson多样性指数与苜蓿青贮营养指标的相关关系均不显著(表7);但对稀有菌群敏感的菌群Shannon多样性指数却与粗蛋白及乳酸含量极显著或显著负相关,与中性洗涤纤维和乙酸含量显著正相关(表7),说明该指数的增加不利于苜蓿青贮品质相关指标的提升。
乳酸菌是一类能够利用碳水化合物发酵产生大量乳酸的细菌,不同乳酸菌在青贮中的作用不同,有的乳酸菌代谢产物可抑制霉菌生长,有的可以抑制氨的产生等[7]。本研究中,凝结芽孢杆菌组与对照组距离较近(图1),说明其对苜蓿青贮菌群结构的影响较小,甚至在一定程度上降低了苜蓿青贮中乳杆菌属的相对丰度(表4);含植物乳杆菌组距离对照组和凝结芽孢杆菌组均较远(图1),说明植物乳杆菌对苜蓿青贮菌群结构的影响较大,无论哪个含有植物乳杆菌的处理,苜蓿青贮后乳杆菌属的相对丰度均较对照组显著增加(表4)。
4 结论
通过对添加6种乳酸菌组合的苜蓿青贮样品细菌群落结构进行分析,各乳酸菌组合的应用均在一定程度上改善了苜蓿青贮的细菌群落结构,其优势乳酸菌群均为厚壁菌门(Firmicates)的乳杆菌属(Lactobacillus)和片球菌属(Pediococcus),二者相对丰度之和为65.4%~79.0%,其中含植物乳杆菌的组合效果优于含凝结芽孢杆菌的组合。
