基于全基因组重测序技术的狮头鹅Indel标记分析
2021-03-30王慧芳周光现孙永峰陈新企雷栩斌张锐毅贾汝敏赵志辉
王慧芳,周光现,孙永峰,陈新企,雷栩斌,张锐毅,贾汝敏*,赵志辉*
(1. 广东海洋大学滨海农业学院,湛江 524088; 2. 吉林农业大学动物科学技术学院,长春 130118)
中国是世界上家鹅品种最多、驯养历史最悠久的国家之一。由于我国幅员辽阔,各地自然生态条件复杂多样,不同时期的经济文化背景不同,对鹅的选择利用目的不同,逐步形成了具有不同遗传特性和生产性能的地方品种,分布遍及全国[1-4]。目前,随着鹅相关产品需求水平的增长,低繁殖力成为了制约鹅产业发展的主要因素[5-6]。影响鹅产蛋性能的因素是多面的,如遗传基因、生理状况、环境条件、饲料营养和使用年限等,各种应激(如停电停水、噪音等)都可显著降低鹅的产蛋量[7-10]。狮头鹅起源于鸿雁,是我国第一批受保护的畜禽品种资源之一,因长期受人类和自然环境的影响,有着高度的遗传多样性,其具有耐粗饲、生长速度快、抗逆性强、耗料少、肉质好等优良特性,被誉为“世界鹅王”[11-12]。狮头鹅成年种公鹅体重为10~12 kg,母鹅为9~10 kg,母鹅年产蛋25~32枚[13-14]。吉林白鹅是由吉林农业大学科技工作者历经20余年精心培育而成,其成年公鹅体重4.1 kg,母鹅3.2 kg,年产蛋数达100枚以上,具有产蛋多、耐粗饲、抗病力强等特点[15]。狮头鹅与吉林白鹅产蛋量相差悬殊,适合作为探索鹅繁殖性状相关研究的素材。
随着基因组学技术的发展,使得大规模、高通量检测狮头鹅基因组内的变异位点,筛选产蛋相关通路以及相关产蛋基因成为可能[16-18]。Indel (insertion deletion) 是影响基因组中生产繁殖性能的关键遗传标记,研究发现,生物学基因组中Indel起着关键性的作用[18-20]。Indel变异一般比SNP变异少,但同样反映样品与参考基因组之间的差异,并且编码区的Indel会引起移码突变,导致基因功能上的变化[21-22]。本研究采用第三代重测序技术,获得了狮头鹅插入/缺失(Indel)位点,经过数据的比较分析发现,大量基因与狮头鹅的产蛋性能相关。经过数据分析发现,GnRH信号通路、雌激素信号通路、催产素信号通路、催乳素信号通路以及孕酮介导的卵母细胞成熟通路与狮头鹅产蛋性状相关。利用比较基因组学筛选鉴定两种鹅群体中特异性Indel位点[23],为狮头鹅的群体遗传分析提供基础,并进一步筛选产蛋相关基因,为狮头鹅生产研究提供新的思路。
1 材料与方法
1.1 试验材料
本研究选取健康成年狮头鹅母鹅(年均产蛋25~32枚) 和吉林白鹅母鹅(年均产蛋≥100枚)各5只(R1~R5:狮头鹅;R6~R10:吉林白鹅),静脉翅下采血3 mL,加入抗凝剂,置于-20 ℃保存备用。
1.2 血液DNA提取
血液样品DNA使用美基生物公司的HiPure Blood DNA Mini Kit试剂盒进行提取。经1%凝胶电泳检测DNA完整性,并使用Nanodrop检测其纯度。合格样品DNA送北京百迈客公司测序。
1.3 全基因组重测序
1.3.1 文库构建与测序 样品基因组DNA检测合格后,用机械打断的方法(超声波)将DNA片段化,然后进行片段纯化,末端修复,3′端加A以及连接测序接头。通过琼脂糖凝胶电泳选择片段的大小,PCR扩增目的片段形成测序文库,对质检合格的文库进行Illumina测序。
1.3.2 测序数据质控 测序数据通过公式Qphred =-10lg(e)(Qphred为碱基质量值;e为测序错误率)转化可以得到碱基测序的错误率。在碱基识别过程中计算Phred数值(一种预测碱基判别发生错误的概率模型),其对应关系如表1所示。

表1 预测碱基判别发生错误概率结果
1.3.3 低质量数据过滤 原始测序序列(Raw Reads)含有带接头的以及低质量的Reads,需对Raw Reads进行过滤得到Clean Reads后,方可用于后续的信息分析。数据过滤的主要步骤:1) 去除带接头(adapter)的reads;2) 过滤N含量超过10%的reads;3) 去除质量值低于10的碱基超过50%的reads。
1.4 Small Indel的检测与注释
将在参考基因组上定位后的Clean Reads结果进行分析,检测是否存在Small Indel变异。使用GATK软件工具包检测Small Indel插入与缺失;使用Picard(http://sourceforge.net/projects/ picard/)过滤冗余reads(MarkDuplicates),以保证检测结果的准确性;然后使用GATK的HaplotypeCaller(局部单体型组装)算法进行Indel的变异检测;通过SnpEff软件实现Small Indel的注释。
1.5 DNA水平的变异基因挖掘
比较筛选狮头鹅与吉林白鹅两个群体特有的差异Indel位点,通过BLAST变异基因与GO、COG、KEGG等功能数据库比对,筛选产蛋相关通路。通过KEGG注释筛选产蛋相关候选基因,用以分析基因功能。
2 结 果
2.1 数据质量控制
由电泳检测结果(图1)可以看出,DNA纯度以及完整性良好。另外检测得出OD260 nm/OD280 nm值均在1.88左右,其浓度、体积和质量平均值分别为:173.4 ng·μL-1、99.1 μL和18.7 μg。
通过对原始数据各碱基比例分布和碱基质量分布的分析来进行质量控制,得到图2。从图中可以看出,AT曲线以及GC曲线均重合,证明AT碱基以及GC碱基比例均相等,无明显偏向性,曲线较平缓,未发生分离,测序结果正常,该数据可用于下一步分析。
通过对狮头鹅和吉林白鹅全基因组重测序,原始数据过滤处理后共得到350.35 Gb的Clean Data,各样本的Raw data在71 553 908~208 410 322 Mb之间, Q20与Q30平均值分别达到96.74%、92.19%。GC含量在43.67%~45.64%之间(表2),结果表明,测序质量良好,可进行后续分析。
R1~R5. 狮头鹅;R6~R10. 吉林白鹅。下同 R1-R5. Shitou goose;R6-R10. Jilin white goose. The same as below图1 琼脂糖凝胶电泳检测结果Fig.1 The detection results of agarose gel electrophoresis
2.2 与参考基因组的比对分析
参考基因组为Anser cygnoides (Swan goose),其版本为AnsCyg_PRJNA183603_v1.0;下载网址:https://www.ncbi.nlm.nih.gov/genome/?term=Anser+cygnoides,参考基因组的大小为1 134 Mb(表3)。由表4可知,所有样本数据与参考基因组的比对率在93.18%~94.96%之间,平均比对率为94.15%,平均覆盖深度为26×,1×覆盖度(至少有1个碱基的覆盖)在99.47%以上,5×覆盖度(至少有5个碱基的覆盖)在96.84%以上。结果表明,测序物种同参考基因组相似度高,证明测序准确,可进行后续的相关分析。
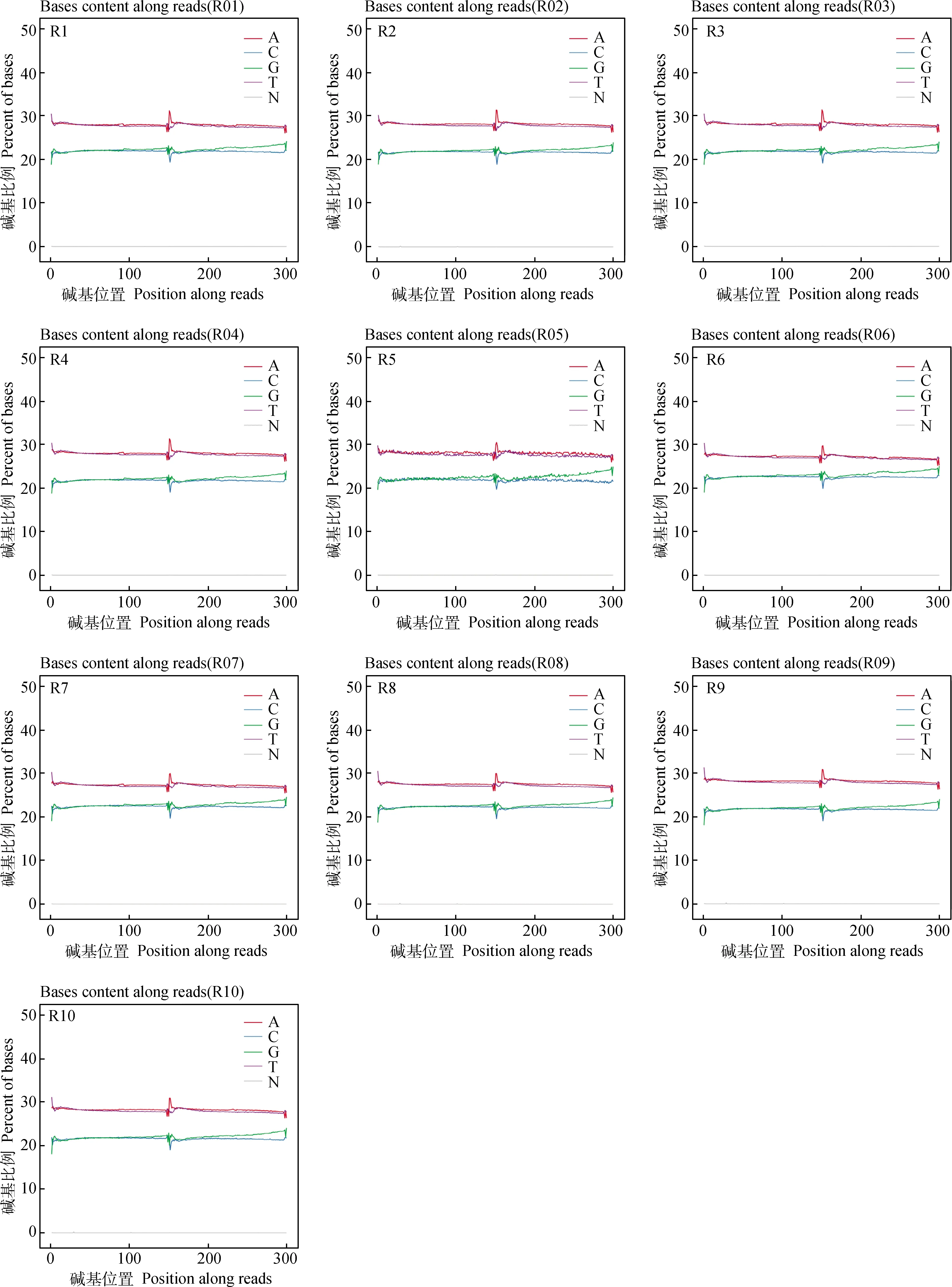
N. 测序中识别不出的碱基 N. The unrecognized bases in sequencing图2 样品各碱基比例分布Fig.2 The proportion of each base of the samples
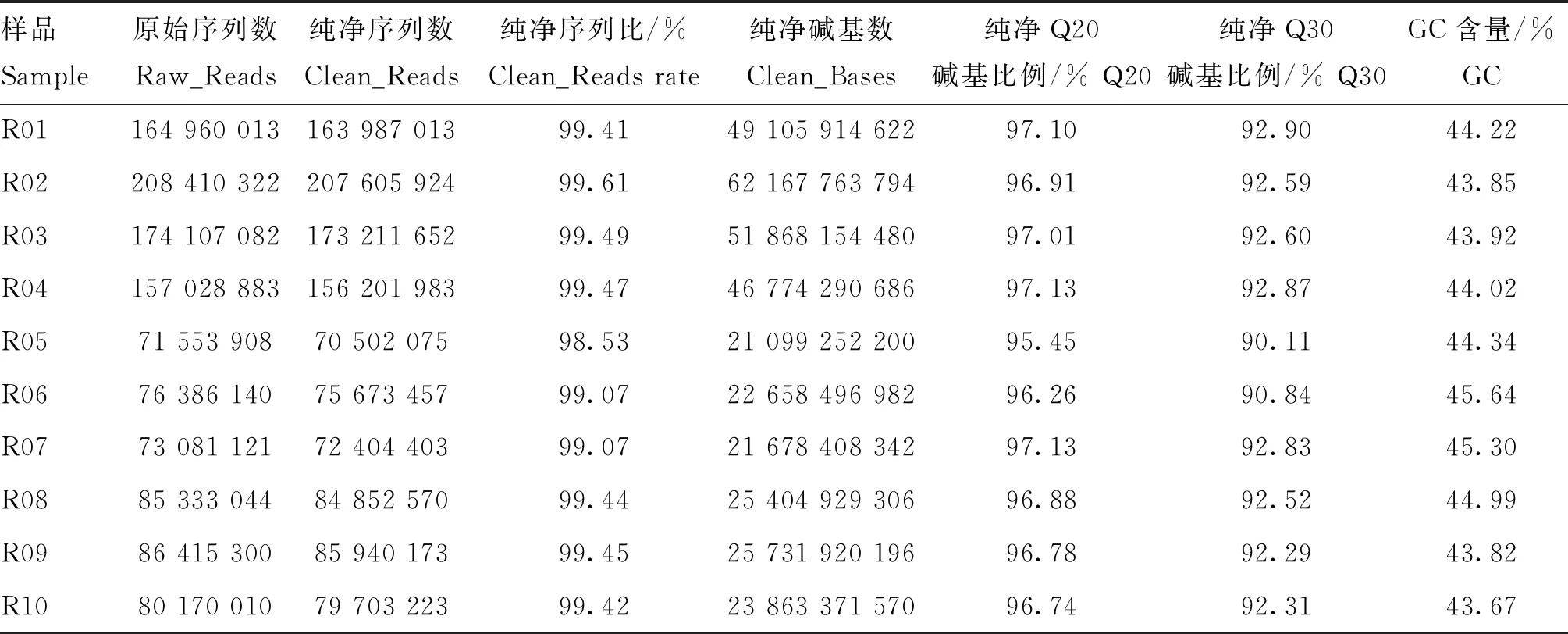
表2 测序数据质量表

表3 参考基因组信息

表4 测序深度及覆盖度统计
2.3 Indel序列长度及分布分析
利用GATK从10只试验鹅与参考基因组比对结果中检测Indel变异(小于50 bp碱基片段的插入与缺失)。然后对Indel序列的分布位置进行注释及分析(表5)。
通过SnpEff软件注释分析Indel位点,分别从10只试验鹅中发现了786 695、782 331、766 853、775 202、718 792、717 274、708 825、738 934、753 315、 739 991个Indel位点(平均有748 821.2个),其数量和分布与参考基因组相似。在这10只试验鹅中,Indel位点主要分布在内含子,吉林白鹅与狮头鹅Indel位点在内含子中分别占有54.18%和52.12%;其次是基因间区,两种鹅分别占有36.05%与33.91%(图3)。大部分的Indel位点分布在内含子与基因间区中,CDS区中分布的Indel位点最少。
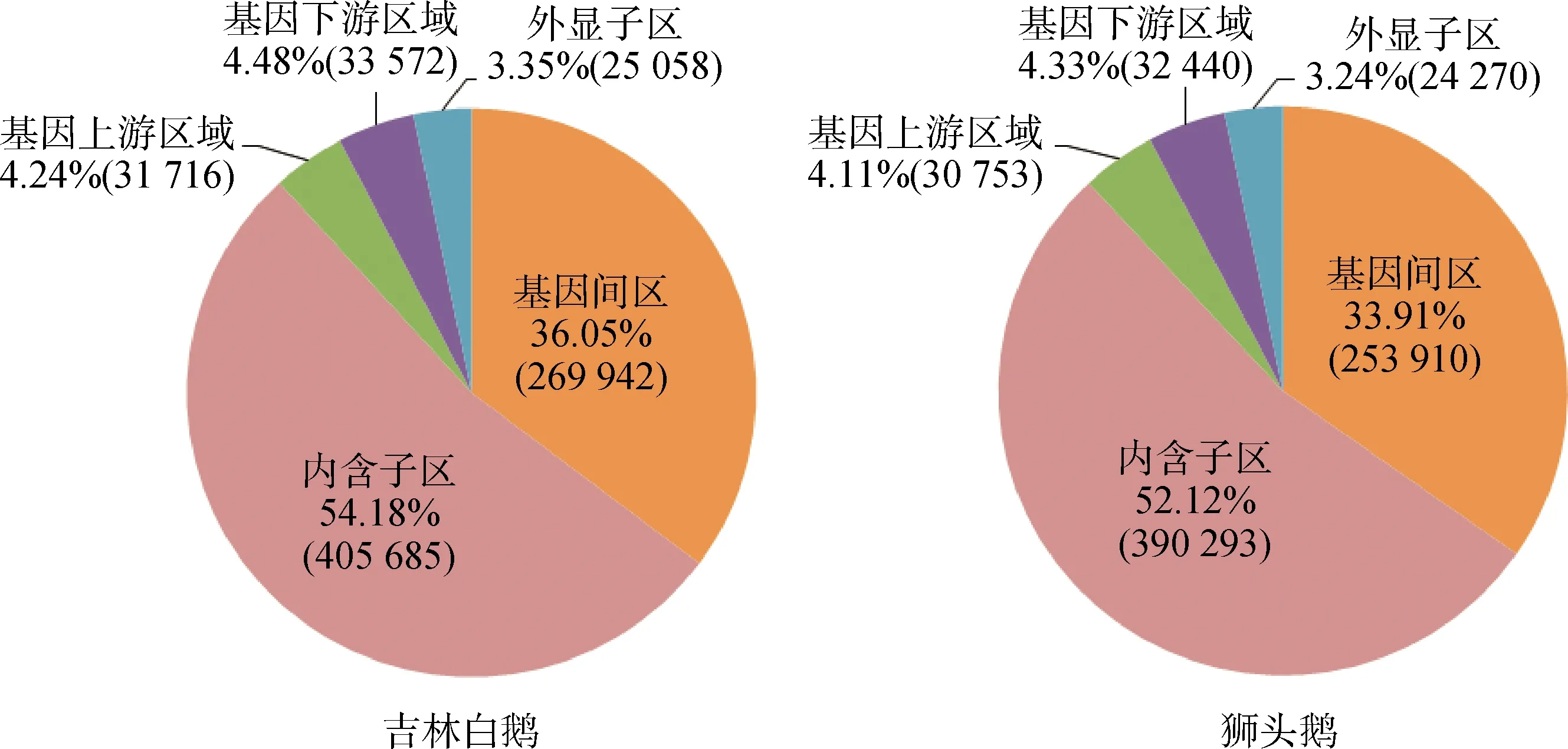
图3 吉林白鹅和狮头鹅相同Indels在基因组上的分布Fig.3 Genomic distribution of the same Indels of Jilin white goose and Shitou goose

表5 Indel 位点的检测及注释
在这10只试验鹅全基因组中,Indel位点的长度分布趋势也基本一致。对群体Indel突变位点的碱基长度进行统计并分析,得到图4,发现全基因组中长度为1 bp的Indel位点数量超过310 000个(约占全基因组所有Indel的43%);长度为2~4 bp的Indel位点数量为2 529 498个;长度为5~9 bp的Indel位点数量为860 657个;长度大于等于10 bp 的Indel位点数量共808 872个。并且在编码区中,长度为1 bp的Indel位点数量共22 754个(约占编码区所有Indel的33%);长度为2~4 bp的Indel位点数量为22 426个;长度为5~9 bp的Indel位点数量为10 080个;长度大于等于10 bp的Indel位点数量共13 491个。另外,图4说明了在片段变异10 bp长度的范围内,随着碱基片段长度增加,发生Indel变异的数量随之减少,且数量波动较大。
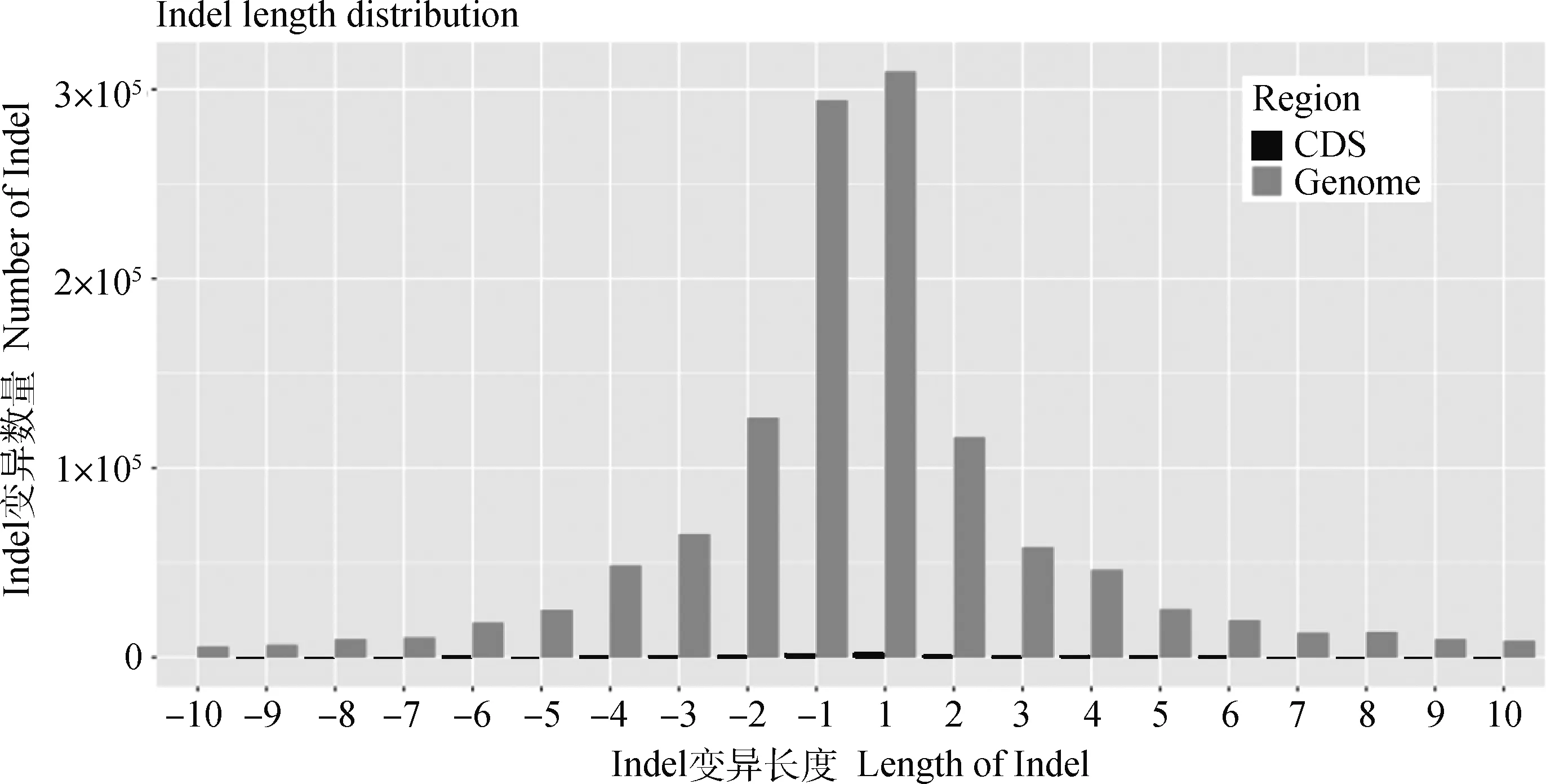
图4 全基因组和编码区的Indel长度分布情况Fig.4 Distribution map of Indel length in the population’s entire genome and coding region
10只试验鹅的Indel位点在CDS区的分布趋势和数量也与参考基因组相似,分别有7 090、7 026、 7 022、7 060、6 696、6 649、6 651、6 862、6 868和6 827个Indel位点(表6)。其中共有11 737个Indel位点导致了插入或删除。
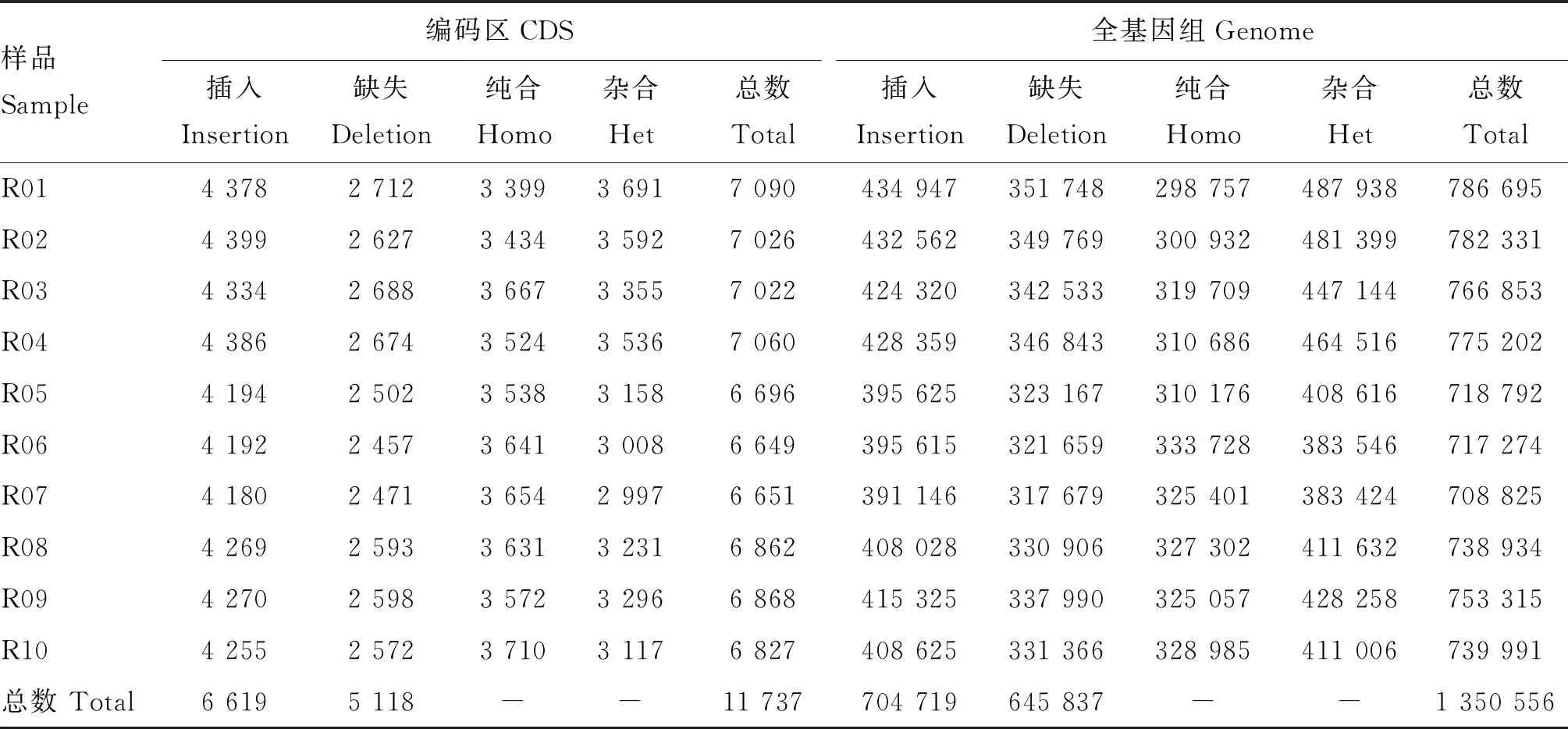
表6 Indel变异在全基因组和编码区的统计情况
2.4 变异基因注释分析
通过GO注释分析富集了到细胞组分(cellular component)17个、分子功能(molecular function)20个和生物过程(biological process)23个,详见表7。
通过KEGG注释分析富集到253条通路,其中筛选出与产蛋相关的通路,分别为GnRH信号通路、雌激素信号通路、催乳素信号通路、催产素信号通路以及孕酮介导的卵母细胞成熟通路。通过比对测序得到的群体基因注释列表发现了36个产蛋相关变异基因,详见表8。这为狮头鹅后期高产基因的筛选奠定了基础。

表7 变异基因功能富集分析的相关注释

(转下页 Carried forward)
3 讨 论
狮头鹅是我国乃至世界著名的大型水禽品种,也是我国南方养殖范围较广的经济家禽,其产蛋期一般为9月至次年4月[24-25]。吉林白鹅是我国北方养殖较广的经济家禽,其产蛋旺季在2~6月,研究表明,两种鹅日照长短不一样[26]。甄霆等[27]研究发现,在浙东白鹅的繁殖季节通过调节光照可影响FSH和INH的表达,从而延长其产蛋时间,缩短抱窝和就巢时间,增加鹅的产蛋量,说明鹅的繁殖性能受光照的影响,合理地控制光照可延长鹅的产蛋期,缩短休产期,提高鹅的产蛋性能。姚颖等[28-31]研究发现,浙东白鹅约83.04%的产蛋是在有光照的情况下,仅有16.96%的产蛋是在无光照的情况下,说明产蛋行为与光照密切相关[28]。产蛋性状的遗传力较低,其由微效多基因控制,三代测序技术的发展为研究狮头鹅产蛋性状提供了新的思路。由于狮头鹅较强的就巢性和产蛋季节性导致其繁殖力较低,从而造成狮头鹅产蛋性能低下。提高狮头鹅的产蛋性能一直是研究的主要目标之一。
本研究利用重测序技术对5只狮头鹅和5只吉林白鹅全基因组测序后共得到350.35 Gb数据,其中Q20与Q30平均值分别达到96.74%、92.19%,测序数据结果可靠。在此基础上,通过注释分析得到Indel位点平均有748 821.2个变异。大部分(53%左右)的Indel位点分布在内含子中,蛋白质编码区中分布的Indel位点最少(11 737个)。研究还发现,Indel位点的长度分布趋势也基本一致,10只 试验鹅的Indel位点在CDS区的分布趋势和数量也与参考基因组相似。在此基础上,通过plink软件对SNPs进行主成分分析(PCA)发现(数据未显示),吉林白鹅与狮头鹅两个群体能够有效地分离。从SNP层次观测,两种鹅具有较大的差异性,这种差异性包含潜在主导其不同性状的SNP位点。吉林白鹅体型小,年产蛋量高,而狮头鹅体型大,年产蛋量低。张泽宾[32]对22只野鸭和56只家鸭全基因组测序得到310万个Indel变异位点,对Indel注释发现,绝大多数的变异位于基因间区及内含子等非编码区域,此研究发现,编码区中的Indel突变比率与其它区间相比较低。刘杰[33]分别选取北京油鸡和科宝肉鸡中高采食量个体48只和低采食量个体48只进行全基因组重测序,得到的Indel变异位点在内含子和基因间区最多,编码区较少。章双杰等[34]对4种鸭群进行全基因组重测序分析,通过检测到的Indel位点发现,其Indel变异大部分存在于基因间或是基因内的内含子区,证明编码区的变异相对较少。以上研究结果均与本试验Indel变异分布结果一致,非编码区的Indel变异多于编码区,这一结果符合生物学特性。对差异分析筛选的候选Indel标记的作用可进一步研究,为鹅生长性状遗传机理的相关研究奠定理论基础。

表8 产蛋相关通路及变异基因
在此基础上,通过数据库比对分析进一步筛选产蛋相关通路,发现在促性腺激素释放激素(GnRH)信号通路、催乳素(PRL)信号通路、催产素(OT)信号通路和孕酮介导的卵母细胞成熟通路均有Indel位点变异。GnRH作为HPG轴的关键信号分子与繁殖周期的变化呈相关性。产蛋性状是由多个相关基因共同表达和相互作用来调控的,并受多种外源因素影响[35-37]。常见的产蛋相关主要基因有促性腺激素释放激素(GnRH)[38]、促性腺激素抑制激素(GnIH)[39]、催乳素(PRL)[40]、促卵泡激素(FSH)[41]、雌激素受体(ESR)[42]等。张克山等[43]研究表明,在产蛋前期或高峰期,四川白鹅的GnRHmRNA表达量高于休产期,并指出在调控鹅产蛋性状中GnRH信号通路发挥重要作用。催产素通路在鹅的繁殖、就巢和产蛋等方面有重要作用。本课题组成功克隆了狮头鹅PRL基因并进行了原核表达,研究发现,狮头鹅与其他禽类PRL具有高度保守性[44]。催乳素通路是细胞生长和分化过程中调控鹅性成熟的主要通路,同时也是鹅产蛋性状的候选遗传标记基因之一[45]。PRL基因的多态性主要与产卵性状相关。催乳素通路在鹅卵巢发育和产卵过程中也发挥重要作用。孕酮介导的卵母细胞成熟通路对卵泡发育起调节作用。卵泡和排卵取决于促性腺激素的释放。在家禽生产中,孕酮的分泌与卵巢的生长和状态紧密相关。在排卵过程中,孕酮作为正反馈信号在排卵前触发GnRH/LH通路[46]。总之,本研究筛选出的产蛋相关变异通路和调节途径,以及进一步筛选出的与产蛋相关的候选基因,为研究狮头鹅繁殖性能提供有效基础和依据。
4 结 论
通过全基因组测序后注释分析Indel变异位点,发现大部分的Indel位点分布在内含子中,蛋白质编码区中分布的Indel位点最少;另外发现,Indel位点长度分布趋势基本一致,在CDS区的分布趋势和数量与参考基因组相似,非编码区的Indel变异数量多于编码区,这一结果符合生物学特性。比对测序得到的群体基因注释列表筛选出了产蛋相关变异基因,本研究突破了传统育种的局限性,有效地为低遗传力产蛋性状的遗传进展加快了步伐,提供研究鹅繁殖性能重要的分子标记理论数据,可以在实际选育工作中提高鹅养殖产业的经济效益。
