大头隆胸长蝽线粒体基因组测序及分析(半翅目:地长蝽科)
2021-03-30王月然王艳会
王月然,叶 飞,门 宇,王艳会,谢 强*
(1. 南开大学生命科学学院,天津 300071;2. 中山大学有害生物控制与资源利用国家重点实验室,广州 510275;3. 中山大学生命科学学院,广州 510275)
大头隆胸长蝽Eucosmetusincisus(Walker, 1872)隶属于半翅目Hemiptera异翅亚目Heteroptera蝽次目Pentatomomorpha长蝽总科Lygaeoidea地长蝽科Rhyparochromidae缢胸族Myodochini隆胸长蝽属Eucosmetus。缢胸族是地长蝽科中包含属最多的族。然而,目前尚无缢胸族相关的线粒体基因组报道。
缢胸族昆虫均为植食性,已知其中部分物种以禾本科植物未成熟的种子为食,是一类重要的经济害虫(郑乐怡和邹环光, 1981)。这类昆虫通过喙直接吸食植物种子,并且会携带产生有毒物质的真菌,间接影响植物的生长(Rosmanaetal., 2014; Aminetal., 2015)。大头隆胸长蝽是东洋界特有且在我国水稻种植地区广泛发生的缢胸族代表性昆虫,常于灌浆期集中在穗部吸食水稻种子,造成稻米苦涩、发黄、易碎,并使作物减产。已有文章报道此类昆虫影响水稻种植,并将其列入水稻害虫名录(宋慧英等, 1986)。
因此无论是基于其地理分布特殊性还是害虫防治,都有必要对大头隆胸长蝽开展线粒体基因组研究,这也可以为地长蝽科昆虫的分子系统发育研究积累更多数据。本研究扩增并测定了大头隆胸长蝽线粒体基因组编码区域的全部基因序列,并且分析了该线粒体基因组的核苷酸组成、密码子使用、组成偏差、rRNA二级结构等特征。除对大头隆胸长蝽线粒体基因组本身的分析外,还联合了蝽次目毛点类(包括蝽总科、红蝽总科、缘蝽总科和长蝽总科)(Tullgren, 1918; Schaefer, 1975; Schuh and Slater, 1995)中其它物种的同源序列,基于13种蛋白质编码基因构建系统发育树,对红蝽总科、缘蝽总科和长蝽总科间的系统发育关系进行了探讨。
1 材料与方法
1.1 标本采集
大头隆胸长蝽的成虫个体,在2018年7月采自于广西来宾市金秀县青山瀑布,标本浸泡在无水乙醇中储存,带回实验室后置于-20℃环境中保存。
1.2 DNA提取
通过解剖获得大头隆胸长蝽胸部肌肉组织,并用液氮迅速冷冻研磨,利用CTAB法(Reinekeetal., 2010)结合试剂盒法提取全基因组,-20℃环境中保存。
1.3 引物设计、PCR扩增和测序
利用前期设计并优化的通用引物扩增若干具有部分重叠区域的DNA片段(Lietal., 2013),扩增失败的DNA片段,根据已扩增的上下游片段的序列,利用Primer premier 5(Lalithaetal., 2000)设计特异性引物,得到互相重叠的线粒体基因的片段(表1)。使用TaKaRa LA DNA聚合酶进行PCR扩增,PCR反应过程:94℃预变性2 min,94℃变性30 s,引物最低扩增温度下退火45 s,72℃延伸1~3 min,共进行35个循环,72℃终延伸7 min。使用1%琼脂糖凝胶电泳检测PCR产物,若条带单一并符合预期大小,则送交测序服务公司测序(BGI, 广州; TSINGKE, 广州)。测序浓度没有达到要求的可通过增加模板量或者凝胶回收进行处理以提高浓度再测序。
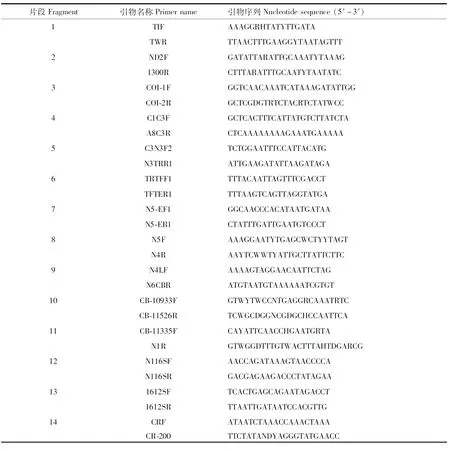
表1 本研究中使用的PCR引物
1.4 DNA序列拼接、注释及分析
通过在GenBank中进行BLAST搜索以初步判断所得序列属于目标类群,用DNA star v7.1中的SeqMan(Swindell and Plasterer, 1997)组件对序列进行拼接,得到大头隆胸长蝽的线粒体基因组序列。
tRNA基因通过Mitos WebServer(http://mitos.bioinf.uni-leipzig.de/index.py)(Berntetal., 2013)和tRNA scan-SE v2.0(http://lowelab. ucsc.edu/tRNAscan-SE/)(Lowe and Chan, 2016)这两种在线工具进行确认并预测二级结构。对于两种方法都无法检测到的tRNA基因,将其和已知亲缘关系较近的昆虫线粒体基因组的tRNA序列进行比对,确定位置和序列,再利用RNA structure v5.8(Mathews and Reuter, 2010)推测二级结构。
蛋白质编码基因利用NCBI中的开放阅读框查找工具ORF Finder (https://www.ncbi.nlm.nih. gov/orffinder/)进行初步注释,再将所得蛋白质编码基因序列与近缘物种的线粒体基因组相应序列进行比对,进一步确定各个蛋白质编码基因的边界,同时获取起始密码子及终止密码子。
核糖体RNA基因通常被认为处于两侧基因间的空缺区域(Boore, 2001; Cameron, 2014b),如tRNA-Leu(UAG)和tRNA-Val之间的序列界定为16S rRNA,tRNA-Val和控制区之间的序列界定为12S rRNA。核糖体RNA的二级结构参考黑腹果蝇Drosophilamelanogaster(双翅目:果蝇科)(Cannoneetal., 2002),白斑地长蝽Panaorusalbomaculatus(半翅目:地长蝽科)(Lietal., 2016a)以及豆突眼长蝽Chauliopsfallax(半翅目:束长蝽科)(Lietal., 2013)的二级结构进行预测,茎环结构的命名参考烟草天蛾Manducasexta(鳞翅目:天蛾科)(Cameron and Whiting, 2008)和意大利蜜蜂Apismellifera(膜翅目:蜜蜂科)(Gillespieetal., 2006)的线粒体rRNA基因。确定线粒体基因组内各个基因的位置后使用CGView Server (http://stothard.afns.ualberta.ca/cgview_server/index.html)(Grant and Stothard, 2008)在线平台绘制线粒体基因组的结构示意图。
线粒体基因组的核苷酸组成和蛋白质编码基因的密码子使用频率通过MEGA v7.0(Kumaretal., 2016)进行分析;计算各基因的核苷酸组成偏向性:AT-skew=(A-T)/(A+T)和GC-skew=(G-C)/(G+C)(Perna and Kocher, 1995)。
1.5 系统发育分析
选取蝽次目内21个物种的蛋白质编码基因序列分析长蝽总科的系统发育地位,其中长蝽总科的10个物种作为内群,蝽总科、红蝽总科、缘蝽总科的11个物种作为外群(表2)。除大头隆胸长蝽外的其它物种线粒体基因组均来自GenBank,按照物种名进行排序形成了13个蛋白质基因的数据集合,在MEGA v7.0中进行多重比对并手工校正,得到包含13个蛋白质编码基因全部三个位点的矩阵PCG123。使用矩阵PCG123通过MEGA v7.0(Kumaretal., 2016)获得去掉蛋白质编码基因第3位密码子的矩阵PCG12。
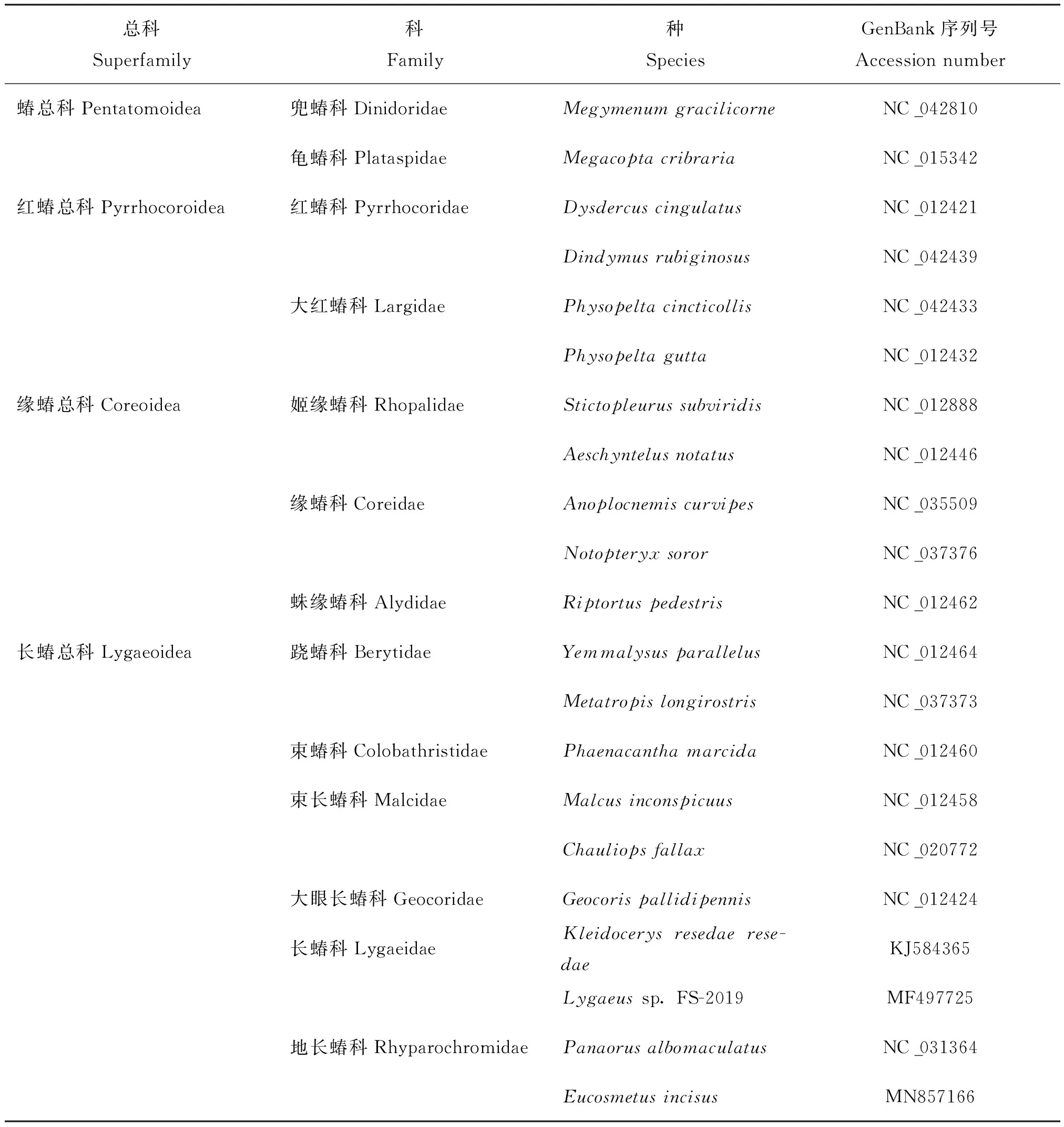
表2 本研究中所使用的类群信息
本研究采用贝叶斯分析(Bayesian inference)和最大似然法(maximum likelihood)重建系统发育树,核苷酸替换模型通过IQ-TREE v 1.0(Lametal., 2015)分析得到。利用MrBayes v3.12(Huelsenbeck and Ronquist, 2001)进行贝叶斯分析,共运行1千万代。每隔1 000代取一次样,舍去收敛之前的数据样本。利用RAxML v8.2.9(Stamatakis, 2006)进行最大似然法分析,自展检验(bootstrap)值设为1 000。
2 结果与分析
2.1 大头隆胸长蝽线粒体基因组结构
大头隆胸长蝽的线粒体基因组是双链闭合环状DNA大分子,GenBank序列号为MN857166。测得的长度为14 562 bp,由于控制区的特殊结构碱基组成,没有测得控制区的全部序列。已测得的序列包含一部分控制区和典型的37个基因,包括22个转运RNA基因,13个蛋白编码基因和2个核糖体RNA基因(图1, 表3)。大头隆胸长蝽线粒体基因组的基因组成和排列方式保守,与果蝇Drosophilayakuba(双翅目:果蝇科)和大多数蝽次目昆虫相同(Clary and Wolstenholme, 1985; Huaetal., 2008)。
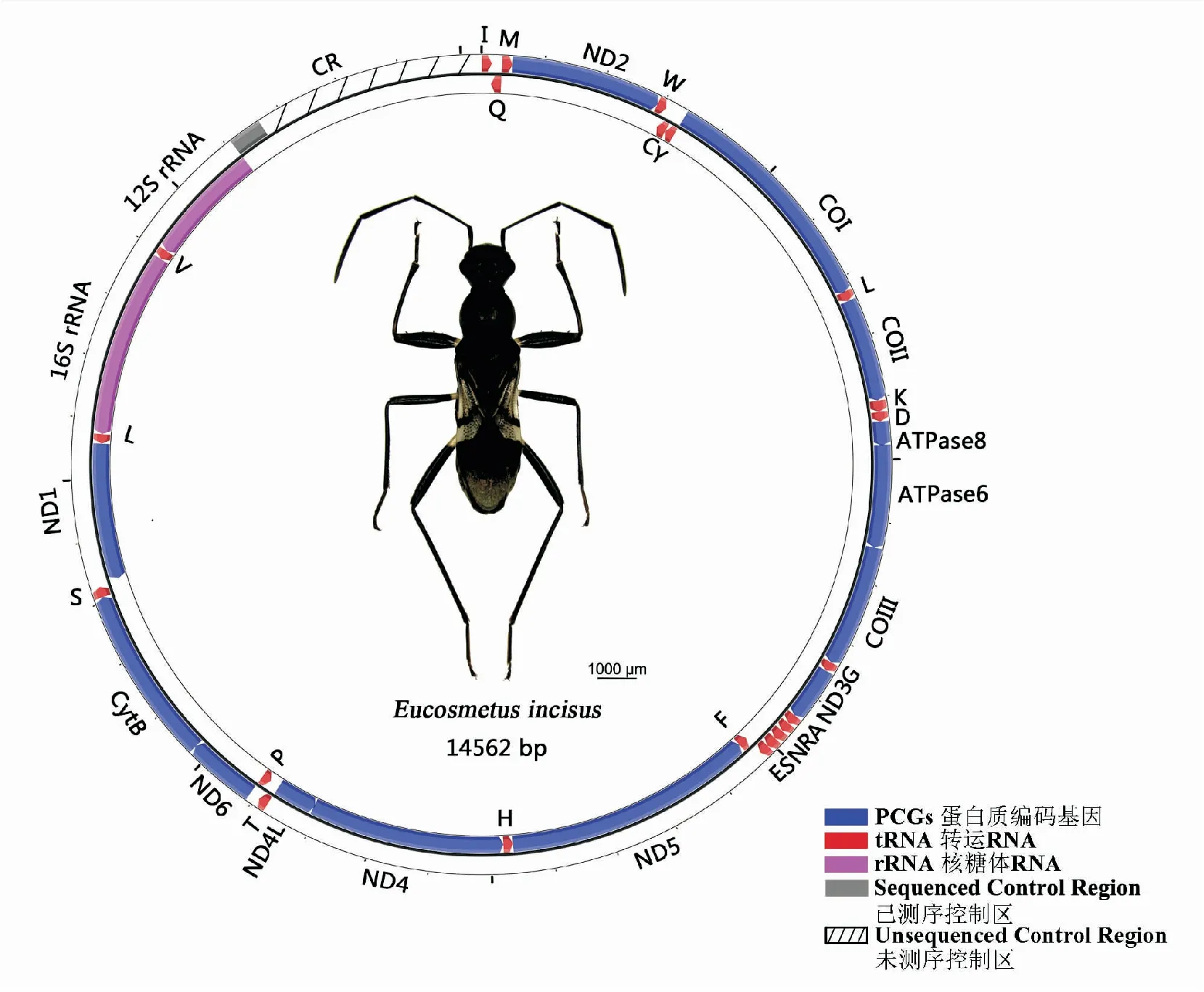
图1 大头隆胸长蝽线粒体基因组结构Fig.1 Mitochondrial genome map of Eucosmetus incisus注:箭头表示基因转录的方向。蓝色代表蛋白质编码基因,红色代表tRNA基因,紫色代表rRNA基因,灰色代表已测得控制区,斜线代表未测得的控制区。Note: Arrows indicated the orientation of gene transcription. PCGs were showed as blue arrows, tRNA genes as red arrows, rRNA genes as purple arrows, the sequenced control region as gray arrows and the unsequenced control region as slash.
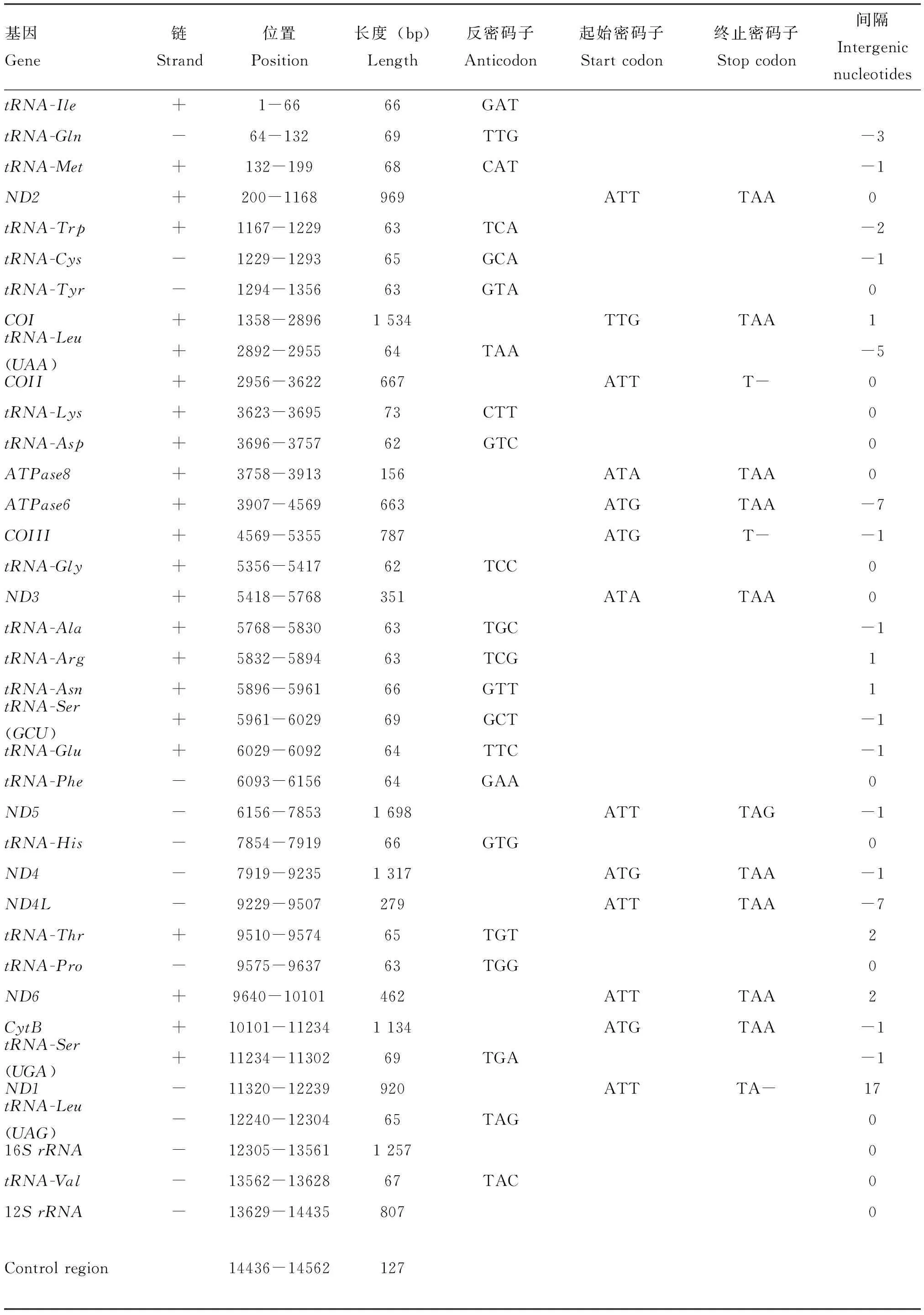
表3 大头隆胸长蝽线粒体基因组结构
大头隆胸长蝽线粒体基因组内发生基因重叠的区域共有15处,总长度为32 bp,大小从1~7 bp不等。最长2处基因重叠为7 bp,分别位于ATP8和ATP6,ND4和ND4L之间,并且这两段重叠序列互为反向互补序列(ATGATAA),这与Cameron(2014a)中报道的重叠序列相同。最短的10处基因重叠为1 bp,分别位于tRNA-Gln和tRNA-Met、tRNA-Trp和tRNA-Cys、ATP6和COIII、ND3和tRNA-Ala、tRNA-Asn和tRNA-Ser(GCU)、tRNA-Ser(GCU)和tRNA-Glu、tRNA-Phe和ND5、ND4和ND4L、ND6和CytB以及CytB和tRNA-Ser(UGA)之间。同时观察到基因间隔区域有6处,总长度为34 bp,大小从1~17 bp不等,最长的基因间隔发生在tRNA-Ser(UGA)和ND1之间,为17 bp。37个编码基因中,有9个蛋白质编码基因和14个tRNA基因由J链编码,4个蛋白质编码基因和8个tRNA基因以及2个rRNA基因由N链编码。
2.2 蛋白质编码基因
大头隆胸长蝽的线粒体基因组包含13个蛋白质编码基因,全长共10 937 bp。除起始和终止子之外,共发现3 622个密码子。其中ND1、ND4、ND4L、ND5位于N链,而其余9个蛋白质编码基因位于J链。A+T含量为77.9%,G+C含量为22.1%。除了COI使用TTG作为起始密码子以外,其它所有蛋白质编码基因都以ATN作为起始密码子。其中,ND2、COII、ND5、ND4L、ND6和ND1使用ATT作为起始密码子,ATP6、COII、ND4和CytB使用ATG作为起始密码子,ATP8和ND3使用ATA作为起始密码子。这种非传统的COI起始密码子在蝽次目昆虫中非常常见。终止密码子方面,有10个蛋白质编码基因使用最常见的三联体密码(TAA和TAG)作为为终止密码子,其中,ND5使用TAG作为终止密码子,其余9个使用TAA作为终止密码子。另外,ND1使用TA作为终止密码子,COII和COIII的则使用单个T作为终止密码子(表4)。以TA或T作为终止密码子的现象在昆虫线粒体基因组中很常见,有研究推测完全终止密码子TAA可以通过转录后多腺苷酸化产生(Ojalaetal., 1981)。

表4 大头隆胸长蝽线粒体基因组中蛋白质编码基因的起始密码子和终止密码子
2.3 tRNA
大头隆胸长蝽线粒体基因组包含典型的22个tRNA基因,其中有14个位于J链上,8个位于N链上,长度范围在62 bp(tRNA-Asp和tRNA-Gly)到73 bp(tRNA-Lys)之间。其tRNA二级结构比较保守,除tRNA-His之外的所有tRNA都能够折叠成经典的三叶草的二级结构(图2)。根据Mitos WebServer的分析,在tRNA-His的二级结构中TψC臂的“环”结构缺失,仅有“茎”结构,这一现象在巨红蝽Macrocheraiagrandisgrandis(半翅目:大红蝽科)(Menetal., 2019)中同样有报道。
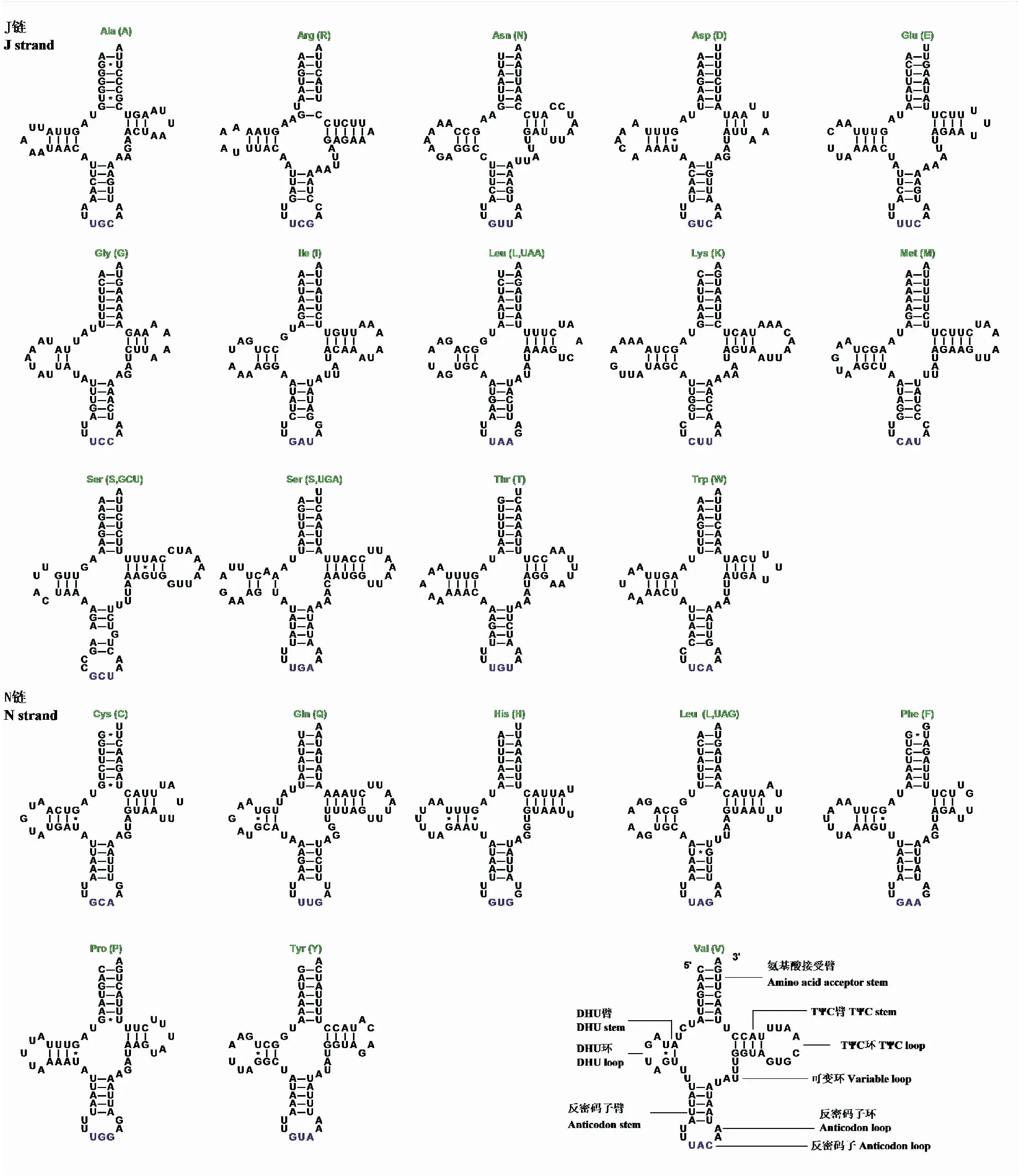
图2 大头隆胸长蝽线粒体基因组中tRNA结构Fig. 2 Predicted secondary structures of tRNAs in Eucosmetus incisus mitochondrial genome注:tRNAs使用相应的氨基酸缩写表示。Watson-Crick经典配对用短线表示,GU配对用星号表示,其他非经典配对用空心圆表示。Note: The tRNAs were labeled with the abbreviations of their corresponding amino acids. Inferred Watson-Crick bonds were illustrated by lines, GU bonds by asterisk and the other non-Watson-Crick interactions were represented by hollow circles.
大头隆胸长蝽的tRNA的氨基酸接受臂和反密码子环具有极低的变异性,长度都为7 bp。反密码子臂长度比较保守,除了tRNA-Arg和tRNA-Glu为4 bp外,其余tRNA的反密码子臂长度都为5 bp。DHU臂长度为2~4 bp,TψC臂长度为3~5 bp。变化量最大的是DHU环和TψC环,分别为4~8 bp和1~8 bp (表5)。
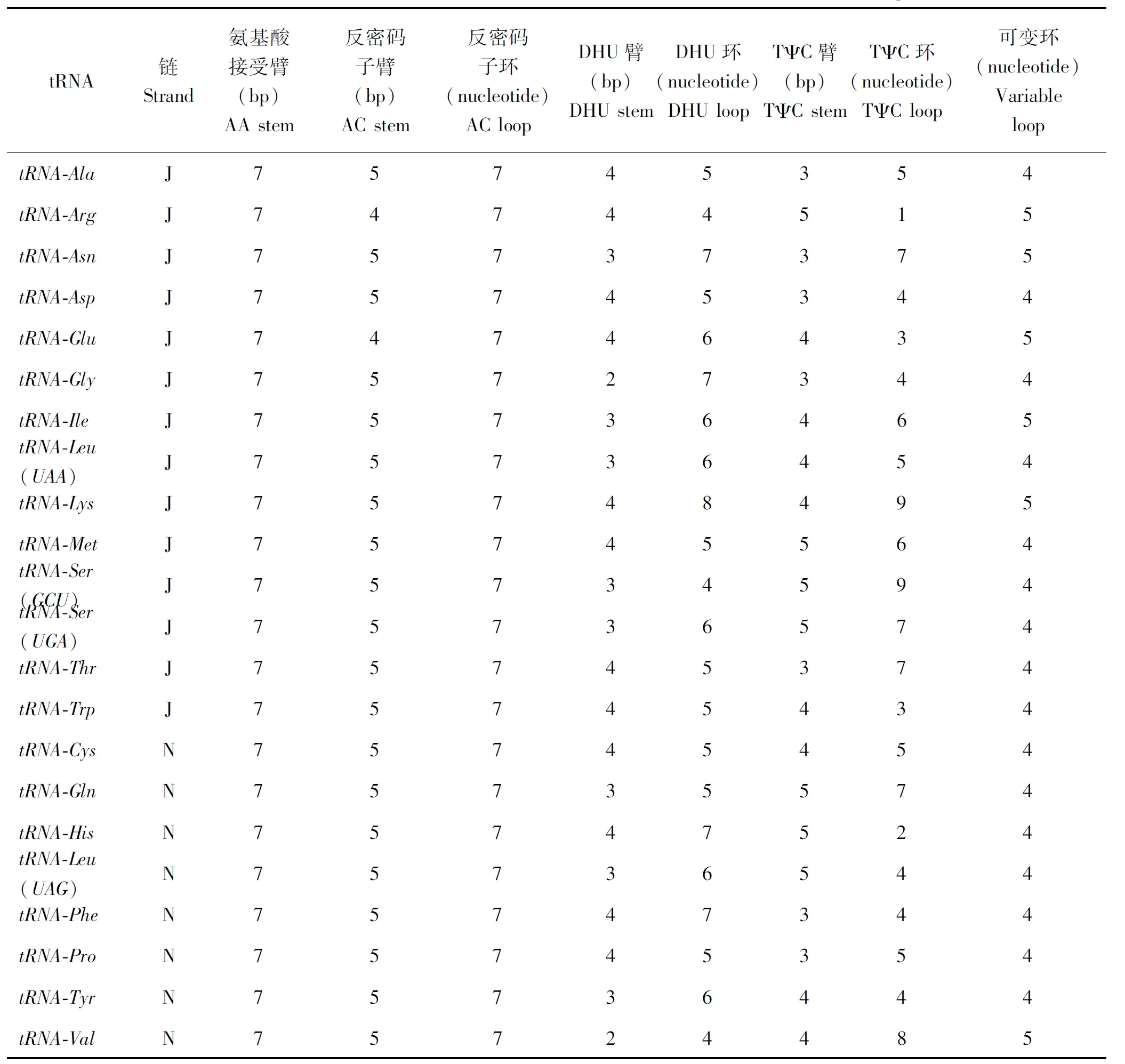
表5 大头隆胸长蝽线粒体基因组中22个tRNA的核苷酸分布情况
此外,共发现17处非Watson-Crick碱基配对存在于大头隆胸长蝽线粒体tRNA基因二级结构中,且都为G=U配对,其中有15个集中在氨基酸接受臂和DHC臂上,剩余2个分别位于反密码子臂和TψC臂上(表6)。
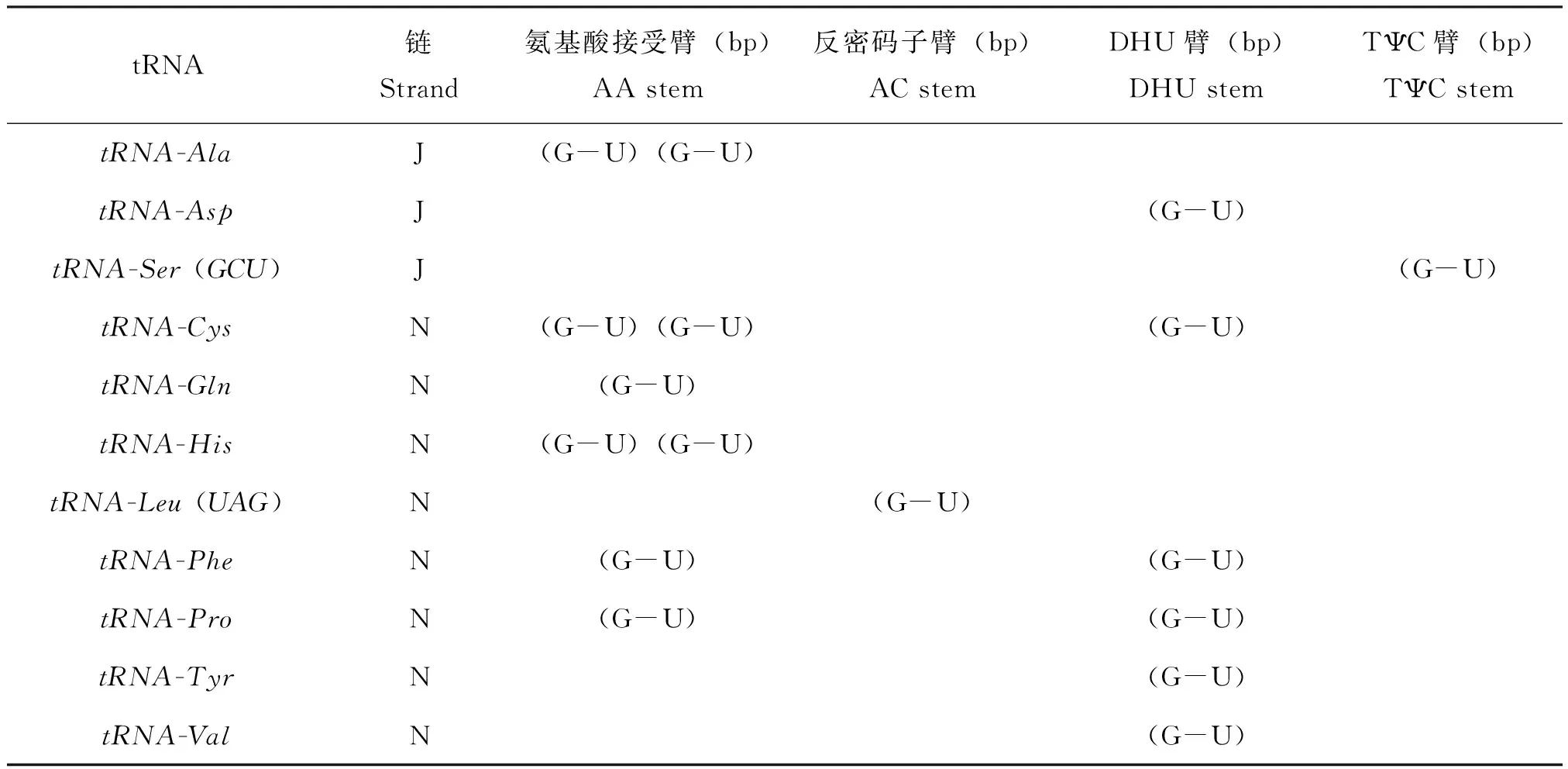
表6 大头隆胸长蝽线粒体基因组tRNA中非典型的碱基对
2.4 rRNA
大头隆胸长蝽的线粒体基因组中的16S rRNA基因长1 257 bp,位于tRNA-Leu(UAG)和tRNA-Val之间,其二级结构包含6个结构域(节肢动物中结构域III缺失)和45个茎环结构(图3)。和长蝽总科内的其它物种相比,大头隆胸长蝽在H991、H1196、H235、H2735以及H183到tRNA-Val之间的茎环结构存在较大变异。而H1775、H2064、H2507等二级结构则和长蝽总科其它物种相比,无论在序列还是在二级结构上都十分保守。这与已报道的长蝽总科线粒体基因组内16S rRNA的结构域IV和V比结构域I、II、VI更保守的研究结果一致(Lietal., 2013; 2016ab)。
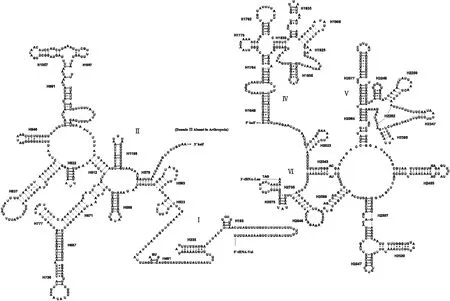
图3 大头隆胸长蝽线粒体基因组中的16S rRNA的二级结构Fig.3 Predicted secondary structure of the 16S rRNA in Eucosmetus incisus mitochondrial genome
12S rRNA基因长807 bp,位于tRNA-Val和控制区之间,其二级结构包括3个结构域和27个茎环结构(图4),发现相比结构域I和II,结构域III更保守。特别是在结构域II中,从H567到H769存在一个拉链状二级结构,大头隆胸长蝽的这部分结构与长蝽总科其它物种相比,不仅在序列长度上存在差异,而且在核苷酸组成上也存在较高的碱基替换,使得这部分茎和环的长度各有不同,但整体上仍保持一个拉链状结构。rRNA的同源性更多的体现在二级结构保守性上,而不是具有某一段保守的序列,说明rRNA结构构成上的生物学意义要大于它的序列组成(陈国忠等, 2005)。
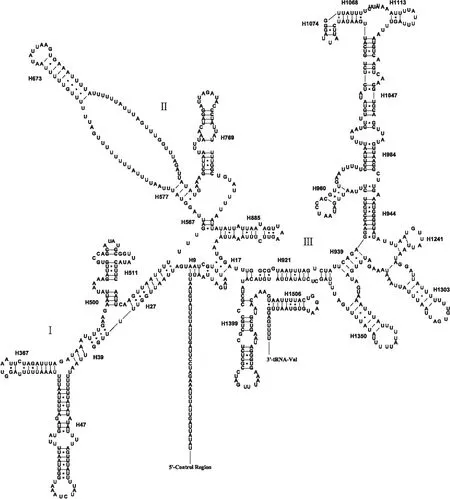
图4 大头隆胸长蝽线粒体基因组中12S rRNA二级结构Fig.4 Predicted secondary structure of the 12S rRNA in Eucosmetus incisus mitochondrial genome
2.5 核苷酸组成和密码子使用
在所测得的大头隆胸长蝽线粒体基因组中,ATCG 4种核苷酸含量分别为45.9%、32.4%、13%和8.6%。AT偏斜率(AT-skew)大小为17%,GC偏斜率(GC-skew)为-20%,具有明显的AT偏向性。这与大部分其它蝽次目昆虫类似,AT-skew基本为正值,即整体上A的含量大于T,GC-skew基本为负值,表明C的含量大于G。大头隆胸长蝽线粒体基因组总体上具有较高的A+T含量,为78.3%,在蛋白质编码基因中为77.9%,在tRNA基因中为79.1%,rRNA基因的A+T含量最高,为80%。在蛋白质编码基因中,A+T含量最高的基因是ND6(87.8%),含量最低的基因是COI(72.1%)。总基因组、蛋白质编码基因J链表现为AT偏移和CG偏移,总蛋白质编码基因及其N链、总rRNA编码基因、tRNA编码基因N链则刚好相反,表现为TA偏移和GC偏移,总tRNA编码基因及其J链表现为AT偏移和GC偏移(表7)。由此可见,线粒体基因组中核苷酸组成在不同链间是不对称的。

表7 大头隆胸长蝽线粒体基因组核苷酸组成分析
值得注意的是,由N链编码的蛋白质基因和rRNA基因都是TA偏移和GC偏移,而除COI之外所有由J链编码的蛋白质基因都刚好相反,都是AT偏移和CG偏移,COI为TA偏移和CG偏移(图5)。昆虫线粒体基因组J链、N链上蛋白质编码基因核苷酸组成偏向性刚好相反的情况在C.fallax(Lietal., 2013)也有发现,有报道称GC-skew和AT-skew的值因复制起点的方向和密码子位置的变化而改变(Hassaninetal., 2005; Weietal., 2010)。为了深入了解这种现象的机制,需要进行更多关于线粒体基因组序列和功能的研究工作。
核苷酸的AT偏向性也反映在密码子使用中,蛋白质编码基因密码子的使用表现出极大的不均质性,其中密码第三位的A+T含量最高,为86.5%,第一位和第二位次之,分别为74.2%和73%。密码子ATT(371)、TTA(356)、ATA(330)以及TTT(309)是使用频率最高的四种密码子,全部由A、T构成,分别转运氨基酸Ile、Leu、Met和Phe。对于大多数氨基酸来说,使用最频繁的密码子是NNA和NNU,而不是与tRNA反密码子严格配对的密码子(图6)。如甲硫氨酸Met对应的密码子是AUA和AUG,使用次数分别为330和29,其中AUG是与tRNA-Met的反密码子严格配对的密码子。
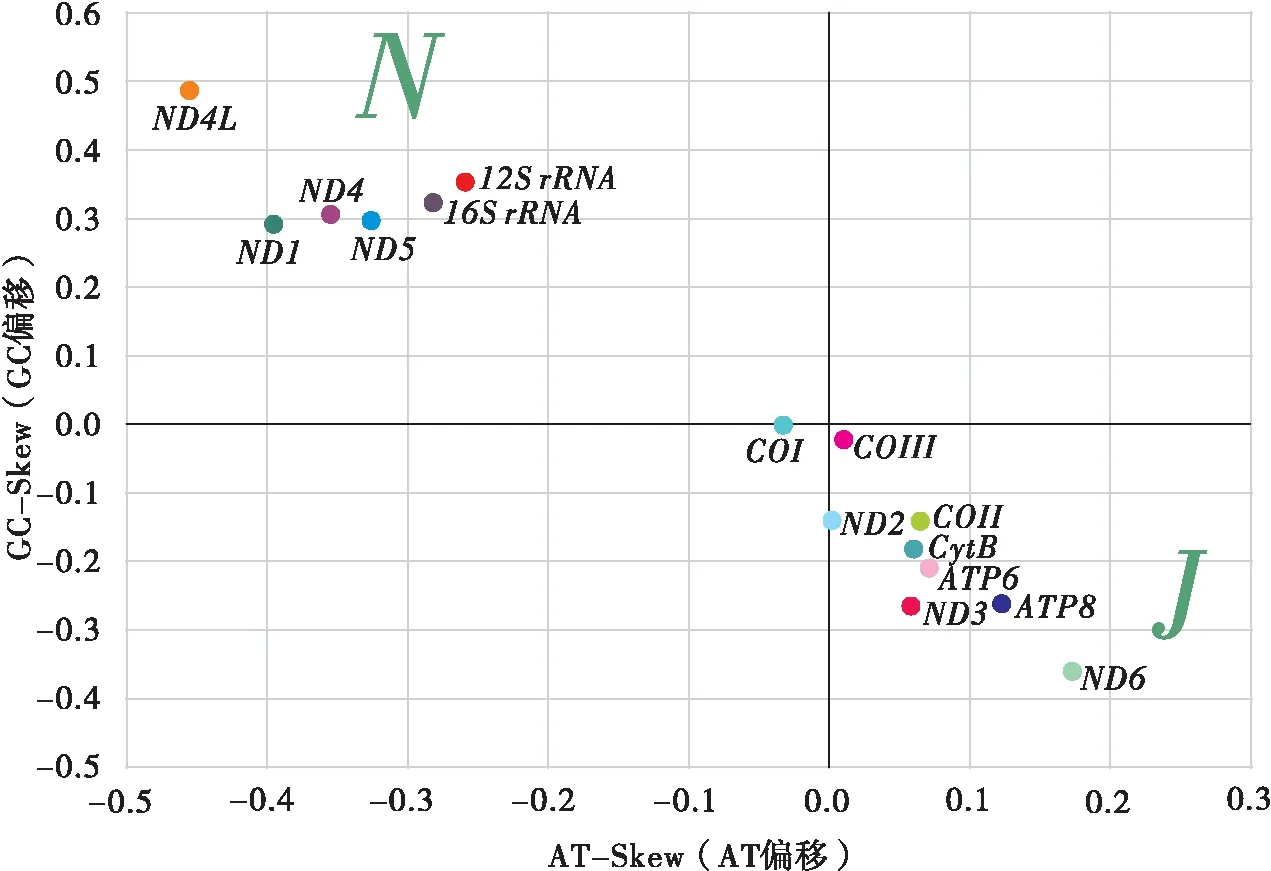
图5 大头隆胸长蝽线粒体基因组的AT偏移和GC偏移Fig.5 AT-skews and GC-skews of Eucosmetus incisus mitochondrial genome注:13个蛋白编码基因和2个rRNA 基因用不同颜色圆环表示。Note: 13 protein coding genes (PCGs)and 2 rRNAs were represented in different color circles. Letter J meant J-strand, N meant N-strand.
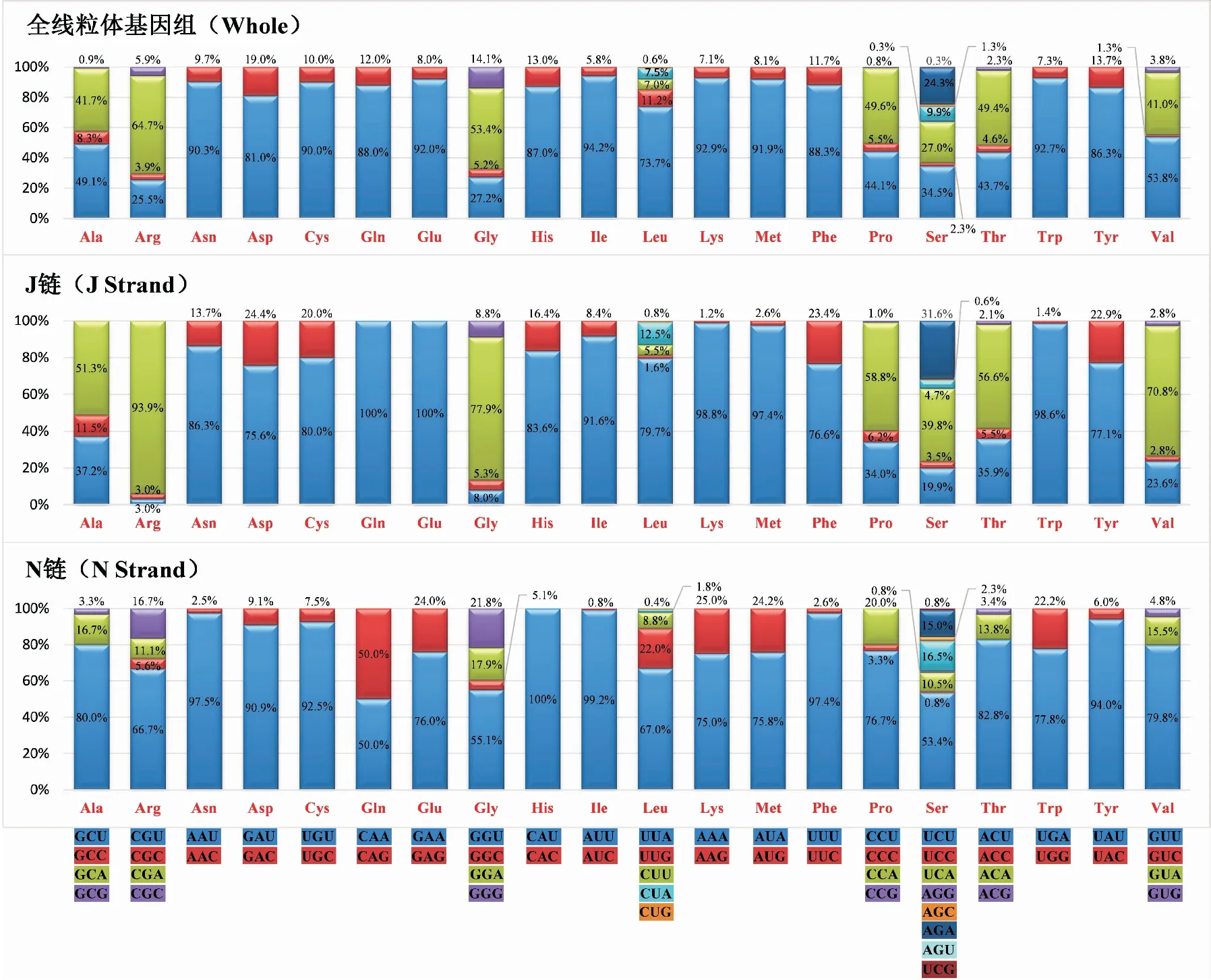
图6 大头隆胸长蝽线粒体基因组中每个氨基酸的同义密码子使用率Fig.6 Percentage of synonymous codon usage of each amino acid in the Eucosmetus incisus mitochondrial genome注:X轴上为密码子家族。Note: Codon families are provided on the x-axis.
2.6 系统发育
本研究选取4个总科共21个物种,根据含有蛋白质编码基因全部123位密码子的矩阵PCG123,和去除第三位密码子的矩阵PCG12,分别进行最大似然法和贝叶斯分析,得到了不同的系统发育结果。其中,通过矩阵PCG123得到的两个系统发育树完全不相同,并且与客观事实不尽相符,而通过矩阵PCG12分析得到具有相同拓扑结构的系统发育树。通过对比有无密码子第3位碱基得到的系统发育结果的拓扑结构是否一致,发现第3位密码子的碱基组成异质性较高,容易形成核苷酸替代饱和,严重影响树形结构,表明密码子第3位的存在与否对于系统发育研究非常重要。
根据矩阵PCG12得到系统发育树:{[(长蝽总科+缘蝽总科)+红蝽总科]+蝽总科},这个结果很好地支持了长蝽总科的单系性,在总科关系方面,长蝽总科和缘蝽总科亲缘关系更近(图7)。这与Xieetal.(2005)和Huaetal.(2008)根据18S rRNA和线粒体基因组数据得到的树形一致。
从贝叶斯分析来看,关于长蝽总科、缘蝽总科、红蝽总科、蝽总科系统发育分支节点处的后验概率均为100%,而在长蝽总科内部,(地长蝽科+长蝽科)与束长蝽的分支节点处后验概率为85%,小于95%这一通常认为的较为可靠的阈值(Leaché and Reeder, 2002; Suzukietal., 2002)。从自展检验的值来看,4个总科的分支节点支持率均高于70%这一通常认为的可靠的阈值(Huelsenbecketetal., 1993),长蝽总科内部部分节点支持率偏低。

图7 基于线粒体基因组蛋白质编码基因所得到的系统发育树Fig.7 Phylogenetic tree inferred from the sequences of PCGs in mitochondrial genome注:贝叶斯和最大似然法分析具有一致的拓扑结构。节点处上方的数字代表贝叶斯后验概率,下方代表bootstrap值。Note: Bayesian analyses and Maximum likelihood showed the same topology. Numbers at the nodes were Bayesian posterior probabilities (up)and Maximum likelihood bootstrap values (down).
这说明各总科间的系统发育关系总的来讲得到了解析,但还需要进一步去验证;而长蝽总科内部关系仍处于未解析的状态。造成这种现象的原因可能有两个,一是长蝽总科内部取样不充分,二是仅根据线粒体基因组进行系统发育推断对于类群选取比较敏感。
3 结论与讨论
本研究测定了大头隆胸长蝽编码区域的基因序列,并分析了该物种线粒体基因组特征,这是对缢胸族昆虫线粒体基因组序列的首次报道。大头隆胸长蝽线粒体基因组已测得部分大小为14 562 bp,包含标准的13个蛋白编码基因,22个转运RNA基因,2个核糖体RNA基因以及部分控制区。其线粒体基因排列顺序同亚库巴果蝇Drosophilayakuba和大多数蝽次目昆虫排列顺序相同。线粒体基因组序列存在基因重叠和基因间隔现象,并发现有两个长度为7 bp的基因重叠片段互为反向互补序列(ATGATAA),一个位于ATP8和ATP6之间,另一个位于ND4和ND4L之间。蝽次目昆虫中大部分物种在tRNA-Ser(UGA)和ND1之间存在长度不等的非编码区。
线粒体基因组中核苷酸组成在不同链间不对称,大头隆胸长蝽线粒体核苷酸组成表现出了很高的AT偏向性,13个蛋白编码基因和2个核糖体RNA中,由N链编码的基因都是TA偏移和GC偏移,而除COI之外所有由J链编码的基因都刚好相反,都是AT偏移和CG偏移,COI为TA偏移和CG偏移,该观察结果很可能归因于复制方向的不对称。此外,核苷酸AT偏向性也反映在密码子的使用中,使用最频繁的密码子均由AT组成,且多数并不与tRNA反密码子严格配对。
非传统起始密码子TTG在蝽次目昆虫中广泛存在,通常发生在蛋白质编码基因COI中,以单个T作为终止密码子的现象多发生在COII和COIII中,而以TA作为终止密码子的情况仅随机出现在少部分基因中。大头隆胸长蝽线粒体基因中,除tRNA-His因缺少TψC环,不能正常折叠外,其它21个tRNA均能折叠成经典三叶草结构。大头隆胸长蝽的rRNA二级结构在一些非保守结构域与长蝽总科其它物种相比,存在长度差异和较高的碱基替换,但并不影响各物种具有大致相同的二级结构,说明rRNA结构构成上的生物学意义要大于它的序列组成。
长期以来,长蝽总科、红蝽总科、缘蝽总科三者间的系统发育关系一直存在争议,且至今尚无较为统一的意见。本研究的系统发育结果是基于蛋白质编码基因的第1、2位密码子构成的数据集得到的,系统发育结果很好地确认了长蝽总科的单系性,并且支持(((长蝽总科+缘蝽总科)+红蝽总科)+蝽总科)这一关系。而长蝽总科内部关系仍处于未解析状态,可能原因是长蝽总科内部取样不充分,另外仅根据线粒体基因组进行系统发育推断对于类群选取比较敏感,因此未来需要尽可能选取完整的科级分类单元类群,并结合更多分子标记(核基因分子标记)和形态学来较为全面的进行探讨和研究。大头隆胸长蝽线粒体基因组的测序不仅为确定长蝽总科、缘蝽总科和红蝽总科的系统发育关系提供更多证据,还为将来开展地长蝽科缢胸族昆虫相关的分子系统发育研究初步提供了基础数据。
