基于高通量测序的菜头肾叶绿体基因组的组装及序列分析
2021-03-17余佳兴秦宇雯任仙樱高海东吴志刚姜程曦
赵 祺,余佳兴,秦宇雯,任仙樱,高海东,吴志刚,姜程曦*
基于高通量测序的菜头肾叶绿体基因组的组装及序列分析
赵 祺1, 2,余佳兴1#,秦宇雯1, 2,任仙樱1, 3,高海东4,吴志刚1*,姜程曦1, 3*
1. 温州医科大学药学院,浙江 温州 325035 2. 浙江省生物医药协同创新中心,浙江 温州 325035 3. 安徽省九华山佛教医药研究所,安徽 池州 242700 4. 南京集思慧远生物科技有限公司,江苏 南京 210033
以药用植物菜头肾为材料,对其叶绿体基因组进行组装和序列分析,为进一步开展菜头肾遗传与鉴定学研究奠定基础。采用高通量测序技术首次对菜头肾叶绿体基因组进行测序,以板蓝叶绿体基因组为参考,进行特征分析和聚类关系研究。生物信息学分析表明,菜头肾叶绿体基因组总长为144 713 bp,具有典型的被子植物叶绿体基因组环状四分体结构,GC含量为38.35%;共注释得到129个基因,包括84个蛋白编码基因,37个tRNA基因和8个rRNA基因;蛋白编码基因对应的密码子偏好使用A/T碱基。系统发育研究表明,菜头肾与板蓝亲缘关系最近,提示它们在进化上具有共线性。建立了适于菜头肾植物完整叶绿体基因组组装及其特征分析的方法,为菜头肾的鉴定、系统发育研究奠定了基础。
菜头肾;板蓝;叶绿体;基因组;高通量测序
菜头肾C. Ling来源于爵床科马蓝亚族黄猄草属多年生草本植物[1],别名土太子参、肉根马蓝,是温州民间著名补肾方剂“七肾汤”的君药,在临床上可与其他药物配伍或单独使用[2-3]。由于菜头肾具有显著的补肾功能,其在民间广泛使用,并已实现了人工种植[4]。目前,菜头肾的相关研究主要集中于栽培、资源调查[5]、化学成分鉴定[6-7]等方面研究,有关该植物叶绿体基因组组装、基因组成和序列分析等未见报道。
叶绿体是植物细胞进行光合作用的重要场所,拥有一套完整的基因组,可进行自主遗传。20世纪60年代初,在烟草L.[8]、地钱L.[9]中首次报道了叶绿体全序列。由于叶绿体基因组包含大量遗传信息,不同物种之间具有良好的共线性,被广泛应用于玉米L.[10]、水稻L.[11]、大豆(Linn.) Merr.[12]等植物分子进化及系统发育的研究,并在药用植物的遗传转化、基因工程和分子育种等方面发挥着重要作用[13-17]。鉴于此,本研究利用高通量测序技术对菜头肾叶绿体基因组进行测序,分析组装后的叶绿体基因组结构、SSR(Simple Sequence Repeat,简单重复序列)位点、IR(inverted repeat,反向重复序列)区间特征比较及近缘物种亲缘关系,为菜头肾的鉴定、系统发育研究奠定基础。
1 材料与方法
1.1 材料
菜头肾C. Ling采自温州市瓯海区大罗山(27°51′N,120°40′E),该地属南亚热带海洋性季风气候区,年均气温17 ℃,无霜期216~317 d,年均降水量1611 mm,年均相对湿度79%。经温州医科大学中药学教研室生药学专家鲍康德副教授鉴定所有样品均来源于爵床科马蓝亚族黄猄草属菜头肾,各样品均采集新鲜幼嫩的叶片,液氮速冻下研磨,−80 ℃冰箱保存备用。
1.2 方法
1.2.1 DNA基因提取 全基因组DNA采用植物基因组DNA提取试剂盒(TIAN⁃GEN,DP305)提取样本的总DNA。
1.2.2 测序 总DNA样品经南京集思慧远生物科技有限公司进行测序,得到测序reads、质控。采用Illumina HiSeq PE150双末端测序策略进行建库测序,建库类型为350 bp DNA小片段文库,测序深度为10倍。
1.2.3 叶绿体基因组组装 通过Illumina HiSeq PE150测序平台对样品进行测序,用MITObim 1.8[18]组装软件进行组装。由于NCBI数据库中暂未收录菜头肾同属植物参考序列,因此选用同为马蓝亚族的板蓝(Nees) Kuntze(NC_037485.1)为参考序列。然后用Gap Closer[19]1.12软件(http://soap.genomics.org.cn/ soapdenovo.html)对组装结果进行内洞修补,最后去除冗余的短序列得到最后的组装结果。
1.2.4 基因组特征分析 先用GeneMarkS (Version 4.17)[20]软件对菜头肾叶绿体基因组进行编码基因预测,tRNAscan-SE软件[21](Version 1.3.1)、rRNAmmer软件[22](Version 1.2)分别对tRNA和rRNA基因进行预测,用OGDRAW软件[23]呈现叶绿体基因组序列图,再用MISAI.0[24]软件分析菜头肾叶绿体全基因组SSR位点,Find repeats工具获得菜头肾、穿心莲等6种基因组的IR序列、大单拷贝区(LSC)和小单拷贝区(SSC),MAFFT v7.394[25]软件进行序列比对,最后使用PhyML v3.0[26]软件基于最大似然法构建系统发育树。
2 结果与分析
2.1 全基因组测序与叶绿体基因组组装
基于高通量测序,发现菜头肾全基因组总长为144 713 bp,碱基为5 196 698 700 bp,具一个典型的4分区域结构,包含4个区段:LSC为92 026 bp、SSC为17 825 bp、2段反向互补重复的IR区(IRA和IRB)17 431 bp。GC含量为38.35%,有效数据20值、30值分别96.58%和90.91%。IR、LSC和SSC区域的GC值存在一定的差异,IR区域的GC值最高,为45.47%;LSC次之,36.49%;SSC区域的GC值最小,仅32.34%(表1)。值得注意的是,部分基因在SSC和IRB交界处重复(图l)。
2.2 叶绿体基因组结构基本特征
菜头肾叶绿体基因组共注释129个基因,包括84个蛋白编码基因,37个tRNA基因和8个rRNA基因。根据其功能可以把它们分为4大类:分别是光合作用相关基因、表达相关基因、未知功能基因,以及成熟酶基因()、囊膜蛋白基因()等其他基因。在这些基因中含有24个内含子和17个双拷贝基因(表2),包括1个光系统亚基()、4个rRNA基因(、、、)、8个tRNA基因(、、、、、、、)、1个NADH脱氢酶亚基基因()、2 个核糖体蛋白大小亚基编码基因(、)及1个未知功能蛋白基因()。值得注意的是,在菜头肾叶绿体基因组中存在一些未知功能的基因,如、、、,还需要做进一步研究才能确定其功能。在SSC区共有12个CDS和1个tRNA基因()存在。然而绝大多数的基因存在于LSC区,有59个CDS和22个tRNA 基因(表2),其中内含子最长,其片段大小为2450 bp,包含基因。
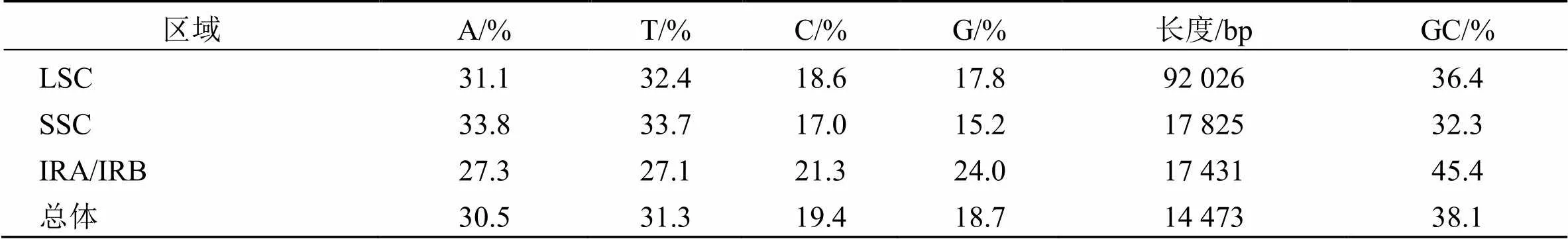
表1 菜头肾叶绿体基因组碱基组成

图1 菜头肾叶绿体基因组
2.3 SSR分析及爵床科IR区边界比较分析
在菜头肾叶绿体全基因组中,共搜索184个符合条件的SSR位点(表3),多分布于LSC区,其中包括92个单核苷酸重复基元,10个二核苷酸重复基元,67个三核苷酸重复基元,9个四核苷酸重复基元,4个五核苷酸重复基元和2个六核苷酸重复基元。在所有SSR中,最多重复基元是A/T,然后依次是AT/TA、TTG/TTA和ATA/ATA。由此可以看出,A/T基元占优势,具有碱基偏好性。这与菜头肾叶绿体全基因组富含AT(含量为61.65%)一致,这种偏好性可能是由于AT比GC更容易解链有关。
通过与爵床亚科板蓝、穿心莲(Burm. F.) Nees、黄花恋岩花(Lévl.) J. R. I. Wood、恋岩花Nees、鳄嘴花(Burm. f.) Lindau 4个属5个物种的叶绿体基因组IR区边界进行比较,发现这6种植物叶绿体基因组长度144 133~152 672 bp,基因具有相对保守性,从大到小依次为黄花恋岩花、恋岩花、穿心莲、鳄嘴花、菜头肾、板蓝。其中板蓝与菜头肾基因结构与种类最相似,与基因都有2份拷贝,基因绝大部分在IRA与IRB区内,板蓝少部分基因(约379 bp)位于LSC/IRA边界,菜头肾基因全部位于IRA区内;基因分别位于IRB/SSC和SSC/IRA边界,这可能与 IR/SSC边界的扩张有关(图2),位于SSC/IRA边界的基因长度为5496 bp和5514 bp,为正常基因,但位于IRB/SSC边界的基因全长仅为840 bp和834 bp,它的3’端发生缺失,该为一个假基因。穿心莲、鳄嘴花等其他物种没有基因,鳄嘴花IR区也没有基因,基因差异最大。

表2 菜头肾叶绿体基因组编码的基因
*具有1个内含子的基因**具有2个内含子的基因△多拷贝基因#假基因
*indicate one intron in the gene**indicate two introns in the gene△multiple copy gene#pseudogene.

表3 菜头肾叶绿体基因组中SSR位点类型及数量

图2 爵床科6个物种叶绿体全基因组IR边界的比较示意图
2.4 基于叶绿体基因组全序列的聚类分析
叶绿体基因组对系统发育研究具有重要意义[27]。为了鉴定菜头肾在爵床科中的进化位置,将测得的菜头肾叶绿体基因组序列与NCBI网站下载的其他10个物种叶绿体全基因组序列构建系统发育树。由于目前暂无黄猄草属植物叶绿体基因组GenBank,且爵床科植物叶绿体基因组研究有限,在此选取了在分类上与菜头肾同一亚族的板蓝,同一族的黄花恋岩花、黄花假杜鹃L.等4个物种,同一亚科的鳄嘴花、穿心莲、孩儿草(L.) Nees 3个物种,同一科的老鼠筋L. Sp.和小花老鼠筋Vahl,及不同科的日本紫茎Sieb. et Zucc.。结果显示(图3),不同科物种与爵床科距离最远,说明亲缘关系较远,此与《中国植物志》植物分类结果相一致[28];包括菜头肾在内的11个爵床科物种集为一支,属间关系比较清晰,支持率为100%;菜头肾与板蓝组成的小分支是姐妹关系,说明其亲缘关系密切,此与《中国植物志》植物分类结果一致,暗示它们在进化上具有共线性。

图3 基于叶绿体全基因组的12个物种的最大似然法(ML)聚类结果
3 讨论
菜头肾是温州民间特色中草药,主要作为验方“七肾汤”组方之一,根据临床病情的不同,可调整药物剂量,当肝炎迁延期、慢性肾炎腰痛时,重用菜头肾[29-30]。此外,菜头肾曾被《中国物种红色名录》列为极危(CR)等级[31],因而具有重要的保护、研究和开发利用价值。本研究利用第2代测序技术,对爵床科植物菜头肾进行了叶绿体基因组测序,获得其全长144 713 bp,注释得到129个基因;鉴定184个SSR 位点,大多数位点具有A/T碱基偏好性。系统进化分析表明菜头肾与已报道的同一亚族植物板蓝的亲缘关系最近,暗示它们在进化上具有共线性。
黄猄草属Bremek. 植物全世界共有10种,我国有7~9种。目前,黄猄草属植物遗传背景工作研究有限,本实验首次报道该属物种叶绿体基因组序列,不仅可以丰富叶绿体基因组序列的数量,还可以为研究菜头肾的鉴定、系统发育奠定基础。然而,与爵床科其它属所具有的物种多样性水平相比,基础研究工作有待加强。
通过对比分析,菜头肾与已发表的爵床科植物叶绿体基因组GC含量相似,基因构成和次序一致,体现了叶绿体基因组结构稳定,总体进化速率较低[32-34]。因为叶绿体基因组在总体上的保守性,在利用叶绿体基因组信息时,通常会筛选其中的高变区如、、等常用的叶绿体片段。值得注意的是,在该基因组中存在一些未知功能的基因,如、、和,还需要做进一步研究才能确定其功能。
SSR广泛存在于原核生物和真核生物基因中,具有共显性:本研究经分析鉴定出184个SSR,多分布于LSC区,主要由碱基A和T组成,这与爵床属叶绿体基因SSR分析结果类似[35-36]。SSR分布还具有物种特异性:二至六核苷酸重复类型SSR 位点中,菜头肾转录组以三核苷酸重复为主,其优势重复基元类型是TTG/TTA和ATA/ATA,共占比22.39%;不同属植物穿心莲和板蓝以二核苷酸重复为主,其优势重复基元类型是AT/AT和AG/CT,穿心莲占比33.60%,板蓝占比90.70%[37-38]。SSR丰富的变异位点,为今后开发SSR标记及进一步揭示菜头肾的系统进化提供了可靠的分子手段。
《中国植物志》记载:菜头肾暂仍保留在黄猄草属,然而其具根茎,侧根肉质增厚,这在马蓝亚族(Strobilanthinae)实不多见,或许为一新属,有待进一步深入研究。通过对比分析,发现菜头肾与板蓝叶绿体基因长度相近(相差580 bp),mRNA、rRNA、tRNA分别均为84、8、37个,IR区间结构相近,基于叶绿体全基因组序列构建的系统发育树中,爵床科内分支支持率全为100%,且菜头肾与同一亚族的板蓝亲缘关系最近,构成姐妹关系。但因受黄猄草属物种叶绿体全基因组序列信息不全的影响,无法利用叶绿体基因组对菜头肾进行属内鉴别。因此,对菜头肾的鉴别和系统发育研究,后期还需从形态学、遗传学等进一步研究、验证。
利益冲突 所有作者均声明不存在利益冲突
[1] 中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 2002: 93.
[2] 甘慈尧. 浙南本草新编 [M]. 北京: 中国中医药出版社, 2016: 324-325.
[3] 林泉. 浙南药用植物中的一新种菜头肾[J]. 植物分类学报, 1975, 13(2): 93-94.
[4] 郑毅, 周建武, 陈小玲, 等. 珍稀药用植物菜头肾的人工栽培技术 [J]. 农业科技通讯, 2016(9): 252-253.
[5] 姚振生, 陈京, 尤志勉, 等. 浙江省永嘉县道志地区菜头肾资源调查 [J]. 江西科学, 2007, 25(4): 397-401.
[6] 张森尧, 潘亚琴, 徐攀, 等. 中药菜头肾中化学成分的初步分析 [J]. 世界中西医结合杂志, 2008, 3(4): 204-205.
[7] 屈玲霞, 王扬, 刘永巧, 等. 菜头肾化学成分研究 [J]. 中国现代中药, 2019, 21(4): 438-440.
[8] Shinozaki K, Ohme M, Tanaka M,. The complete nucleotide sequence of the tobacco chloroplast genome: Its gene organization and expression [J]., 1986, 5(9): 2043-2049.
[9] Ohyama K, Fukuzawa H, Kohchi T,. Chloroplast gene organization deduced from complete sequence of liverwortchloroplast DNA [J]., 1986, 322(6079): 572-574.
[10] Arévalo-Gallegos S, Varela-Rodríguez H, Lugo-Aguilar H,. Transient expression of a green fluorescent protein in tobacco and maize chloroplast [J]., 2020, 45: 1-9.
[11] Badro H, Furtado A, Henry R. Relationships between Iraqi rice varieties at the nuclear and plastid genome levels [J].(Basel), 2019, 8(11): E481.
[12] Jin L, Wan Z P, Wei D,. Characterization of the complete chloroplast genome of black soybeans,(L.) Merr. (legume) [J]., 2019, 4(2): 3309-3311.
[13] 李双双, 薛龙飞, 苏爱国, 等. 高等植物线粒体基因组测序和序列分析 [J]. 中国农业大学学报, 2011, 16(2): 22-27.
[14] 张韵洁, 李德铢. 叶绿体系统发育基因组学的研究进展 [J]. 植物分类与资源学报, 2011, 33(4): 365-375.
[15] 崔柳青, 李一帆, 潘卫东. 叶绿体基因工程研究进展 [J]. 生物技术通报, 2012(6): 1-6.
[16] Bock R. Plastid biotechnology: Prospects for herbicide and insect resistance, metabolic engineering and molecular farming [J]., 2007, 18(2): 100-106.
[17] 范李强, 胡欢, 郑洪蕾, 等. 响叶杨(杨属)叶绿体基因组测序与比较分析 [J]. 四川大学学报: 自然科学版, 2018, 55(1): 165-171.
[18] Freyer R, Hoch B, Neckermann K,. RNA editing in maize chloroplasts is a processing step independent of splicing and cleavage to monocistronic mRNAs [J]., 1993, 4(4): 621-629.
[19] Luo R B, Liu B H, Xie Y L,. Erratum: SOAPdenovo2: An empirically improved memory-efficient short-read de novo assembler [J]., 2015, 4: 30.
[20] Besemer J, Lomsadze A, Borodovsky M. GeneMarkS: A self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions [J]., 2001, 29(12): 2607-2618.
[21] Lowe T M, Eddy S R. tRNAscan-SE: A program for improved detection of transfer RNA genes in genomic sequence [J]., 1997, 25(5): 955-964.
[22] Lagesen K, Hallin P, Rødland E A,. RNAmmer: Consistent and rapid annotation of ribosomal RNA genes [J]., 2007, 35(9): 3100-3108.
[23] Lohse M, Drechsel O, Bock R. OrganellarGenomeDRAW (OGDRAW): A tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes [J]., 2007, 52(5/6): 267-274.
[24] Biggeri A, Baccini M, Bellini P,. Meta-analysis of the Italian studies of short-term effects of air pollution (MISA), 1990-1999 [J]., 2005, 11(1): 107-122.
[25] Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability [J]., 2013, 30(4): 772-780.
[26] Guindon S, Dufayard J F, Lefort V,. New algorithms and methods to estimate maximum-likelihood phylogenies: Assessing the performance of PhyML 3.0 [J]., 2010, 59(3): 307-321.
[27] Hu Y H, Woeste K E, Zhao P. Completion of the chloroplast genomes of five Chineseand their contribution to chloroplast phylogeny [J]., 2017, 7: 1955.
[28] Ding P, Shao Y H, Li Q,. The complete chloroplast genome sequence of the medicinal plant[J]., 2016, 27(4): 2347-2348.
[29] 胡欣欣. 浅议温州民间验方七肾汤的应用 [J]. 浙江中医杂志, 2015, 50(2): 131.
[30] 张蔷蓉. 温州民间补肾草药和验方“七肾汤” [J]. 中草药, 2008, 39(6): 954-955.
[31] 汪松, 解焱. 中国物种红色名录: 第1卷[M]. 北京: 高等教育出版社, 2004: 393-395.
[32] Jansen R K, Raubeson L A, Boore J L,. Methods for obtaining and analyzing whole chloroplast genome sequences [J]., 2005, 395: 348-384.
[33] Yaradua S S, Alzahrani D A, Albokhary E J,. Complete chloroplast genome sequence of: Genome comparative analysis and phylogenetic relationships among Acanthaceae [J]., 2019, 2019: 4370258.
[34] Yaradua S S, Alzahrani D A, Albokhary E J,. Corrigendum to “complete chloroplast genome sequence of: Genome comparative analysis and phylogenetic relationships among Acanthaceae” [J]., 2020, 2020: 2049759.
[35] Yaradua S S, Alzahrani D A, Albokhary E J,. Complete chloroplast genome sequence of: Genome comparative analysis and phylogenetic relationships among Acanthaceae [J]., 2019, 2019: 4370258.
[36] 钮峥洋. 亚洲广义爵床属叶绿体系统发育基因组学研究 [D]. 南京: 南京林业大学, 2020.
[37] 李俊仁, 陈秀珍, 汤小婷, 等. 穿心莲转录组SSR位点信息分析 [J]. 中国中药杂志, 2018, 43(12): 2503-2508.
[38] Chen H M, Shao J J, Zhang H,. Sequencing and analysis of(Nees) Kuntze chloroplast genome revealed the rare simultaneous contraction and expansion of the inverted repeat region in angiosperm [J]., 2018, 9: 324.
Assembly and sequence analysis of chloroplast genome ofbased on high-throughput sequencing
ZHAO Qi1, 2,YU Jia-xing1, QIN Yu-wen1, 2, REN Xian-ying1, 3, GAO Hai-dong4, WU Zhi-gang1, JIANG Cheng-xi1, 3
1. School of Pharmacy, Wenzhou Medical University, Wenzhou 325035, China 2. Zhejiang Biomedical Collaborative Innovation Center, Wenzhou 325035, China 3. Jiuhua Mountain Buddhist Medicine Research Institute of Anhui Province, Chizhou 242700, China 4. Nanjing Jisihuiyuan Biotechnology Co., Ltd., Nanjing 210033, China
The chloroplast genome ofwas assembled and sequenced, laying a foundation for the further development of the genetics and identification of.The chloroplast genome ofwas sequenced using high-throughput sequencing technology for the first time, taking the chloroplast genome ofas a reference, the feature analysis and clustering relationship study were carried out.Bioinformatics analysis showed that the total length of the chloroplast genome ofis 144,713bp, containing a typical angiosperm chloroplast genome circular tetrad structure, with a GC content of 38.35%. A total of 129 genes were annotated, including 84 protein-coding genes, 37 tRNA genes and eight rRNA genes. The codons corresponding to protein coding genes are preferred with A/T bases. Phylogenetic studies showed that the relationship betweenandwas the closest, suggesting that they were evolutionarily co-linear.The research provided a method suitable for the assembly of the complete chloroplast genome and the analysis of characteristics of the whole chloroplast genome of the, which also could lay the foundation for the research on the identification and phylogeny of.
C. Ling;(Nees) Kuntze;chloroplast; genome; high-throughput sequencing
R282.12
A
0253 - 2670(2021)06 - 1744 - 07
10.7501/j.issn.0253-2670.2021.06.024
2020-12-01
国家中医药管理局中药饮片标准化建设项目(ZYBZH-Y-SC-40);云南大理药业股份有限公司课题(KJHX1603);温州医科大学人才科研启动项目(OTJ18037)
赵 祺(1994—),女,湖北襄樊人,硕士,研究方向为中药鉴定学。Tel: (0577)86591685 E-mail: 1297909219@qq.com
姜程曦,博士,研究员,硕士生导师,研究方向为中药学。Tel: 18969715696 E-mail: jiangchengxi@126.com
吴志刚,副教授,研究方向为中药次生代谢产物研究。Tel: 15867176058 E-mail: wuzhigang177@126.com
#并列第一作者:余佳兴(1997—),女,四川成都人,硕士,研究方向为中药学。Tel: (0577)86591685
[责任编辑 时圣明]
