黄酮类化合物对CYP1A1酶抑制活性的3D-定量构效关系研究
2021-03-09陈萍萍吕沐瀚梁思成齐晓怡葛广波
何 庆,陈萍萍,李 昊,吕沐瀚,梁思成,齐晓怡,熊 霞,葛广波
1.西南医科大学附属医院 皮肤科,四川 泸州 646000
2.西南医科大学附属医院 消化内科,四川 泸州 646000
3.上海中医药大学交叉科学研究院,上海 201203
细胞色素P450 酶1A1(cytochrome P450 1A1,CYP1A1)是人体内重要的细胞色素P450 酶。CYP1A1 结构中共含有512 个氨基酸,相对分子质量为5.816×104,包含12 个α 螺旋和3 个β 折叠。其中,α 螺旋部分形成了6 个底物识别位点[1],可选择性识别、结合和催化不同类型的化合物[2]。研究表明,CYP1A1 的活性与急慢性炎症、肺损伤、动脉粥样硬化、肿瘤等多种疾病的发生、发展密切相关[3]。CYP1A1 能够代谢和活化杂环芳胺、工业芳胺、多环芳烃类[4-5]等多种环境致癌和致突变物质,促进DNA 突变并最终形成肿瘤[6]。CYP1A1 介导的前致癌物活化与癌症的发生发展关系密切,抑制其催化功能是癌症化学预防的潜在靶点[7-8]。黄酮属于天然多酚类化合物,广泛分布于蔬菜、水果、草药中,至今已发现上千种不同类型的黄酮类化合物[9]。黄酮类化合物具有保护心血管、抗氧化、抗衰老、调血脂、调节免疫等作用[10]。黄酮类化合物可以抑制CYP1A1 酶的活性[11-14],降低CYP1A1 介导的前致癌物活化,预防癌症[15]。然而,黄酮类化合物与CYP1A1 抑制活性的构效关系少见报道。
定量构效关系(quantitative structure-activity relationship,QSAR)是基于配体的计算机辅助药物设计的主要方法之一,通过对化合物结构特征和生物活性进行定量分析,建立合理的数学模型,广泛用于新化合物的活性预测、筛选及设计。3D-QSAR 不仅能够考察药物分子不同的构型或构象对其活性及性质的影响,还能够反映受体与配体的相互作用信息[16]。CoMFA 和CoMSIA 是目前应用最广泛的三维定量构效方法[17-18]。本研究采用第二代CoMFA(Topomer CoMFA)[19-20]构建黄酮类化合物与CYP1A1 抑制活性的3D-QSAR 模型,为黄酮类化合物与CYP1A1 抑制活性的构效关系提供参考,并为筛选CYP1A1 抑制剂提供更加快速有效的方法。
1 材料
1.1 药品与试剂
7,4′-二羟基黄酮(7,4′-dihydroxyflavon,批号AB151809,质量分数为97%)购自德国ABCR 公司;4′,5′-二羟基黄酮(4′,5′-dihydroxyflavone,批号L14161,质量分数为 98%)、5,6-二羟基黄酮(5,6-dihydroxyflavone,批号H27039,质量分数为97%)购自 Alfa Aesar 公司;7,8-二羟基黄酮(7,8-dihydroxyflavone,批号D1916,质量分数>98%)、6-甲氧基黄酮(6-methoxyflavone,批号M1346,质量分数>99%)购自TCI 公司;异黄腐酚(isoxanthohumol,批号JOT-11423,质量分数≥98%)、芫花素(5,4′-dihydroxy-methoxyflavone,批号JOT-10632,质量分数≥98%)购自成都普菲德生物技术有限公司;5,6,7-三甲氧基黄芩素(5,6,7-trimethoxyflavone,批号B29309,质量分数≥97%)购自上海源叶生物科技有限公司;石吊兰素(lysionotin,批号MB4495,质量分数≥98%)购自美仑生物。
7-乙氧基试卤灵为课题组自制;葡萄糖-6-磷酸(glucose 6-phosphate,G-6-P)购自美国 Sigma-Aldrich 公司;葡萄糖-6-磷酸脱氢酶(glucose-6-phosphate dehydrogenase,G-6-PDH)、β-烟酰胺腺嘌呤二核苷酸磷酸( β-nicotinamide adenine dinucleotide phosphate,β-NADP)、MgCl2购自美仑生物;PBS 缓冲液购自中国医药集团有限公司;CYP1A1 酶(批号8191004)购自Gentest 公司。
1.2 仪器
SpectraMaX M4 酶标仪(上海美谷分子仪器有限公司);MS-100 型恒温混匀仪(杭州奥盛仪器有限公司)。
2 方法
2.1 数据集来源与能量优化
通过查阅相关文献,选取50 种已报道的对CYP1A1 有抑制作用的黄酮类化合物,其化学结构与活性数据见图1和表1。CYP1A1 抑制活性采用半数抑制浓度( half maximal inhibitory concentration,IC50)表示,为更好地获得黄酮类化合物的结构与活性之间的线性拟合情况,将其负对数值pIC50作为模型的因变量。
pIC50=−lgIC50
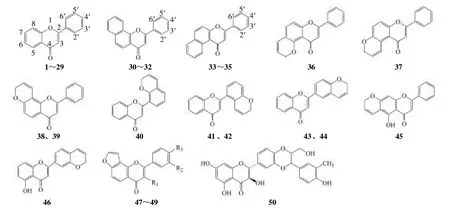
图1 50 种黄酮类化合物的结构Fig.1 Structures of 50 flavonoids
采用Chemoffice 16.0 构建化合物的三维结构,导入SYBYL-X2.0 软件中,在Compute Minimize模块对化合物进行能量优化[21-22],采用Powell 能量梯度法,选择Tripos 分子力场,进行优化,Gradient设置为 21 J/mol,分子电荷设置为 Gasteiger-Hunckel,最大迭代次数设置为1000。优化完成后将其导入分子表单。
2.2 Topomer CoMFA 模型构建
Topomer CoMFA 方法是对传统CoMFA 方法的一种改进,是CoMFA 与Topomer 技术的联合,能够在短时间内快速、准确预测化合物的活性并构建3D-QSAR 模型。将50 个黄酮类化合物按4∶1 分成训练集和测试集。根据化合物取代基不同,分为不同类型的结构层,从不同结构层中随机挑选化合物组成测试集,剩下的化合物为训练集。以黄酮作为公共骨架,随机选择一个分子作为模板,切割分子的方式为“Split into two”,如图2所示,切出2 个取代基(R1、R2)片段。软件自动识别剩余的化合物并以相同的方式进行切割,遇到程序不能自动识别的分子,选择与模板分子尽可能相同的方式手动切割,直至所有的训练集分子被切割完为止。
Topomer CoMFA 采用偏最小二乘法(partial least squares method,PLS)[23]建模分析,当所有训练集分子切割完成后,程序会自动计算每个分子片段的立体场和静电场性质,再以立体场和静电场性质描述符为自变量,以训练集化合物分子的pIC50为因变量构建模型。测试集不参与QSAR 模型的建立,仅用于模型的外部验证,以检测模型的实际预测能力。
2.3 实验验证
采用构建的Topomer CoMFA 模型预测9 种黄酮类化合物的CYP1A1 抑制活性。96 孔板中加入100 mmol/L PBS 缓冲液(pH 7.4)及还原型烟酰胺腺嘌呤二核苷酸磷酸( nicotinamideadenine dinucleotide phosphate,NADPH)产生体系(1 mmol/L NADP+、10 mmol/L G-6-P、1 U/mL G-6-PDH、4 mmol/L MgCl2)和CYP1A1 酶,总体积为200 μL,轻轻震荡混合。分别加入样品或纯乙腈,于恒温混匀仪37 ℃预孵育3 min;加入反应辅因子(β-NADP)起始反应;采用酶标仪连续监测反应10 min。产物试卤灵激发波长560 nm,发射波长590 nm。
2.4 分子对接
采用SYBYL-X2.0 中的Surflex-Dock[24]进行分子对接,对接所使用的蛋白酶晶体结构来源于RCSB PDB 数据库(http://www.rcsb.org/structure/4I8V),晶体结构的PDB ID 为4I8V[1],对接模式为Surflex-Dock(SFXC)。在对接前对蛋白进行处理,从复合物中提取配体A/BHF602,删除不需要的配体及所有水分子,给蛋白加氢加Gasteiger-Hückel电荷,设定距离配体分子0.5 nm 范围内的所有氨基酸残基为活性口袋进行叠合,然后将模板分子1 号化合物与蛋白晶体进行对接。用总打分函数、配体与受体对接的不适当程度和极性打分函数评价对接结果。总打分函数表示受体与配体的亲和能力,打分越高越好;不适当程度的绝对值越接近零,表示配体与受体对接时的不适当程度越小;当结合位点位于分子表面时,极性打分函数越高越好,当结合位点位于分子内部时,极性打分函数越低越好。
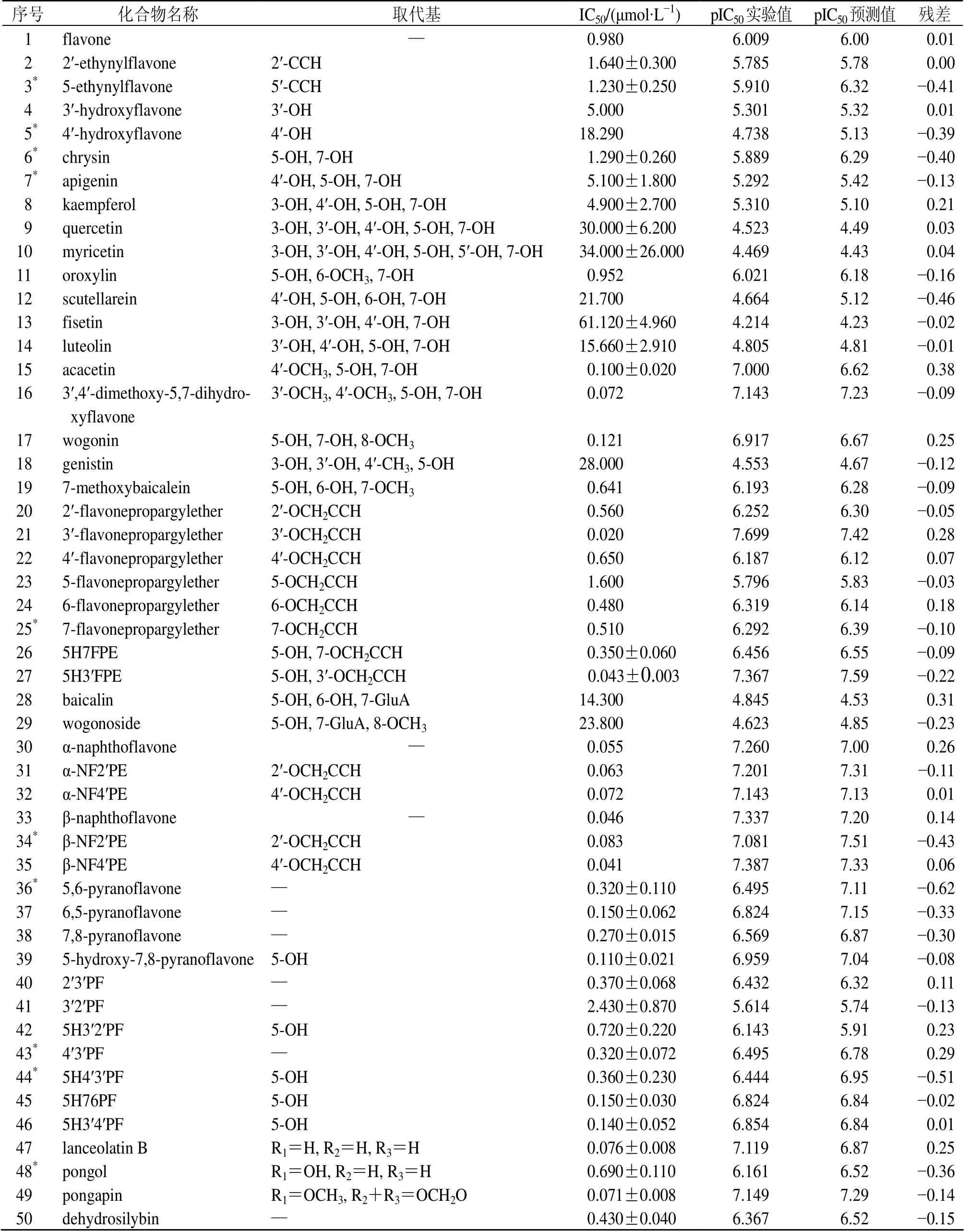
表1 50 种黄酮类化合物的结构及活性数据Table 1 Structures and pIC50 values of inhibitors used for modeling
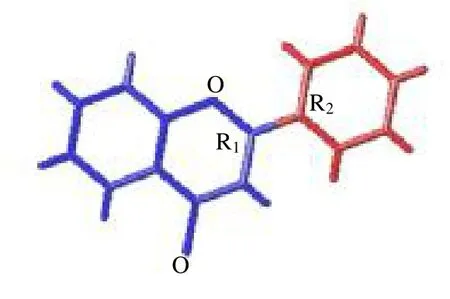
图2 黄酮的分子片段切割示意图Fig.2 Cutting of flavonoids
3 结果
3.1 Topomer CoMFA 模型统计结果分析
对于所建Topomer CoMFA 模型,交互验证复相关系数(q2)、拟合复相关系数(r2)和外部验证相关系数(rpred2)越大表示模型的相关性越好,预测能力越强;q2的标准误差(q2stderr)、r2的标准误差(r2stderr)和标准估计偏差(SEE)越小证明模型质量越高。当q2>0.500、r2>0.600 时,表明建立的模型具有显著的统计学意义[25-26],当rpred2>0.5 表明模型具有较强的外部预测能力[27]。本研究所建模型各参数如表2所示,模型q2>0.500,r2>0.600,表明模型具有较好的拟合与内部预测能力,模型rpred2=0.956 7,表明模型具有较好的外部预测能力。

表2 Topomer CoMFA 的分析结果Table 2 Statistical results of Topomer CoMFA
采用该模型对训练集和测试集化合物的活性进行预测,结果见表1。化合物训练集和测试集pIC50实验值与预测值的线性回归图如图3所示,所有样本均匀分布在45°直线附近,表明所建模型具有良好的可靠性和预测能力。
3.2 3D-QSAR 分析
Topomer CoMFA 方法构建的3D-QSAR 模型构建不仅可以获得模型评价参数,还可以获得各取代基的静电场和立体场三维等势图。其中,静电场等势图用红色和蓝色表示,立体场等势图用绿色和黄色表示。静电场中,红色代表在此区域引入吸电性基团有利于增大化合物分子的预测活性值,蓝色代表在此区域引入供电性基团有利于增大化合物分子的预测活性值;立体场中,绿色代表在此区域引入大体积取代基有利于增大化合物分子的预测活性值,相反,黄色代表在此区域引入小体积取代基有利于增大化合物分子的预测活性值。
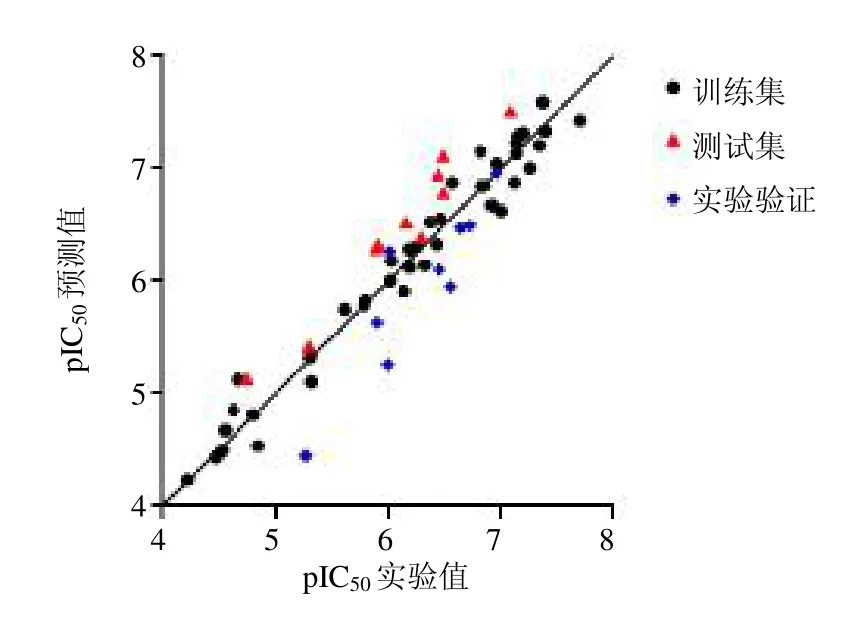
图3 实验数据与预测的pIC50 值之间的线性回归分析Fig.3 Linear regression between experimental and predicted pIC50 values
以化合物 1(pIC50=6.01)为模板生成的Topomer CoMFA 模型的各基团静电场和和立体场三维等势图如图4所示。图4-A 中,R1分子片段的C-3 附近有一小块黄色色块图,表明在此处引入小体积取代基有利于活性值升高。通过比较化合物48(pIC50=6.16)、化合物47(pIC50=7.12)的结构及活性值关系可以看出,当其他基团不变时,将化合物48 R1分子片段C-3 位上的羟基换成氢原子,此位置上取代基体积逐渐减小,化合物活性值随之升高。同时,R1分子片段的C-5、C-6、C-7、C-8 位在立体场中被大片绿色区域围绕,当其上的氢原子被更大体积的基团取代时,有利于CYP1A1 抑制活性值的升高。如化合物24(pIC50=6.32)、化合物25(pIC50=6.29)与化合物1(pIC50=6.01)相比,分别在C-6、C-7 位用体积更大的氧丙炔基取代了氢原子,其CYP1A1 抑制活性升高。图4-C 中,R2分子片段的C-3′、C-4′、C-5′位在立体场中被绿色区域围绕,如化合物21(pIC50=7.70)、化合物22(pIC50=6.19)分别在化合物1(pIC50=6.01)的基础上,在C-4′、C-5′位用体积更大的氧丙炔基取代氢原子,活性显著提高。
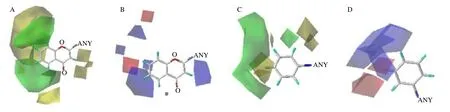
图4 化合物1 的Topomer CoMFA 模型的等势图Fig.4 Contour map of Topomer CoMFA model of compound 1
由图4-B 可以看出,R1分子的C-3、C-6 位在静电场被蓝色区域所覆盖,当引入供电性基团时其CYP1A1 抑制活性提高。对比化合物6(pIC50=5.89)和化合物11(pIC50=6.02),保持其他基团不变,化合物6 C-3 位上的氢原子被甲氧基替换,成为化合物11,供电性增强,化合物活性增高。同理,由图4-D 可以看出,R2分子的C-4′位在静电场中被蓝色区域所覆盖,对比化合物6(pIC50=5.89)和化合物15(pIC50=7.00),保持其他基团不变,化合物6 C-4′上的氢原子被甲氧基替换,成为化合物15,供电性增强,化合物活性增强。
3.3 实验验证结果分析
用于实验验证的化合物结构与活性数据见表3,化合物pIC50实验值与预测值的线性回归关系如图3所示,其相关系数为0.832 8,从图中可以看出化合物取值点基本分布在45°直线附近,表明所建模型具有良好的可靠性和预测能力。
3.4 对接结果分析
分子对接结果见表4和图5,棒状表示配体,线状表示氨基酸残基,虚线表示氢键给体,图5-A为配体分子与CYP1A1 酶蛋白受体的对接位点。由图5-B 可知,模板分子配体与晶体结构中的主要氨基酸残基共形成了1 个氢键,与THR497 形成氢键相互作用,总打分函数、配体与受体对接的不适当程度和极性打分函数分别为4.837 5、−1.730 6、0.046 2。图5-C 为实验活性最高的化合物E 与蛋白质氨基酸残基氢键相互作用的示意图,与ASN222 和ASN225 残基各形成1 个氢键,总打分函数、配体与受体对接的不适当程度和极性打分函数分别为7.930 5、−0.991 8、1.920 4。相较于模板分子而言,化合物5 的2 个氢键作用力对其抑制活性十分重要,化合物5 与CYP1A1 的亲和性明显高于模板分子,这可能是其活性更强的原因。图5-D 为实验活性最低的化合物B 与蛋白质氨基酸残基氢键相互作用的示意图,化合物B 与配体分子分别在SER116、ASP313 残基各形成1 个氢键,总打分函数、配体与受体对接的不适当程度和极性打分函数分别为6.065 8、−0.324 2、1.213 9。相较于模板分子而言,这两个氢键的作用力可能是其活性更强的原因;而与活性最高的化合物E 对比可以发现,形成氢键的残基位点不同,化合物的抑制活性也有差异。
化合物I 不与配体形成氢键,其总打分函数、配体与受体对接的不适当程度和极性打分函数分别为6.671 7、−0.466 7、0.013 3。相较于模板分子而言,其抑制活性稍强,表明氢键的形成并不是增加化合物抑制活性的唯一因素。
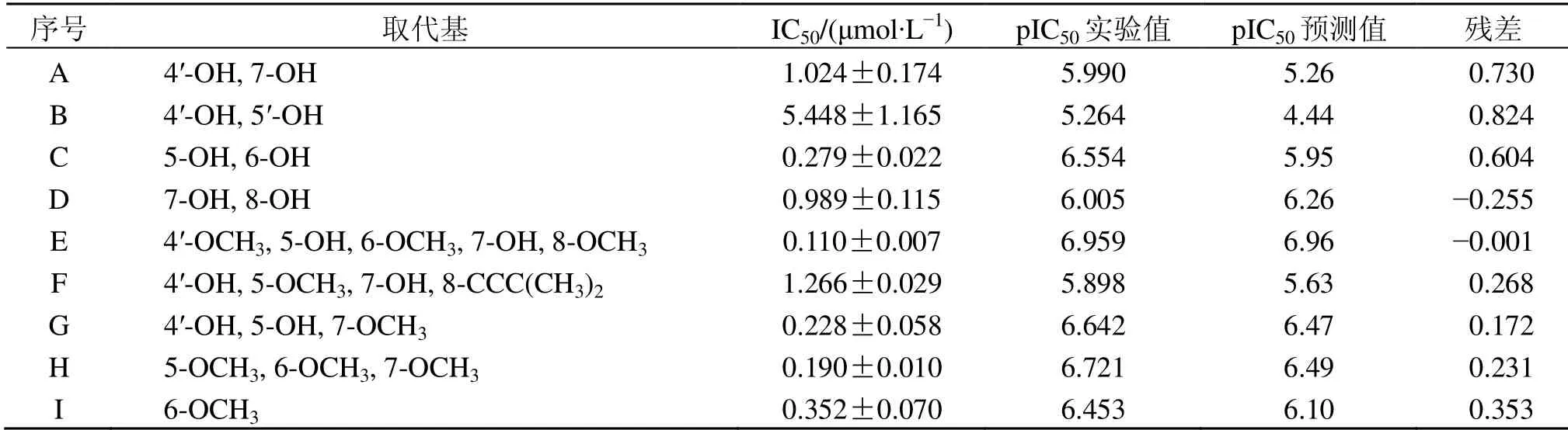
表3 实验验证所用黄酮类化合物的结构与活性数据Table 3 Experimental values,predicted values and residual of flavonoids used for experimental verification
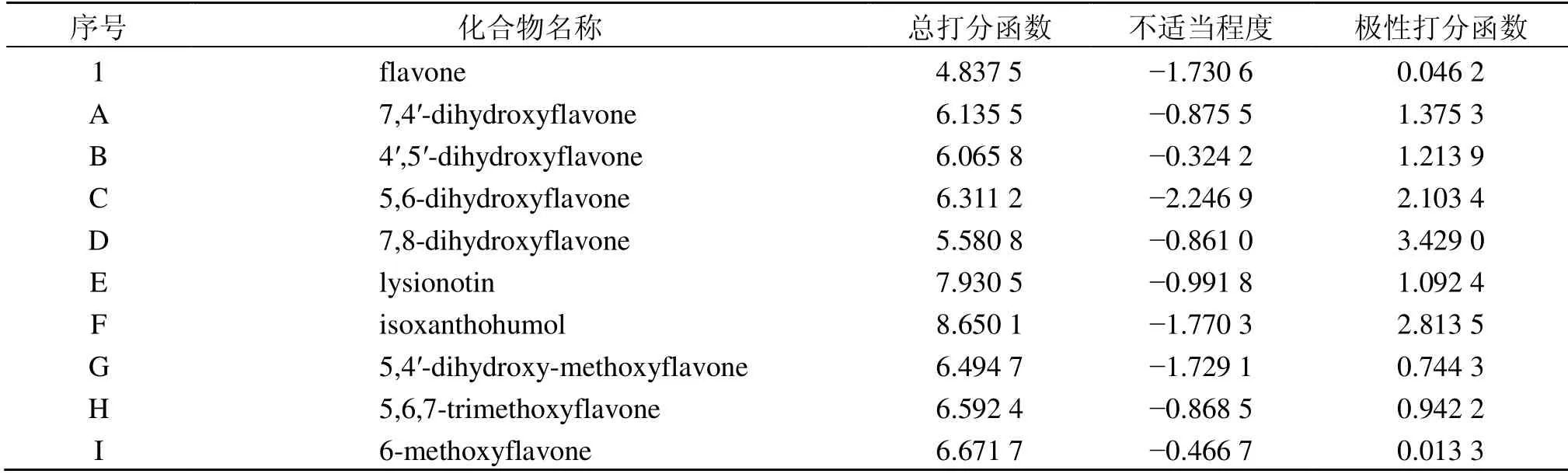
表4 实验验证所用的黄酮类化合物的分子对接结果Table 4 Results of molecular docking of flavonoids used for experimental verification
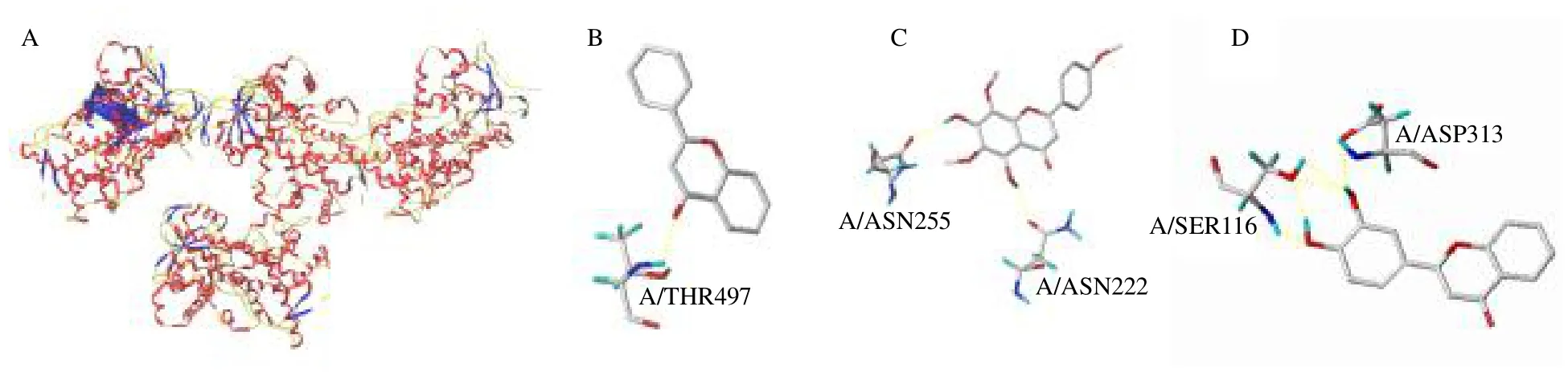
图5 配体与CYP1A1 的对接模型 (A) 及化合物1 (B)、化合物E (C)、化合物B (D) 与CYP1A1 分子的氨基酸残基之间形成的氢键示意图Fig.5 Docking mode between reference ligand and CYP1A1 (A),hydrogen bond interaction diagram of compound 1 (B),compound E (C),and compound B (D) with amino acid residues
4 结论
本研究利用Topomer CoMFA 方法对50 个黄酮类化合物进行了建模分析。该模型通过提供立体场和静电场的可视化图像,直观地揭示了化合物中不同的取代基结构对其活性的影响。统计结果q2=0.800、r2=0.965 0,表明所建立的模型具有较高的预测能力和良好的稳定性。利用模板分子和实验测出的活性分子进行分子对接,探究配体和受体蛋白之间的关系,对接结果表明小分子与大分子蛋白的氨基酸残基THR497、ASN222、ASN255、SER116和ASP313 可以形成氢键,表明所建模型具有良好的可靠性,可为设计高活性分子提供理论参考。本研究为分析黄酮类化合物的CYP1A1 抑制活性提供了高效的方法,为新型CYP1A1 酶抑制剂的开发提供新的思路。
利益冲突所有作者均声明不存在利益冲突
