主要禾本科作物ASR 基因家族全基因组鉴定及表达分析
2021-02-16孟亚轩孙颖琦赵心月王凤霞瓮巧云刘颖慧
孟亚轩,孙颖琦,赵心月,王凤霞,瓮巧云,刘颖慧
(河北北方学院 农林科技学院,河北 张家口 075000)
ASR(Abscisic acid-,stress-,ripening-induced protein)蛋白是植物特异的胁迫反应关键调控因子[1],在植物糖类代谢、果实成熟、激素响应等过程中发挥重要调控作用[2]。ASR 蛋白属固有无序蛋白质,具有高亲水性、热稳定性等特点[3],可作为保护蛋白维持细胞离子平衡,也可入核作为调控蛋白参与信号转导、激活下游基因表达[4]。研究表明,ASR基因可提高植物对逆境的耐受能力[5-9]。二穗短柄草(Brachypodium distachyon)BdASR1-1基因通过诱导非酶抗氧化剂的产生提高二穗短柄草的抗旱能力[6];海蓬子(Salicornia europaea)SbASR1-1基因可激活活性氧(ROS)清除相关基因表达,清除过量积累 的H2O2,抵 御 盐 胁 迫 伤 害[7];番 茄(Solanum lycopersicum)SlASR1 蛋白具有分子伴侣活性,能够防止冻融引起的蛋白质失活[8];玉米(Zea mays)ZmASR3基因通过调节抗氧化系统以及降低胞内离子外渗提高转基因拟南芥的干旱耐受性[9]。ASR 基因在果实发育过程中发挥重要作用,过量表达草莓(Fragaria ananassa)FaASR1基因可促进草莓花色苷积累,影响果实发育进程[10]。
随着基因组学的发展,利用分子生物学技术挖掘优质抗旱基因成为提高作物抗旱丰产能力的主要途径之一。谷子(Setaria italica)、水稻(Oryza sativa)、高粱(Sorghum bicolor)、玉米、小麦(Triticum aestivum)是我国主要禾本科粮食作物,具有资源广、产量高等特点,对其进行比较基因组学研究是禾本科粮食作物品质改良与分子育种的关键环节[11]。目前,关于ASR 基因家族全基因组鉴定的研究主要集中于二穗短柄草[6]、草莓[10]上,尚未见关于上述5种禾本科作物ASR基因家族成员全基因组鉴定和表达的对比研究。为此,利用生物信息学方法对上述5 种禾本科作物ASR 基因家族成员进行鉴定,并对其序列、系统进化、结构域、启动子及表达模式进行分析,以揭示ASR 家族成员序列特点和表达特性,为进一步研究ASR蛋白功能提供参考。
1 材料和方法
1.1 ASR家族基因鉴定和序列分析
利用ASR 隐马模型(PF02496)对谷子、水稻、高粱、玉米、小麦进行全基因组扫描,通过SMART 进行筛选,去除冗余基因,确定候选ASR 家族成员[12]。使用在线软件ExPASy、SOPMA 进行ASR 蛋白序列解析[13]。利用BatchPrimer 进行简单重复序列(SSR)分析。
1.2 ASR蛋白系统进化树构建和共线性分析
利用Clustal 进行多序列比对,模式植物烟草(Nicotiana tabacum)、蒺 藜 苜 蓿(Medicago truncatula)作为外类群组参与构树,使用MEGA 7.0软件绘制ASR 蛋白系统进化树(NJ 法,bootstrap 设置为1 000)[14]。利用DNAMAN 进行氨基酸序列比对。使用Ensembl Plants 数据库进行共线性分析,采用TBtools 软件计算同源基因的非同义替换率与同义替换率的比值,即Ka/Ks。Ka/Ks 用来衡量每个基因对的选择压力,Ka/Ks<1 为纯化选择,Ka/Ks>1为正选择,Ka/Ks=1为中性选择[15]。
1.3 ASR家族蛋白基序(Motif)、结构域分析和启动子预测
利用MEME 在线工具分析ASR 氨基酸基序组成(最小宽度6,最大宽度60,基序检索上限10)。利用Prosite 分析并绘制ASR 基因家族结构域。使用GSDS 2.0 分析谷子、水稻、高粱、玉米、小麦基因组注释文件(gff),分析ASR 家族基因结构并进行可视化处理[16]。使用Tbtools 软件将ASR 蛋白进化树、基因结构、基序分布进行整合。将ASR 基因转录起始位点上游1 500 bp 核苷酸序列提交至PlantCARE进行顺式作用元件预测[17]。
1.4 ASR蛋白亚细胞定位预测
使用WoLF PSORT Ⅱ进行ASR 蛋白亚细胞定位预测,利用PSOET 分析核定位信号(Nuclear localization signal,NLS)类型[18]。
1.5 ASR家族基因表达模式分析
利用GEO 数据库检索水稻、高粱转录组数据。通过筛选,选择水稻GSE21494 转录组数据,获得日本晴根、茎、叶、穗的转录组数据;选择高粱GSE48205 转录组数据,获得R16 盆栽控水干旱14 d、高温50 ℃处理3 h、盆栽控水3 d 后50 ℃处理3 h 的干旱高温复合处理的叶片转录组数据。小麦转录组数据来自WheatExp数据库,获得中国春幼苗20%PEG 干旱处理1 h 和6 h、40 ℃高温处理1 h 和6 h、高温干旱复合处理1 h 和6 h 的转录组数据及根、茎、叶、穗的转录组数据。利用Phytozome 数据库获得谷子根在氨、尿素、干旱、硝酸盐胁迫处理时的转录组数据。利用qTeller 数据库,获得玉米B73自交系幼苗冷、热、盐、紫外线(UV)胁迫处理的转录组数据。将检索到的数据进行归一化处理[19],使用TBtools绘制热图。
为进一步分析ZmASR 基因的表达模式,将B73玉米自交系种植于温室(16 h 光/8 h 暗,辅以人工照明),温 度 为 白 天25 ℃/黑 夜18 ℃。 参 考TROUVERIE等[20]的方法对玉米根系进行100 μmol/L脱落酸(ABA)、20%PEG6000 处理,分别于0、6、12、18、24 h取根和叶片,液氮速冻,-80 ℃保存备用。将材料研磨后提取总RNA,利用FastQuant RT Kit反转录为cDNA 用于实时荧光定量PCR(qRT-PCR)检测。使用DNAMAN 设计引物,选actin为内参基因,反应体系参考LIANG等[9]的方法。试验设3次重复,采用2-ΔΔCT法计算目的基因相对表达量。
2 结果与分析
2.1 ASR家族基因鉴定及序列分析
2.1.1 ASR 家族基因鉴定及蛋白质理化性质分析
经分析发现,谷子、水稻、高粱、玉米、小麦ASR 家族成员数量分别为7、6、8、9、29 个。ASR 家族基因在染色体上不均匀分布,同时存在成簇分布现象,如SiASR2、SiASR3、SiASR4在谷子5 号染色体中成簇存在。蛋白质理化性质分析结果(表1)表明,ASR 蛋白分子质量小,脂肪系数小,呈碱性,具有亲水性、稳定性等特征,二级结构总体以α-螺旋为主,无规则卷曲次之,β-转角及延伸链较少。谷子、水稻、高粱、玉米、小麦ASR 家族成员中,开放阅读框最长的为SbASR1(2 570 bp),氨基酸最多的为TaASR2(279 aa),分 子 质 量 最 大 的 为TaASR1(28.83 ku),理论等电点最高的为ZmASR4(10.56),不稳定系数最高的为ZmASR4(69.65),脂肪系数最高的为OsASR4(56.95),亲水性总均值最低的为TaASR2(-1.760)。

表1 ASR家族成员信息Tab.1 Information about ASR gene family members
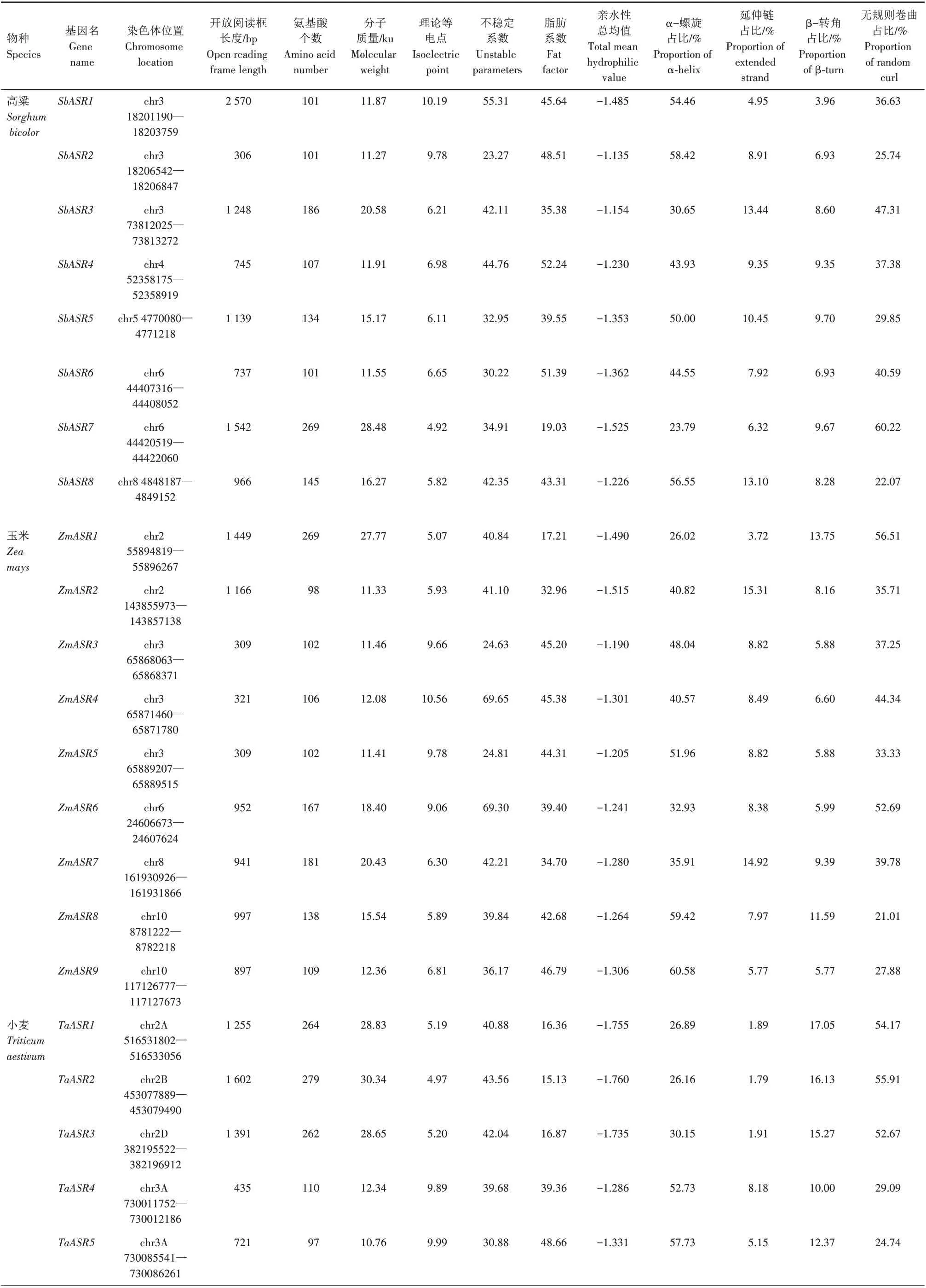
续表1 ASR家族成员信息Tab.1(Continued) Information about ASR gene family members

续表1 ASR家族成员信息Tab.1(Continued) Information about ASR gene family members

续表1 ASR家族成员信息Tab.1(Continued) Information about ASR gene family members
2.1.2 ASR 家族基因SSR 分析 SSR 分子标记因其多样性和稳定性,广泛应用于基因挖掘、分子育种等领域。基于此,本研究利用BatchPrimer 对鉴定到的ASR 家族成员进行SSR 位点挖掘。结果显示,ASR 家族成员中有16 个成员鉴定到SSR(表2)。其中,SbASR3基因具有二核苷酸重复、三核苷酸重复、四核苷酸重复3种SSR类型,ZmASR8基因鉴定到二核苷酸重复、三核苷酸重复2种SSR 类型,其余成员具有二核苷酸重复或三核苷酸重复1种SSR类型。

表2 ASR家族基因SSR分析Tab.2 SSR analysis of ASR family genes
2.2 ASR蛋白进化分析
2.2.1 ASR 蛋白系统进化树构建及氨基酸序列分析 为明确ASR 蛋白进化模式,选用模式植物烟草、蒺藜苜蓿作为外类群组参与进化树构建(图1)。根据进化树拓扑情况,ASR 蛋白可分为Ⅰ-1、Ⅰ-2、Ⅱ-1、Ⅱ-2 四个亚组,分别占43%、9%、17%、31%。其中,烟草、蒺藜苜蓿ASR 家族成员聚类到Ⅰ-2 亚组,禾本科作物ASR 家族成员聚类到其他亚组。相比烟草、蒺藜苜蓿,禾本科ASR 家族成员关系更密切,说明ASR 蛋白在进化过程中具有种属特异性特点。
为进一步了解ASR 蛋白亚组之间的差异,利用DNAMAN 对ASR 氨基酸序列进行比对。由图2A 可知,Ⅰ、Ⅱ亚组ASR 蛋白氨基酸序列具有较高一致性,均含有1 个ABA/WDS 结构域(a)和1 个C-末端核靶向信号位点(b)。外类群聚类成的Ⅰ-2亚组与其他亚组相比并未出现明显差异,但Ⅱ亚组中ZmASR2、OsASR5 蛋白发生了氨基酸位点缺失现象。氨基酸位点保守性分析结果(图2B)表明,Ⅰ、Ⅱ亚组ASR 蛋白氨基酸位点具有一定保守性。综上所述,ASR 蛋白在进化过程中具有保守性的同时也存在变异现象。
2.2.2 ASR 家族蛋白基序及结构域分析 由图3A可知,ASR 基因含有1~3 个外显子,与目前发现的其他植物ASR 基因拥有相似的剪切方式。基序分析结果显示,亲缘关系较近的成员具有相似的基序组合方式,但也存在基序缺失现象,如Ⅰ-1 亚组SbASR1 缺 失 蛋 白 质 保 守 基 序2(Motif 2)、CDS(Coding sequence)保守基序3(Motif 3);Ⅱ-2亚组成员TaASR15 缺失蛋白质保守基序4(Motif 4)、CDS保守基序6(Motif 6)。与其他亚组相比,Ⅰ-1 亚组成员蛋白质保守基序和CDS 保守基序相似度较高,说明Ⅰ-1 亚组成员进化较保守。外类群聚类成的Ⅰ-2 亚组与禾本科作物ASR 家族成员相比并无明显差异,说明ASR 家族成员在不同物种间的进化轨迹具有一致性。ASR 家族蛋白4 个亚组中,Ⅰ-1、Ⅰ-2、Ⅱ-1 亚组的基序组成及分布具有较高一致性,Ⅱ-2亚组相较于其他亚组差异明显。结构域预测结果显示,ASR 家族成员均只含有ABA/WDS 结构域,位于C-末端(图3B)。
2.2.3 ASR 家族基因共线性分析及进化选择压力分析 为进一步阐述ASR 基因的进化起源关系,对ASR 家族基因进行共线性分析,并计算Ka/Ks,部分结果见表3。进化选择压力分析结果表明,大部分ASR 共线基因对Ka/Ks<1,说明ASR 基因在进化过程中主要受纯化选择。

表3 ASR家族基因进化选择压力Tab.3 Evolutionary selection pressure of ASR family genes
2.3 ASR家族蛋白亚细胞定位预测
蛋白质亚细胞定位与其参与的生化过程密切相关,为进一步研究ASR 蛋白功能,利用WoLF PSORT Ⅱ进行亚细胞定位预测,利用PSOET 进行NLS 预测,部分结果见表4。鉴定到的59 个ASR 家族成员中,有68%ASR 蛋白定位到细胞核,32%定位到线粒体,说明大部分ASR 成员为转录因子,可调控下游基因表达。鉴定到的ASR 家族成员中46%的成员没有任何NLS,其余成员只含有1 种或2种NLS。

表4 ASR蛋白亚细胞定位及NLS预测Tab.4 Subcellular location and NLS prediction of ASR protein

续表4 ASR蛋白亚细胞定位及NLS预测Tab.4(Continued) Subcellular location and NLS prediction of ASR protein
2.4 ASR基因启动子分析
利用PlantCARE 在线软件分析ASR 转录起始位点上游1 500 bp 核苷酸序列。结果(图4)显示,ASR 基因启动子区域含有多种顺式作用元件,包括组织特异性元件(如胚乳特异性表达元件、种子特异性调控元件)、应激响应元件(如光响应元件、低温响应元件)、激素响应元件(如ABA 响应元件、茉莉酸响应元件),暗示ASR 基因参与了植株建成和激素、应激等响应过程。同时,不同物种ASR 家族成员具有相似的顺式作用元件分布情况,如响应元件、厌氧诱导元件、光响应元件、茉莉酸响应元件在ASR 家族成员中均具有较高的分布。另外,一部分同源基因之间,如TaASR1、TaASR3基因具有类似的顺式作用元件;不同亚组基因差异较大,如SbASR1基因较SiASR1基因拥有更多的顺式作用元件。
2.5 ASR基因表达模式分析
不同物种ASR 基因在植株建成过程中表达差异较大(图5A—B),水稻ASR 家族成员在叶和根中表达量较高,小麦ASR 家族成员在叶中表达量较高。此外,发现同亚组中部分旁系同源基因具有相似表达模式,如Ⅱ-2 亚组中TaASR13基因和TaASR20基因。由图5C可知,玉米ASR家族成员在遭遇不同胁迫时具有不同的表达模式和响应程度。如在遭遇低温、盐胁迫时大部分成员上调表达,在遭遇高温、UV 胁迫时大部分成员下调表达。同时存在部分特殊基因,如ZmASR3基因在不同胁迫条件下表达量均较低。在谷子受到氨、干旱等不同胁迫时,ASR基因同样存在差异表达现象(图5D)。由图5E—F 可知,经高温、干旱及二者复合胁迫处理后高粱、小麦ASR 家族成员差异表达,且表达模式在一定程度上具有相似性。高粱、小麦部分ASR 家族成员在遭遇高温胁迫时表达量下调,在遭遇干旱、干旱和高温复合胁迫时表达量上调。
为明确ZmASR 基因在不同胁迫条件下的表达模式,挑选6 个ZmASR 基因进行qRT-PCR 分析,结果(图6)显示,ZmASR 基因均受ABA、干旱诱导表达,但具有表达特异性。其中,ZmASR7、ZmASR8基因在叶中受到干旱、ABA 诱导时均具有较高表达量;ZmASR2基因在根中受到ABA 诱导时具有较高表达量,18 h 达到峰值;ZmASR3基因在根中受ABA诱导表达,18 h 达到峰值;ZmASR1、ZmASR9基因相比其他成员则表达量较低。说明玉米ASR 家族成员在参与植物响应干旱、ABA 胁迫过程中具有不同的表达策略,且表达具有组织特异性,存在一定功能分化。
3 结论与讨论
利用生物信息学方法,在谷子、水稻、高粱、玉米、小麦基因组中分别鉴定到7、6、8、9、29个ASR家族基因,对比鹰嘴豆(10个)、草莓(4个)、苹果(5个)ASR 家族成员[21],推测ASR 基因在进化过程中发生了全基因组加倍、串联复制现象[22]。通过构建系统进化树发现,不同物种ASR 基因聚类为不同分支,说明ASR 基因在进化过程中不同物种间存在分歧,具有种属特异性特点。染色体定位结果显示,ASR基因存在成簇分布现象,在草莓[10]、苹果[21]中也出现了此类现象。基因结构是研究基因演变的重要依据,基因结构的多样性在进化过程中扮演重要角色[23]。本研究发现,ASR基因结构具有多样性,说明这些基因在演变过程中具有不同进化轨迹。保守基序分析结果表明,Ⅰ-1、Ⅰ-2、Ⅱ-1亚组成员的氨基酸基序组成及分布具有较高一致性,Ⅱ-2亚组成员相较于其他亚组成员差异明显,结合其基因结构分析结果,说明这2类基因具有相对独立的进化史,这与LIANG 等[9]、ZAN等[5]相关研究结果一致。亚细胞定位结果显示,ASR 蛋白大多定位在细胞核中,但部分ASR 蛋白无NLS,说明ASR 蛋白在没有NLS的情况下也可通过扩散作用进入细胞核作为转录因子发挥作用。
本研究发现,ASR 基因分布在不同器官中,在不同物种中具有不同的表达模式,如TaASR 基因在叶中高表达,OsASR 基因在叶、根中高表达,说明ASR 家族成员广泛参与植物的发育过程,不同物种间具有特异性,这与LI 等[2]、SACHDEVA 等[1]研究结果一致。在植物遭遇逆境胁迫时,大部分ASR 基因可被检测到表达差异,但响应程度不同。如遭遇干旱、高盐、低温胁迫时上调表达,遭遇UV、高温胁迫时下调表达,但也存在部分基因特异响应某种胁迫的情况(如ZmASR5基因在低温胁迫条件下高表达,在遭遇其他胁迫时表达量较低)。qRT-PCR 结果同样证明ASR 基因存在表达差异。植物在响应干旱、高盐等渗透胁迫因子刺激时,内源ABA 与PYL(Pyrabactin resistance 1-like)相互作用并激活PP2C(Type 2C protein phosphatases),形 成PYL-ABAPP2C,通过释放SnRK2(Sucrose non-fermenting 1-related protein kinase 2)诱导下游胁迫响应基因表达[24]。启动子分析结果显示,ASR 基因具有大量ABA 响应元件,结合其在不同植物中受胁迫诱导后表达模式的相似性,推测ASR蛋白通过响应ABA信号改变基因表达模式参与植物对逆境胁迫的响应。
