噬菌体合成生物学研究进展和应用
2021-01-21袁盛建马迎飞
袁盛建,马迎飞
(1 中国科学院深圳先进技术研究院,深圳合成生物学创新研究院,中国科学院定量工程生物学重点实验室,广东省合成基因组学重点实验室,深圳市合成基因组学重点实验室,广东 深圳 518055; 2 中国科学院大学,北京 100049)

噬菌体(bacteriophage, phage)是专一感染细菌、古菌和藻类等微生物的病毒的总称,在土壤、河水、空气或生物体内都广泛存在。噬菌体于1915—1917 年被发现[1],一个多世纪以来,由于噬菌体基因组较小(5~735 kb,中值约为50 kb[2-3])、繁殖速度快和易于遗传操作,噬菌体研究一直在推进生物学许多领域的发展,例如遗传学、分子生物学和合成生物学。
遗传学方面,科研工作者们利用噬菌体证实了DNA 是遗传物质[4],并确定了三联体密码子[5];分子生物学方面,噬菌体为科学家提供了许多分子生物学工具酶,如DNA 聚合酶、限制性内切酶、连接酶等[6],现在全球各地实验室还都在使用着噬菌体来源或者噬菌体相关的工具酶。
噬菌体研究对合成生物学领域也有重要贡献。第1个被测序的基因组就是φX174噬菌体,2003年这个噬菌体全基因组又被Ⅴenter 等[7]从头合成并拯救成为有活性的噬菌体颗粒;也正是在研究细菌如何防御噬菌体的过程中,使得科学家发现了CRⅠSPR/Cas 系统[8],该发现获得 2020 年诺贝尔化学奖,并且正在推动一场重大的生物技术革命[9];噬菌体在基因元件开发方面具有很大潜力,许多噬菌体衍生的基因元件已被改编用于构建基因电路。同时,噬菌体在生物圈的数量非常多,达到1031数量级[10],而且进化出多种方式接管宿主细胞的资源来产生更多的噬菌体后代。因此,对于噬菌体的基因多样性和功能多样性的研究将为合成生物学研究提供极其丰富的基因元件库。
尽管如此,我们对于噬菌体的认识还极其缺乏。比如:对于自然界中绝大多数噬菌体和宿主的关系及其在自然生态系统中的作用还不清楚。
采用合成生物学方法和思路理性设计、合成人工噬菌体,可以加强、改变或利用噬菌体某方面的特性,有望帮助我们回答噬菌体基础研究和应用研究中一些普遍存在的问题[11-13]。通过在噬菌体基因组上添加功能基因(如抗菌肽和荚膜解聚酶基因)可增强噬菌体的侵染效率;尽可能去除毒力蛋白基因和未知功能的基因,甚至设计出底盘噬菌体基因组,以达到生物安全性高度可控的目的;在噬菌体基因组上添加抗细菌抗性的基因元件,解决细菌对噬菌体产生抗性的问题[14];噬菌体可搭载各种基因线路,用于改造细菌群落,敲除菌群中特定基因;人工改造噬菌体尾部蛋白基因,替换或拓展宿主谱[15];温和噬菌体改造为烈性噬菌体策略的开发,可以丰富噬菌体资源库。同时,噬菌体强大的定向进化平台、噬菌体展示技术、噬菌体DNA载体等功能也正在蓬勃发展。
高通量测序技术、DNA 大片段合成技术和合成生物学等方面的发展使科研工作者能够合成出第一个最小基因组细菌(支原体)[16]。这些用于合成细菌基因组的新策略和方法,可以加速噬菌体基因组设计构建的能力,更深入挖掘噬菌体各方面的潜力。本文作者讨论了噬菌体合成生物学研究最新进展、潜在的方向、如何应用合成生物学产生具有特定功能的噬菌体以及人工噬菌体的各种用途(图1)。
1 噬菌体合成生物学研究方向
1.1 提高噬菌体侵染效率
沙眼衣原体(Chlamydia trachomatis)是专性的细胞内寄生病原体,因此细胞外环境中的噬菌体无法进入细胞内对其进行攻击。Bhattarai 等[17]为了抑制沙眼衣原体,将噬菌体人工改造使其能被真核细胞内吞。在M13 噬菌体衣壳上融合两个不同的功能肽:整合素结合肽(RGD)和沙眼衣原体部分膜蛋白肽段(PmpD),RGD 诱导细胞的内吞作用,使噬菌体进入真核细胞,PmpD 能中断沙眼衣原体的感染和增殖。用改造后的噬菌体预防或治疗HeLa 细胞的衣原体感染,荧光显微镜检查结果显示改造噬菌体能明显抑制沙眼衣原体的生长[17]。因此,改造噬菌体可用于有效防控沙眼衣原体或其他胞内病原体。
生物膜(biofilm)通常与持续的慢性细菌感染有关,由各种生物大分子如蛋白质、多糖、脂类和DNA 等组成。生物膜可以限制分子和颗粒的扩散从而保护病原菌不易受到噬菌体和抗生素等抗菌剂的攻击,是细菌防治中的一个重要阻碍因素,使许多临床感染难以根治[18]。2007 年,Lu 等[18]在T7 噬菌体上整合生物膜降解基因DspB,改造后的噬菌体清除生物膜的效率比野生型强约3倍,能够减少4~5 个数量级的生物膜细菌[图1(a)]。这类研究还可以扩展到其他类似研究之中,例如在噬菌体上添加蛋白质表面活性剂、抗菌肽和荚膜解聚酶[19-20]等。
淀粉欧文菌(Erwinia amylovora)是一种常见的植物病原体,其会产生荚膜以对抗噬菌体的侵染,因此很少有噬菌体能有效预防或治疗淀粉欧文菌感染。将欧文菌噬菌体L1 的荚膜解聚酶基因dpoL1-C 克隆到噬菌体Y2 的基因组中,改造后的Y2 噬菌体能产生更大、更清晰的噬菌斑。尽管每个被感染细胞产生的噬菌体数量变少了,但是经过改造的噬菌体能更有效地杀死欧文菌,与野生型相比,改造的噬菌体能减少欧文菌约3 个数量级[13]。噬菌体治疗在花卉细菌感染中也得到了类似的结果,说明表达解聚酶可以帮助噬菌体更好地发挥作用[13]。

图1 噬菌体合成生物学的应用[15,18,28]Fig.1 Application of synthetic phages[15,18,28]
丝状噬菌体M13 通常被用作DNA 线路载体,以便将DNA转导给细菌,这些类似的噬菌体改造载体被称为噬菌粒(phagemids)[21-22]。在M13 噬菌粒上搭载小型抗菌肽基因(AMPs)和蛋白毒素基因可以将大肠杆菌的数量降低4个数量级,从而将无毒的M13噬菌体改造为有效的杀菌剂。在使用EMG2大肠杆菌作为病原体的腹膜炎感染模型中,该改造的M13可以有效保护小鼠免于死亡[23]。展示了噬菌体作为DNA线路传递载体的潜在应用价值。
工程噬菌体表达生物膜降解酶、解聚酶、抗菌肽或其他功能基因的例子表明合成生物学可用于大大改善天然噬菌体的性状,更能有效对抗细菌感染。
1.2 改变噬菌体宿主范围
噬菌体侵染细菌的第1个步骤是识别细菌表面的特异性受体,细菌表面受体可能是多糖、蛋白质甚至是细胞器,例如菌毛或鞭毛[24]。这些受体是高度可变的,因此大多数噬菌体仅能侵染特定病原菌种的部分菌株和变体。这种专一性限制了噬菌体在耐药菌感染防治上的应用。可能需要由多种噬菌体组成的鸡尾酒制剂来覆盖足够的病原菌种类。随着病原菌种群在噬菌体侵染过程中不断进化,这些噬菌体鸡尾酒制剂需要定期更新换代,这可能会给实践和监管带来挑战。
有些噬菌体也能够侵染一种以上的细菌。例如,噬菌体SH7 能同时侵染大肠杆菌、痢疾志贺菌、弗氏志贺菌和伤寒沙门菌[25],噬菌体Mu能同时侵染大肠杆菌、弗氏柠檬酸杆菌、索氏志贺菌和欧文菌[26]。但是,广宿主范围噬菌体数量较少。因而,噬菌体的宿主谱改造具有重要的研究和应用价值。
解决宿主范围的一种方法是开发噬菌体基因模块,对已经被批准为安全有效的噬菌体基因模块进行替换,以扩大噬菌体宿主范围。2015 年,Ando 等[15]合理替换了T7-like 噬菌体的尾部基因,改变了其宿主范围[图1(b)]。在T7-like噬菌体中,宿主范围主要由gp17编码的尾部蛋白控制。当把尾部蛋白基因与其他相关噬菌体的类似基因进行替换时,出现了可预测的宿主范围变化。而序列相对差异较大的克雷伯菌噬菌体,需要将整个尾部(gp11、gp12和gp17)设计成一个模块,把整个尾部模块相互替换,让大肠杆菌的T7 噬菌体能够侵染克雷伯菌。该方法需要在研究比较透彻的噬菌体(噬菌体已进行测序、分类和结构基因清晰)之间才能进行,而且结构差异越大,需要替换的基因可能越多,将急剧增加改造难度。
同样,Yosef 等[27]建立了筛选替换 T7-like 噬菌体不同尾部蛋白的平台,从而有效转导DNA 到多种不同的细菌中。具体而言,将来自15 种不同的T7-like 噬菌体对应尾部蛋白基因gp11、gp12和gp17克隆到质粒中,同时在这些基因片段两端添加T7噬菌体基因组的同源DNA 片段,可同源重组至T7 噬菌体基因组上。用T7(缺失尾部蛋白基因)侵染携带这些质粒的细菌时,质粒表达的尾部蛋白可能组装至T7 噬菌体头部颗粒上,同时尾部蛋白基因也可同源重组至T7 噬菌体基因组,对子代噬菌体进行DNA 转导测试,成功的转导表明该质粒编码的噬菌体尾部蛋白可以组装至T7 噬菌体颗粒上。同时,对成功替换尾部的质粒进行诱变形成突变库,可以筛选出更高转导效率的噬菌体。这种方法能比较高效率扩展噬菌体转导DNA的宿主谱。
T3 噬菌体通过尾丝蛋白上的4 个不同区域结合其宿主受体脂多糖(LPSs),进而侵染细菌,当把T3 噬菌体用于抑制宿主菌时,细菌的LPS 合成途径基因能够快速产生突变,导致LPSs 缩短,进而使细菌产生抗噬菌体抗性。2019 年,美国麻省理工学院团队[28]通过对这4 个DNA 区域设计随机简并引物[图1(d)],并整合至噬菌体尾丝蛋白基因上,从而产生出了一个多样性非常大的突变噬菌体库(105~107)。用这个突变噬菌体库,可以侵染耐受细菌,并且可以侵染进一步产生噬菌体抗性的细菌,而且这些策略也可以产生能够感染新宿主的噬菌体。开发高通量方法来调节噬菌体宿主范围对于噬菌体检测与治疗具有非常重要的应用价值。
1.3 温和噬菌体的改造
噬菌体按生活周期分为温和噬菌体(lysogenic)和烈性噬菌体(lytic),烈性噬菌体能在宿主菌体内扩增并裂解细菌而使细菌死亡,而温和噬菌体的主要存在方式是将自身的基因组整合至宿主菌基因组中,随细菌基因组的复制而进行复制。前噬菌体所携带的大部分基因受到噬菌体本身表达的阻遏蛋白(repressor)的阻遏作用而不能表达,只在某些理化因素的影响下,阻遏物失活而进入裂解周期。
温和噬菌体通常应避免直接用于治疗用途,因为它们可能会把自身基因组整合至宿主菌内,从而可能会把一些毒力基因整合至宿主菌中,并且使目标菌株免疫其他噬菌体的侵染[29]。但是,某些细菌尤其厌氧菌似乎很难分离到严格烈性的噬菌体[30]。另一方面,随着宏基因组测序和生物信息学的飞速发展,科研工作者们发现溶原性细菌是广泛存在的。细菌染色体中普遍存在温和噬菌体基因组:目前已在约40%~50%的微生物基因组中鉴定到了温和噬菌体[31-32]。
合成生物学的最新进展,使得科研工作者们开发出把温和噬菌体转变为烈性噬菌体的策略[33-34],或者利用温和噬菌体携带基因线路,从而干扰细胞内的重要生命过程导致死亡[35]或使细菌对抗生素重新敏感[36]。
CRⅠSPR、BRED(bacteriophage recombineering with eletroporated DNA)等技术可用于改造温和噬菌体将其转化为可用于治疗的烈性噬菌体。比如完全删除其阻遏进入裂解周期的阻遏蛋白基因[37],去除毒力因子和整合酶等。2019 年,Spencer 团队[38]改造了1 株分枝杆菌噬菌体ZoeJ,删除了ZoeJ 噬菌体进入溶原周期的基因gp45,使其杀菌能力明显增强,研究人员给患者静脉注射由这株改造噬菌体和其他2 株噬菌体组成的鸡尾酒制剂,经过6 个月治疗后,患者的临床症状明显有所改善,并且没有产生其他副作用。Kilcher 等[33]构建了李斯特菌(Listeria monocytogenes)噬菌体P335的一株缺失主要裂解阻遏物的突变体,并通过表达另外一种不同的溶菌酶,提高了噬菌体的裂解活性。产生的改造噬菌体能有效杀灭单核细胞增生李斯特菌,并且能够抑制细菌抗性的产生。
1.4 工程溶菌酶用于杀死细菌
噬菌体蛋白除了用作生物技术工具外,还可以作为治疗用途。噬菌体的溶菌酶(lysins)是一种高度进化的蛋白,能够特异性降解细菌细胞壁肽聚糖,从而使细胞裂解。溶菌酶目前已成功用于多种动物模型中,以控制在血液、感染组织和黏膜表面等发现的致病性超级耐药菌。相对抗生素而言,溶菌酶的优点是对病原体具有特异性,对正常菌群的影响小,并且具有杀死黏膜表面上定殖病原体的能力[39]。同时,溶菌酶令人激动的地方在于细菌似乎并未对它们产生有效的抗性。纯化的溶菌酶对许多革兰氏阳性细菌具有杀菌作用,因为它们的细胞壁直接暴露在外部而可以直接与溶菌酶接触[40],目前成功的例子包括肺炎链球菌(Streptococcus pneumoniae)[41-42]、化脓链球菌(Streptococcus pyogenes)[43-44]、金黄色葡萄球菌 (Staphylococcus aureus)[45-47]和 分 枝 杆 菌(Mycobacteria)[48]等。但是,革兰氏阴性细菌的细胞壁夹在内膜和外膜之间,因此大多数天然溶菌酶无法进入。
现在有不少策略正在解决这个问题。比如Kim等[49]用广宿主谱噬菌体SS3e的溶菌酶基因表达纯化的溶菌酶,能同时抑制沙门菌(Salmonellasp.)、鲍曼不动杆菌(Acinetobacter baumanii)、大肠杆菌(E.coli)、肺炎克雷伯菌(Klebsiella pneumoniae)和铜绿假单胞菌(Pseudomonas aeruginosa),甚至对革兰氏阳性菌如金黄色葡萄球菌有一定抑制作用。Briers 等[50]开发了称为 Artilysins 的人工合成溶菌酶,该溶菌酶融合了能渗透革兰氏阴性细菌外膜的肽,能够被主动转运摄取进入外膜而发挥作用。Artilysins 对铜绿假单胞菌、鲍曼不动杆菌、大肠杆菌和鼠伤寒沙门菌都具有杀菌作用,能将细菌数量降低4~5个数量级[51]。
溶菌酶的发展潜力似乎是无限的。最近已经使用生物信息学技术鉴定了数千种不同的溶菌酶,其中许多具有新颖的蛋白结构域[52]。新型溶菌酶的鉴定,溶菌酶晶体结构的解析以及细菌细胞壁结构的深入研究,将有力推动设计和构建可能针对任何病原体的“量身定制的溶菌酶”的发展。
1.5 噬菌体展示
噬菌体展示(phage display)是一种强大的、多功能的平台技术,能在突变库中筛选出具有优良特性的多肽或蛋白[53]。在所有可用的分子展示技术中,噬菌体展示已被证明是最受欢迎的方法。2018 年,噬菌体展示技术相关科学家获得了诺贝尔化学奖。噬菌体展示技术可以迅速把表型选择与基因型联系起来,比如将多肽与丝状噬菌体M13 的外壳蛋白pⅠⅠⅠ融合,筛选出所需功能多肽,通过测序就能轻松得到对应多肽的基因型。目前已开发了T7、T4、Fd 和λ 噬菌体等展示系统,其中一些可用于筛选大型蛋白。
噬菌体展示技术可用于筛选:①B 细胞和T 细胞表面抗原决定簇。噬菌体展示法提供了一种经济、快速的方法来绘制抗原决定簇,目前,已经绘制了包括严重急性呼吸综合征(SARS)冠状病毒[54]、人乳头瘤病毒(HPⅤ)[55]和禽流感病毒(AⅠⅤ)[56]等的抗原决定簇;②选择与受体或蛋白质结合的生物活性肽,疾病特异性抗原类似物,与非蛋白质靶标结合的肽,细胞或器官的特异性肽;③肽介导的药物递送系统的开发和其他应用[57]。
抗体药物现在变得越来越重要,在不同的抗体发现方法中,噬菌体展示技术已成为最强大的方法之一[58]。截至2016 年初,通过噬菌体展示发现或进一步开发的6 种人类抗体已批准用于治疗。2002 年,阿达木单抗(Humira)成为首个获得市场批准的噬菌体展示衍生抗体,也是首个获批的人类抗体,并且它是目前市场上最畅销的抗体药物。目前,更多噬菌体展示来源的抗体正在进行临床实验[59]。可以看出,噬菌体展示技术能促进基础研究和转化医学的发展。
1.6 噬菌体辅助的持续进化
噬菌体辅助持续进化(PACE,phage-assisted continuous evolution)是将噬菌体的生长与蛋白生物学功能的进化联系起来,只有蛋白生物功能朝设计目标进化,噬菌体才能扩增,并且通过对成功扩增噬菌体的基因组进行测序来轻松获取进化序列。该系统还可以通过使用低保真的DNA 聚合酶来提高突变频率而加速进化。
在Esvelt 等[60]的开创性论文中,选择的是T7 RNAP(RNA 聚合酶)。为了迫使T7 RNAP 定向进化,将其基因克隆到没有外壳蛋白基因pIII的缺陷株M13中。而把pIII基因单独克隆一个质粒上,并用T3 启动子进行启动,这样M13 的产生取决于T7 RNAP 获得识别T3 启动子的能力。在这样的系统中,只有T7 RNAP 基因获得功能性突变才能产生后代。该系统可以全自动进行,并在不到200 h的进化过程中成功获得突变的T7 RNAP[60]。通过创造性地设计pIII表达的启动子以及伴随的选择压力,可以选择获得多种功能[61]。例如,PACE被用于进化新的氨基酰基tRNA合成酶[62],改变目标基序的蛋白酶[63]、转录因子[64],甚至使苏云金芽孢杆菌δ的突变体进化,降低昆虫对毒素的抗性[65]。
抗生素生物合成基因簇(BGC,antibiotic biosynthetic gene clusters),长度范围为15~100 kb,包含抗生素生物合成酶基因、调节基因、抗生素输出基因和自身的耐药性基因。Johnston 等[66]用PACE来实现BGC对抗生素双环霉素(BCM)的生物活性依赖适应,BGC产生具有生物活性的代谢产物BCM,从而为其生产者带来竞争优势,促进异源宿主中BCM 的产量提高,推动BGC 的进化。这一研究表明,依赖PACE,可以改善代谢途径和产品产量。
1.7 噬菌体基因元件的开发
细胞工厂的设计很大程度上依赖于模块的正交设计,即各模块各自执行自身的功能,减少模块之间和底盘细胞原有的代谢和调控网络的干扰,以简化人工设计的复杂性。噬菌体为合成生物学提供了丰富的正交元件来源,因为它们已经进化出许多不同的基因线路来控制相同的基因网络,以接管宿主细胞的资源来产生噬菌体子代。
对生物体进行编程需要对基因表达进行精确控制。一种常见的策略是通过使用启动子特异性RNA 聚合酶(RNAP)调节mRNA 的产生来执行逻辑功能。一些噬菌体自身表达RNAP基因,并且只识别噬菌体本身的启动子而不能识别宿主菌的启动子。这些酶中研究最多的是噬菌体T7、SP6和N4[67-68]。而且,许多噬菌体彼此之间的RNAP只识别自身的启动子,这种特异性使RNAP本质上彼此正交。噬菌体RNAP 在很大程度上不依赖宿主,可以在大多数细胞环境中起作用,具有应用的普遍性。另外,基因工程可以增加噬菌体RNAP的多样性。例如,通过替换或突变RNAP的特异性识别模块来改变T7-like RNAP 识别启动子的特异性[69-70]。
另外,噬菌体的转录调控基因元件也被广泛用于合成生物学中,λ阻遏物CⅠ已参与许多合成生物学应用,例如第1 个振荡器(oscillator)的设计[71]、切断开关(kill switch)[72]和脉冲检测基因线路(pulse-detecting circuit)[73]。这些例子展示了如何通过噬菌体基因元件来实现对基因表达的模块化和正交控制。
2 噬菌体合成生物学研究技术
2.1 噬菌体基因组编辑技术
噬菌体已经被发现了一个多世纪[1],但大部分噬菌体基因的功能仍然未知。在大肠杆菌T4 噬菌体中,其278 个基因仍有128 个(46%)为未知功能基因[74]。在2016年,Kyrpides课题组[75]从全球各地超过5T 的测序数据中,分析了270 多万条病毒蛋白序列,其中75%功能是未知的。大量的未知功能基因给噬菌体基因组编辑和人工合成设计带来很大阻碍。噬菌体基因编辑可以鉴定必需基因与非必需基因。
2.1.1 同源重组进行噬菌体编辑
温和噬菌体可以在细菌中稳定地保持,从而可以使用与细菌基因工程相同的方法来修改其基因组,但是烈性噬菌体的基因组由于细胞毒性等无法完整克隆到细菌中以进行后续的基因操作。
因此,烈性噬菌体基因组通常通过同源重组(等位基因交换)进行编辑,首先,将要编辑的DNA 整合到质粒上并转化至宿主细菌,然后用野生型噬菌体侵染该菌株,使噬菌体基因与质粒上设计的DNA 片段进行同源重组。然而许多非模式菌的宿主(尤其革兰氏阳性菌),没有有效的转化方案。其次重组频率通常很低,等位基因交换产生重组体的频率通常不超过1%,分离重组噬菌体工作量巨大[26]。为了增加噬菌体DNA 和重组模板(带有设计DNA片段和上下游同源臂)之间的重组频率,一个常用的策略是在宿主菌中高水平表达异源的重组功能蛋白,例如RecE/ RecT-like 蛋白质,并通过将噬菌体基因组和重组模板共电转化到重组菌株中来改造噬菌体,这种策略称为电转化DNA 的噬菌体重组(BRED,bacteriophage recombineering of electroporated DNA)[37]。电转化后,回收细菌细胞,与野生型细菌宿主混合并铺板。然后通过PCR 筛选平板中的突变噬菌斑。此技术首先由Marinelli 等[37]应用于编辑分枝杆菌噬菌体,并且此后被扩展到其他宿主菌的噬菌体,例如大肠杆菌和肠炎沙门菌[76-77],BRED 可用于删除、插入和替换基因,以及在噬菌体基因组中进行点突变。通过使用这种方法,已获得了较高频率(10%~15%)的突变噬菌体,但是需要高效率的宿主感受态细胞。
2.1.2 CRISPR/Cas:突变噬菌体的反筛系统与噬菌体编辑
CRⅠSPR/Cas 是细菌和古细菌在长期的演化过程中出现的一种对抗噬菌体入侵或外源DNA 的适应性免疫防御系统。CRⅠSPR/Cas 系统有两个主要组成部分:①CRⅠSPR 全称为成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeat),转录的RNA 用于指导识别外来DNA 序列;②Cas 充当系统的催化核心并负责切割 DNA,导致 DNA 双链断裂。CRⅠSPR/Cas 系统的作用方式包括3 个主要过程,即CRⅠSPR 适应、RNA 表达和 CRⅠSPR/Cas 干扰[8,78-79]。2020 年诺贝尔化学奖授予了CRⅠSPR/Cas9 的发现者。由于噬菌体是CRⅠSPR/Cas 系统的天然靶标,因此它们可以被用作突变噬菌体的反筛,消除野生型噬菌体并富集稀有突变体。例如,嗜热链球菌CRⅠSPR/Cas 系统能有效用于嗜热链球菌噬菌体2972 基因组中的点突变、基因缺失和基因替换筛选[80]。同样,使用 CRⅠSPR/Cas 系统来促进大肠杆菌噬菌体T7突变体的筛选[81]。
结合CRⅠSPR/Cas 系统和基因重组技术可以有效删除、插入基因。Martel和Moineau[80]利用嗜热链球菌的 ⅠⅠ-A 型 CRⅠSPR/Cas 系统在嗜热链球菌噬菌体2972基因组中,有效引入了甲基转移酶基因;Lemay 等[82]成 功 地 将 化 脓 性 链 球 菌 ⅠⅠ-A 型CRⅠSPR/Cas 系统用于编辑乳酸乳球菌的烈性噬菌体p2的基因组,实现了orf 47的单碱基突变、插入和orf 24、orf 42和orf 49的删除。Tao 等[74]通过CRⅠSPR/Cas 系统成功对大肠杆菌T4 噬菌体的基因组进行单点和多点突变、插入和缺失,在T4 噬菌体DNA 受糖基化和羟甲基化修饰保护时,编辑效率有所降低。同时,在CRⅠSPR/Cas9核酸酶的选择压力下,短至50 bp 的同源臂足以筛选到目标突变体。另外,删除了T4 噬菌体RNA 连接酶基因rnlB中的294 bp,仍然观测到了噬菌斑,证明了这种编辑策略可以用于鉴定噬菌体基因必需基因和非必需基因。Shen等[83]应用化脓性链球菌的CRⅠSPR/Cas系统在肺炎克雷伯菌噬菌体phiKpS2中,成功删除了1个启动子和9个基因。应用短至30~60 bp的同源臂足以引入点突变、移码突变、基因缺失等,有趣的是,可以删除holin基因,对phiKpS2扩增影响很小,其中移码突变可有效评估噬菌体基因是否必需。Schilling 等[84]应用 CRⅠSPR/Cas 系统在枯草芽孢杆菌噬菌体中能高效地插入、删除或突变基因。除了使用先前表征的 ⅠⅠ-A 型系统外,Hupfeld 等[85]于2007 年已经鉴定了李斯特菌的新型CRⅠSPR/Cas系统,该系统随后被改装并用于李斯特菌大型烈性噬菌体的基因改造。
以上这些研究无不表明,CRⅠSPR/Cas 系统使得噬菌体基因编辑变得简单而高效,同时,也可以用于获得未知功能基因的新信息,快速鉴定噬菌体的必需基因与非必需基因,扩展我们对噬菌体-宿主相互作用的了解[图2(a)]。
2.2 噬菌体基因组的设计与合成
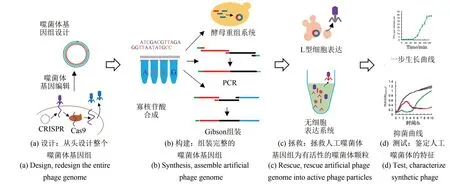
图2 噬菌体的合成生物学研究方法总结Fig.2 The methods for synthetic phages
人工噬菌体基因组的合成,分为4个步骤:设计、构建、拯救与测试(图2)。设计:去除部分非必需基因,并添加基因元件,或模块化设计整个噬菌体基因组,把设计的基因组序列拆分成利于合成的100 nt左右的Oligo序列[图2(a)];构建:合成Oligo 序列后,利用PCR 仪通过退火与延伸,把Oligo 片段组装成1 kb 长度的双链,通过酵母重组平台、PCR 或Gibson 组装成完整的噬菌体基因组[图2(b)];拯救:应用电转化、无细胞表达体系或L型细胞把合成的人工噬菌体基因组拯救成为有活性的噬菌体颗粒[图2(c)];测试:通过一步生长曲线和抑菌曲线对人工噬菌体进行初步表征[图2(d)]。
2.2.1 人工噬菌体基因组的设计
有尾噬菌体(the talled phage,Caudovius)具有较大的基因组,已知的这类噬菌体其基因组大小在16~735 kb,携带基因数量从十几个至几百个[3]。有尾噬菌体的核心基因(core gene)包括[86-87]:①结构蛋白基因模块,衣壳蛋白基因(capsid)、尾丝蛋白基因(tail fiber)、尾梢蛋白基因(tail sheath)、尾板蛋白基因(base plate)、领蛋白基因(neck)等;②DNA 复制、修复和包装基因模块,DNA 聚合酶基因(DNA polymerase)、末端酶基因(terminase)、DNA 连接酶基因(DNA ligase)、DNA 解旋酶基因(DNA helicase)、外切酶基因(exonuclease)等;③基因转录和翻译基因模块,RNA 聚合酶基因(RNA polymerase)、tRNA 等;④其他基因模块[88-89]。有尾噬菌体基因组的这些特点,使得模块化设计、合成人工噬菌体成为可能。考虑到噬菌体基因组的时序性表达,基因模块化设计时,按照早期、中期和晚期基因顺序,同时相应基因的启动子、核糖体结合位点(RBS)和终止子等也必须同时进行移动[16]。还可以通过在不同基因功能模块之间的区域整合其他功能基因,如生物膜解聚基因等,赋予人工噬菌体新的特性。如在人工噬菌体的基因组中引入PCR-tags 和在非关键区域中引入其他功能性序列如多酶切位点等[90],可方便后续的基因测序和其他工程化的处理。
同时,在自然界中,生物体的竞争优势直接取决于其存活和繁衍的能力及进化并持续进化以适应环境变化的能力,天然生物系统是自然选择进化的结果,而进化可能会导致系统复杂使其难以理解和操纵。Chan 等[90]重新设计了T7 噬菌体的基因组。设计目标是在DNA 序列上分开重叠的基因元件,实现对主要遗传元件的独特操作并简化生物系统。通过酶切酶连方式将12 179 bp 的工程DNA 替换了野生型基因组的左边11 515 bp,转化至ⅠJ1127细胞中并得到了有活性的噬菌体,所得的嵌合基因组编码的T7 噬菌体似乎保持了原始噬菌体的关键特征,同时更易于建模和操作。初步设计构建的可行性表明,可以对天然生物系统的基因组进行系统的重新设计和构建以服务于科学理解或人类意图。我们可以根据已有的知识和理解,优化设计噬菌体基因组,人工合成促使噬菌体更快地进化以满足噬菌体应用的要求。
2.2.2 人工噬菌体基因组的合成
根据设计的噬菌体基因组序列,在体外合成短的Oligo 片段(100 nt),随后通过PCR 扩增、Gibson 组装[91]或者酵母重组平台[16]进行组装,形成完整的噬菌体基因组[图2(b)]。近些年来,人工设计合成大片段噬菌体基因组的技术已经成熟,可分为体内组装和体外组装两种方式。
体外组装即通过PCR、酶切酶连、Gibson 组装和Golden Gate等方法进行噬菌体基因重组组装,然后转化至宿主菌中进行噬菌体基因组拯救。2003 年,Ⅴenter 课题组[7]人工合成了大肠杆菌的φX174 噬菌体(5386 bp),先通过人工合成42 nt长度Oligo 序列,随后通过PCR 连接成更长序列并得到其全基因组,环化后,电转入大肠杆菌中,拯救出有活性的φX174噬菌体。
Gibson 组装为一种无缝克隆的方法,通过核酸外切酶,将有重叠序列的DNA 双链消化出黏性末端,通过黏性末端序列互补,使两条链相互连接起来。在DNA 聚合酶的作用下,补全缺口,在连接酶的作用下形成完整的双链。Gibson 组装在体外进行,50 ℃反应1 h 即可完成多片段的拼接,速度快,操作方便,但是大片段拼接的通量和长度都不如酵母重组平台高[91]。
一些噬菌体(例如丝状噬菌体M13 或RNA 噬菌体MS2)的基因组足够小(<10 kb),可以在体外轻松进行组装。但是,大多数有尾噬菌体的基因组太大(>20 kb)[2],在体外操作并不容易,且在细菌体内扩增噬菌体基因片段时,可能抑制细菌生长[92]。在国内外,已有不少科研工作者对体内组装噬菌体基因组进行了探索。酵母容易摄取外源DNA 及本身拥有强大的同源重组系统,使酵母成为一个高效拼接大片段DNA 的平台[93],同时可避免噬菌体基因相关的细胞毒性。
Jaschke等[94]在酵母中从头合成了φX174 噬菌体5.5 kb长的基因组。他们还重构了基因组,分开重叠基因,调节RBS(核糖体结合位点)和启动密码子以简化噬菌体的基因组成[94]。利用这一原理,Ando 等[15]应用 PCR 方法,把整个 T7-like 噬菌体的基因组片段化,并在每个DNA 片段末端加上30 bp 长的同源臂,转化进入啤酒酵母后,可以重建各种T7-like 噬菌体基因组。同时可以在PCR过程中将各种所需的突变添加到基因组中的任何位置,从而为基因组设计提供了前所未有的灵活性。随后从酵母中提取合成的噬菌体基因组,并将其转化至大肠杆菌菌株中,从而产生突变型噬菌体颗粒[图2(b)]。
2015 年,Ando 等[15]通过 PCR 和酵母重组系统,把T7 噬菌体的尾部识别基因模块替换成K11噬菌体相应基因模块,使大肠杆菌噬菌体能侵染克雷伯菌,人工创造出了新的能侵染克雷伯菌的噬菌体;2017 年,Oldfield 等[95]以酵母重组方法从头合成了具有较大基因组的单纯性疱疹病毒(herpes simplex virus,152 kb)。
这些进展无不表明合成生物学的进步对噬菌体基因组人工合成的巨大推动,使我们从头设计、从头合成噬菌体变得可行和简便。
2.3 噬菌体基因组拯救与功能鉴定
将合成的噬菌体基因组直接化学转化或电转化导入到宿主细胞中进行拯救,需要很高的转化效率,尤其是对于大型噬菌体基因组。尽管针对某些细菌(例如大肠杆菌和铜绿假单胞菌)已经设计了高效的转化方案,但是许多其他细菌物种却极难转化,很难产生有活性的噬菌体颗粒。这成了噬菌体基因组体外合成或基于酵母合成的限制瓶颈。目前,无细胞表达体系和L 型细胞表达[图2(c)]为该问题提供了潜在的解决方案。
由于革兰氏阴性和革兰氏阳性菌的转录和翻译机制之间存在巨大差异,革兰氏阳性菌的噬菌体无法用大肠杆菌机器进行拯救。针对此限制,Kilcher 等[96]使用 L 型细菌作为拯救宿主。L 型细菌是经过改造或人工选择而不含细胞壁的细菌。当存在聚乙二醇时,细胞非常脆弱,但易于摄取外源DNA。使用单核细胞增生性李斯特菌L 型,Kilcher 等[33]拯救了常见感染革兰氏阳性细菌(如单核细胞增生李斯特菌、枯草芽孢杆菌或金黄色葡萄球菌)的多种噬菌体。作者还拯救了由Gibson 组装[91]合成的噬菌体基因组。为了证明这种新的拯救宿主的功能多样性,还拯救了多个李斯特菌温和噬菌体的基因组[图2(c)]。
无细胞表达体系(cell free system)是一种高效、快速的体外合成蛋白手段[97],即在细胞抽提物的基础上,加入外源补充成分,如RNA聚合酶、氨基酸、tRNA 等,体外把DNA 转录成RNA 并翻译成蛋白。通过对无细胞表达体系的不断优化,能用于蛋白的高通量表达及体外基因线路的验证[98]。近几年,应用大肠杆菌无细胞表达体系,Noireaux 课题组将包括 T7(dsDNA,40 kb)[77]、φX174 (ssDNA, 5.6 kb)[99]和 T4 (dsDNA,169 kb)[100]等在内的一系列噬菌体基因组,成功拯救成为有活性的噬菌体颗粒,其中T4 噬菌体颗粒组成来自大约50 个不同基因的1500 多种蛋白质。其中头部涉及24 种蛋白质组件,在尾部组件中为22 种,尾丝结构中为6 种[101],说明了无细胞表达体系在噬菌体基因组拯救方面应用的潜力。
在未来的工作中,优化这两种方法用于更广泛的噬菌体基因组拯救,扩展其普适性,将变得非常重要。
人工合成的噬菌体基因组,拯救出有活性的噬菌体颗粒后,主要检测其一步生长曲线、抑菌曲线及形态[图2(d)],进一步鉴定合成的人工噬菌体的功能。一步生长曲线,即噬菌体一个子代生长的时间和繁殖的数量。根据抑菌曲线可以初步判断噬菌体对宿主菌的抑制能力。利用透射电镜观察人工噬菌体的形态,结构蛋白模块替换后,噬菌体形态也会发生相应变化。
3 噬菌体合成生物学研究应用
由于噬菌体对宿主具有极高的特异性,因此它们在诊断和治疗细菌感染方面有着巨大的医学潜力,而我们不断提高的合成生物学研究能力进一步增强了这种潜力。通过基因工程改造噬菌体可以增加噬菌体的杀菌效率,而且噬菌体作为“致死因子”“靶向药物”“抗生素辅助剂”或“裂解酶”等的基因载体,可增加杀菌方式的多样性[92,102],同时,可以利用噬菌体的精准靶向性实现对菌落调控。人工噬菌体还被用于疫苗开发、纳米材料等。人工改造噬菌体的优势在于能够设计各种快速组装的模块化组件,可以降低技术开发成本,提高灵活性,创建新性能的噬菌体以满足不断变化的市场需求[103]。
3.1 人工噬菌体用于病原菌感染预防和治疗
噬菌体发现伊始,就被成功用于细菌感染性疾病的治疗[104],抗生素的出现,使噬菌体治疗受到阻碍,但是随着超级耐药菌的威胁日益严重[105],噬菌体治疗又重新受到重视[106-107]。实际上,噬菌体疗法在俄罗斯等国家已有近百年的历史[108]。应用噬菌体预防和治疗细菌感染具有独特的优势[10,109]:①噬菌体具有极高的宿主特异性,识别特定的宿主细菌,对整个菌群的影响比抗生素小很多;②噬菌体能利用宿主菌进行繁殖,扩增自身数量,效率高;③在成功应用噬菌体治疗耐药菌感染的案例中,还未发现噬菌体对于人体有明显的副作用。
噬菌体在预防与治疗细菌感染方面,已取得了非凡的成果。已有报道的噬菌体治疗成功案例包括口腔、肺部、生殖道、肠道、心脏和皮肤等感染;给药方式从口服、外敷到静脉注射、雾化给药等;目前,多个国家有噬菌体治疗案例的发表,如美国、法国、比利时、英国、德国、荷兰、意大利、澳大利亚、以色列、波兰和格鲁吉亚[110],中国也有成功治疗案例的报道[111]。
波兰噬菌体治疗中心2008—2010 年期间,对153 例感染了超级耐药菌的患者进行了噬菌体治疗,40%的患者根除了病原菌,获得了良好的治疗效果[112]。2016 年,大学教授 Tom Patterson 在埃及旅游时感染多重耐药鲍曼不动杆菌而处于昏迷,医生束手无策,使用鲍曼不动杆菌的噬菌体鸡尾酒制剂进行治疗,完全治愈。2019 年,Spencer 团队[38]给分枝杆菌患者静脉注射噬菌体鸡尾酒制剂,患者的临床症状明显有所改善,并且没有产生其他副作用等。
国内近几年也出现了几例应用噬菌体成功治愈超级耐药菌感染的患者。
上海噬菌体与耐药研究所已应用噬菌体治愈了数位超级细菌感染患者。2018年8月成功治愈了一例多重耐药肺炎克雷伯菌全尿路感染的患者。2019 年7 月应用非敏感型抗生素-噬菌体联用方案(non-active antibiotic and bacteriophage synergism,NABS)成功治愈多重耐药肺炎克雷伯菌尿路感染。抗生素复方新诺明单独使用,即使浓度再高,也无法抑制细菌生长,仅使用噬菌体时,抗噬菌体细菌很快出现,但是,高浓度复方新诺明和噬菌体联用时,可彻底抑制超级细菌的生长[111]。本文作者团队和深圳市人民医院重症医学科陈怀生团队、深圳市呼吸疾病研究所黄维团队共同开展深圳市第1例应用噬菌体治疗耐药鲍曼不动杆菌肺部感染的临床试验。采取抗生素和针对耐药鲍曼不动杆菌的专一性噬菌体联合雾化给药的方式,经过连续两个星期的治疗,成功清除了患者肺部感染的耐药鲍曼不动杆菌(待发表)。
目前,噬菌体治疗还处于小规模应用阶段,要满足噬菌体的大规模实际应用,仍需要解决噬菌体本身的一些限制因素[113-114]:①经验证据表明,噬菌体是安全的,但是一些研究表明,野生型噬菌体可以参与细菌毒力的进化[115]并引起免疫反应[116],这使得噬菌体的应用有着不可预知的风险;②细菌对噬菌体产生抗性的可能性;③噬菌体具有较高的宿主特异性,噬菌体治疗必须有一个足够大的噬菌体库,才能快速找到相关病原菌的噬菌体用于治疗;④噬菌体的最适应用条件,如最佳治疗时间、最佳治疗剂量、细菌被裂解时的副作用及最佳治疗方式等都需要进行系统的研究来确定。⑤为了规范噬菌体治疗方法,必须根据噬菌体产品的复杂性而更新监管策略[117]。合成生物学可以潜在解决这些限制因素,如上文提到的,通过人工改造噬菌体,可以表达生物膜降解蛋白或荚膜解聚酶提高噬菌体的侵染效率,替换或突变尾部蛋白改变噬菌体的宿主范围等。此外,人工噬菌体可应对细菌对噬菌体抗性[11,118]。细菌快速获得噬菌体抗性的最常见方式是通过突变、遮蔽噬菌体受体、CRⅠSPR 系统或调整基因表达[119]。可将噬菌体受体设计为病原菌高度保守的或者毒力因子所在的区域[120],提高细菌进化的代价。噬菌体与细菌相互对抗,已经发现了大量抗性系统的对应对抗基因元件,通过在噬菌体基因组中人工整合这些对抗元件,能够应对细菌相应的噬菌体抗性[图1(c)]。
噬菌体治疗的优势之一是噬菌体在杀死它们目标细菌的同时复制自己。然而,噬菌体不受控制的自我扩增,可能使细菌大量裂解,导致内毒素突然释放而引起相关的副作用[121]。为了解决这个问题,可以将噬菌体设计为非复制性的,仅作为纳米注射器将基因线路注入到特定细菌种群中。与噬菌体裂解细菌并产生子代相比,噬菌体预期可以将DNA注入更多种类的细菌中,因为噬菌体要产生子代,不仅要将基因组注入到细菌中,还要能抵抗诸如限制性酶、CRⅠSPR系统和毒素-抗毒素等细菌防御系统[122-123],从而扩大噬菌体治疗范围。
3.2 合成噬菌体用于病原菌检测
对病原菌进行快速而有效的检测,不仅能指导疾病诊断,减少药物的使用,同时也是感染性疾病预防控制的重点。传统的病原菌检测方法建立在对细菌的选择性培养和标准生化试验及免疫学试验之上。过程相当耗时,开发一种快速有效检测病原菌的方法具有重大的意义。
由于噬菌体能指数扩增信号且读数简单(噬菌斑),这使得噬菌体用于细菌诊断成为可能。表达报告基因的人工噬菌体提供了一种更独特而简单的方法。常见的报告基因有绿色荧光蛋白基因(gfp)[124]、细菌荧光素酶基因(lux)[125-126]和发光酶基因(luxAB)[16]等,通过噬菌体的扩增同时引起报告基因的表达,目标菌体可快速通过检测报告基因的表达而得到检测。
荧光蛋白基因较小,很容易克隆到噬菌体基因组中。被噬菌体侵染的细菌发出荧光而被显微镜观察到,而且荧光信号可被细胞分选仪识别。
从分枝杆菌噬菌体TM4 开始,已经开发了几代具有荧光表达功能的噬菌体,用于检测结核分枝杆菌和评估相应的抗生素耐药谱[127-128]。研究人员在T4 噬菌体基因组上整合绿色荧光蛋白基因(gfp),能在数小时内检测污水中的大肠杆菌[124]。
尽管如此,由于生物样品中的天然荧光容易产生背景信号,荧光噬菌体系统信噪比较差。生物发光是一种通过化学底物的酶促氧化产生光的过程,它不会遭受与自发荧光有关的背景问题,并且通常比荧光系统更亮。比如荧光素酶是一种生物发光酶,已被用于各种噬菌体系统中,以检测广泛存在的病原菌,其敏感性通常低至单个CFU。目前已有的研发包括结核分枝杆菌[129-130]、副溶血弧菌(Vibrio parahaemolyticus)[131]、鼠伤寒沙门菌(Salmonella typhimurium)[132]、鼠疫耶尔森菌(Yersinia pestis)[133-134]、痢疾志贺菌(Shigella dysenteriae)[135]、 炭 疽 芽 孢 杆 菌 (Bacillus anthracis)[126,136-137]和 致 病 性 大 肠 杆 菌(Escherichia coliO157:H7)[138]等。
更重要的是,把检测样本与合适浓度的抗生素作用后,抗生素杀死药物敏感菌而耐药菌则存活下来,然后重组噬菌体即可指示这些活的病原菌的存在,进而检测出细菌的耐药性[139]。
应用携带报告基因的噬菌体能快速、有效地指示病原菌的存在。该种方法操作简单,检测迅速,成本低,然而噬菌体检测病原菌的报道大都限于实验室而没有进入临床,其主要原因是噬菌体识别病原菌株过于单一,且噬菌体检测敏感性没有达到某些样本(如血液)的检测阈值[140]。
另外,噬菌体只能在特定的活菌内繁殖,具有特异性和避免死菌干扰的特点。然而,当很大一部分细菌种群处于持久休眠状态或者病原菌存在防御机制而噬菌体不能正常侵染时,噬菌体检测有可能产生假阴性。为了避免这种局限性,Schmidt 等[141]利用 P22 和 9NA 噬菌体尾丝蛋白对沙门菌O 抗原的特异性结合来研发类似ELⅠSA 的诊断工具,他们将其称为ELⅠSA-like 尾丝吸附测定(ELⅠTA)。ELⅠTA 可以检测和区分 O4、O4、O5 和O9 血清型的沙门菌[141]。通过使用识别不同表面多糖的尾丝蛋白或其他噬菌体蛋白,可以很容易地对其他沙门菌或其他细菌进行检测[142-143]。
3.3 噬菌体对菌群的调控
微生物群落(microbial community)遍布我们的整个星球,并在生物圈、农业、生物技术和人类健康中发挥着至关重要的作用。而我们编辑细菌群落的工具非常有限。抗生素通常是广谱的,不仅能杀死有害细菌,也能杀死益生菌。噬菌体具有高度特异性的宿主范围,因此有望能精准地敲除微生物群落中某个菌株或编辑微生物群落中的特定基因[144]。
Wang 等[145]研究了不同噬菌体组合对番茄青枯雷尔菌感染的生物防治。在单季作物中,利用田间分离的4株噬菌体组合可使青枯病发病率降低80%。发病率降低的原因是病原体密度降低,虽然病原菌产生了对噬菌体的抗性,但生长变得缓慢,同时出现了拮抗青枯菌的细菌种类,增加了群落多样性。另外,噬菌体处理不影响其他的微生物菌群。特定的噬菌体组合有潜力成为控制植物病原细菌的精密工具。
Hsu 等[146]研究了噬菌体对微生物组的动态影响。小鼠体内被明确种属的人类肠道共生细菌定殖,并通过噬菌体处理,发现噬菌体捕食不仅直接影响靶细菌,而且还通过菌群间相互作用导致对其他细菌的级联效应。代谢组学分析表明,由噬菌体捕食引起的微生物组变化对肠道代谢组有直接影响。
噬菌体M13 的基因组也可以构建包含CRⅠSPR 系统的噬菌粒,以很高的准确性和效率从微生物群中特异性去除抗生素抗性基因或毒力基因[147]。Yosef 等[36]利用 λ 噬菌体传递 CRⅠSPR/Cas 系统,并将CRⅠSPR 系统整合至细菌基因组上,这个CRⅠSPR 系统能同时针对抗生素抗性基因和该细菌的烈性噬菌体基因组。CRⅠSPR/Cas 系统一旦被输送到病原体中,便可以清除耐药菌体内的抗性基因,从而使其再次对抗生素敏感。此外,CRⅠSPR/Cas 系统保护被清除耐药基因的病原体不受烈性噬菌体侵染,从而可有效减少抗生素耐药病原体而为抗生素敏感细菌提供生态位,为医院提供了一种潜在的防控超级耐药菌的生物系统。
噬菌体合成生物学研究的进展,加上噬菌体生态学的定量表征,将允许对微生物群落进行更复杂的原位操作,使噬菌体精准调控微生物群落具有治疗疾病的潜力。开发有效的载体平台,高通量地将非致命性基因线路传递给细菌物种,对于微生物组工程将非常有价值,尤其是增加微生物组的特定细菌菌株的代谢能力将非常有用。
3.4 基于噬菌体的药物递送
噬菌体衣壳可用于将药物运输到特定组织并防止药物降解。通常使用单链RNA 噬菌体MS2,因为它的外壳蛋白在大肠杆菌中表达时能自发组装成不含遗传物质的病毒样颗粒(ⅤLP)。同时MS2 外壳蛋白可以耐受多个位置插入标签,从而能搭载的有效成分包括药物、酶、核酸、归巢肽等,使其能靶向特定动物组织部分以及提高其抗降解能力[148-150]。
噬菌体M13 是另一种常用的药物递送载体。Ⅴaks 和 Benhar[151]通过酯键将抗生素附着到 M13颗粒表面,使M13 颗粒挂满氯霉素,而血清酯酶能触发其缓慢释放。当存在葡萄球菌的抗体片段时,这些生物纳米颗粒会聚集在病原体附近,并释放出较大浓度的氯霉素,甚至杀死耐氯霉素的细菌[151]。这种靶向的,噬菌体介导的局部高浓度抗生素的递送可普遍用于治疗其他抗生素耐药性感染,这种方法使抗生素变得有靶向性,并且大大降低了所需剂量和相关毒性,在临床上非常有意义。噬菌体颗粒也可用于其他类型的药物递送。沙门菌噬菌体P22的衣壳被人工改造携带止痛肽齐考诺肽。随后对所得的ⅤLP 进行化学修饰,以在衣壳表面上连接来自人免疫缺陷病毒Tat 的穿膜肽。经过改造的ⅤLP 能够穿越血脑屏障并在各种动物和人类脑细胞模型中释放活性肽[152]。通过将Cas9 蛋白融合到噬菌体衣壳蛋白的一个片段上,ⅤLP-Cas9-gRNA 复合体能成为一个具有DNA 导向性的药物载体[153]。这些例子凸显了噬菌体在药物传递中的广泛用途。
3.5 基于噬菌体的疫苗
疫苗开发的一个重要方面是选择合适的抗原和佐剂,人体疫苗佐剂容易引起副作用,从注射部位疼痛到肝功能不全等。噬菌体疫苗作为一种新型的基因工程疫苗,其具有强大的免疫优势,噬菌体的表面结构规则、大小合适,使其能同时激活先天免疫系统和适应性免疫系统以引发免疫反应,而且在血清中的半衰期较长[154-155],因此可以将编码目的抗原的基因整合到噬菌体基因组中,从而与噬菌体衣壳蛋白融合来展示抗原性蛋白质或肽。最近,对丝状噬菌体f88 进行了工程改造,以展示来自甲型流感病毒的免疫抗原,f88-抗原复合物可以由肠道定植的益生菌大肠杆菌连续产生。该大肠杆菌菌株经过改造后可表达假结核耶尔森菌invA基因,该基因可促进其与肠黏膜的黏附,使其能够长期定居,该系统可有效预防甲型流感病毒感染[156]。这种方法的好处包括易于口服不用注射,并且由于益生菌的定植和持续递送抗原而不用二次加强接种,这使得该方法非常适合于贫困地区的疫苗接种。目前,已经开发了用于预防口蹄疫[157]、乙型肝炎[158]、EB 病毒[159]等的噬菌体疫苗。
另外,由于噬菌体已经经历了数亿年的选择过程,因此它们在侵染细菌时倾向于最大可能地扩增自己,可能引起过度免疫刺激。因此,开发人工噬菌体以控制噬菌体的扩增,对于疫苗载体有着重要意义。受RNA 噬菌体MS2 等的启发,Baker 及其同事[160]在计算机上设计了一种完全人工合成的衣壳,并包装了自己的mRNA。随后从随机突变文库中,选择出具有更高包装效率的衣壳,同时能更好地模块化插入外源片段,从而证实完全合成噬菌体颗粒也能获得类似的功能。
3.6 噬菌体纳米材料
因为易于在噬菌体表面展示具有特定功能的肽,丝状噬菌体引起材料科学家的极大兴趣。例如,筛选噬菌体展示文库,鉴定出能够将金属结晶到丝状噬菌体上以生产用于生物电子学的纳米线的肽[161-162]。
同时,M13 噬菌体外壳蛋白 gpⅤⅠⅠⅠ被修饰以显示带负电荷的氨基酸的肽,并且能够与阳离子金属(例如钴和锰)相互作用,从而形成高产量的过度氧化物。这些氧化物形成了LiO2电池电极,与碳电极相比,它们具有更高的循环寿命和比热容[163]。
噬菌体M13 也可以通过改造其外壳蛋白而能与碳纳米管结合[164]。M13 pⅤⅠⅠ蛋白第 381 位的天冬酰胺突变为精氨酸使噬菌体能选择性地识别并结合碳纳米管,从而能使碳纳米管形成特定的三维结构[165]。现在,M13 已被工程化为纳米金颗粒以及由其他金属制成的纳米颗粒的载体,展现了人工噬菌体在新型生物纳米材料中应用的功能多样性[166]。
噬菌体衣壳也可以用来增强酶的活性。把酶紧密地排列在噬菌体衣壳上,可以保护酶级联反应中的不稳定中间体,从而增加代谢通量。在噬菌体MS2 上编码SpyTag 并使用其连接两种不同的酶,将其变成了一个微反应器[167]。所得的ⅤLP 复合物使酶至少保持7 天活性,并且与用游离酶相比,产量增加了60%。由于某些底物和产物在富含脂质的环境中更易溶解,因此同一研究人员使用含脂质的噬菌体φ6 作为载体,成功开发了含脂质的纳米反应器[168]。
4 总结与展望
噬菌体一直是生物学发展中不可或缺的参与者,并为生物技术的发展做出了巨大贡献。噬菌体许多基因元件是构建合成生物学中基因回路的重要组成部分,通过噬菌体基因组学,我们看到了噬菌体惊人的多样性[169],噬菌体基因组中许多有用的功能元件有待我们去发现,如温和噬菌体裂解阻遏蛋白、终止因子和整合酶等,可以帮助我们建立精细调节的遗传回路。噬菌体溶菌酶是一个必将增长的研究领域,不少公司也正在把噬菌体溶菌酶推入临床或兽医测试的各个阶段[170]。噬菌体的衣壳结构还推动了各种有机和无机化合物的合成。
近十年来,由于大量耐药菌的出现,针对细菌感染性疾病的噬菌体疗法又重新受到人们的重视。作为抗菌剂和细菌诊断试剂,噬菌体表现出巨大的医疗潜力。2014 年,噬菌体疗法被美国国家过敏与传染病研究所列为应对抗生素抗性的重要武器之一。然而,噬菌体疗法临床上的大规模应用受到各种因素的制约。噬菌体的人工改造可能突破这些制约因素,因此,合成生物学家关注的另一个领域是创建用于预防和治疗的人工噬菌体。噬菌体基因组添加荧光基因后,可以用来检测病原菌;尾部蛋白基因的替换可以扩展噬菌体宿主谱;添加生物膜降解基因可降解生物膜,添加抑菌药物或裂解酶可以提高天然噬菌体治疗潜力等[92],并且我们预见到,随着多重耐药菌对全球健康威胁的日益增加,人们对噬菌体的临床应用需求日益迫切,噬菌体治疗将继续作为一种抗生素的补充应用而增长。我们希望人工合成的噬菌体将能够缓慢替代天然噬菌体。与天然噬菌体相比,人工构建的噬菌体更容易模块化。另外,我们必须防止这种为获得最大杀灭效率而构建的噬菌体扩散到环境中,因为这些人工噬菌体可能会使自然环境中的菌群产生对噬菌体的抗性或破坏自然菌落结构。
前文综述了噬菌体合成生物学研究的进展和应用,人工噬菌体不仅用于防控细菌感染,还用于病原体检测、食品安全、菌群调控、疫苗开发和纳米材料等,但是,自然界中噬菌体类型和基因库的巨大多样性尚未得到充分利用[10],其潜力还远没有被挖掘,噬菌体研究仍处于起步阶段。实际上,大多数天然噬菌体尚未在实验室得到培养。在已知的天然噬菌体中,许多尚未表征或没有适合的遗传操作平台。因此,到目前为止,噬菌体合成生物学研究仅涉及现有噬菌体类型的一小部分。随着DNA 测序技术的不断发展,噬菌体基因数据将急剧增加,而我们合成DNA 的能力也在不断增强,使得我们无需在实验室中分离这些天然噬菌体,可以直接通过合成这些DNA 来挖掘这些噬菌体的功能基因元件,以重构天然噬菌体或设计出结合了各种噬菌体基因元件的新型噬菌体。噬菌体基因组的人工合成必将更好地解决噬菌体大规模应用的各种限制问题,推动噬菌体应用更快发展。
