合成微生物群落研究进展
2021-01-21曲泽鹏陈沫先曹朝辉左文龙陈业戴磊
曲泽鹏,陈沫先,曹朝辉,左文龙,陈业,戴磊
(中国科学院深圳先进技术研究院,深圳合成生物学创新研究院,中国科学院定量工程生物学重点实验室,广东 深圳518055)
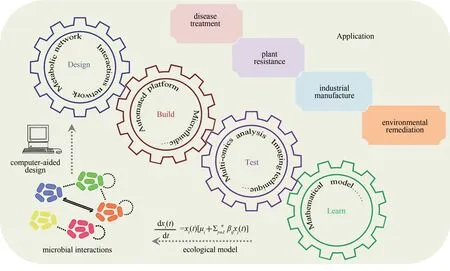
1 合成微生物群落
合成微生物群落是人工合成的多个物种共培养的微生物体系(包括野生型和基因组改造的微生物)[1-3]。微生物群落数量庞大、分布广泛,在自然生态系统中扮演着重要角色[4]。近年来,研究发现微生物群落与人体健康[5-6]、植物抗逆[7]息息相关,在天然产物生产、食品、酿造业、生物能源[8]、环境治理[9]等多个领域也有着重要作用。相比于单一菌株,微生物群落可以更好地适应环境变化,在代谢上分工协作,催化更为复杂的反应。合成微生物群落可以作为简化的模型系统,来研究微生物群落的结构和功能。随着DNA 测序技术的发展和测序成本的降低,关于微生物群落的研究不断增加[10],但是我们仍然缺乏对微生物群落功能的分子机制和生态机制的理解[11]。合成微生物群落未来的研究方向,是运用建物致知、建物致用的理念和合成生物学、定量生物学的方法,对微生物群落进行精准的调控和改造,解决人类健康、农业生产等领域的重要问题。
合成微生物群落属于合成生物学和微生物组学的交叉领域,是新兴的研究方向。根据Web of Science 检索结果和统计分析发现,近十年来,合成微生物群落的相关文章逐年增加[图1(a)]。目前为止,在合成微生物群落研究领域,美国研究者发表的文章数量占比34.3%,中国研究者占比12.7%,位居第二[图1(b)]。
本文介绍了微生物群落的生态研究,合成微生物群落的研究方法和技术,合成微生物群落的应用。
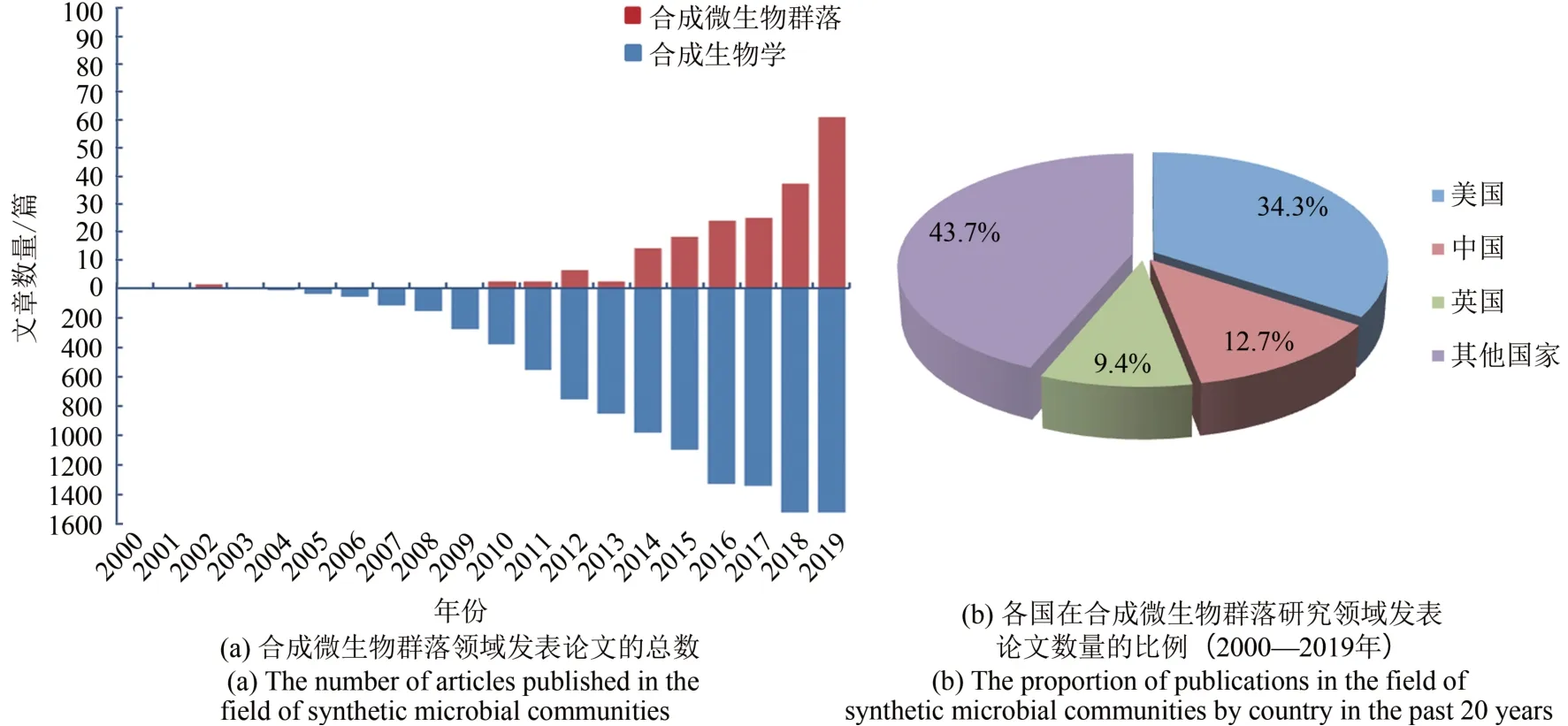
图1 合成微生物群落领域的研究趋势Fig.1 Research trends in synthetic microbial communities
2 微生物群落的生态研究
微生物群落并不是许多各自独立的微生物凑在一起,它是一个在资源竞争、营养共生、群体感应、基因水平转移等相互作用机制下形成的复杂生态系统。微生物群落结构的形态是微生物之间、微生物与宿主和环境共同作用的结果[图2(a)]。在复杂的自然微生物群落中研究这些相互作用,以现有的技术和工具十分困难;组成明确的合成微生物群落在研究物种相互作用、群落组装、生态稳定性等问题方面有着明显的优势。
2.1 微生物之间的相互作用
微生物与微生物的相互作用决定了群落结构[12-13]。通过合成菌群建立起的菌群互作网络可用于预测群落的生态稳定性[14],干预和调控群落结构,预测关键物种[15]等。根据菌群相互作用的利弊关系[图2(b)],有利(+)、不利(−)和中立(0),可分为6种不同的相互作用情况[1,16-17](表1):互利共生(+/+),偏利共生(+/0),偏害共生(−/0),寄生或捕食(+/−),竞争(−/−)和中立(0/0)。
群落中关键物种的变化,将会造成菌落结构的剧烈变化。Niu 等[27]在玉米根际人工构建了由7 个菌组成的合成菌群,通过经典的移除或添加实验,发现当阴沟肠杆菌(Enterobacter cloacae)被移除时群落会完全解体,并且由极小短小杆菌(Curtobacterium pusillum)代替,进而发现了阴沟肠杆菌为该合成群落的关键物种。
微生物之间的相互作用可以通过合成生物学的工具进行改造。目前,改造相互作用关系的研究主要利用大肠杆菌和酵母菌等模式微生物,包括[28]:
①基于群体感应系统(quorum sensing,QS)改造相互作用模式[29-32]。细菌之间可以通过QS 信号分子,比如高丝氨酸内酯(Acyl-HSL)、自诱导物-2(autoinducer-2,AⅠ-2)、寡肽类分子(AⅠ-P)等进行交流。在合成微生物群落中,可以通过改造QS 系统来调控菌群行为。例如,Balagadde等[29]通过在两株大肠杆菌建立QS 双向通讯,调控细胞自杀基因及其抑制途径的表达水平,来模拟微生物之间的捕食或寄生的关系。Kong等[33]利用群体感应分子乳酸链球菌肽(nisin)和抗菌肽(lcnA) 的模块化合成途径在乳酸链球菌(Lactococcus lactis)模拟了菌群的6 种相互作用模式。此外,Liu 等[13]在两株乳酸链球菌中分别表达抗菌肽(lcnG)的α 亚基和β 亚基,模拟了菌群间的合作关系,并且证明了相互作用的改变是菌群结构变化的关键因素。
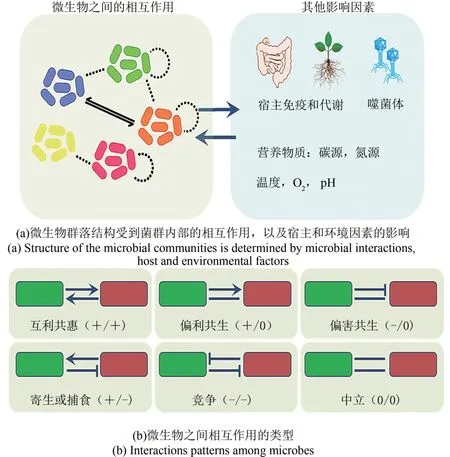
图2 影响合成微生物群落结构的重要因素[14]Fig.2 Ecological factors that shape the structure of microbial communities[14]

表1 微生物相互作用类型的研究案例Tab.1 Examples of different types of microbial interactions
②基于营养互补关系改造相互作用模式[18,34-35]。例如,Shou 等[18]通过在赖氨酸营养缺陷型酿酒酵母中引入腺嘌呤合成途径、同时在腺嘌呤营养缺陷型酿酒酵母中引入赖氨酸合成途径,构造营养互补的酵母菌群,来模拟微生物之间的互利共生关系。
2.2 影响微生物群落的其他因素
定植于宿主的微生物群落受到宿主代谢产物和免疫系统的影响。例如,三萜类化合物是植物产生的次级代谢物中结构最为丰富的化合物家族之一,Huang 等[36]发现其在调节拟南芥根际微生物群落中起着关键作用。此外,植物免疫系统对微生物组的组装具有积极作用,并通过响应环境变化控制微生物稳态[37]。在动物宿主的肠道微生物研究中,发现了宿主对微生物组成的选择作用[38]。Roeselers等[39]比较了自然栖息地生长的斑马鱼和实验室设施中世代饲养的斑马鱼的肠道细菌群落,发现它们的肠道菌群成员惊人地相似,都有一个共同的核心肠道菌群。
微生物群落的结构还受到所处的环境中诸多因素的影响,比如碳源、氮源、pH、氧气含量、温度等[40]。大量环境样本的测序数据表明微生物的群落结构和功能基因的表达受到环境因子的影响[41-43]。这些环境因子可以直接影响某些物种的生长,也可以改变微生物相互作用关系[44],进而驱动群落结构的改变。Hu等[30]通过改变环境因子造成了合成生态系统的灭亡、互利共生和偏利共生等动力学行为。Garcia 等[45]发现环境温度的变化会造成微生物群落多样性和功能关系的变化。
此外,微生物群落与噬菌体密切相关。噬菌体具有高度宿主特异性,可以降低特定宿主菌的丰度,释放生态位空间和营养物质,从而改变微生物群落的组成和多样性。Wang等[46]利用田间分离的4株噬菌体组合,显著降低了青枯病病原菌的丰度,并且增加了群落多样性。Hsu 等[47]在携带组成明确的菌群的小鼠体内,研究了噬菌体对肠道菌群的影响,发现噬菌体可以直接减少靶细菌的数量,并通过菌群互作产生级联效应,影响其他物种和肠道代谢组。
3 合成微生物群落的研究方法
Lawson 等[48]提出以设计-构建-测试-学习(design-build-test-learn)循环为核心(图3),开发新的实验和数据分析技术,促进微生物群落生态学的基础研究,推动合成微生物群落的发展,进而实现对微生物群落组成和功能的精准调控和改造。以下将从设计、构建、测试、学习4个方面,介绍微生物群落研究中重要的方法和技术。
3.1 设 计
微生物群落互作网络高度复杂,传统微生物群落设计通常采用自上而下的方法,即通过在生物反应器中优化物理化学参数,实现所需群落功能的最大化。然而,自上而下的方法忽略了微生物群落的代谢网络和成员之间的相互作用,限制了我们通过分子尺度对群落功能进行优化。随着测序技术和多组学分析手段的出现,使得基于菌群相互作用和代谢网络的自下而上的设计方法成为可能。
基于对菌群互作和代谢网络模型的理解,设计合成菌群来获得具有特定功能的微生物群落,例如代谢产物表达、宿主互作等。Bucci 等[49]构建的微生物组-免疫系统模型,用于预测特定肠道菌群的稳态组成以及其诱导Treg 免疫细胞的能力,可以辅助设计最优的诱导Treg 细胞的合成菌群。Zhao 等[50]从产丁醇的工程大肠杆菌出发,通过自下而上的设计方法以及基因工程的手段,构建出对己糖和戊糖高效协同利用的“Y-型”合成菌群。在该研究中,为了实现菌群共存和代谢分工的目的,分别构造了特异性利用己糖和戊糖的菌株,两菌株产生相同的中间代谢产物,最后经过丁醇合成途径形成丁醇。设计合成菌群来研究微生物生态学的基本原理,进而指导对复杂微生物群落的精准改造。Liu 等[13]通过构建相互作用可调节的合成菌群来模拟自然界菌群相互作用的变化,发现相互作用的可变性在生态系统演替中的关键作用。进而将相互作用的可变性引入到数学模型中,成功解释了实验结果。然而,自下而上的设计方法在对非模式生物的研究中还存在各种问题,包括代谢网络模型的准确性和完整性,大量基因和代谢产物的功能未知,对生态系统和代谢调节的理解有限[51]。
3.2 构 建
自下而上构建合成微生物群落的前提是微生物可培养。培养组学的发展使得自然微生物群落中有更多的微生物可以被培养,使得构建组成更加多样的微生物群落成为可能。培养组学利用多种培养条件,结合菌落快速鉴定技术,实现菌种资源的发掘和鉴定[52-54]。Lagier 等[55]利用多种培养方法,结合飞行时间质谱(MALDⅠ-TOF-MS)和16S rRNA 测序分析,对30 000 个菌落进行快速鉴定,从人体粪便样品中分离出分布在117个属中的340种菌。Bai等[56]通过改良微生物培养条件和分类鉴定技术,分离了12 500 个拟南芥根系菌,占自然界生长的拟南芥根际菌的64%。此外,常规培养基难以培养微生物菌群中的所有菌种,基于16S rRNA 或者基因组序列预测的特定培养基,大大增加了可培养的微生物数量[57-58]。
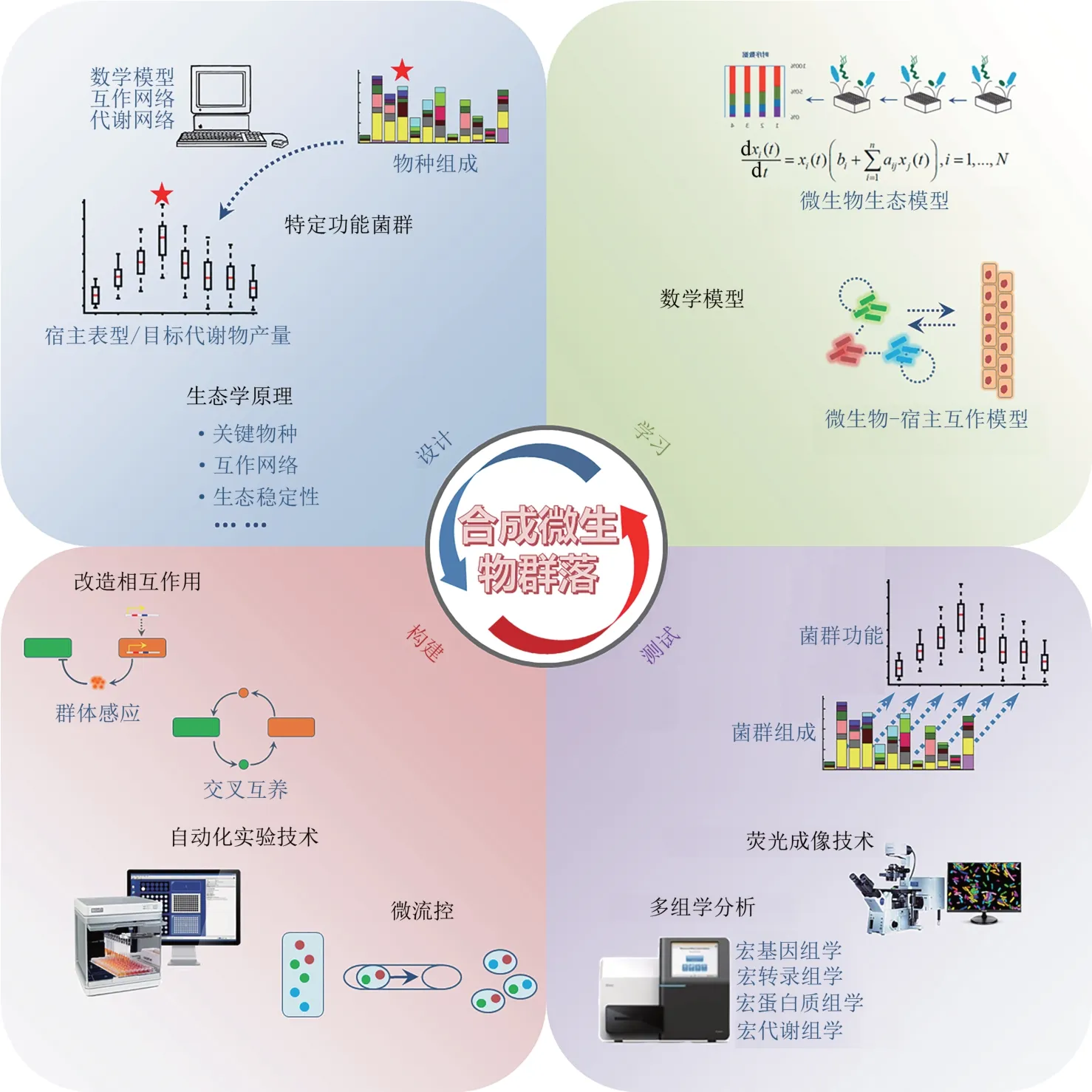
图3 “设计-构建-测试-学习”循环[12]Fig 3 The schematic diagram of Design-Build-Test-Learn principle[12]
为了提高构建的通量和精准度,构建合成微生物群落需要借助自动化实验技术。Ⅴenturelli等[12]通过自动移液工作站构建了12个人体肠道菌种的两两组合菌群,通过拟合广义Lotka-Ⅴolterra生态模型,发现可以通过两两相互作用来预测群落组装的过程。自动移液工作站可以构建不同物种数量和比例的组合,但是实验通量(约103个群落/天)有一定局限。微流控技术可同时组装和测量数以万计的微生物群落的活动,能够对微生物相互作用进行高通量筛选,大大提高了构建和测试的通量[59-61]。Kehe 等[62]开发的 kChip 技术,达到每天组装10 万个合成群落的规模,可以在不同环境条件下,如抗生素、天然产物、碳源等,对荧光标记和非荧光标记菌株进行快速功能分析。研究人员等[63]通过kChip 技术,高通量构建了>15 万个成对菌群,发现了正相互作用的普遍性及其和环境中碳源的关系。然而,微流控技术的应用仍存在一些问题,包括精准检测液滴中的微生物、避免交叉污染等[59,61]。
3.3 测 试
目前用于研究微生物群落组成和功能的测试方法包括:多组学分析(宏基因组学、宏转录组学、宏蛋白质组学和宏代谢组学),用于鉴定微生物群落的物种和功能[64-66];荧光成像技术,用于研究微生物群落的时空分布[67];同位素示踪技术,用于测量微生物组的代谢通量[68-69];质谱成像技术,用于研究微生物群体之间的化学相互作用[70-72]。
多组学分析技术,包括扩增子测序、宏基因组学、宏转录组学、宏蛋白质组学和宏代谢组学的进步,有助于我们打开微生物系统的黑盒子,了解特定的基因、物种和代谢途径在生态系统中扮演的角色,对潜在调控机制和功能基因进行研究和发掘[73]。随着对人体微生物组、环境微生物组的多组学研究不断深入,与特定疾病或环境相关的微生物物种、基因、蛋白质和代谢产物正在被发现。宏基因组数据可以有效互补扩增子测序中所得到菌种的功能基因信息,为预测微生物群落的潜在功能特性和分离培养提供了重要依据[74]。宏转录组学和宏蛋白质组学提供了微生态系统中存在的RNA 和蛋白质丰度[75-76]。将宏基因组、宏转录组学和宏蛋白质组的数据相结合,可以分析微生物群落在特定环境下的基因组成和基因表达信息,为进一步的功能研究提供基础[77-79]。
自然界中的微生物群落存在明显的空间分布异质性[80-83]。使用荧光成像技术对微生物进行原位观察,可帮助我们获取微生物群落的丰度以及空间结构信息。比如,通过研究定植于宿主的微生物群落的空间结构,可以揭示微生物之间的相互作用以及宿主-微生物的相互作用。现有的荧光成像技术主要包括:通过改造特定的微生物表达荧光蛋白,通过化学修饰的方法对特定微生物进行代谢标记,以及基于16S rRNA 的荧光原位杂交技术[84-87]。对活细菌进行标记的荧光蛋白和代谢成像,可以实时示踪微生物在时空上的动态变化[85-86]。荧光原位杂交技术可以定位微生物在微米尺度上的生物分布,有助于充分理解微生物群落的组装、生理生态和相互作用[88];根据所设计探针的特异性高低,荧光原位杂交可以实现从细菌种水平到门水平等不同层次的标记。结合高通量的测序技术,基于荧光成像技术获取的空间结构信息为理解复杂微生物群落的组成、代谢、与宿主的互作等提供了重要的基础[89-90]。
3.4 学 习
通过对测试阶段获得的实验数据进行定量分析和数学建模,完成对微生物的生态网络、微生物代谢、微生物与宿主的互作等机制的学习,来指导合成菌群的调控和优化,开启新的设计-构建-测试-学习循环。
数学模型可以帮助我们理解微生物群落的组成和相互作用[91],预测抗生素、益生元等干预对群落结构和功能的影响,进而指导合成菌群的设计[73]。通过对比实验结果与数学模型预期的差异,可以帮助我们发现新的作用机制、通路或者互作关系。例如,如果基于全基因组的代谢通路模型的预测结果与实验得到的群落的生理现象不符,则说明代谢网络不完整或基因标注有错误,进而可以增加我们对代谢通路的认识[92]。数学模型可以研究常规实验难以研究的生态系统特性,帮助我们理解实验现象背后的机制。基于生态网络的数学模型,发现了群落稳定性与负相互作用在群落中的比例有关[14];揭示了群落的多稳态[93-94]和优先效应[95-96]等现象的机制。
目前,合成微生物群落研究中比较常见的数学模型之一是广义Lotka-Ⅴolterra 模型(generalized Lotka-Ⅴolterra model,简称 gLⅤ),用于描述不同物种的丰度随时间的变化[12,97-101]。例如,Bucci等[102]开发的MDSⅠNE软件利用菌群丰度的时序数据拟合gLⅤ模型的参数,可以准确预测合成微生物群落的动态变化,识别维持群落稳定性的关键菌种,优化用于抑制病原菌生长的合成群落设计。此外,常见的数学模型包括:代谢网络模型,用于描述群落中每个成员的代谢流[103];消费者-资源模型,用于描述物种丰度和关键代谢物浓度随时间的变化[104-106]。
4 合成微生物群落的应用
4.1 疾病治疗
人体肠道菌群是一个动态的复杂微生物群落[107-108],在新陈代谢、抵御病原体入侵和调控免疫系统反应等方面发挥着多种作用[109-111]。人体肠道菌群与宿主疾病密切相关,如肥胖[112-115]、糖尿病[116-119]等。调控肠道菌群被认为是治疗菌群失衡相关疾病的最有潜力的方法之一[120-121]。粪菌移植(fecal microbiota transplantation)已被用于治疗艰难梭菌感染[122]、炎症性肠病(inflammatory bowel disease)[123]等。例如,Zhang等[124]利用FMT临床治疗了一例严重克罗恩病患者。然而,健康的粪菌移植菌群资源有限,而且组成复杂,治疗机理不清晰,存在一定风险。相比于粪菌移植,合成微生物群落具有明显优势:组成明确,与宿主的相互作用相对清晰,并可重复制备[125]。近年来,合成微生物群落被用于改善艰难梭菌感染、自身免疫病[126]、炎症性肠病治疗[127]和辅助癌症免疫治疗[128]等,在动物模型中取得了良好的效果,部分已经进入临床试验阶段。Honda 团队[126]根据梭状芽孢杆菌在增加Treg细胞的细胞丰度和诱导重要抗炎分子方面的作用,分离并筛选了17 株梭状芽孢杆菌,通过口服这些菌株,改善了成年小鼠的结肠炎和过敏性腹泻症状。基于这项研究构建的合成菌群ⅤE202,被用于治疗炎症性肠病,目前已进入临床试验。此外,Honda 团队[128]从健康的人体粪便中分离出11 株可以诱导γ-干扰素产生的CD8 T细胞的菌株,构建的合成菌群被用于癌症的免疫辅助治疗,目前也已进入临床试验。
4.2 植物生长和抗逆
定植于植物宿主的微生物群落,如根际微生物群落,在调节植物健康生长和胁迫抗性方面发挥着重要作用[129]。在植物营养方面,Zhang 等[130]发现水稻根系微生物群落有利于植物对有机氮的利用;Finkel 等[131]运用合成微生物群落揭示植物磷胁迫响应与其共生微生物的互作关系;研究人员通过比较植物突变株与野生型的微生物群落,发现水杨酸介导的植物先天免疫[132]和磷酸饥饿途径[133]对微生物群落结构的影响。同时,植物共生微生物可以有效预防病原菌的入侵[134]。例如:Durán Paloma 等[135]发现拟南芥根际微生物可使其免受丝状真菌的侵害;南京农业大学微生态与根际健康团队[136]利用合成菌群增强细菌间竞争性互作,以更好地预防土传病害青枯菌的入侵;Carlström 等[96]运用合成群落,鉴定出影响植物叶面微生物组的关键菌株以及其调控微生物群落抵御外来入侵菌种的生态学理论。此外,通过组成明确的合成微生物群落和植物基因型以及成分确定的营养基质和培养条件,使得实验具有更高的可重复性,便于揭示微生物群落结构与植物表型的因果关系[137]。目前合成微生物群落的研究主要是模式植物中的基础研究,在农业领域应用的主要肥料和农药产品为单菌制剂[138],简单群落如3~5 个合成菌群制剂在国外公司的少许产品中也有推广[139]。例如,美国的Pivot Bio以及Joyn Bio生物公司,将合成生物学应用于农业生产,对植物共生固氮微生物进行改造和应用,以减少氮肥对环境的影响。
4.3 在其他领域的应用
此外,合成微生物群落在酿造业、生物能源、天然产物合成、环境治理、生态恢复等多个领域有着广阔的应用空间[140]。
4.3.1 酿造业
在酒类饮料以及食品调味物质酿造中,可以通过合成微生物群落增加产量和风味物质。王丽娟[141]利用酒醪对醋醅微生物进行批次传代驯化,形成了醋酸菌WS8(Acebactersp.WS8)、乳杆菌L7(Lactobacillussp.L7)、乳杆菌L8(Lactobacillussp.L8)和不可培养的乳杆菌clone22(Lactobacillussp.clone22)为主的功能微生物群落,其产醋酸和乙偶姻能力显著提高。Wu 等[142]将芝麻香型白酒酿造体系中的5个优势种共培养后,积累了更多的风味物质。
4.3.2 生物能源
利用可再生的木质素和纤维素生产的乙醇被认为是未来化石燃料的替代品,然而纤维素的降解效率低限制了其应用[143]。Patle 等[8]构建了由运动发酵单胞菌(Zymomonas mobilis)和假丝酵母(Candida tropicalis)组成的合成菌群,将来自果蔬的废弃物转化为乙醇,乙醇得率高达97.7%。
4.3.3 天然产物合成
基于合成菌群的混合发酵体系,通过引入微生物之间的相互作用,激活沉默基因的表达,是诱导次生代谢的重要方法[144]。Schroeckh Ⅴolker等[145]将构巢曲霉(Aspergillus nidulans)和吸水链霉菌(Streptomyces hygroscopicus)共培养,激活了构巢曲霉中聚酮合酶PKS的基因簇。Zhou等[146]利用酿酒酵母和大肠杆菌分工合成了抗癌药物紫杉醇前体,同样的方法也用于合成丹参酮前体和功能化倍半萜。
4.3.4 环境治理和生态恢复
农药的过度使用已经对环境造成了严重破坏甚至威胁到公众健康[147]。Dejonghe 等[148]发现农药利谷隆降解菌群中,贪噬菌WDL1(Variovoraxsp.WDL1)可以用利谷隆为碳氮源生长,并将其降解为中间产物,然后食酸戴尔福特菌WDL34(Delftia acidovoransWDL34)和睾丸酮丛毛单胞菌WDL7(Comamonas testosteroniWDL7)可以降解这些中间产物。通过设计具有特定污染物降解功能的合成菌群,可以对环境污染进行原位或异位治理。陆地生态系统功能和稳定性取决于植物的组成和多样性,而丛枝菌根真菌(arbuscular mycorrhizal fungi)对于维持植被的多样性至关重要[149]。合成菌群可应用于退耕还林、草场生物多样性的恢复。
5 展 望
合成微生物群落是合成生物学和微生物组学两个领域交叉形成的新兴前沿研究[150]。在本文中,我们总结了合成微生物群落的研究进展,主要聚焦微生物群落生态学、设计-构建-测试-学习的研究方法、合成菌群在多个不同领域的应用。然而,合成微生物群落研究仍然存在很多重要的问题有待解决:
①如何构建可控、稳定的微生物互作网络。目前针对微生物群落中物种相互作用的改造仍然局限于模式生物的体系,未来的研究需要构建更多的细胞间交流元件和调控网络。同时,需要进一步理解微生物相互作用网络和群落稳定性的生态学规律。
②如何表征和控制微生物群落的空间结构。目前对不同环境下微生物群落的三维结构进行原位分析存在很多局限,需要开发表征和重塑微生物群落三维结构的方法。
③如何实现对微生物群落功能的精准控制。许多疾病和共生微生物群落的失调相关,但是目前缺乏精准调控微生物群落组成和功能的手段。
随着微生物组学的发展,我们对微生物群落功能的分子机制和生态机制的理解不断深入;同时,合成生物学、定量生物学的发展将为微生物群落的精准调控和改造提供重要工具。合成微生物群落未来的研究,将坚持建物致知、建物致用的理念,探索复杂微生物群落的基本科学规律,同时致力于解决人类健康、农业生产等领域的重要问题。
