新疆哈萨克族传统风干肉中真菌多样性分析
2021-01-20王俊钢李宇辉刘成江郭安民岳建平
王俊钢,李宇辉*,刘成江,郭安民,岳建平
1(新疆农垦科学院 农产品加工研究所,新疆 石河子,832000) 2(农产品加工重点实验室(新疆农垦科学院),新疆 石河子,832000) 3(额敏县新大同创生物工程有限责任公司,新疆 额敏,834699)
风干牛肉是新疆哈萨克族传统的发酵肉制品,也是山区牧民过冬必备食品[1]。新疆北疆牧区的牧民通常在11月到来年1月制作风干肉,牛肉经过分割切条、腌制后自然风干,制作时间约为2~8个月不等,其产品类型和我国的火腿[2]、土耳其的pastirma[3]和意大利的bresaola[4]相类似。近年来,随着新疆旅游业的发展,风干肉作为新疆的特色旅游食品,其消费量逐年上升。传统哈萨克族风干肉的发酵主要依靠天然多菌种混合发酵,包括乳酸菌、微球菌、酵母菌、霉菌等各种微生物[5],发酵体系中微生物菌群结构复杂,其中真菌所产生的酶可以对肉中的糖类、脂肪和蛋白质进行分解,生成酮醛酯等风味物质提高产品品质、赋予产品特殊的风味和色泽[6-9]。FLORESM等[10]研究发酵香肠发现德巴利酵母菌(Debaryomycesspp.)对成熟过程中挥发性物质的产生有重要影响,通过抑制脂质氧化产物的产生、促进乙酯的生成,可以产生香肠特有的香气。SIRINE等[11]研究表明青霉菌具有抗氧化、防止酸败和保持色泽的作用,还可以防止香肠表面形成黏性物质。
传统食品中的微生物菌落结构更加复杂[12-14]。为了更加清楚地了解传统发酵食品中微生态体系,可借鉴生态学中对微生物多样性研究技术和方法[15]。高通量测序技术是微生态学研究方法的一种,能够快速全面的分析微生物结构复杂的样品[16],QUIJADA等[17]使用高通量测序技术对西班牙发酵香肠“Chorizo de León”中微生物菌群多样性研究发现,不同地区发酵香肠中微生物在亚属分类等级上有明显差异;REBECCHI等[18]采用Illumina高通量测序研究猪肉、牛肉和羊肉腊肠中微生物的多样性,结果显示不同动物来源的香肠微生物多样性存在显著差异。
由于地理环境的差异,使不同来源的风干肉中微生物多样性有所不同[19]。但目前对新疆风干肉中微生物菌群多样性还没有系统的研究,尤其是真菌,其对环境的要求更低,适应性更强,但在风干肉中的群落结构还不清楚,因此很有必要对新疆风干肉中的真菌多样性进行研究,明确真菌结构组成,为风干肉安全性研究和下一步挖掘专用发酵剂提供理论基础,同时也可为后期产品的工业化生产提供依据。
1 材料与方法
1.1 样品
选取新疆北疆4个哈萨克族聚居的牧区为采样对象,全部为自然发酵。制作时间都为当年11月份制作的新鲜风干肉,且自然风干时间都为40 d,采集的样品单独装入采样袋,放在车载冰箱(0~4 ℃)条件下24 h内运回实验室,存贮在-80 ℃冰箱,备用。详细分组情况如表1所示。其中采样地方相同的用相同的大写字母,数字代表来自于不同的家庭。
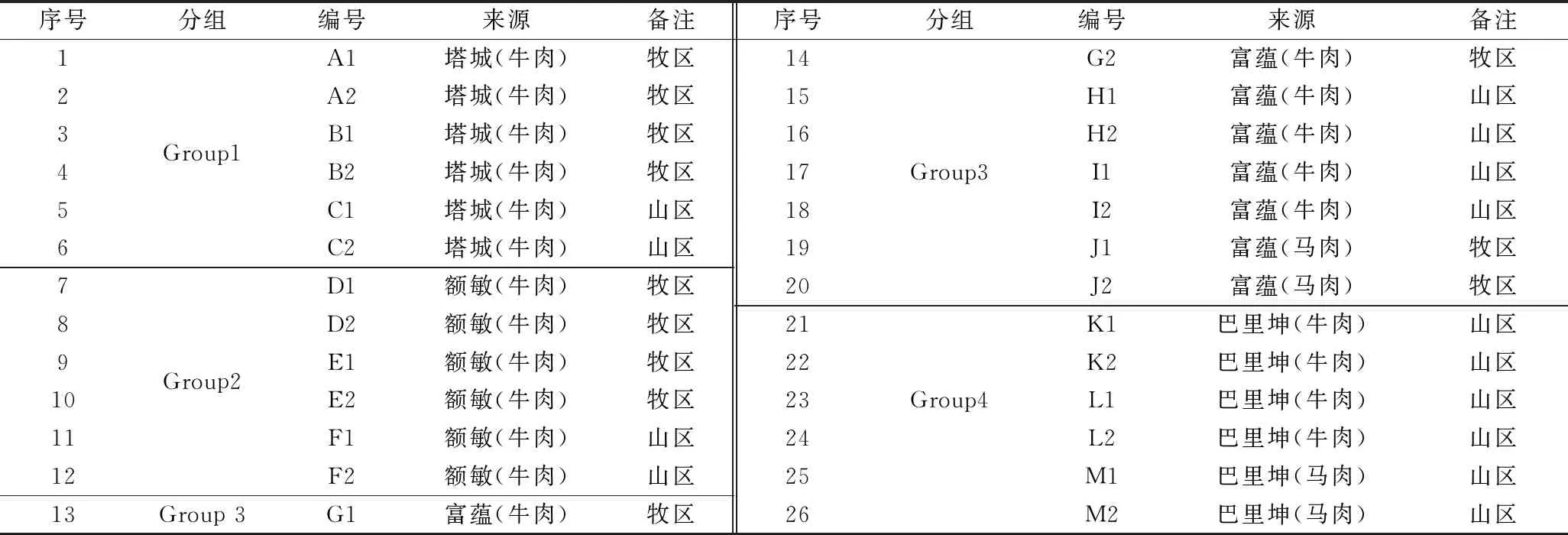
表1 样品来源及分组Table 1 Source and grouping of air-dried meat samples
1.2 试剂
强力土壤®DNA提取试剂盒,美国OMEGA公司;2×TaqMaster Mix (Dye Plus),南京诺唯赞生物科技有限公司;Magnetic Soil and Stool DNA Kit试剂盒,天根生化科技(北京)有限公司;Axygen凝胶回收试剂盒,Axygen美国;琼脂糖,西班牙Biowest公司。
1.3 仪器与设备
高速冷冻离心机(Eppendorf 5810R),德国Eppendorf AG公司;精密电子天平(BSM-220.4),上海卓精;Bio-Rad电泳仪、PCR仪(T100)、凝胶成像系统(HP1020),美国Bio-Rad伯乐公司;立式压力蒸汽灭菌器(LDZX-30KBS),上海申安医疗器械厂。
1.4 方法
1.4.1 真菌DNA提取与检测
直接使用DNA提取试剂盒提取样品中真菌的总DNA。提取方案按照试剂盒说明书进行,每个样品提取3次,提取的DNA用乙醇沉淀并提纯。DNA浓度用分光光度法检测,之后在1.2%琼脂糖凝胶电泳上检测DNA的完整性。
1.4.2 真多样性分析
选取真菌的ITS1 区序列进行高通量测序分析。使用PCR仪进行PCR扩增,引物选择ITS1 区通用引物ITS1F:5′-CTTGGTCATTTAGAGGAAGTAA-3′;ITS2:5′-GCTGCGTTCTTCATCGATGC-3′,扩增出的片段用1.2%琼脂糖凝胶电泳检测,使用凝胶成像系统进行观察,效果较好的样本于2%琼脂糖凝胶电泳切胶回收,以回收产物为模板进行1次8 循环的PCR 扩增,将Illumina平台测序所需要的接头、测序引物、标签序列添加到目的片段两端。全部PCR 产物采用Axy Prep DNA 凝胶回收试剂盒进行回收,并用FTC-3000TMReal-Time PCR 仪进行荧光定量,均一化混匀后完成文库构建,在IlluminaMiSeq 2×300 bp平台上完成测序。
PCR反应体系为:4 μL 5×FastPfu Buffer,2 μL 2.5 mmol/L dNTPs,0.8 μL 5 μmol/L Forward Primer,0.8 μL 5 μmol/L Reverse Primer,0.4 μL FastPfu Polymerase,0.2 μL BSA,10 ng Template DNA补dd H2O至20 μL。程序如下:95 ℃预变性5 min,95 ℃变性60 s,50 ℃退火60 s,72 ℃延伸60 s,循环数为25,72 ℃最终延伸7 min。扩增结束后,采用2%琼脂糖凝胶电泳检测扩增产物的纯度。使用凝胶回收试剂盒切胶回收PCR产物进行文库构建后,在IlluminaMiSeq PE 300平台上进行高通量测序分析。
1.5 数据处理及分析
使用QIIME 中的PyNAST[20]对代表性序列进行比对并去除低质量、接头污染和低复杂的序列,用FLASH[21]对有效序列进行拼接,根据序列首尾两端的barcode 和引物序列区分样品。再用UPARSE[22]软件将序列聚类到97%的操作分类单位(operational taxonomic unit,OTU)[23]。筛选重复序列,使用Uchime软件去除嵌合体[24]。所有数据分析使用R语言[25]和Python(https://www.python.org/)进行。结合热图或聚类分析计算物种丰度与环境因子的相关性及其显著性[26]。
2 结果与分析
2.1 样品ITS1基因序列质量评估及Alpha多样性分析
26个样品测序共获得 2 059 240对有效的基因序列条带,平均每个样品产生35 455条有效序列。为了得到更高质量及更准确的生物分析结果,对双端 Reads拼接、过滤后共产生1 936 392条优化序列,每个样品平均产生74 477条优化序列。
根据97%相似性水平下的OTU信息,采用Alpha多样性指标的Chao1、Ace、Shannon、Simpson 指数以及群落覆盖度指数Coverage等[27]对样品中物种丰度和多样性进行评估,结果如表2所示。所有样品的OTU覆盖率为99.98%~100.00%,说明样品中真菌种类几乎都能被检测到,测序结果可以很好的反应样品中真菌的真实情况。OTU数量越多说明样品中的真菌丰度越高[28],由表2可知,样品G1、G2、J1、J2、L1和L2的OTU数量在100以上,其他样品中的OTU数量基本都在100以下。这说明G1、G2、J1、J2、L1和L2这6个样品中真菌种类较多,其中G1、G2、J1、J2源自于富蕴县,L1和L2属于巴里坤地区,这可能是由于当地环境所引起的。来源于富蕴县的4个样品均采自水草丰富、环境相对较好的牧区,微生物更加容易生长,因此真菌的种类相对较高。来源于巴里坤山区的2个风干牛肉样品采自牧民家,这可能是和牧民的制作方式有关,卫生条件直接关系到产品中的微生物情况。图1-a表示真菌群落OTU 数目的Venn
图,该图可用于统计多个样本中共有和独有的OTU 数目,4组共有的OTU 数目84个,26个样品种共有的OUT数目仅有13个(图1-b),其中塔城(Group1)、额敏(Group2)、富蕴(Group3)和巴里坤(Group4)地区样品中OTU数目分别为125、133、261和316, Group1和 Group2之间差异不显著(P>0.05),其他组之间差异显著(P<0.05)。
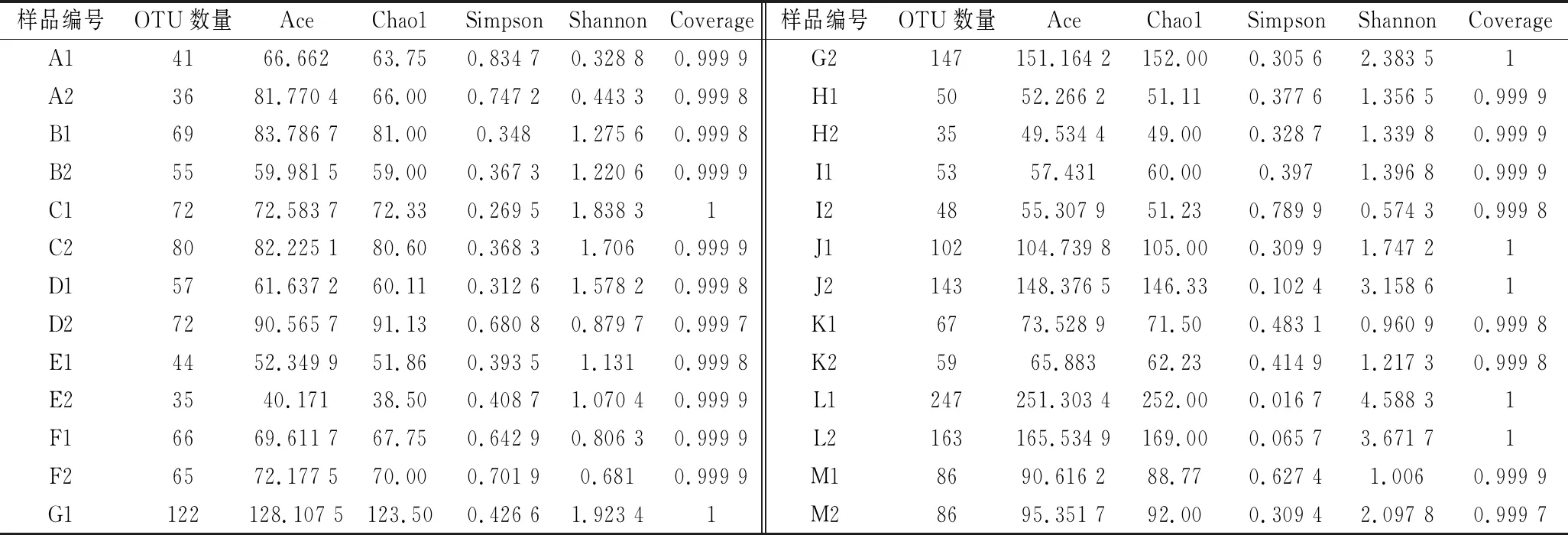
表2 Alpha多样性指数统计Table 2 Alpha diversity index statistics
稀释曲线反映了持续抽样下新OTU(新物种)出现的速率;在一定范围内,随着测序条数的加大,若曲线表现为急剧上升则表示群落中有大量物种被发现;当曲线趋于平缓,则表示此环境中的物种并不会随测序数量的增加而显著增多(图2-a)。Shannon指数越大,则表示OTU种类越多,物种越丰富,表明样品中已涵盖绝大多数的微生物信息,当曲线趋于平坦时,说明测序数据量足够大,OTU种类不会再随着测序量的增加而增加。由图2-b 可知,对26种风干肉制品中的真菌进行的高通量测序分析表明,当测序量<40 000时,OTU数呈明显上升趋势,表明此时仍未检测到更多种类的菌种。当测序数量逐渐增加时,样品的OTU数量增加,但趋势趋于平稳。当测序深度达到60 000时,所有样品的OTU变化均趋于平稳。随着测序量的增加,真菌的多样性几乎不变,这表明现有的测序量已经可以反映样品中真菌的丰度信息。采用随机抽样的方法对序列进行测序,以绘制出绘制的序列数和可代表的OTU数的曲线。即可知多数样本在序列数接近60 000时,OTU已经接近饱和,说明现有测序量已经可以反映出样品中真菌丰度信息。
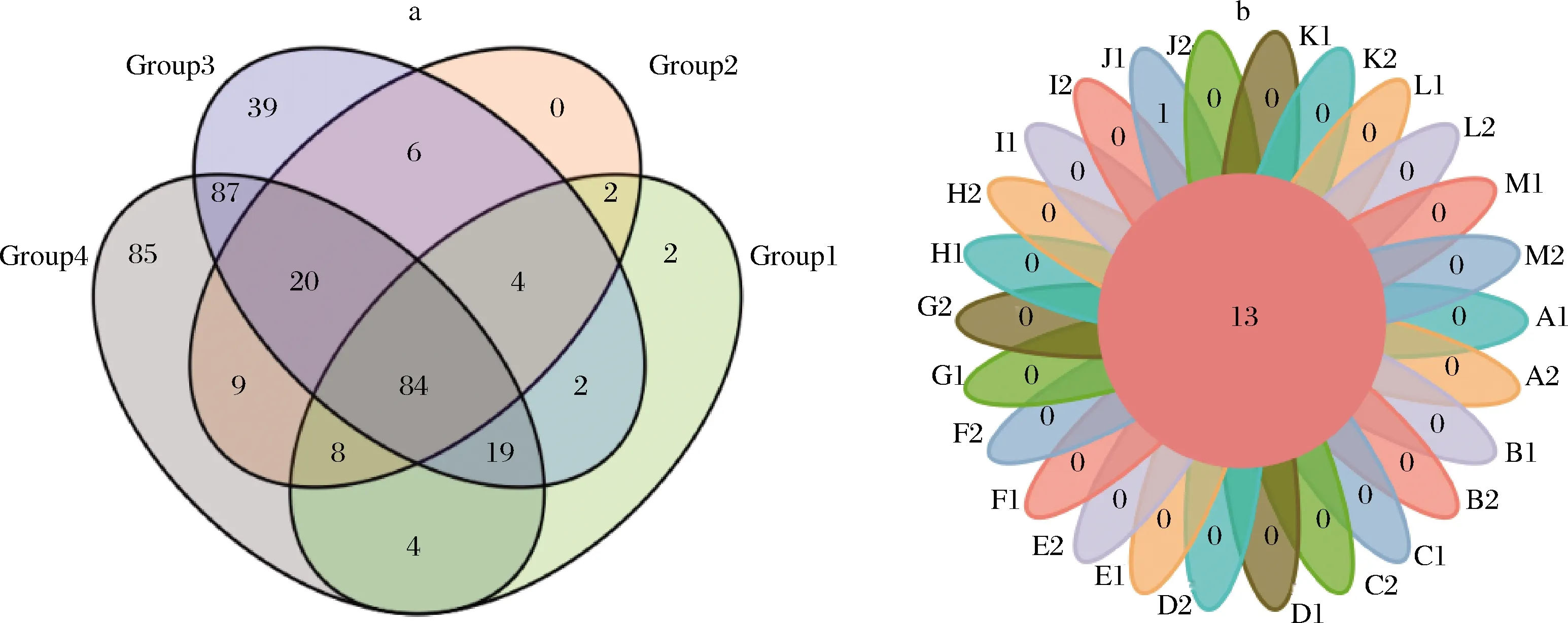
图1 样品中真菌群落OTU的Venn图Fig.1 Venn diagram of OTU of fungal community in samples
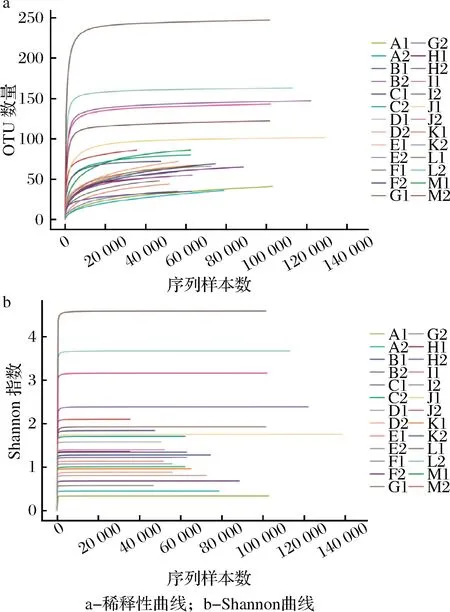
图2 样品稀释性、Shannon指数曲线Fig.2 Sample dilution and Shannon index curve
2.2 不同来源风干肉中真菌群落组成分析
根据分类学分析结果,可以得到不同样品在不同分类水平上的群落组成情况。图3-a表示的是在门水平下,样品中真菌丰度在0.1%以上的真菌用群落组成表示,丰度<0.1%的真菌用Other表示。由图3-a可知,样品中的优势真菌门有子囊菌门(Ascomycota)73.13%、担子菌门(Basidiomycota)24.77%和被孢霉菌门(Mortierellomycota)2.76%。这3个菌门真菌含量占到测序序列总数的99%。不同地区风干肉中真菌主要优势菌群略有差异,塔城地区优势真菌门为Basidiomycota,占该组真菌菌群的50.65%,而其他3个地区(额敏、富蕴、巴里坤)主要优势真菌门为Ascomycota,分别占该组真菌菌群的70.33%、85.65%和80.47%。高庆超等[29]研究发现,同属于自然发酵的黑果枸杞酵素,通过高通量测序在门分类水平下,子囊菌门(Ascomycota)、担子菌门(Basidiomycota)占整个真菌门水平的99%。陈新[30]在对新疆新疆天山雪岭土壤中微生物种群的研究中也发现,土壤真菌中被孢霉菌门(Mortierellomycota)、担子菌门(Basidiomycota)和子囊菌门(Ascomycota)的相对丰度约占总和的70%,属于优势种群。以上结果可以看出,虽然研究对象不同,但真菌群落结构和优势菌群的差异不大,这是因为自然发酵产品中的微生物主要来自于当地的土壤、空气和水,在门的分类水平上来看,自然发酵食品的真菌类型和土壤当中的真菌基本一致。
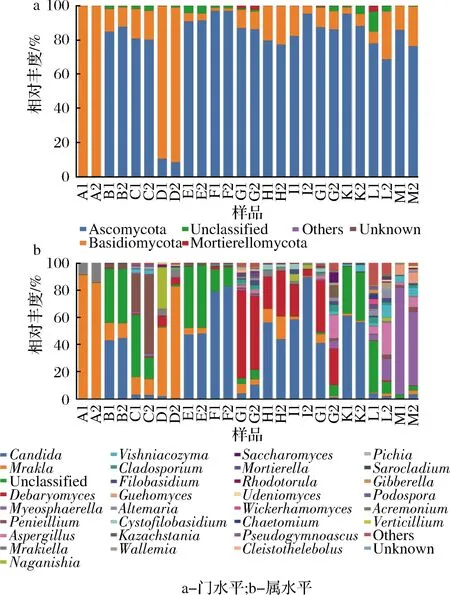
图3 各样品在门、属水平下真菌群落结构分布Fig.3 Relative abundance of the major fungus phylum and genus in samples
不同样品中真菌在分类属水平上的分布情况如图3-b所示。在26份风干肉样品中共鉴定出32个属,相对含量>1%的10个属分别为假丝酵母属(Candida) 30.09%、Mrakia16.26%、Unclassified15.82%、德巴利酵母属(Debaryomyces)11.14%、球腔菌属(Mycosphaere)6.37%、青霉属(Penicillium)4.23%、曲霉属(Aspergillus)2.39%、(Mrakiella)2.88%、Naganishia1.71%和维希尼克氏酵母(Vishniacozyma)1.55%。其中,塔城牛肉(A1、A2)相较于其他几个样品所含真菌属比较单一,优势真菌属为Mrakia,占到85%左右。额敏牛肉(F1、F2)和托里牛肉(K1、K2)在真菌属组成较为相似。不同地区风干肉中真菌主要优势菌群不同,样品塔城牛肉(C1、C2)中优势真菌属为青霉属(Penicillium),占40%左右;样品富蕴牛肉(G1、G2)中优势真菌属为德巴利酵母属(Debaryomyces),占60%左右;样品巴里坤马肉(M1、M2)中优势真菌属为球腔菌属(Mycosphaere),占65%左右。不同地区真菌在属水平上有着一定差异(图4-b),塔城地区和额敏地区菌群结构及种类比较类似,这可能是由于塔城和额敏都属于塔额盆地,地理环境和气候都比较类似。其他2个地区样品中真菌多样性则更加丰富,可能是由于这2个地区都属于山区牧区,降水量多,物种相对丰富,因此,菌群多样性也更加丰富。
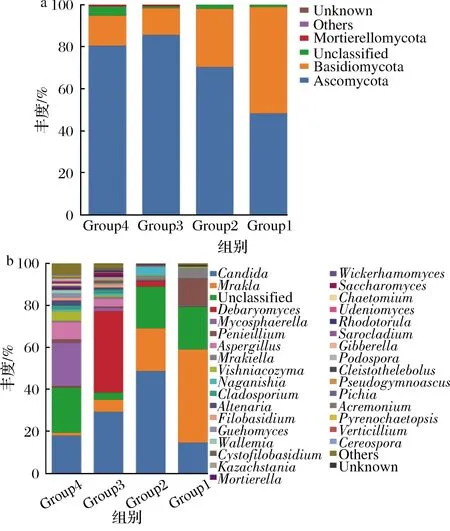
a-门水平;b-属水平图4 四组样品在门、属水平下真菌群落结构分布Fig.4 Relative abundance of the major fungus phylum and genus in four groups of samples
有研究表明,汉逊德巴利酵母(Dabaryomyces)和假丝酵母(Candida)是发酵肉制品的生产中最常用的酵母菌[31],因为其可促进发酵香肠的呈色,改善发酵香肠的风味,另外,有些酵母菌还能抑制腐败菌的滋生[32],赵永强等[33]用米曲霉和鲁氏酵母对合浦珠母贝肉进行发酵发现,其可以有效改善产品风味及游离氨基酸含量。另外,文开勇等[34]对四川传统腊肉研究发现,曲霉属(Aspergillus)为四川腊肉中优势真菌属。发酵肉在长时间的发酵过程中,真菌的生长繁殖可以产生多种酶类化合物,将肉中大分子的蛋白质、脂肪分解成小分子的醇酮醛等化合物,并能增加氨基酸的种类和游离脂肪酸的形成,促使亚油酸转化成共轭亚油酸,有利于风干肉中特殊风味的形成,显著提升发酵肉的风味品质和营养价值[35]。另外,酵母菌和霉菌还可以促进发酵肉中乳酸菌的生长,加速肉制品中水分含量的降低,并且能够降低风干肉的酸度,抑制发酵肉中脂质氧化、过氧化物的生成及腐败微生物生长繁殖,从而提高风干肉的安全性能[36-37]。本实验中优势的真菌属有假丝酵母属(Candida,30.09%)、德巴利酵母属(Debaryomyces,11.14%)和曲霉属(Aspergillus,2.39%),说明这3个属的真菌在风干肉的发酵过程中属于优势菌群,对风干肉品质形成有一定的影响。
为了能够更加直观的看出不同来源不同样品之间真菌属水平的相似程度,通过物种丰度聚类热图(Heatmap)表示(图5)。Heatmap是一颜色梯度来代表数据矩阵中数值的大小,并根据物种丰度相似性进行聚类,将高丰度和低丰度的物种分类聚集,通过颜色梯度及相似程度反映多个样品群落组成的相似性和差异性。由图5可知,在真菌属水平,Group1、Group2、Group3和Group4中样品的聚类分析结果相互交叉,并有差异,其中L2、J2和D1样品和其他样品的群落组成差异较大,剩下的其他样品之间菌群聚类结果比较相似。
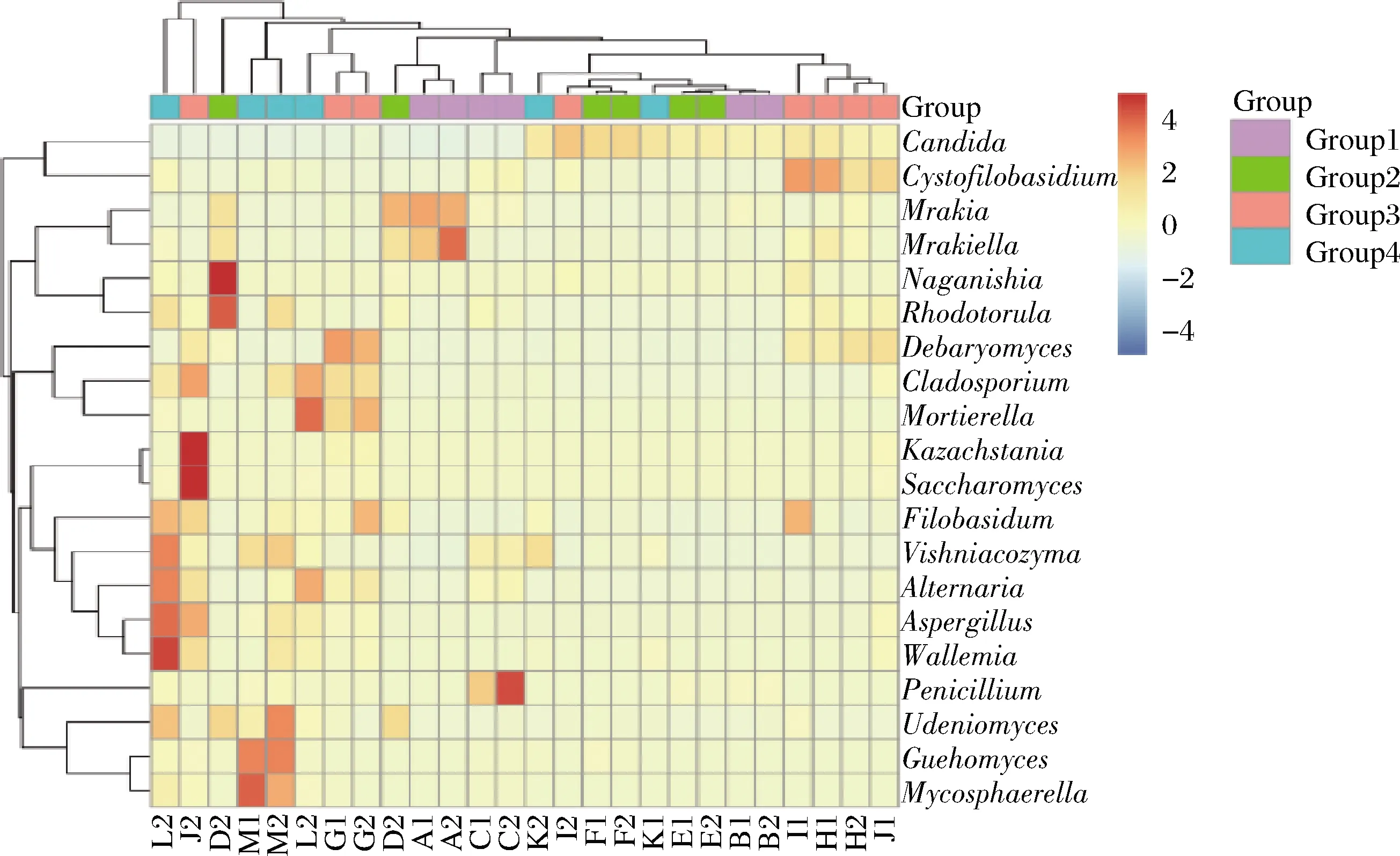
图5 样品在属水平物种丰度聚类热图Fig.5 Cluster heatmap of species abundance at genus level
26个样品都属于哈萨克族传统手工制作,其中4个是马肉,22个是牛肉,全部没有添加发酵剂,都属于自然发酵,但由于制作地区环境、风干温度以及制作人员的操作不同等原因导致了不同样品的真菌群落组成差异。实验结果表明,风干马肉和风干牛肉菌群多样性差异不大。从PCoA图可以看出(图6),26 个样品的真菌群落组成大致可以分为2 个类型,其中Group1和Group2样品分布相对较集中,说明这2组样品真菌群落结构比较相近,这与图5的结果相互印证。Group3和Group4组中样品分布在2个不同的位置,且相互之间距离较远,其中Group3组中J1、J2和G1、G2分别来自于富蕴的牧区,而其他样品则是山区,地理环境造成了其真菌菌群的差异。Group4组中K1、K2和其他样品真菌群落差异较大则是因为L1、L2和M1、M2采自于临近村落的2种不同的原料肉,而K1、K2则是采自于另外较远的村庄。
3 结论
本研究采用IlluminaMiSeq第二代测序技术研究了新疆北疆4个地区26个传统风干肉中真菌多样性,并结合高通量测序结果揭示了风干肉中真菌的多样性,不仅可以检测到可培养的真菌,而且还检测出了大量的不可培养真菌,尤其是对自然发酵肉制品,可以更加全面的反应样品中的菌群多样性。
高通量结果表明,4组26 个样品测序共获得真菌原始序列 2 059 240条,过滤后得到的有效序列为 1 936 392 条。26份风干肉样品中真菌在真菌分类门水平上共鉴定出5个门,平均丰度值>1%的3个真菌门分别为:子囊菌门(Ascomycota,73.13%)、担子菌门(Basidiomycota,24.77%)和被孢霉门(Mortierellomycota,2.76%)。这3个菌门真菌含量占到测序序列总数的99%。不同地区风干肉中真菌主要优势菌群不同,塔城地区优势真菌门为Basidiomycota,而其他3个地区(额敏、富蕴和巴里坤)主要优势真菌门为Ascomycota。另外,在26份风干肉样品中共鉴定出32个属,相对含量>1%的属分别为假丝酵母属(Candida,30.09%)、Mrakia16.26%、德巴利酵母属(Debaryomyces,11.14%)、球腔菌属(Mycosphaere,6.37%)、青霉属(Penicillium,4.23%)、曲霉属(Aspergillus,2.39%)、(Mrakiella) 2.88%、Naganishia1.71%和维希尼克氏酵母(Vishniacozyma,1.55%)。丰度聚类热图和Beta多样性分析表明,L2、J2和D1样品和其他样品的群落组成差异较大。
