α干扰素抗病毒相关全基因组表达谱的生物信息学分析及其对新型冠状病毒肺炎潜在药物研究的意义
2020-12-31陈浩然陈熙勐迟小华张钧栋李卓阳刘格良张皓旻王毅兴卢学春
智 鹏,杨 波,陈浩然,陈熙勐,迟小华,张钧栋,郭 斌,李卓阳,刘格良,张皓旻,王毅兴,卢学春
2019年12月31日,研究人员在武汉报道的数个不明原因肺炎病例中发现了一种未知的冠状病毒,即新型冠状病毒(2019-novel coronavirus,2019-nCoV)[1]。感染2019-nCoV后,绝大多数患者初始症状多为发热、乏力和干咳,引起传染性疾病-新型冠状病毒肺炎(coronavirus disease 2019,COVID-19),但目前尚缺乏疫苗,也没有特效的抗病毒药[2]。国家卫健委颁布的诊疗指南中明确提出,可用α干扰素雾化吸入进行抗病毒治疗[3]。本研究通过分析α干扰素抗病毒相关全基因组表达谱,探索其对COVID-19的潜在治疗意义。
1 资料与方法
1.1 数据来源 本研究以“interferon-alpha”为关键词在基因表达数据库(gene expression omnibus,GEO)进行检索,并以①物种为人;②至少2个生物重复;③实验设计思路清晰、数据质量好为标准进行筛选。
1.2 实验设计 本研究所使用的数据集均为经GEO数据库预处理后的转录组表达谱矩阵。根据数据来源实验描述以及各样本类型,将数据设定为对照组与处理组。
1.3 差异表达基因识别 在Bioconductor(http://www.bioconductor.org/)网站下载生物信息分析的R语言程序[5]。利用Impute程序包对已获取的表达谱进行归一化处理[6]。同时利用注释包对数据进行注释,将探针对应到基因上,随后利用Limma程序包以|logFC|>1,FDR<0.05作为阈值筛选处理组与对照组的差异表达基因。
1.4 富集分析 利用基因本体学(gene ontology,GO)和京都基因与基因组百科全书(the kyoto encyclopedia of genes and genomes,KEGG)通路分析[7]。采用Fisher确切概率法,以FDR<0.05,P<0.05作为筛选条件,以获得差异表达基因富集的生物学进程(Biological Processes,BP)、细胞组分(cellular Component,CC)、分子功能(Molecular Function,MF)和KEGG通路。
1.5 构建显著性基因的蛋白质互作网络 使用STRING开源数据库对前述显著性基因构建蛋白质互作(protein-protein interaction,PPI)网络[8]。以可靠性阈值大于0.4作为截断值并将数据下载。随后将数据导入Cytoscape可视化分析软件对PPI网络进行可视化,去除游离节点,使用Degree算法评估网络中基因节点的重要性,选取degree值前十位的基因作为核心基因[9]。
1.6 疾病关联分析 本研究前期基于“系统生物学”和“比较功能基因组学”理论设计了“整合多组学分析”算法,并以此为基础建立了涵盖所有疾病、9 000多种临床常用药物、1 000多种中药以及近10万种化合物的人体功能基因组学大数据的临床生物信息学表观精准治疗预测平台(epigenomic precision medicine prediction platform,EpiMed)[10-11]。应用该平台对上述差异表达基因进行多组学关联分析,寻找α干扰素的适应证及为后续优化含α干扰素的新冠病毒病治疗联合方案提供理论依据。
2 结果
2.1 数据筛选 共计检索出2 478条结果,根据筛选标准在结果中再次筛选包含α干扰素处理组与对照组实验数据的全基因组RNA表达芯片数据集。最终选择并下载符合本研究要求的GSE5542数据集。该数据集来源于GPL96平台(affymetrix human genome U133A array)。作用对象为A549细胞,细胞培养条件为:DMEM(西格玛,圣路易斯,美国)与10%胎牛血清(FBS)(欧米茄科学公司,圣弗南度谷,加拿大)。将未经α干扰素处理组数据设为对照组,经α干扰素处理6 h和24 h的数据分别设为6 h处理组和24 h处理组。
2.2 差异表达基因识别 共发现6 h处理组有311个差异表达基因,其中上调差异表达基因212个,下调差异表达基因99个;24 h处理组有303个差异表达基因,其中上调差异表达基因194个,下调差异表达基因109个,图1。
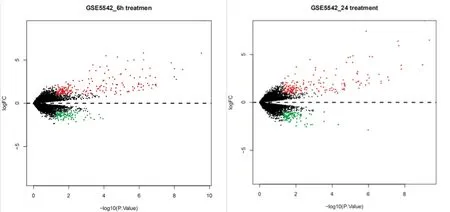
图1 差异表达基因
2.3 富集分析 GO 富集分析结果显示,6 h处理组共富集BP 44条,CC 1条,MF 2条,24 h处理组共富集BP 50条,CC 12条,MF 3条,图2。
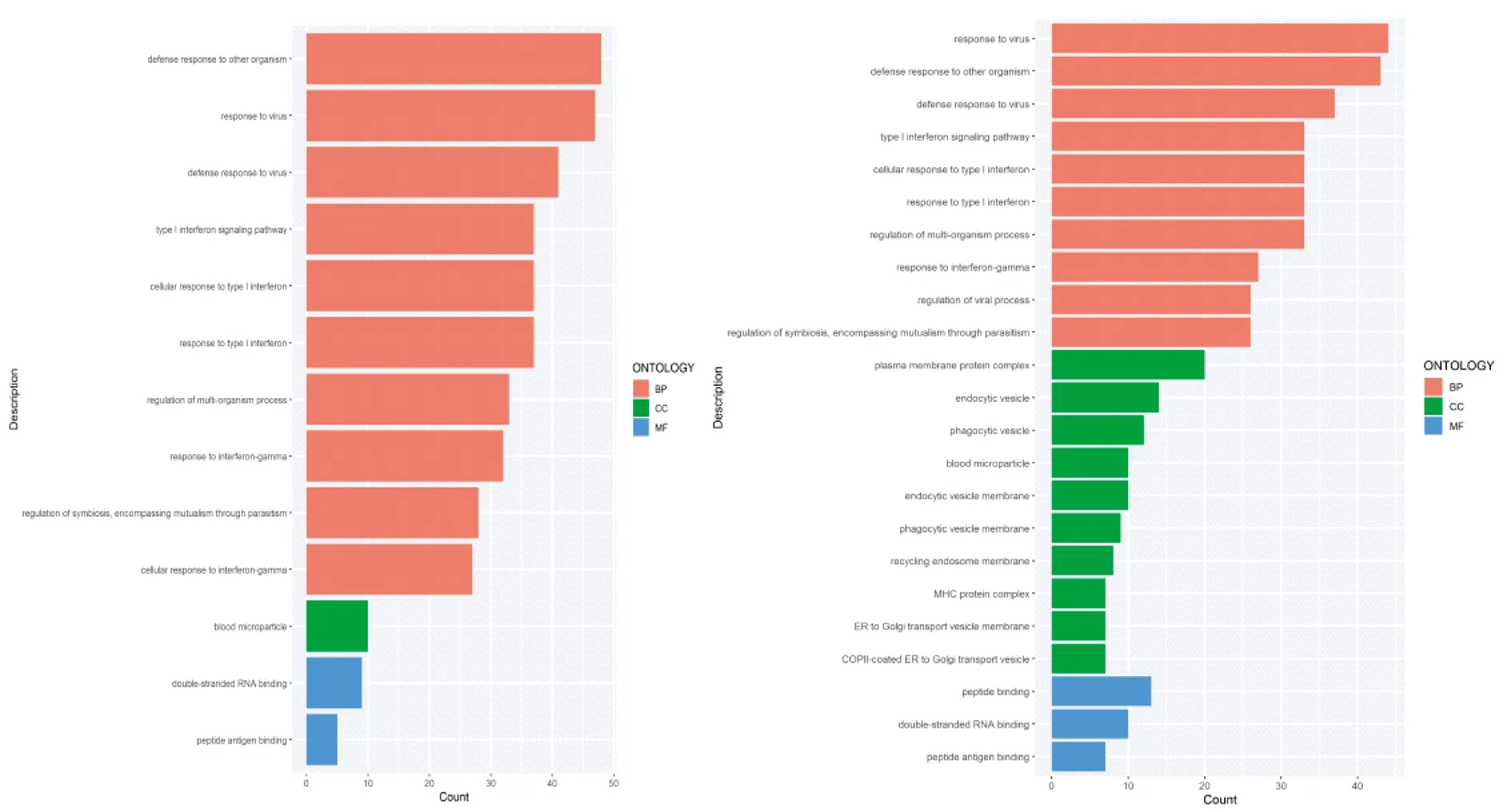
图2 GO富集分析
KEGG通路富集分析显示,6 h处理组共富集217条KEGG通路,24 h处理组共富集209条KEGG通路(图3),选取部分通路图展示,图4。分别为RIG-I样受体信号通路、JAK-STAT信号通路、Th17细胞分化和人免疫缺陷病毒感染信号通路图这些基因在红色区域表达上调,在绿色区域下调。
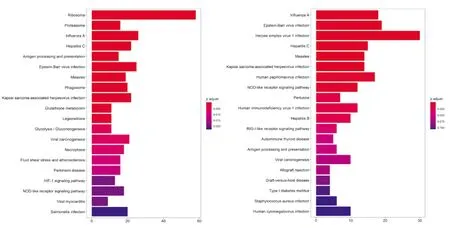
图3 KEGG通路分析
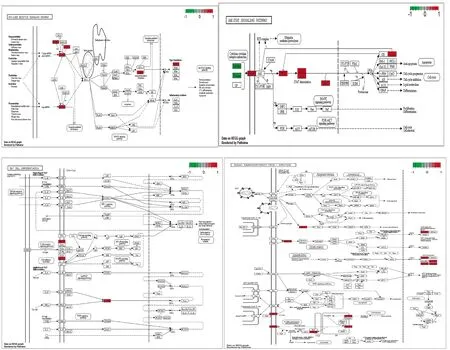
图4 部分信号通路图
2.4 PPI网络分析及核心基因筛选 6 h处理组共得到294个蛋白,876条相互关系,平均点度5.96。24 h处理组281个蛋白,698条相互关系,平均点度4.97(图5),对PPI网络中的基因进行节点得分计算,筛选得到排名前10位的核心基因,图6。
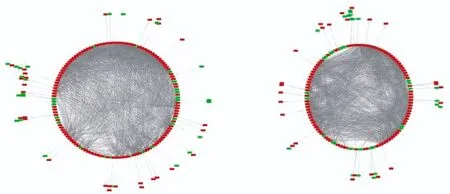
图5 蛋白互作网络
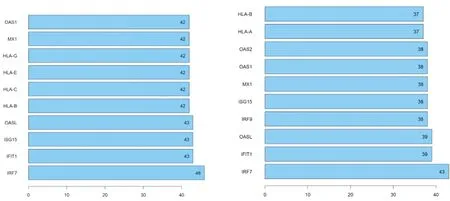
图6 核心基因
2.5 疾病关联分析 以EpiMed平台为依据,选取结果中6 h处理组和24 h处理组中与α-干扰素作用负相关的疾病和正相关的药物,筛选得到疾病和药物以及匹配靶点。部分结果见表1和表2。
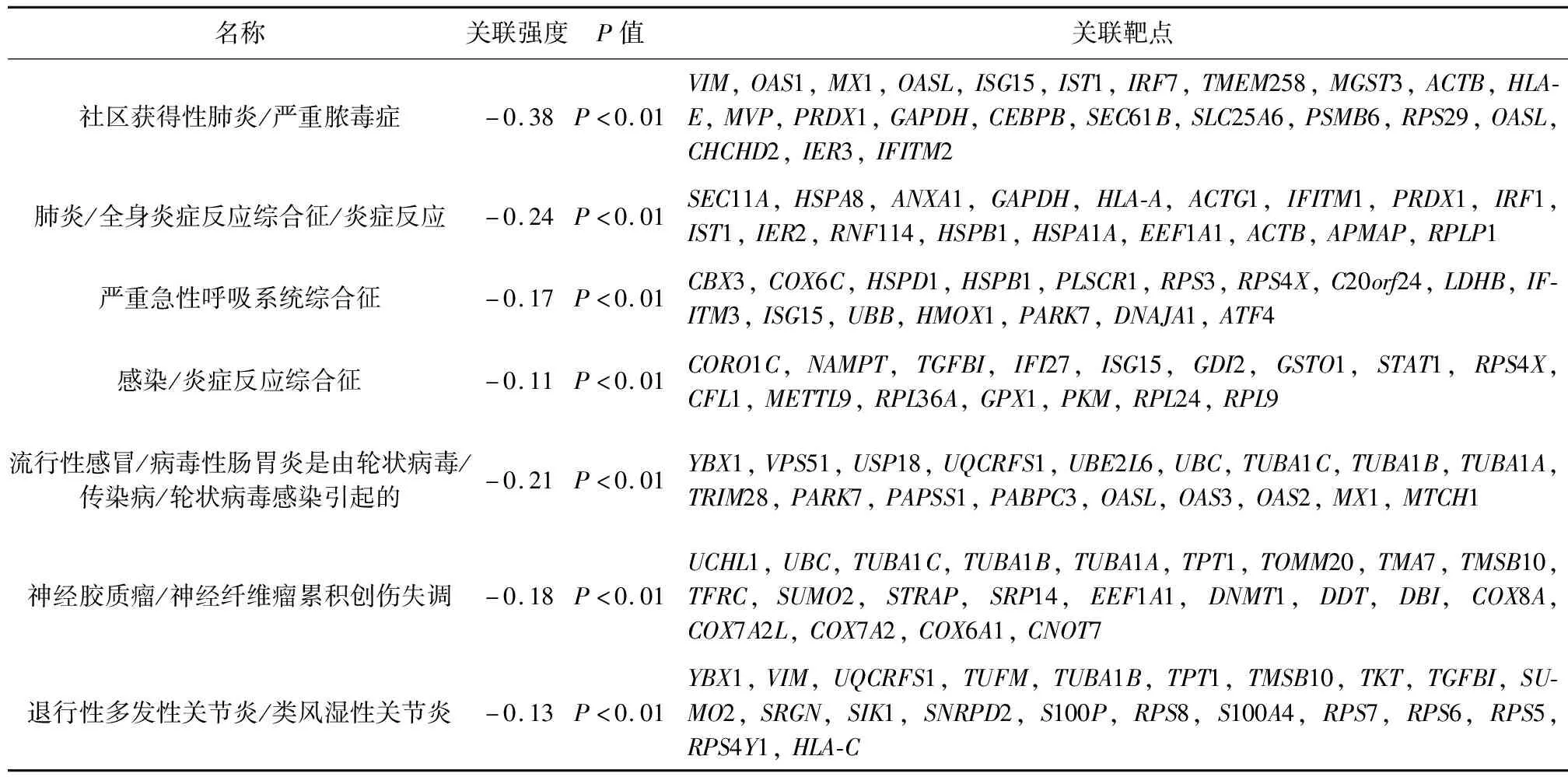
表1 EpiMed疾病关联分析
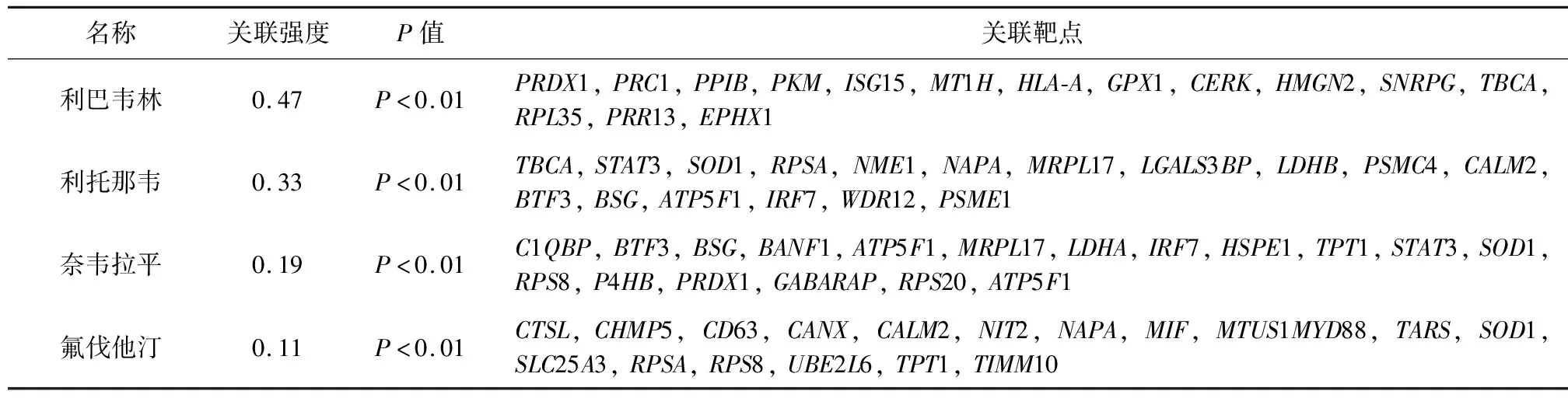
表2 EpiMed药品关联分析
3 讨论
现阶段,针对COVID-19的治疗还处于研究阶段,α干扰素虽已作为临床治疗的药物已写入诊疗方案并取得初步疗效,但其治疗机制尚不明确。本研究应用临床生物信息学方法,对α干扰素的药物功能基因组学数据开展研究,发现α干扰素发挥抗病毒作用不仅仅通过单一靶点或通路发挥作用,其作用是全基因组范围的。
本研究发现,在α干扰素影响的基因中,大量基因与抗病毒有关,包括IRF7、OASL、ISG15等在内的基因已经有明确的文献证实[12-14]。同时,还有大量基因与免疫激活、抗炎性反应以及抗肿瘤有关[15]。在信号通路方面,本研究发现,α干扰素的作用不仅限于抗病毒,如RIG-1样受体能识别病毒核酸成分,此类RIG-I样受体都定位在细胞内膜结构上,病毒侵入细胞后,在酸性环境中释放核酸物质,进而被RIG-I样受体识别,发挥抗病毒作用[16];α干扰素可通过对JAK-STAT受体信号通路的抑制作用,影响细胞增殖、分化、凋亡以及炎症等生物学过程,推测在2019-nCoV感染的治疗中,α干扰素可能通过此通路减少正常细胞的凋亡,减轻对靶器官的损伤作用[17]。同时α干扰素还具有抗炎性作用,如Th17细胞信号通路,它是一类由原始CD4+T细胞分化而来的辅助性T细胞。Th17细胞可以分泌多种重要的细胞炎症因子,其中最重要的是白介素17(interleukin-17,IL-17)与其受体结合后可诱导细胞产生IL-8、IL-6和粒细胞-巨噬细胞集落刺激因子GM-CSF、趋化因子CXCL,趋化并激活中性粒细胞在局部浸润,推测α干扰素可通过抑制炎性因子的产生而发挥对2019-nCoV感染的治疗作用[18]。
利用EpiMed平台进行多组学比对分析发现,α干扰素对社区获得性肺炎等疾病具有治疗作用,且潜在治疗靶点中有大量基因发挥了关键作用,如MX1,OASL,ISG15等;同时,α干扰素针对脓毒症等感染性疾病同样具有潜在治疗作用,且治疗靶点中存在大量免疫相关基因[19],提示α干扰素可通过影响免疫相关分子及信号通路发挥免疫调节作用,而2019-nCoV感染可激活先天性和适应性的免疫反应,但不受控制的先天反应和受损的适应性免疫反应可能导致局部和全身组织损伤[20-21]。不仅如此,还与病毒性疾病、传染性疾病和退行性关节炎等负相关,而这些疾病与本次新型冠状病毒肺炎的临床病理相似,据此推测α干扰素对COVID-19具有潜在治疗作用。在药物关联分析中,与α干扰素呈正相关药物包括利巴韦林、利托那韦、奈韦拉平、西多福韦、氟伐他汀等,在国家卫健委办公厅最新印发的诊疗方案中明确指出:利托那韦和利巴韦林为抗病毒治疗药物,且建议利巴韦林和干扰素联合应用,与本研究结果一致。同时,西多福韦、氟伐他汀等药物的发现对COVID-19的潜在药物研究具有一定的借鉴意义。
综上,本研究发现,α干扰素具有抑制炎症因子产生、免疫调节等方面的作用,为其用于COVID-19治疗提供了初步的分子机制认识;筛选出的潜在药物还有待于体内外实验的验证。
