牦牛低氧适应相关基因启动子甲基化程度与组织差异表达关系研究
2020-12-21胡明军杨远潇赵文仕江明锋
柏 玛,胡明军,杨远潇,赵文仕,江明锋
(1.云南香格里拉市畜牧兽医局饲料草山站,云南香格里拉 674499;2.西南民族大学青藏高原研究院,四川成都 610041;3.西南民族大学生命科学与技术学院,四川成都 610041)
低氧适应性是指机体在低氧环境、高原低氧以及疾病导致机体氧获得和运送出现障碍时,为维持基本生命活动所建立的一套保护性机制。机体对低氧的适应除器官功能的变化外,还可能通过调整低氧诱导因子和细胞代谢来完成[1]。调控低氧信号传导的主要转录因子是低氧诱导因子-1(Hypoxia-inducible Factors-1,HIF-1),该因子先由Semenza 等[2]于1992 年在缺氧诱导的细胞核抽提物中发现。HIF-1 是一个小转录因子家族,高度保守,主要介导细胞在低氧环境下的应答,可以促进血管分化,对胚胎和肿瘤中血管的形成十分重要[2]。HIF-1 转录因子主要有HIF-1α、HIF-1β、HIF-2α、HIF-2β、HIF-3α、HIF-3β共6 个成员。其中,HIF-1α和HIF-3α被认为与低氧诱导反应直接相关。在氧气含量正常的组织中HIF-1α的表达水平较低,但在组织缺氧时,HIF-1α表达水平显著上调且其表达水平受其富含GC 序列元件的启动子调控[3];HIF1AN 是HIF-1α的抑制蛋白,可降低HIF-1α的表达水平。HIF-3α也被认为与低氧适应反应有关,是低氧诱导基因表达的负抑制因子[4]。
研究发现,HIF-1 起着核信号的作用[5],共有100多种低氧相关基因受到HIF-1 的调节。如促红细胞生成素(Erythropoietin,EPO)作为HIF-1的一个下游基因,其表达受到HIF-1 的调控。在缺氧缺血的体外模型中,EPO 以剂量依赖的方式抑制HIF-1基因的表达[6]。缺氧作为一种普遍刺激,影响着许多生理过程,如许多生活在海平面上的人患有慢性间歇性缺氧(IH),这是一种由睡眠呼吸暂停导致的心血管和呼吸系统疾病。研究表明,DNA 甲基化改变是由慢性IH 引起的病理变化,并可能影响缺氧诱导因子信号[7]。Dewi 等[8]研究还发现,在正常(非低氧)情况下,EPO启动子增强子区的甲基化降低了EPO基因的转录活性。Xiong[9]等研究表明,牦牛心、肝、脾、肾和肺等组织中的HIF-1α和HIF-2α基因表达量均显著高于普通牛,牦牛这些组织中HIF-1α和HIF-2α基因增强子区的甲基化水平显著低于普通牛。青藏高原是天然的低氧环境,世居的哺乳动物经过长期的进化形成特有的耐低氧机制[10]。但导致这一系列现象的分子机制仍不清楚,需要进行深入研究。本项目研究了牦牛心、肝、肺、脑、肌肉等组织中EPO、HIF-1α、HIF-3α和HIF-1AN4 个基因启动子区域的甲基化程度对其表达的影响,旨在进一步探明牦牛低氧适应的分子机制。
1 材料与方法
1.1 样品采集 在四川省红原县龙日种畜场采集3 头健康成年公牦牛的心、肺、脑、肝脏和肌肉组织,迅速放入液氮中保存。
1.2 牦牛不同组织总DNA 的提取 采用AxyPrep 基因组DNA 小量试剂盒(AXYGEN,Union city,USA),按说明书步骤提取牦牛各组织样品总DNA,用紫外分光光度计进行定量。
1.3 亚硫酸盐测序 采用甲基化试剂盒EZ DNA Methyl ation-KitTM(ZYMO RESEARCH,Los Angeles,USA)对DNA 进行处理,详细步骤参见试剂盒说明书,处理后的DNA 于-20℃保存备用。
1.4 牦牛EPO、HIF-1α、HIF-3α和HIF-1AN基因启动子克隆 根据普通牛EPO基因(NM_173909)、HIF-1α基因(NM_174339)、HIF-3α基因(NM_001105342)以及HIF-1AN基因(NM_001083443)的启动子序列设计扩增相应基因启动子的引物(表1)。以亚硫酸盐修饰后的DNA 为模板进行扩增,25 µL PCR 体系:模板2 µL(80 ng)、10×buffer 2.5 µL、EX-Taq(TaKaRa)0.2 µL(1 U),上下游引物各1 µL(10 pmol/L)。扩增条件为:94℃预变性3 min,94℃变性30 s、退火30 s、72℃延伸2 min 进行35 个循环后72℃继续延伸7 min、4℃保存。PCR 产物用1%琼脂糖凝胶电泳进行检测,回收相应目的DNA 片段。
1.5 牦牛EPO、HIF-1α、HIF-3α和HIF-1AN基因启动子甲基化分析 利用www.urogene.org/methprimer 网站在线生物信息学软件分析克隆的4 个基因的启动子序列,对其核心启动子序列及位置进行分析。用不同组织提取的基因组DNA 作模板进行PCR 扩增,PCR 产物用1% 琼脂糖凝胶进行电泳检测。回收相应目的DNA片段,并连接到PMD19-T 载体上。挑取重组子测序,然后进行序列分析,检测各个核心启动子区段的甲基化数目。
1.6 总RNA 的提取与cDNA 合成 将采集的牦牛心、肺、脑、肝脏和肌肉组织各0.6 g 放入预先用液氮冷却的研钵内,研磨成粉,然后加入Trizol Reagent(Invitrogen,Cakifornia,USA)提取总RNA。采用PrimeScript®RT reagent Kit With gDNA Eraser DRR047A(TaKaRa,中国大连)试剂盒进行反转录反应,以Oligo dT 为引物合成第一链cDNA,反转录反应程序为37℃ 15 min,85℃5 s,然后4℃保存,得到的cDNA 作为qPCR 实验的模板,置于-80℃冰箱备用。
1.7 牦牛EPO、HIF-1α、HIF-3α和HIF-1AN基因的组织表达研究 根据GenBank 数据库中普通牛EPO、HIF-1α、HIF-3α、HIF-1AN、β-actin、GAPDH基因序列(登录号见1.4)设计qRT-PCR 引物用于相应基因的组织表达研究(表2)。用BIO-RAD CFX96 荧光定量PCR 仪筛选出RT-qPCR 的最适温度后,以牦牛的心脏、肝脏、肺、脑、肌肉组织cDNA 为模板,采用10 µL反应体系:SsoFastTM EvaGreen®Supermix 5 µL、上下游引物各0.5 µL(10 pmol/L)、cDNA 1 µL(0.5 μg)、ddH2O 3 µL 进行qRT-qPCR,每个样本均设置3 个重复,检测各基因的表达量。反应程序:95℃ 10 min、95℃15 s、最适温度1 min,40 个循环;95℃ 15 s、65℃ 5 s。以β-actin 基因和GAPDH基因作为内参基因,取平均值,用2-△△Ct法计算各基因表达量。
1.6 统计分析 数据采用SPSS 软件进行分析。不同组织间mRNA 相对表达量差异采用方差分析进行检验,甲基化率差异采用χ2检验。
2 结果与分析
2.1 牦牛EPO、HIF-1α、HIF-3α和HIF-1AN4 个基因启动子克隆 图1 及测序结果表明,EPO基因启动子长870 bp,HIF-3α基因启动子长1 067 bp,HIF-1α基因启动子长1 067 bp,HIF-1AN基因启动子长1 669 bp。EPO、HIF-1α、HIF-3α及HIF-1AN核心启动子序列分析结果显示,EPO基因启动子中含有1 个核心区段,HIF-1α、HIF-3α、HIF-1AN基因启动子各含有2 个核心启动子区段。

表1 牦牛EPO、HIF-1α、HIF-3α 和HIF-1AN 基因启动子克隆引物

表2 牦牛EPO、HIF-1α、HIF-3α 和HIF-1AN 基因qRT-PCR 引物
图1 牦牛低氧适应相关基因启动子PCR 产物琼脂凝胶电泳图
2.2 牦牛EPO、HIF-1α、HIF-3α和HIF-1AN基因核心启动子甲基化检测 牦牛EPO基因核心启动子序列全长127 bp,包含8 个CpG 岛,在脑组织中的甲基化水平显著高于心脏和肌肉组织(图2)。由表3 可知,EPO基因在心脏、肌肉和脑组织中,CpG 岛发生甲基化的位点分别有4、10 个和18 个,EPO基因在牦牛心、肌肉、脑的甲基化率分别为8.3%、20.8%、45.0%,牦牛脑中的甲基化率显著高于心脏和肌肉组织。
牦牛HIF-1α基因核心启动子有2 段序列,长度分别为221 bp 和117 bp,包含24 个CpG 岛。HIF-1α基因在肝、心脏和肌肉组织中甲基化的CpG 岛数目为:3个、12 个和17 个(表3)。HIF-1α基因的核心启动子在肝脏、心脏和肌肉的甲基化率分别为2.5%、8.5%、12.1%,且在牦牛肌肉中的甲基化率极显著高于心脏和肝脏。
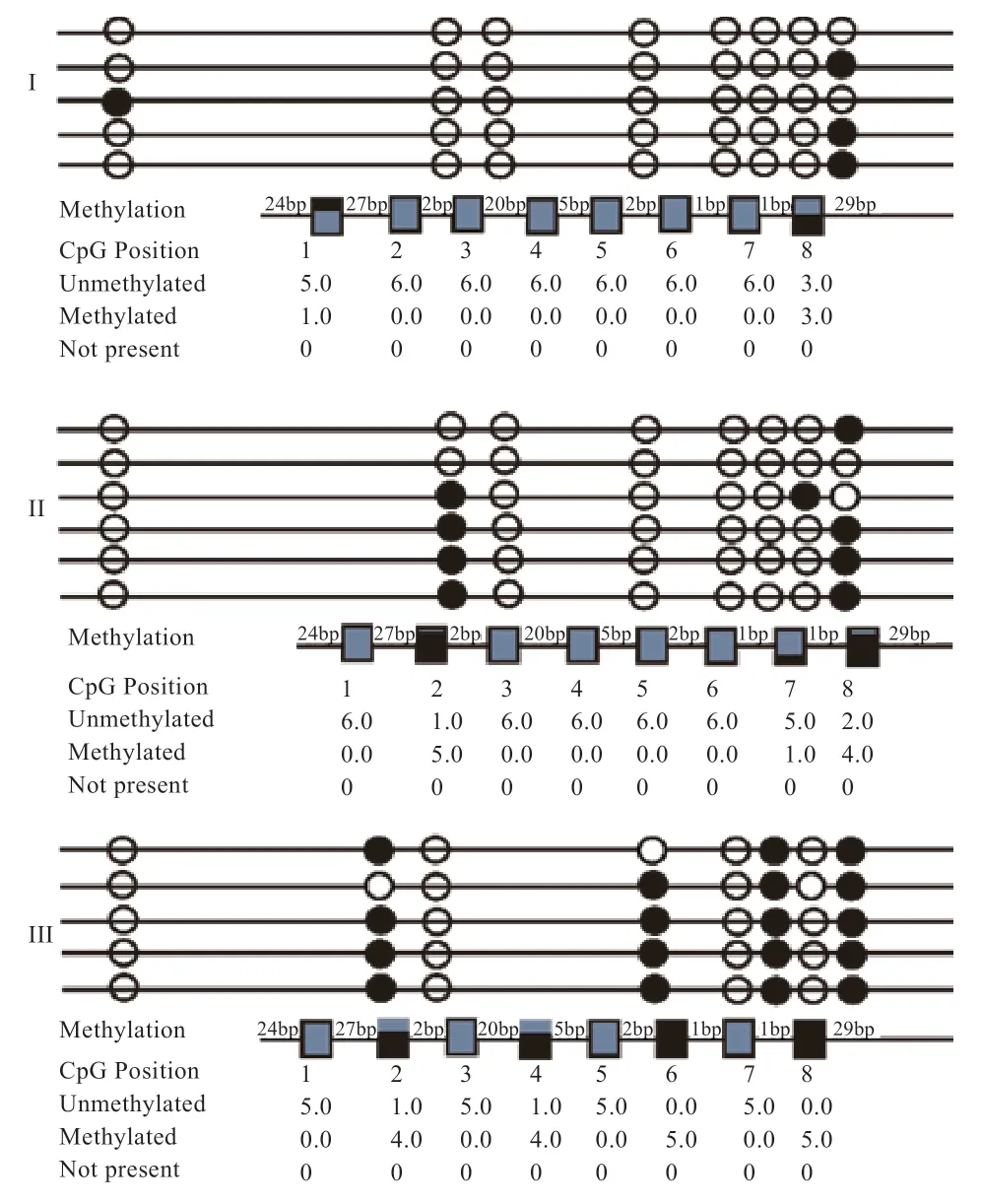
图2 EPO 基因核心启动子在组织中的甲基化位点图
牦牛HIF-3α基因核心启动子有2 段序列,长度分别为252 bp 和163 bp,包含22 个CpG 岛。HIF-3α基因在心脏、脑和肝组织中甲基化的CpG 岛数目为:6 个、10 个和14 个(表3),其核心启动子在心脏、脑和肝脏中的甲基化率分别为5.5%、9.1%、12.7%,且在牦牛肝脏中的甲基化率极显著高于心脏和脑。
牦牛HIF-1AN基因核心启动子有2 段序列,长度分别为271 bp 和167 bp,包含20 个CpG 岛。HIF-1AN基因在心脏、脑和肺组织中甲基化的CpG 岛数目为:21、68 个和87 个(表3)。HIF-1AN基因的核心启动子在心、脑和肺中的甲基化率分别为22.8%、69.4%、87.0%,并且在牦牛肺中的甲基化率极显著高于心脏和脑。
2.3 牦牛EPO、HIF-1α、HIF-3α和HIF-1AN基因的组织差异表达分析 由图3 可知,EPO基因的表达水平除在肺和肌肉之间差异不显著外,在其余组织间差异极显著;牦牛HIF-1α基因在肝组织和脑组织间差异显著,其余组织间差异极显著;HIF-3α基因在肺和肌肉组织间差异显著,其余组织间差异极显著;HIF-1AN基因在肝脏和肌肉组织差异不显著,肝脏和肌肉组织与脑组织间差异显著,其余组织间差异极显著。
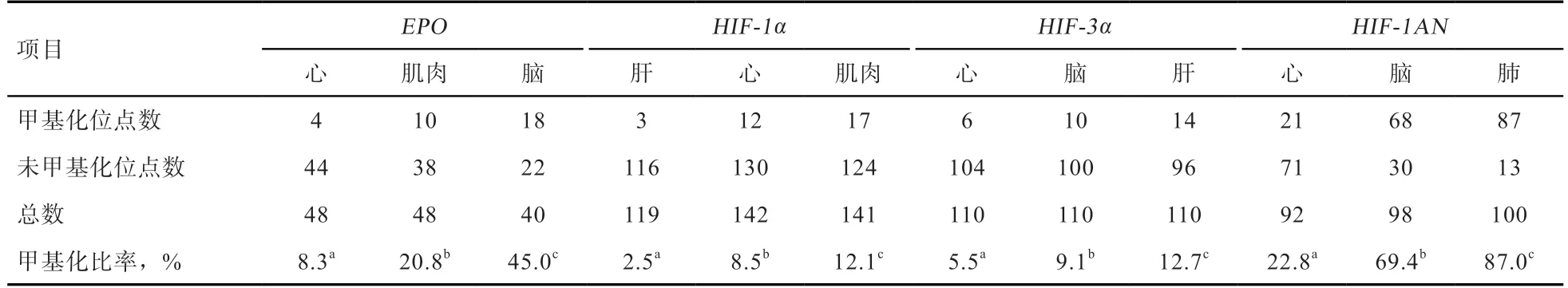
表3 牦牛EPO、HIF-1α、HIF-3α 和HIF-1AN 基因核心启动子的甲基化率分析
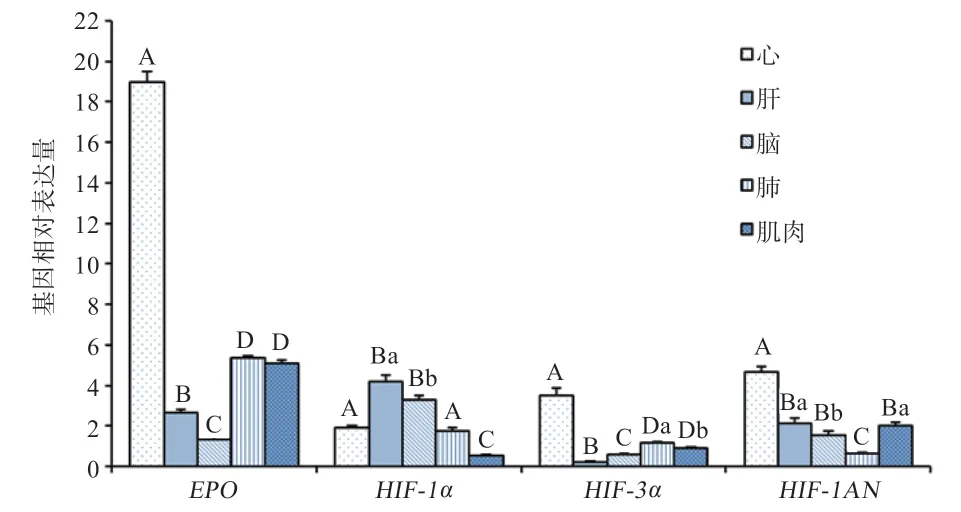
图3 EPO、HIF-1α、HIF-3α 和HIF-1AN 基因在牦牛不同组织的表达量
3 讨 论
对低海拔地区的动物而言,长期暴露在较严重的缺氧环境中对机体是有害的。高原世居动物则不受低氧环境的损害,表明高原世居动物已经完全适应高原低氧的生态环境。本研究发现,EPO基因在牦牛心脏的表达量最高,其次是肺、肌肉和肝脏,在脑组织中表达量最低。缺氧是EPO基因转录水平增加的主要原因。对EPO基因的表达和调控起最主要作用的是[A/G]CGTG 序列,其编码基因vegf和EPO启动子区域已被证实均含有低氧效应原件(HRE),表明EPO基因是受HIF-1 调控的靶基因[10-11];HIF-1α、HIF-3α和HIF-1AN基因在牦牛不同组织表达差异显著。在缺氧环境中,HIF-1α亚基的降解途径被抑制,大量的HIF-1α蛋白积累并结合HIF-1β形成具有活性的HIF-1 因子。目前,已经发现3种HIF-α/β亚基的类型。HIF-1α/β被认为是介导缺氧诱导因子表达的主要调控因子。HIF-1 广泛存在于大多数组织中,而HIF-2 主要在高度血管化的器官和组织中表达[12]。Naik 等[13]研究发现HIF-1AN作为HIF-1α抑制因子,能够抑制HIF-1α基因的转录活性,同时形成体外小管和迁移细胞。Li 等[14]在人的Ⅱ型肺细胞研究过程中发现,HIF-1α与HIF-3α基因在表达模式和应答方式上具有一致性,这说明二者在功能上可能起着互补的作用,共同确保细胞在低氧的环境中正常运转。
造成这3 个基因在不同组织表达出现差异的原因可能是相关基因启动子区域甲基化程度不一。由于启动子区域的高度甲基化,使它们在有些组织中沉默。本研究发现,HIF-1α和HIF-1AN基因在不同组织间的表达量存在差异。HIF-1能够诱导靶基因在红细胞和组织中表达,同时抑制如EPO等有关基因的表达。EPO作为HIF-1的靶基因,当低氧时HIF-1α表达水平增加,HIF-1α从胞浆移入胞核,与胞核内HIF-1β结合产生HIF-1,并与靶基因HRE 中HIF-1 结合点结合,促进EPO转录,引起一系列细胞对缺氧的反应[15]。长时间处于低氧环境下,上调HIF-1 活性可以使细胞在低氧状况下的生存能力提高,并生成大量组织血管[16]。组织中氧供给和利用在达到平衡之前,HIF-1基因的mRNA一直被表达,直到新的平衡建立。在新平衡建立后,若给予更进一步的低氧刺激,HIF-1基因的mRNA 水平又明显增加[17],这说明HIF-1基因的mRNA 与缺氧程度存在明显的依存关系。
为了研究牦牛组织中EPO、HIF-1α、HIF-3α和HIF-1AN基因差异表达的遗传学机制,本文扩增了牦牛4 个基因启动子区序列并进行了甲基化检测,结果发现EPO、HIF-1α、HIF-3α和HIF-1AN等低氧相关基因的启动子区域在不同组织中的甲基化率存在显著差异。牦牛HIF-1AN基因启动子区域的甲基化率显著高于其他3 个基因。结合4 个基因在相关组织的表达量来看,表达量高的组织如心脏,甲基化率低。与此相反,表达量低的组织相关基因启动子区域的甲基化率高,这说明基因启动子甲基化影响转录因子与启动子区域结合,进而抑制其表达。本实验还发现HIF-1AN基因在组织中的表达显著高于其他3 个基因,进一步说明甲基化可能是导致牦牛组织中低氧适应相关基因表达下调的机制之一。这与Xiong 等[9]和Reik[18]发现的基因甲基化水平与表达量的关系一致。在人类癌症中,肿瘤与正常的细胞相比,甲基化水平发生很大的变化,在肿瘤的发生过程中基因组DNA 呈现整体低甲基化和局部高甲基化特点[19]。这可能是HIF-1甲基化导致原癌基因的过度表达和正常基因沉默所产生的后果。随着缺氧组织适应性研究的进展,低氧核心调节因子HIF-1所诱导的一系列低氧反应效应基因及其有关甲基化的研究,将为缺氧组织的适应性及人类疾病带来福音。
4 结 论
本研究首次发现牦牛EPO、HIF-1α、HIF-3α和HIF-1AN基因在心、肝、肺、脑、肌肉等5 个组织的表达量与其启动子的甲基化率存在负相关,基因表达量受甲基化水平的影响,本研究结论为牦牛低氧适应领域的研究奠定了基础。
