新疆野苹果和栽培苹果遗传分化与变异特征
2020-12-09于少帅赵文霞姚艳霞张学超淮稳霞
于少帅 赵文霞 姚艳霞 张学超 淮稳霞


摘要:利用15对特异性SSR引物对290份新疆野苹果和栽培苹果样品进行扩增,并通过分子生物学方法分析其遗传变异特征与系统发育关系。15对SSR引物在290份新疆野苹果和栽培苹果样品中共获得69个等位基因,平均每个SSR位点获得4.6个等位基因。新疆野苹果和栽培苹果亲缘关系较近,新疆野苹果种群遗传多样性丰富,遗传分化系数为0.25,基因流为0.76。新疆新源和巩留2个地区的新疆野苹果聚于一个进化分枝,栽培苹果与采自新疆的红肉苹果聚于一个进化分枝。编码转录因子RAX3的SSR位点在枯枝率大于90%的新疆野苹果样品中有特异条带;编码类TMV抗性蛋白质N端的SSR位点在新疆野苹果中有特异性条带;编码类TMV抗性蛋白质N端、转录因子bHLH74的SSR位点在栽培苹果中有特异性条带。
关键词:新疆野苹果;微卫星标记;遗传多样性;系统发育;抗逆性
中图分类号:S661.1文献标识码:A文章编号:1000-4440(2020)05-1274-08
Abstract: 15 pairs of specific simple sequence repeats(SSR) primers were used to amplify 290 samples of Malus sieversii and cultivated apples, and their genetic variation and phylogenetic relationships were analyzed by molecular biology method. 69 alleles were obtained in 290 samples of M. sieversii and cultivated apples based on 15 pairs of SSR primers, with an average of 4.6 alleles per SSR locus. There was a close genetic relationship between M. sieversii and cultivated apples. The genetic diversity of M. sieversii groups was rich, with a genetic differentiation coefficient of 0.25 and a gene flow of 0.76. M. sieversii from Xinyuan and Gongliu of Xinjiang clustered in an evolutionary branch, cultivated apples and the red-flesh apple collected from Xinjiang clustered in an evolutionary branch. The SSR locus that coding transcription factor RAX3 showed a specific band in M. sieversii samples whose deadwood rate was above 90%. The SSR locus that coding the N end of the TMV-like resistant protein showed a specific band in M. sieversii samples. The SSR loci that coding the N end of the TMV-like resistant protein and the transcription factor bHLH74 showed specific bands in cultivated apples.
Key words:Malus sieversii;microsatellite marker;genetic diversity;phylogeny;stress resistance
新疆野苹果(Malus sieversii)是蔷薇科苹果属的濒危二级重点保护植物,是研究世界苹果遗传多样性和基因进化的重要天然基因库,主要分布于中国新疆西部、中亚的哈萨克斯坦和吉尔吉斯斯坦[1]。新疆野苹果由于受自然选择的长期作用,形成了丰富的遗传特征和较强的环境适应性,具有抗病、抗虫等优良性状[2]。近年来,分布于中国的新疆野苹果枯死严重。影响新疆野苹果生存状态的原因较多,比如人类活动、苹小吉丁虫害和苹果树腐烂病等因素[3-5]。但关于引起新疆野苹果枯死的具体原因及枯死机制尚不清楚。因新疆野苹果胁迫因子的多样性和复杂性,在新疆野苹果保育过程中采取单一的防治手段较难起到理想的保护效果。在寻找引起新疆野苹果种群退化的关键因子和保护新疆野苹果的同时,保护、选育和栽培具有抗性特征的遗传资源应该是控制其种群衰退最为经济有效和根本的措施。
关于新疆野苹果种质遗传变异和系统发育的研究结果表明,新疆巩留县、新源县等地的新疆野苹果种群存在丰富的遗传多样性[6-7],种群内部遗传分化程度较高[8]。在新疆野苹果遗传多样性和系统发育关系研究中常用的分子标记有SSR[9]、SRAP[7]、RAPD[10]、ITS[11],其中应用较多的分子标记为SSR标记[2]。作为一种分辨率高、遗传信息量大、操作相对简单的标记方法,SSR被广泛应用于濒危植物遗传变异研究中[12-14]。SSR标记可以与植物的一些表型、抗性等性状关联在一起。Klabunde等[15]用11对SSR引物对152份具有不同苹果叶斑病抗性的巴西栽培苹果的遗传多样性进行评估发现:不同抗性栽培苹果间存在丰富的遗传多样性,苹果抗叶斑病品种与感叶斑病品种间遗传结构差异显著,有120个等位位点是抗病品种独有的。新疆野苹果研究结果显示,SSR标记可以很好地揭示新疆野苹果种群遗传多样性和遗传结构[6],新疆野苹果种内不同个体的叶形等表型性狀的变异与SSR标记的位点有一定的相关性[2,16]。目前关于新疆野苹果抗病虫等抗性差异在新疆野苹果个体、种群间的遗传变异尚未见报道。
新疆野苹果和栽培苹果遗传关系较近,可能是现代栽培苹果的祖先[1]。目前关于新疆野苹果种质遗传变异和系统发育研究应用较多的分子标记是SSR标记[6,9,16],但现在大部分标记均为非编码区分子标记,扩增片段的功能未知,在NCBI上无明确比对结果。基于栽培苹果基因组,筛选了15对可以在栽培苹果、新疆野苹果中扩增出目的条带的的特异性SSR引物,通过这些特异性引物对不同种群新疆野苹果和栽培苹果进行遗传变异分析,旨在揭示新疆野苹果的遗传信息及变异规律。本研究用新开发的15对特异性SSR引物,对新疆野苹果和栽培苹果290份样品进行遗传多样性和系统发育分析,揭示其遗传变异规律和亲缘关系,为新疆野苹果的生态保育,苹果优质新品种培育和产业的可持续健康发展等提供一定的理论依据。
1材料与方法
1.1试验样品
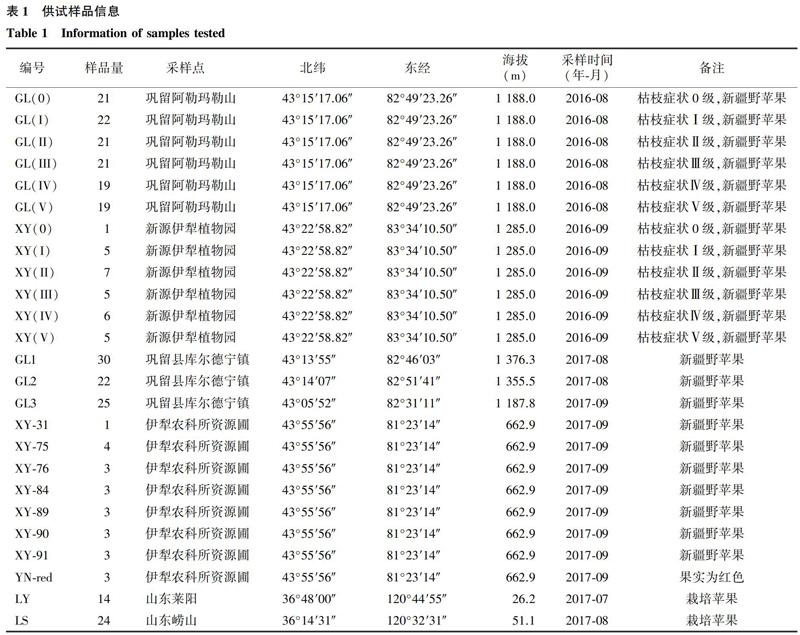
2016年至2017年对新疆维吾尔自治区伊犁哈萨克自治州巩留县和新源县的新疆野苹果健康状况进行调查并采样。根据新疆野苹果树上的枯枝率将新疆野苹果按级别进行划分[17],共分6个级别,0级:单株枯枝率为0;Ⅰ级:单株枯枝率为0~10%;Ⅱ级:单株枯枝率为11%~40%;Ⅲ级::单株枯枝率为41%~60%;Ⅳ级:单株的枯枝率为61%~90%;Ⅴ级:单株枯枝率为91%~100%。新疆维吾尔自治区伊犁州农业科学研究所基于前期调查研究筛选了一些疑似抗逆资源,本研究样品于2017年9月采自新疆维吾尔自治区伊犁州农业科学研究所资源圃。栽培苹果样品于2017年7月至8月采自山东,样品信息如表1所示。
1.2仪器和试剂
植物基因组DNA提取试剂盒、SYBR Green I染料购自北京艾德莱生物技术有限公司,2×Taq Mix购自北京博迈德基因技术有限公司,Hi-Di去离子甲酰胺、POP-7 Polymer、GS-500LIZ相对分子质量内标购自美国ABI公司,A200型基因扩增仪购自杭州朗基科学仪器有限公司, DYY-6C型电泳仪购自北京市六一仪器厂,超纯水由 MILLI-Q 超纯水纯化系统制得,乙醇等其他化学试剂为国产分析纯。
1.3DNA提取
取样品叶片0.1 g,采用CTAB法提取新疆野苹果和栽培苹果的基因组总DNA,用1.0%琼脂糖凝胶电泳检测,-20 ℃保存备用。
1.4PCR扩增
根据已报道的栽培苹果品种金冠苹果基因组[18],获得15对新疆野苹果特异性SSR引物,如表2所示。DNA测序PCR反应体系25.0 μl:1.0 μl DNA模板,0.5 μl 上游/下游引物 (10 μmol/L), 12.5 μl 2×PCR Master Mix (包括 0.05 U/μl Taq DNA polymerase, 4.0 mmol/L MgCl2, 0.4 mmol/L dNTPs),最后用ddH2O补齐。PCR扩增条件是95 ℃ 5 min;95 ℃ 30 s, 52 ℃ 30 s和72 ℃ 30 s,循环35次;72 ℃延伸10 min。PCR产物用SYBR Green I荧光染料染色在1% (质量体积比)的琼脂糖凝胶电泳中检测。毛细管电泳PCR反应体系是30 μl,包括1 μl DNA模板、1 μl上游/下游引物(10 μmol/L,加荧光),15 μl 2×PCR Master Mix (包括 0.05 U/μl Taq DNA polymerase、4.0 mmol/L MgCl2、0.4 mmol/L dNTPs),用ddH2O补齐。PCR反应条件为95 ℃ 5 min;95 ℃ 30 s,52 ℃ 30 s和72 ℃ 30 s,循环35次;72 ℃延伸10 min。PCR扩增结束后,取0.3 μl PCR产物用ABI 3730×l DNA analyzer进行毛细管电泳分析。
1.5数据处理
将上机结果原始文件导入Genemarker 2.2.0,按位点导出峰图、Excel位点信息表。利用GeneMarker V2.2.0和POPGENE V1.32计算引物扩增的等位基因数、基因多样性指数、多态性信息量;使用GenAlEx V6.501软件计算各引物扩增的等位基因的PIC值;利用NTSYSpc V2.10e和MEGA V7.0软件计算品种间的遗传距离,用UPGMA法进行聚类分析,绘制树状图;根据遗传多样性系数(Na)、Neis基因多样性指數、香农指数(I)、期望杂合度(He)和观察杂合度(Ho)进行遗传多样性分析。
2结果与分析
2.1遗传多样性参数和遗传分化
用15对特异性SSR引物扩增290份不同地区、不同枯枝症状级别的新疆野苹果和栽培苹果样品,对其遗传多样性和系统发育关系进行分析,共扩增出69个等位基因,平均每个位点扩增出4.6个等位基因,如表3所示。本研究结果表明,不同类群的等位基因数为1.40~3.67,有效等位基因数为0.98~2.28,Neis基因多样性指数为0.20~0.46,香农指数为0.28~0.83,观察杂合度为0.33~0.73,期望杂合度为0.25~0.76。根据这些遗传多样性参数评估可知,检测的新疆野苹果和栽培苹果样品遗传多样性较为丰富。遗传分化系数(Fst)为0~0.05时,一般认为遗传分化低;Fst值为0.05~0.25时,一般认为中等程度遗传分化;Fst值>0.25时,一般认为显著遗传分化[19]。新疆野苹果和栽培苹果不同类群290份样品的Fst为0.25,基因流(Nm)为0.76,由遗传分化系数和基因流可知,不同类群间具有一定程度的分化,在总的遗传变异中,种群间变异为24.86%。
2.2新疆野苹果和栽培苹果SSR位点特异性分析
15对特异性SSR引物在290份样品中共扩出69个等位基因,平均每个位点扩增出4.6个等位基因。每个位点扩增的片段长度及等位基因数如表4所示。SSR引物XS71-1-4和XS72-1-1在290份样品中扩增的等位基因数最多,为10个;引物XS62-1-4扩增的等位基因数最少,为2个。特异性SSR位点分析结果表明,XS24-1-5-2(转录因子RAX3)位点获得的等位基因是GL(V)、GL1、XY(V)特有的,由此可知编码转录因子RAX3的SSR位点在枯枝率大于90%的新疆野苹果样品中有特异条带。XS2-1-2(组氨酸激酶5)位点246等位基因是YN-red特有的。XS71-1-4(类TMV抗性蛋白质N端)位点440等位基因是GL(0)、GL1、GL2、XY-84特有的。XS71-1-5(类TMV抗性蛋白质N端)位点367等位基因是YN-red特有的。XS71-1-5(类TMV抗性蛋白质N端)位点422等位基因是GL1、GL2、GL3特有的,由此可知编码类TMV抗性蛋白质N端的SSR位点在新疆野苹果中有特异性条带。XS63-1-10引物(扩增的基因序列编码的蛋白质为类TMV抗性蛋白质N端)获得的等位基因是山东烟台栽培苹果特有的[18],XS25-1-8引物(转录因子bHLH74亚型X1)获得的等位基因是LS、LY特有的,由此可知编码类TMV抗性蛋白质N端、转录因子bHLH74的SSR位点在栽培苹果中有特异性条带。
每个位点平均等位基因数,也称为等位基因多样性(通常用A表示)。GL(0)、GL(I)、GL(II)、GL(III)、GL(IV)、GL(V)、GL1、GL2、GL3、LS、LY、XY(I)、XY(II)、XY(III)、XY(IV)、XY(V)、XY-75、XY-76、XY-84、XY-89、XY-90、XY-91、YN-red见表1。
2.3新疆野苹果和栽培苹果不同种群系统发育关系
由Neis无偏差异遗传特征和遗传距离可知,新疆野苹果和栽培苹果亲缘关系较近,新疆野苹果内部具有较高相似性,栽培苹果内部也具有较高相似性。基于Neis无偏差异遗传距离构建的UPGMA进化树可以将新疆野苹果和栽培苹果不同类群或不同个体清晰、准确地分开,形成不同的进化分枝。由新疆野苹果和栽培苹果不同类群的UPGMA进化树可知,栽培苹果LY和LS类群与采自新疆伊犁农业科学研究所资源圃的红肉苹果聚于一类,采自巩留、新源不同样点、不同枯枝症状级别的新疆野苹果样品聚于不同的分枝,但与采样地点、症状级别等无明显关系。巩留、新源不同样点、不同枯枝症状级别的新疆野苹果样品难以区分。由此可知,巩留、新源两地新疆野苹果的亲缘关系较近。基于新疆野苹果和栽培苹果不同个体的Neis无偏差异遗传距离构建的UPGMA进化树和遗传距离结果表明,新疆野苹果和栽培苹果遗传关系较近,但两者可以准确分开,新疆野苹果聚于一个大的进化分枝,栽培苹果聚于另一个大的进化分枝,如图1所示。
3讨论
本研究发现新疆野苹果和栽培苹果亲缘关系较近,新疆野苹果种群遗传多样性丰富,不同类群间存在较高程度的分化水平,遗传分化系数为0.25,基因流为0.76。研究结果与已有研究结果[20-21]基本一致。而揭示种群间遗传分化水平的遗传分化系数与所采用的分子标记和种群自身的遗传变化有一定的关系[6,22]。新疆野苹果和栽培苹果不同种群间基因流相对较低,分化水平较为显著。新疆新源和巩留2个地区的新疆野苹果聚于一个进化分枝,栽培苹果与采自新疆的红肉苹果聚于一个进化分枝。关于新疆野苹果遗传多样性的研究相对较多,但SSR标记大部分为非编码区且功能未知,对于差异标记的功能作用等认知不足[6,21]。
通过特异性SSR引物,不仅可以揭示新疆野苹果和栽培苹果的遗传多样性和系统发育关系,而且对变异位点的抗逆特性也会有一定的了解。在不同SSR位点扩增的等位基因数有所不同,同一SSR位点不同等位基因在不同新疆野苹果或栽培苹果类群中出现的频率也有所不同,如编码转录因子RAX3的SSR位点在枯枝率大于90%的新疆野苹果样品中有特异条带;编码类TMV抗性蛋白质N端的SSR位点在新疆野苹果中有特异性条带;编码类TMV抗性蛋白质N端、转录因子bHLH74的SSR位点在栽培苹果中有特异性条带。SSR引物XS71-1-4和XS72-1-1在290份样品中扩增的等位基因数最多,为10;SSR引物XS62-1-4扩增的等位基因数最少,为2。不同新疆野苹果或栽培苹果种群具有不同的特异性SSR变异位点。这一现象可能与新疆野苹果或栽培苹果进化过程中对生境的适应性有一定的关系[1]。15对SSR特异性引物可以对新疆野苹果和栽培苹果进行遗传多样性、多态性或系统发育分析,揭示新疆野苹果和栽培苹果的遗传多样性和系统发育关系及获得的遗传变异对新疆野苹果和栽培苹果在生物学功能等方面的影响。通过这些新开发的SSR引物,可以很好地揭示新疆野苹果和栽培苹果之间的遗传变异规律和系统发育关系,这些研究结果将为新疆野苹果生态恢复、优质种源选育和优良栽培品种的创制提供一定的参考。新疆野苹果在长期的进化过程中形成了许多抗寒、抗病虫害等的优良性状[23-24]。新疆野苹果种内变异类型丰富,但缺乏系统遗传多样性的系统调查与评估,抗逆特性突出但基因资源深入挖掘与开发利用研究环节相对较为薄弱。目前新疆野苹果无全基因组序列,对国内外新疆野苹果起源、分化、扩散情况,以及新疆野苹果抗病虫害、抗寒和风味形成的遗传构成、基因调控机制等研究有一定的影响,从而限制新疆野苹果抗逆相关基因克隆和新型抗逆新疆野苹果种质资源的创制[2]。全基因组关联分析技术用于新疆野苹果抗病虫害基因的筛选,在全基因组范围内找出存在的变异序列(SNP),以差异强表达候选基因为目标来开发多态性SNPs。该技术一次可定位多个性状,而且定位精度高,可直接获得与目标性状相关的基因。因此,后续研究中有待于通过全基因组测序、转录组测序、全基因组关联分析技术等对新疆野苹果抗病虫害(抗苹果树腐烂病和苹小吉丁虫)、抗寒以及风味形成的相关遗传基因进行挖掘,分析其遗传构成和基因调控机制。通过全基因组测序、转录组测序、全基因组关联分析等技术,充分挖掘和利用新疆野苹果基因资源,通过杂交育种与配套抚育等方法,创制营养价值高、经济价值高的“功能性”新型苹果品种,通过“利用保存”这一方式更好地保存新疆野苹果遗传资源,并基于优质的新疆野蘋果种质资源,解决中国栽培苹果的遗传背景狭窄、种质退化等相关问题,促进中国苹果产业健康、可持续发展。
参考文献:
[1]束怀瑞. 苹果学[M]. 北京: 中国农业出版社, 1999.
[2]张艳敏,冯涛,张春雨,等. 新疆野苹果研究进展[J]. 园艺学报, 2009, 36(3): 447-452.
[3]刘华,臧润国,丁易,等. 天山西部新疆野苹果种群特征[J]. 林业科学, 2010, 46(11): 1-7.
[4]王春晓,赵褔,赵健桐,等. 新疆发生苹果小吉丁虫[J]. 新疆农业科学, 1995(5): 225-226.
[5]刘爱华,张新平,温俊宝,等. 天山野苹果林苹果小吉丁与苹果树腐烂病复合危害研究[J]. 新疆农业科学, 2014, 51(12): 2240-2244.
[6]ZHANG C, CHEN X, HE T, et al. Genetic structure of Malus sieversii population from Xinjiang, China, revealed by SSR markers[J]. Journal of Genetics and Genomics, 2007, 34(10): 947-955.
[7]张春雨,陈学森,林群,等. 新疆野苹果群体遗传结构与遗传多样性的SRAP分析[J]. 园艺学报, 2009, 36(1): 7-14.
[8]DONG Y, ZHANG J, REN Y C, et al. Stduy on genetic diversity of natural population in Malus sieversii with microsatellite[J]. Journal of Plant Genetic Resources, 2013, 14(5): 771-777.
[9]OMASHEVA M, CHEKALIN S, GALIAKPAROV N. Evaluation of molecular genetic diversity of wild apple Malus sieversii populations from Zailiysky Alatau by microsatellite markers[J]. Russian Journal of Genetics, 2015, 51(7): 647-652.
[10]ZHOU Z Q, LI Y N. The RAPD evidence for the phylogenetic relationship of the closely related species of cultivated apple[J]. Genetic Resources and Crop Evolution, 2000, 47(4): 353-357.
[11]赵偑,杨美玲,龙鸿,等. 基于ITS序列的新疆野苹果系统发育分析[J]. 生物学杂志, 2013, 30(5): 19-22.
[12]CHE Y H, YANG Y P, YANG X M, et al. Phylogenetic relationship and diversity among Agropyron Gaertn germplasm using SSRs markers[J]. Plant Systematics and Evolution, 2015, 301(1): 163-170.
[13]GHARGHANI A, ZAMANI Z, TALAIE A, et al. Genetic identity and relationships of Iranian apple (Malus×domestica Borkh.) cultivars and landraces, wild Malus species and representative old apple cultivars based on simple sequence repeat (SSR) marker analysis[J]. Genetic Resources and Crop Evolution, 2009, 56(6): 829-842.
[14]SEGARRA-MORAGUES J G, IRIONDO J M, CATALAN P. Genetic fingerprinting of germplasm accessions as an aid for species conservation: a case study with Borderea chouardii (Dioscoreaceae), one of the most critically endangered Iberian plants[J]. Annals of Botany, 2005, 96 (7): 1283-1292.
[15]KLABUNDE G H F, JUNKES C F O, TENFEN S Z A, et al. Genetic diversity and apple leaf spot disease resistance characterization assessed by SSR markers[J]. Crop Breeding and Applied Biotechnology, 2016, 16(3). http://dx.doi.org/10.1590/1984-70332016v16n3a29.
[16]左力辉,张文林,邱彤,等. 新疆野苹果叶形性状变异及其与SSR标记关系分析[J]. 园艺学报, 2015, 42(4): 759-768.
[17]阿格里斯(美). 植物病理学[M]. 沈崇尧,译.北京: 中国农业大学出版社, 2009:3.
[18]VELASCO R, ZHARKIKH A, AFFOURTIT J, et al. The genome of the domesticated apple (Malus×domestica Borkh.)[J]. Nature Genetics, 2010, 42(10): 833-839.
[19]FREELAND J R, KIRK H, PETERSEN S D.分子生态学[M]. 戎俊,杨小强,耿宇鹏,等译.北京: 高等教育出版社, 2015:9.
[20]ZHANG H X, ZHANG M L, WANG L N. Genetic structure and historical demography of Malus sieversii in the Yili valley and the western mountains of the Junggar Basin, Xinjiang, China[J]. Journal of Arid Land, 2015, 7(2): 264-271.
[21]RICHARDS C M, VOLK G M, REILLEY A A, et al. Genetic diversity and population structure in Malus sieversii, a wild progenitor species of domesticated apple[J]. Tree Genetics and Genomes, 2009, 5(2): 339-347.
[22]MAGUIR T I, PEAKALL R, SAENGER P, et al. Comparative analysis of genetic diversity in the mangrove species Avicennia marina (Forsk.) Vierh. (Avicenniaceae) detected by AFLPs and SSRs[J]. Theoretical and Applied Genetics, 2002, 104: 388-398.
[23]梅闖,闫鹏,韩立群,等. 新疆野苹果不同类型单株对苹果小吉丁虫抗性差异[J]. 新疆农业科学, 2015, 52(10): 1859-1865.
[24]刘君,刁永强,陈淑英,等. 伊犁河谷不同苹果品种苗木冻害调查及分析[J]. 北方园艺, 2013(23): 34-37.
(责任编辑:陈海霞)
