基于数据挖掘分析TOP2A在胰腺癌中的表达及预后意义
2020-11-26孙如丽
梅 雯,孙如丽,李 云,熊 伟,赵 一*
(1.大理大学第四附属医院,云南楚雄 675000;2.楚雄彝族自治州第二人民医院,云南楚雄 675000;3.大理大学基础医学院,云南大理 671000)
胰腺癌(pancreatic adenocarcinoma,PAAD)是恶性程度较高的消化道肿瘤,5 年生存率不足8%〔1〕。临床上90%以上的胰腺癌为胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC),胰腺癌起病隐匿,多以黄疸或腹痛为首发症状,就诊时超过80%的患者已进入中晚期,出现局部或远处转移,失去最佳手术时机,仅有少数可行根治性手术治疗〔2〕。近年来胰腺癌相关诊疗方法取得了快速发展,对胰腺癌的治疗采取外科手术、敏感的化疗和放疗联合应用,但患者术后远处转移和局部复发仍然严重影响着患者生存率。随着肿瘤个体化治疗和靶向治疗技术的发展,从分子水平研究胰腺癌发生、发展相关机制有利于发现新的分子靶点,提供新的治疗手段和提高患者生存率,对临床治疗具有重要现实意义。
DNA 拓扑异构酶(topoisomerase,Topo)是一种存在于细胞核内,调节DNA 空间动态变化的核内蛋白质。DNA 拓扑异构酶通过结合并水解ATP,催化DNA 链的断裂和结合,影响DNA 的拓扑结构〔3〕。拓扑异构酶具有两种同工酶,拓扑异构酶Ⅰ(TopoⅠ)和拓扑异构酶Ⅱ(TopoⅡ),TopoⅡ型在哺乳动物细胞中的催化活性由两种不同的同工酶介导,分别为TopoⅡα和TopoⅡβ,这两种酶在结构和功能上是相似的,但在细胞周期的核定位和相对浓度上都不同,两种酶编码不同的基因产物,表达TopoⅡα 的基因是TOP2A(topoisomeraseⅡalpha)。TOP2A基因位于17q12-21染色体,在DNA 复制、重组、转录、染色体浓缩及染色单体分离等过程发挥重要作用〔4〕。TOP2A表达水平的高低和数量变化可导致细胞内物质和结构发生改变。TOP2A主要在增殖细胞中发挥重要作用,其表达水平与肿瘤细胞增殖与转移密切相关。文献报道,TOP2A是活跃增殖细胞的敏感特异标记物,TOP2A在肿瘤细胞增殖的不同时相其表达水平有所差异,在S期晚期和G2∕M 期表达增加,在有丝分裂后下降,且TOP2A以细胞周期依赖的方式磷酸化〔5〕。有研究发现,TOP2A在多种不同类型的肿瘤中均呈现高表达,如肠癌、食管癌、肝癌、胃癌等多种类型癌症,且TOP2A表达水平与肿瘤细胞增殖水平呈正相关〔6-9〕。TOP2A的异常表达最常发生在乳腺癌中,被视为乳腺癌发展进程中的重要预后分子指标〔10〕。TOP2A在肿瘤的发生、发展中起着重要的调控作用,并与肿瘤的预后密切相关,但目前关于TOP2A与胰腺癌的相关研究鲜有报道。从分子水平深入研究胰腺癌发生、发展机制,寻找高灵敏度的分子标志物,对胰腺癌的早期筛查和临床诊断具有重要意义。本研究旨在利用现有的各种肿瘤生物信息数据库,通过数据挖掘分析TOP2A基因在胰腺癌中的表达及其预后意义,为TOP2A基因在胰腺癌发生、发展中的作用机制提供线索和思路。
1 资料与方法
1.1从ONCOMINE数据库提取数据在ONCOMINE v4.5 数据库中设定筛选和挖掘数据的条件为:①Gene:TOP2A;②Analysis Type:Cancervs.Normal Analysis;③Cancer Type:Pancreatic Cancer;④Data Type:mRNA;⑤Sample Type:Clinical Specimen;⑥临界值设定条件(Pvalue<1E-4,fold change 2,gene rank=top 10%,data type=all)。
1.2从GEPIA数据库提取数据在GEPIA 数据库(http:∕∕gepia.cancer-pku.cn∕)中 设定 筛选 和 挖 掘数据的条件为:①Gene:TOP2A;②Cancer name:PAAD(pancreatic adenocarcinoma);③“Expression on Box Polts”框中设置条件|Log2FC |Cutoff:1,P-value Cutoff:0.01;④“Pathological Stage Plot”输入TOP2A;⑤“Similar Genes Detection”选 择PAAD Tumor 和PAAD Normal;然后输入相应基因进行分析。
1.3 MethHC数据库提取数据在MethHC 数据库(http:∕∕methhc.Mbc.Nctu.Edu.tw)中设定筛选和挖掘数据的条件为:①Gene Reach:PAAD;②“Elect a gene region”中选择Promoter;③List your interested genes:TOP2A。
1.4 String数据库提取数据String数据库(https:∕∕string-db.org ∕)中设定筛选和挖掘数据的条件为:①Protein name:TOP2A;②Organism:Homo sapiens;③Munber of nodes:11;④PPI enrichmentP-value:1.3E-10;⑤High confidence:0.700;⑥Max number of interactors to show:10。
1.5从Kmplot数据库提取数据在Kmplot 数据库(http:∕∕kmplot.com∕analysis∕)中设定筛选和挖掘数据的条件为:①RNA-seq;②Gene:TOP2A;③Cancer symbol:pancreatic ductal adenocarcinoma;④Survival:分别选择总体生存率(overall survival,OS)和无复发生存期(relapse-free survival,RFS)。
1.6 The Human Protein Atlas数据库提取数据The Human Protein Atlas 数据库中设定筛选和挖掘数据的条件为:TOP2A。数据库共有3 种TOP2A 免疫组化抗体(No:HPA006458,No:HPA026773,No:CAB002448),对正常胰腺组织和胰腺癌组织进行免疫组化实验。选择克隆号为No:HPA006458 的抗体对1 例正常胰腺组织和11 例胰腺癌组织的TOP2A蛋白表达程度进行分析。
1.7统计学分析正常组织与肿瘤组织之间TOP2A基因表达差异采用t检验。TOP2AmRNA 表达水平与胰腺癌患者病理分期的关系采用单因素方差分析(one-way ANOVA)。TOP2A基因表达量与患者预后的关系采用Kaplan-Meier 和Log-Rank检验。P<0.05为差异有统计学意义。
2 结果
2.1TOP2A在部分肿瘤类型中的表达ONCOMINE数据库共有462 项研究结果涉及TOP2A在肿瘤组织与对应非肿瘤组织中表达差异的比较。其中差异有统计学意义的有132 项,TOP2A高表达的研究有125 项,低表达的研究有7 项(P<0.05)。TOP2A在消化系统肿瘤的表达情况见表1。

表1 ONCOMINE数据库中TOP2A表达有差异的消化系统肿瘤
2.2TOP2A在胰腺癌中的表达结果提取ONCOMINE v4.5 数据库中的信息发现,从2003 年至2009年,共有10 个子数据集涉及TOP2A在胰腺癌组织和正常组织中的表达,共包括322 个样本。文章分 别发表于Hepatogastroenterology〔11〕、Oncogene〔12〕、Neoplasia〔13〕、Cancer Sci〔14〕、Cancer Cell〔15〕、Clin Cancer Res〔16〕和Am J Pathol〔17〕。对10 个子数据集荟萃分析发现,与正常胰腺组织(对照组)相比,TOP2A在胰腺癌中呈现高表达,中位表达数值为1 169.0,P=0.008,差异有统计学意义。
2.3TOP2A在不同胰腺癌研究芯片的表达差异在ONCOMINE数据库中,获取TOP2AmRNA在胰腺癌研究芯片中的表达结果,对相关数据作箱图比较分析,有7 项研究(Lacobuzio-Donahue Pancreas 2,Pei Pancreas, Grutzmann Pancreas, Logsdon Pancreas,Segara Pancreas, Buchholz Pancreas, Badea Pancreas)均显示,相较于正常胰腺组织,TOP2AmRNA 在胰腺癌中呈现高表达,且差异具有统计学意义(P<0.05)。见图1A~G。但其中1 项研究(Ishikawa Pancreas)中,TOP2AmRNA 在胰腺癌组织和正常胰腺组织中的表达量差异无统计学意义(P=0.109)。见图1H。

图1 ONCOMINE芯片数据库TOP2A mRNA在正常胰腺组织和胰腺癌组织研究芯片中的表达差异
2.4TOP2A与多个基因的相关性分析基于GEPIA数据库基因相关性分析结果显示,胰腺癌组织TOP2A的mRNA表达水平与CDK1、HDAC1、NFE2L3、MTERF3、OLA1和RRM1基因mRNA 表达水平呈显著正相关(P<0.05,R>0)。见图2。
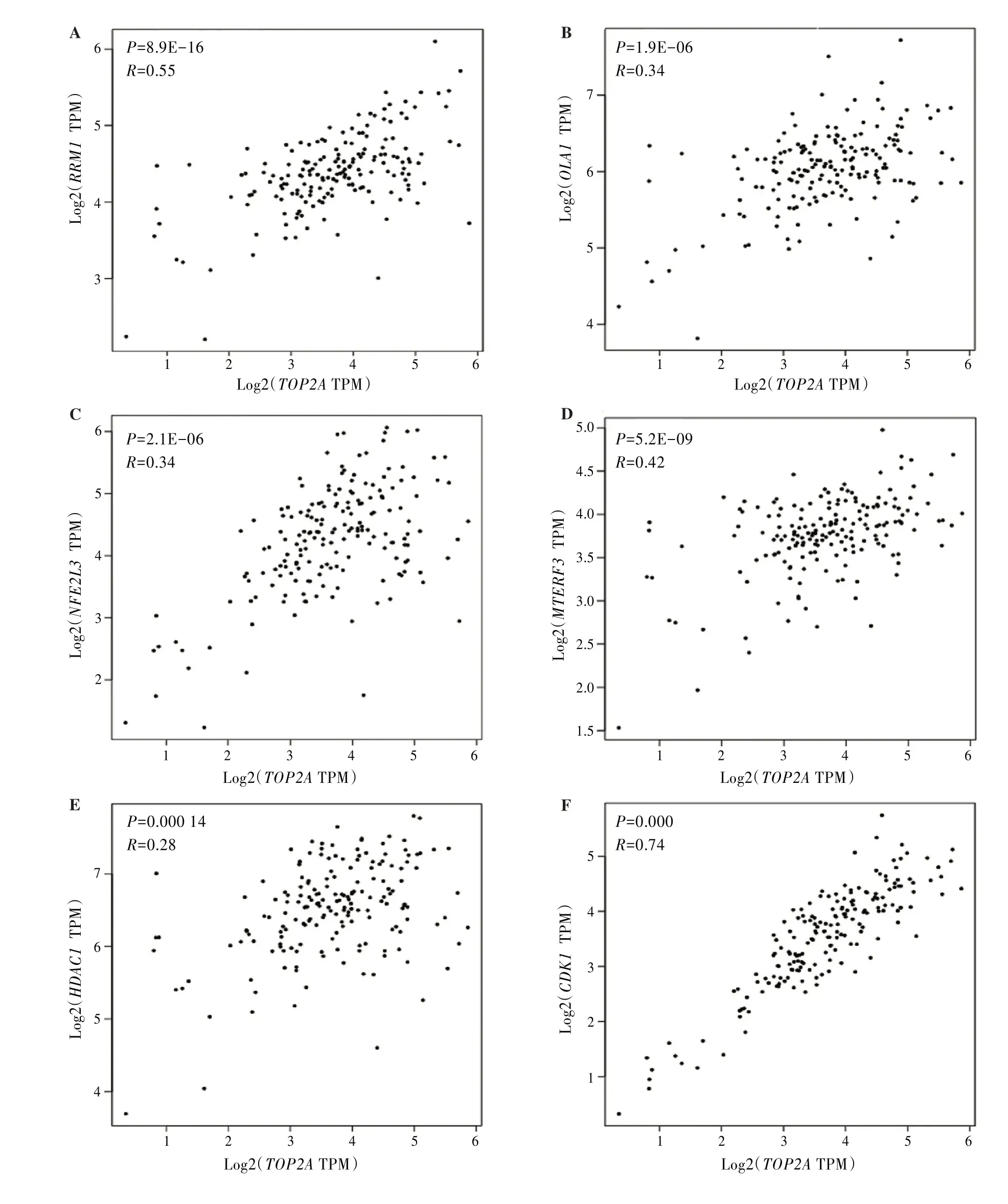
图2 TOP2A mRNA表达水平在胰腺癌患者中与其他基因mRNA表达水平的相关性
2.5TOP2AmRNA表达水平与胰腺癌患者病理分期的关系从GEPIA 数据库下载TOP2AmRNA 表达水平与胰腺癌患者病理分期的相关数据,结果显示TOP2AmRNA 表达水平在胰腺癌患者不同病理分期中的表达差异无统计学意义(P>0.05),单因素方差分析得出F值为1.64,其对应P值为0.183。见图3。
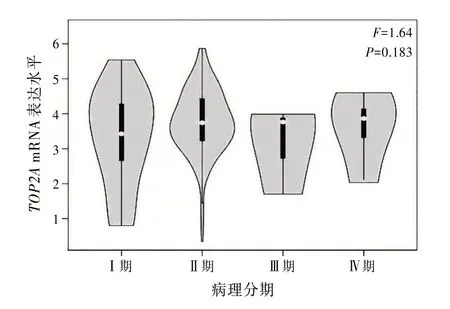
图3 TOP2A mRNA表达水平与胰腺癌患者病理分期的关系
2.6TOP2AmRNA表达与胰腺癌患者预后的相关性为了进一步明确TOP2AmRNA 表达与胰腺癌患者预后之间的关系,在Kmplot 数据库中对TOP2AmRNA 表达水平与胰腺癌患者预后相关的数据进行分析。结果表明,TOP2AmRNA 表达量与胰腺癌患者的OS和RFS均有显著相关性,与TOP2AmRNA低表达组相比,TOP2AmRNA 高表达组胰腺癌患者总体生存时间和无复发生存时间显著缩短(OS:LogrankP=4.9E-0.5;RFS:LogrankP=0.000 75)。见图4。
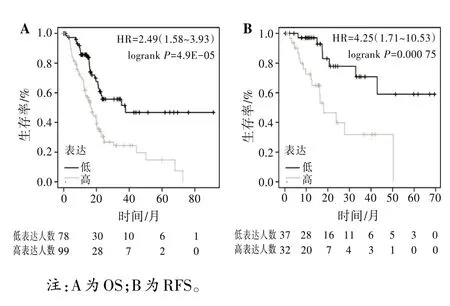
图4 胰腺癌患者的预后与TOP2A mRNA表达水平的关系
2.7TOP2A启动子区DNA甲基化水平分析从Meth-HC数据库下载相关数据,结果显示TOP2A共有1个转录模板,编号为NM_001067,在胰腺导管腺癌中TOP2A启动子区甲基化与正常胰腺组织甲基化水平差异无统计学意义(P>0.05)。见图5。

图5 正常胰腺组织和胰腺导管腺癌TOP2A启动子区甲基化水平比较
2.8 TOP2A相互作用蛋白网络在String 数据库中按照筛选条件,筛选出相互作用强度排位靠前的10 个与TOP2A 有相互作用的蛋白质,分别是NCAPG、CDC20、UBE2C、PBK、TPX2、DLGAP5、CCNB2、TOP1、CDK1、BUB1。见图6。这些蛋白相互作用,可能参与了胰腺癌细胞增殖、分化及细胞周期调节等功能,在胰腺癌发生、发展中发挥着重要的调节作用。

图6 与TOP2A相互作用的蛋白网络图
2.9 TOP2A蛋白在胰腺癌中的表达情况在The Human Protein Atlas 数据库中,选择克隆号为No:HPA006458 的抗体,分析TOP2A 蛋白在1 例正常胰腺组织和11 例典型胰腺癌组织中的表达情况。其中有5 例胰腺癌组织蛋白表达及抗体染色程度为“高等”水平;6例为“中等”水平;0例为“低等”水平。TOP2A 蛋白在胰腺癌组织中较正常胰腺组织高表达(左侧正常组患者id 2032,蛋白表达水平低;右侧肿瘤组患者id 1320,蛋白表达水平高)。见图7。

图7 TOP2A蛋白在正常胰腺组织和胰腺癌中的表达(免疫组织化学染色)
3 讨论
ONCOMINE 数据库是目前世界最大的肿瘤基因芯片数据库,提供癌症全面的基因表达数据及相关临床信息,整合了TCGA、GEO 和已发表文献来源的DNA 和RNA-seq 数据。GEPIA 是由北京大学开发的基因表达谱数据动态分析网站,其数据来自于TCGA 和GTEx 两大数据库。MethHC数据库其数据来自于TCGA,用于分析18 种肿瘤和对应正常组织DNA 甲基 化和mRNA∕miRNA 相 关信 息。String 数据库可用以提取蛋白质-蛋白质之间相互作用网络,预测蛋白质-蛋白质之间的关系。The Human Protein Atlas 数据库采用特制抗体检测每种蛋白在人正常组织、肿瘤组织、细胞系和血液内的分布和表达情况,结果用免疫组织化学染色图表示。为了更好地了解TOP2AmRNA 和蛋白在胰腺癌组织中的作用及其机制,通过数据挖掘,分析并探讨其在胰腺癌组织中的表达水平及预后意义,为进一步研究TOP2A在胰腺癌发生、发展中的作用机制提供理论依据。
目前关于TOP2A与肿瘤进展的研究大多集中在其对DNA 结构的调控上。TOP2A通过调控DNA链的断裂和重新连接,从而影响DNA 的拓扑状态和复制。研究表明,肿瘤的增殖、转移和化疗药物耐药性等主要是通过调节DNA 拓扑状态和DNA 复制来实现〔18〕。有研究表明,TOP2A通常在增殖活跃的细胞中过度表达,并且高表达可能与癌细胞侵袭性有关〔5〕。据报道,TOP2A过表达与脂肪肉瘤和肝细胞癌等癌症的侵袭性密切相关〔19-20〕。另外有研究报道TOP2A在胰腺癌中呈现高表达,且高表达患者生存率较低,TOP2A可激活Wnt-β-catenin 通路,诱导上皮-间充质转换,从而增加癌基因的转录,TOP2A的高表达可能与癌细胞生长速度、浸润程度和远处转移能力等有关〔21〕。通过比较TOP2A在胰腺癌和正常胰腺组织中的表达,对ONCOMINE 数据库和The Human Protein Atlas 数据库中的TOP2A分析发现,mRNA 水平和蛋白水平在胰腺癌中均呈高表达(P<0.05)。此外,TOP2A在乳腺癌、前列腺癌、鼻咽癌等恶性肿瘤中的预后价值已得到证实〔22-24〕。然而,目前TOP2A在胰腺癌中的预后价值尚未得到证实。通过Kmplot 数据库中下载TOP2AmRNA 表达水平与胰腺癌患者预后相关的数据,结果表明,TOP2AmRNA 与胰腺癌患者OS 和RFS 均有相关性,TOP2AmRNA 高表达者比低表达者预后差。因此,推测TOP2A可能成为胰腺癌预后的潜在生物标志物。另外通过基因相关性分析发现TOP2A与CDK1、HDAC1和NFE2L3等基因有显著相关性,这些基因可能相互作用参与了胰腺癌的发生与发展过程。在String 数据库中按照筛选条件,筛选出相互作用强度排位靠前的10 个与TOP2A 有相互作用的蛋白质,这些蛋白相互作用,导致TOP2A在调节DNA 结构方面的活性发生改变,可能参与了胰腺癌细胞增殖、分化及细胞周期调节等功能,在胰腺癌发生、发展中发挥着重要的调节作用,而具体机制需进一步实验验证加以阐明。值得注意的是,TOP2AmRNA 表达水平与胰腺癌临床分期差异无统计学意义,分析可能原因:①上述研究结论仅仅是在转录水平上得出的,有一定局限性;②TOP2AmRNA 表达水平与胰腺癌临床分期的关系受众多因素共同参与调节,绝非某一个促癌因子能起到决定性作用,TOP2A的作用势必被其他拮抗因子影响,而具体原因需进行后续研究加以验证。
总之,通过肿瘤基因数据库相关基因信息的深入挖掘发现,在mRNA 水平和蛋白水平上,TOP2A在胰腺癌组织中呈高表达,其表达水平与胰腺癌的预后有明显关联,通过对数据库进行大量数据挖掘为发现胰腺癌新的分子靶点奠定理论基础,也为今后胰腺癌的研究提供重要线索和数据支持。与此同时,数据库高效能的分析能力和大量的样本使分析结果具有极大的参考价值,避免了由于研究样本量过小产生的误差,合理利用已有信息资源,缩短科研周期,降低科研成本。
