银杏ERF转录因子家族的全基因组学鉴定及表达模式分析
2020-11-11张斌唐满生
张斌,唐满生*
银杏ERF转录因子家族的全基因组学鉴定及表达模式分析
张斌1,2,唐满生1,2*
(1.湖南科技学院化学与生物工程学院,湖南 永州 425199;2.湖南省银杏工程技术研究中心,湖南 永州425199)
以银杏基因组数据库为基础,通过序列比对,并经过鉴定筛选,共获得112个ERF转录因子家族成员,对其进行生物信息学及表达模式分析。结果显示:银杏112个ERF成员在系统进化树上分为10个亚家族;基因结构分析表明,大部分家族基因结构简单,仅少量基因含有1~4个内含子;基因表达分析显示,39个基因在雄蕊中表达量较高,20个基因在幼苗中表达量较高,40个基因在胚中表达量较高;部分基因在雄蕊、胚和幼苗中具有相同的表达模式,说明其可能具有类似的功能。
银杏;基因家族;生物信息学;基因结构;基因表达
转录因子在植物生长发育、响应生物胁迫和非生物胁迫方面发挥着重要作用。转录因子通过结合到DNA序列的特定位置来激活或抑制下游基因的表达,间接发挥作用[1]。目前,在植物中研究较多的转录因子家族主要包括WRKY、MYB、MADS、AP2/ERF、bZIP、NAC和bHLH等[2-5]。ERF转录因子家族是植物中较大的1个家族,属于AP2/ERF超家族,其家族成员均含有由60~70个氨基酸残基组成的AP2/ERF保守结构域(由3个β-折叠和1个α-螺旋组成)[6]。根据氨基酸序列上存在的AP2/ERF结构域的数量和含有的其他结构域,可将AP2/ERF超家族分为3个亚家族,即ERF、AP2和RAV,其中AP2家族成员包含2个重复的AP2/ERF结构域,RAV家族成员包含1个AP2/ERF结构域和1个B3结构域,ERF家族成员含有1个AP2/ERF结构域[7]。ERF家族转录因子可通过结合GCC-box顺式作用元件,参与一些激素信号途径(如乙烯、水杨酸和茉莉酸途径)和植物生长过程[6];ERF转录因子也可调节种子生长、花的发育等过程[6],参与抵抗干旱、盐、冷冻等非生物胁迫以及生物胁迫[8],在植物次生代谢产物的合成,尤其是在药用植物的活性成分生物合成途径中发挥重要作用[9]。
银杏()原产于中国,经济价值较高。董金金等[10]对银杏MADS-box家族基因进行了系统的分析;冯磊等[11]对银杏bHLH家族转录因子进行了生物信息及表达模式分析;李泽宏等[12]从银杏中克隆到1个家族基因,并对其表达模式作了分析。但是,关于银杏ERF转录因子家族的全基因组学分析和鉴定的研究尚少见报道。本研究从银杏基因组中鉴定出ERF家族成员,并对基因家族成员的理化性质、系统进化关系、基因和蛋白结构、保守结构域以及在不同组织的表达模式进行了分析,旨在为进一步探究银杏家族基因的功能提供依据。
1 材料与方法
1.1 材料
试验所使用的银杏()基因组数据以及银杏雄株开花期的雄蕊、雌株的胚和银杏幼苗的表达数据均从银杏基因组数据库(http://gigadb. org/dataset/100209)获取[13]。
1.2 方法
1.2.1银杏ERF家族成员的查找与筛选
根据已报道的拟南芥ERF转录因子家族成员的氨基酸序列[8]在银杏基因组数据库GigaDB (http://gigadb.org/dataset/100613)中进行检索,将检索得到的氨基酸序列在NCBI-CDD (https://www. ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)在线数据库中进行保守结构域的预测,去除不含有ERF转录因子的家族保守结构域或结构域不完整的序列,得到具有完整保守结构域的序列,认定为银杏ERF转录因子家族成员,用于生物信息学分析。
1.2.2银杏ERF家族理化性质及进化树的构建
使用在线数据库ExPASy分析银杏ERF转录因子家族成员的理化性质,如分子质量、等电点和不稳定系数。参考已有报道[8],从TAIR数据库(https://www.arabidopsis.org/index.jsp)下载拟南芥ERF家族成员的氨基酸序列。利用Clustal X对拟南芥和银杏ERF家族成员的氨基酸序列进行多重比对后存储为FASTA格式文件,采用邻接法(NJ)绘制系统进化树[14]。
1.2.3保守结构域、保守基序及基因结构分析
根据ERF家族氨基酸序列在NCBI-CDD数据库中的预测结果,导出各个成员的AP2/ERF保守结构域氨基酸序列,利用Clustal X对氨基酸序列进行比对,结合在线工具WebLogo(http://weblogo.berkeley. edu/logo.cgi)分析银杏ERF家族AP2/ERF结构域中不同氨基酸残基的保守性[15]。使用在线工具GSDS (Gene Structure Display Server,http://gsds.cbi.pku.edu. cn/index.php)绘制ERF编码基因结构图,分析不同基因所含有的外显子、内含子数量。运用MEME在线工具(http://meme-suite.org/)分析氨基酸序列包含的保守基序(Motif)[16]。
1.2.4银杏家族基因的表达
从银杏基因组数据库中下载银杏基因表达数据,即FPKM值,筛选出银杏ERF家族成员的FPKM值,进行Z标准化处理,在Heml 1.0软件中绘制基因的表达热图[17]。根据表达热图,分析家族基因在银杏雄株开花期的雄蕊、雌株的胚和银杏幼苗中的表达情况。
2 结果与分析
2.1 银杏ERF转录因子家族的系统进化分析
根据已知的拟南芥ERF转录因子家族成员的氨基酸序列,在银杏数据库中检索到147条可能为AP2/ERF转录因子家族成员的氨基酸序列。进一步采用NCBI-CDD在线工具对氨基酸序列上的保守结构域进行检测,剔除保守结构域中不完整的序列,最终得到112个银杏ERF转录因子家族成员。根据下载得到的121个拟南芥ERF转录因子家族成员的氨基酸序列,运用MEGA 7.0绘制系统进化树;参考拟南芥ERF家族中的亚家族分类方法[8],将银杏ERF转录因子家族分为10个亚族(图1)。其中,第Ⅱ和第Ⅲ亚家族中包含的ERF转录因子数量最多,均为20个;在第Ⅴ、第Ⅵ和第Ⅹ亚家族中包含的家族成员数量较少,第Ⅴ家族仅包含Gb 03368和Gb 31312,第Ⅵ亚家族包含Gb 10817、Gb 36136、Gb 06377、Gb 27883和Gb 06312,第Ⅹ亚家族中包含有4个成员Gb 32532、Gb 12965、Gb 12588和Gb 16683。在拟南芥中,ERF家族的第Ⅴ、第Ⅵ和第Ⅹ亚家族成员数量也较少,说明ERF转录因子在植物中相对保守。可通过分析银杏ERF转录因子与拟南芥ERF成员的进化关系,推测其功能,为深入研究ERF编码基因在银杏中的功能提供线索。

图1 银杏ERF家族的系统进化分析结果
2.2 银杏ERF家族成员的理化性质
利用ExPASy在线工具对银杏ERF家族蛋白的理化性质进行了分析。银杏ERF转录因子家族成员所含的氨基酸残基数为140~730,平均为343;在整个家族中Gb 24891具有最大的相对分子质量(83.17),Gb 37057的相对分子质量最小,为15.54,银杏ERF家族平均相对分子质量为38.16。银杏ERF第Ⅱ和第Ⅲ亚族成员的相对分子质量较低,平均相对分子质量分别为24.19和29.93;而第Ⅵ和第Ⅹ亚家族成员的平均相对分子质量较高,分别为54.25和57.83。预测得到的理论等电点为4.54~10.50,等电点大于7的银杏ERF成员仅有34个(占30.4%),其余78个(占69.6%)转录因子的等电点均小于7,说明大部分的ERF蛋白质分子中含有较高比例的酸性氨基酸。通过对蛋白质的不稳定系数进行分析,绝大多数的银杏ERF转录因子的不稳定系数大于40,为不稳定蛋白质;仅有5个ERF家族成员(Gb 17212、Gb 19320、Gb 17211、Gb 35015和Gb 24048)的不稳定系数小于40,预测为稳定蛋白质。
2.3 银杏ERF蛋白的保守结构域分析
利用Clustal X对银杏ERF转录因子家族成员的氨基酸序列进行多重比对,发现112个成员中均含有1个由60~70个氨基酸残基组成的保守区域,即AP2/ERF结构域,在其N端含有3个β-折叠,C端含有1个α-螺旋(图2-A)。利用在线工具WebLogo分析银杏ERF家族所有成员AP2/ERF结构域中氨基酸残基的保守性,结果如图2-B所示。G-10(Gly-10)、R-12(Arg-12)、R-14(Arg-14)、G-17(Gly-17)、E-22(Glu-22)、W-34(Trp-34)、G-36(Gly-36)和A-45(Ala-45)等8个氨基酸残基在银杏ERF家族中完全保守,推测这些位点可能在ERF家族蛋白功能中起重要作用;W-16(Trp-16)、R-24(Arg-24)、R-36(Arg-36)、L-35(Leu-35)、T-40(Thr-40)、A-44(Ala-44)、A-47(Ala-47)、V-48(Val-48)、D-49(Asp-49)、A-51(Ala-51)和G-57(Gly-57)等11个氨基酸残基位点在90%以上的ERF保守结构域中均存在,说明ERF家族成员具有保守性。

A为AP2/ERF保守结构域氨基酸序列的多重比对结果;B为利用AP2/ERF保守结构域氨基酸序列生成的WebLogo分析结果。
2.4 银杏ERF家族基因的基因结构分析
利用在线工具GSDS对银杏ERF家族112个成员的基因结构进行了分析,结果如图3所示。大部分的基因不含有内含子,有19个基因成员含有1个内含子,4个基因成员含有2个内含子,5个基因含有3个内含子,仅有2个基因含有4个内含子。其中,含有3个内含子的基因主要分布在第IV、VII、VIII亚家族中,含有4个内含子的2个ERF家族成员Gb 10817、Gb 12965分别属于第VI、第X亚家族。通过分析不同亚家族的基因长度,发现第II和第III亚家族中的大部分基因长度相近,但短于其他家族成员的基因长度。
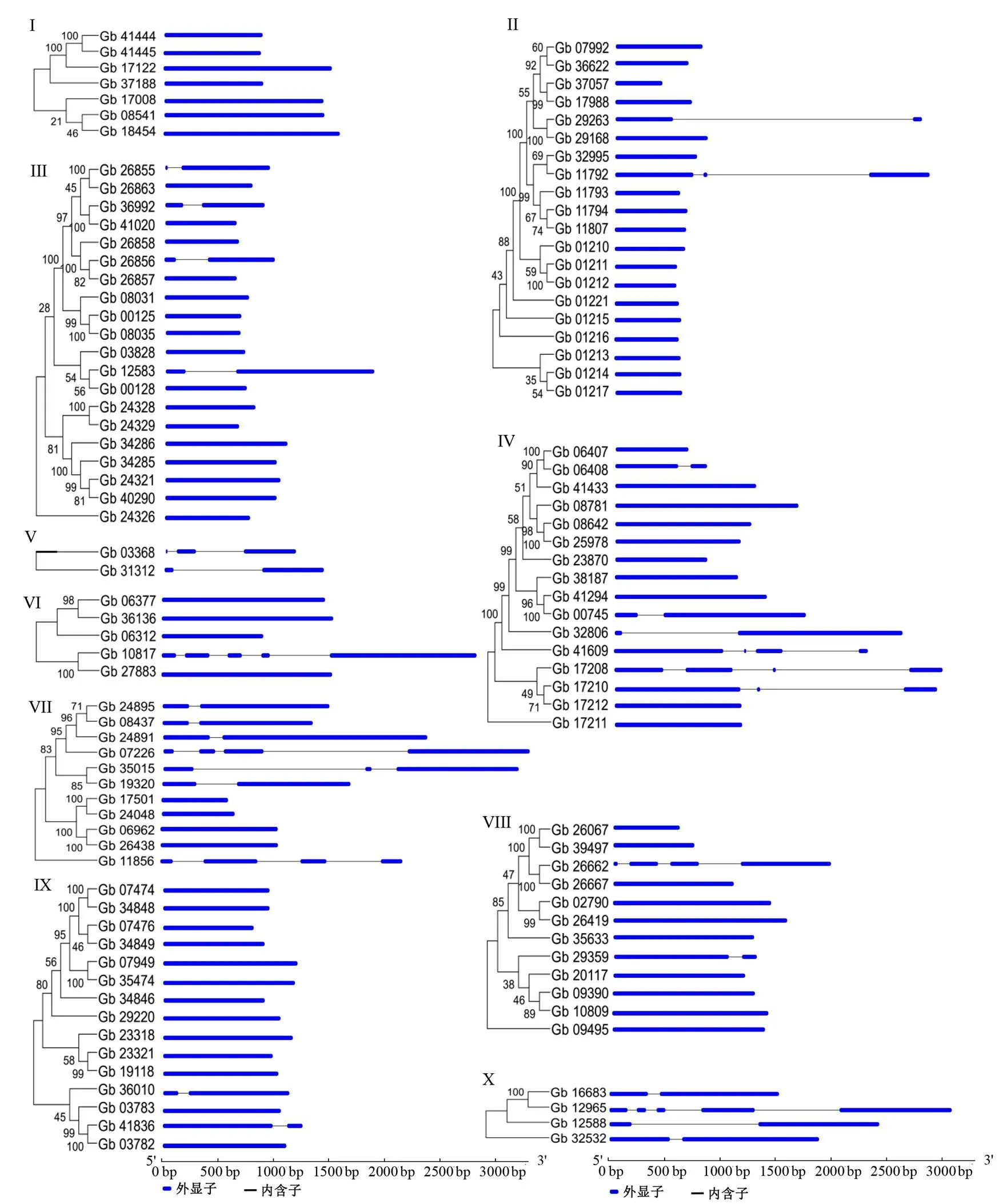
I,Ⅱ,…,X分别表示银杏ERF家族的10个亚家族,粗线部分代表外显子。
2.5 银杏ERF编码基因的表达分析
利用Heml 1.0软件绘制基因表达热图(图4)。结果显示:112个银杏家族基因在不同的组织中表达水平存在差异,39个基因成员在银杏雄株开花期的雄蕊中的表达水平较高;20个基因在银杏幼苗中的表达水平较高;40个基因在银杏雌株的胚中表达量较高;剩余的13个家族基因在3个不同组织中的表达水平一致,且总体表达量较低。基因表达的组织差异性,说明不同的家族基因具有不同的生物学功能。有部分基因表现出类似的组织表达模式,如和的表达水平在雄蕊、幼苗、胚中依次降低,说明这些基因可能具有类似的生物学功能。

图4 银杏不同组织ERF编码基因的表达分析结果
3 结论与讨论
本研究利用生物信息学手段,从银杏全基因组中鉴定出112个ERF转录因子家族成员,并对其理化性质、系统进化关系、保守结构域及保守氨基酸残基、基因结构、保守基序以及在不同组织中的表达模式进行了详细分析。根据拟南芥ERF家族在进化树上的分类,可将银杏ERF家族112个成员分为10个亚家族,与桃[9]、黄瓜[10]、马铃薯[12]中ERF家族包含的亚家族数量一致。进一步对银杏ERF家族成员AP2/ERF保守结构域中的氨基酸进行多重比对发现,Gly-10、Arg-12、Arg-14、Gly-17、Glu-22、Trp-34、Gly-36和Ala-45共8个氨基酸残基是完全保守的。而在拟南芥AP2/ERF结构域上存在7个保守的氨基酸残基(Gly-4、Arg-6、Glu-16、Trp-28、Leu-29、Gly-30)[8],在桃中只有3个完全保守的氨基酸残基(Gly-4、Leu-28和Gly-29)[9],说明家族基因在不同物种中存在一定的差异。有研究[18-20]表明,内含子在植物进化过程中起重要作用,可通过转录后不同剪切方式产生不同的转录本,以增强植物的适应性;在植物基因组扩张的初级阶段,出现了大量的内含子,随着时间的推移逐渐减少。由此推断,越高级的植物物种,其基因组中的内含子数量越少。银杏是著名的活化石森林树种,起源于2.5亿年前,现存的银杏与2.5亿年前的形态性状非常相似,说明其在进化中十分保守,这可能是其基因组中内含子数量相对较少的原因。
对银杏家族基因在不同组织中的表达分析结果表明:39个基因在雄蕊中表达水平较高;20个基因在幼苗中表达水平较高;40个基因在胚中表达水平较高;13个基因在3个不同组织中的表达水平总体较低,并且有部分基因具有相同的表达模式。说明ERF家族基因在功能上有特异性,但同源性较高且表达模式一致的基因可能具有类似的生物学功能。
一般来自不同物种的同源基因可能在功能上是保守的。有研究表明,第Ⅴ亚族拟南芥基因()在桑树中表达可以提高叶片表面的蜡质含量,减少水分的散失,增强耐旱性[21];而第VIII亚族的拟南芥家族基因在茉莉酸防御反应中起负调控作用[22];第IX亚族的和则是抗病基因,参与植物防御反应[23]。由此推断,与它们在进化树上关系较近的银杏家族基因可能具有类似的功能。
[1] ZHANG B,LIU J,YANG Z E,et al.Genome-wide analysis of GRAS transcription factor gene family inL.[J].BMC Genomics,2018,19:348.
[2] ALVES M S,DADALTO S P,GONÇALVES A B,et al. Plant bZIP transcription factors responsive to pathogens:a review[J].International Journal of Molecular Sciences,2013,14(4):7815-7828.
[3] THEIßEN G,MELZER R,RÜMPLER F.MADS-domain transcription factors and the floral quartet model of flower development:linking plant development and evolution[J].Development,2016,143(18):3259-3271.
[4] WANG Y X,LIU Z W,WU Z J,et al. Transcriptome-wide identification and expression analysis of the NAC gene family in tea plant[(L.) O.kuntze][J]. PLoS One,2016,11(11):e0166727.
[5] HOANG X L T,NHI D N H,THU N B A,et al. Transcription factors and their roles in signal transduction in plants under abiotic stresses[J].Current Genomics,2017,18(6):483-497.
[6] LI H,WANG Y,WU M,et al.Genome-wide identification of AP2/ERF transcription factors in cauliflower and expression profiling of the ERF family under salt and drought stresses[J].Frontiers in Plant Science,2017,8:946.
[7] SONG X M,LI Y,HOU X L.Genome-wide analysis of the AP2/ERF transcription factor superfamily in Chinese cabbage(ssp.)[J].BMC Genomics,2013,14:573.
[8] WAN L Y,WU Y S,HUANG J Q,et al.Identification of ERF genes in peanuts and functional analysis ofandin abiotic stress response[J]. Functional & Integrative Genomics,2014,14(3):467-477.
[9] 季爱加,罗红梅,徐志超,等.药用植物转录因子AP2/ERF研究与展望[J].科学通报,2015,60(14):1272-1284. JI A J,LUO H M,XU Z C,et al.Research and perspectives on AP2/ERF transcription factors in medicinal plants[J].Chinese Science Bulletin,2015,60(14):1272-1284.
[10] 董金金,刘伟,李萌,等.银杏MADS-box基因家族的表达及系统发育分析[J].植物生理学报,2018,54(6):1055-1063. DONG J J,LIU W,LI M,et al.Gene expression and phylogenetic analysis of MADS-box family genes in[J].Plant Physiology Communications,2018,54(6):1055-1063.
[11] 冯磊,石元豹,汪贵斌,等.银杏bHLH家族转录因子生物信息学及表达分析[J].江苏农业学报,2019,35(2):400-411. FENG L,SHI Y B,WANG G B,et al.Bioinformatics and expression analysis of transcription factors ofbHLH family[J].Jiangsu Journal of Agricultural Sciences,2019,35(2):400-411.
[12] 李泽宏,袁红慧,程华,等.银杏转录因子基因的克隆及亚细胞定位分析[J].北方园艺,2018(3):92-100. LI Z H,YUAN H H,CHENG H,et al.Cloning and subcellular localization analysis oftranscription factor inL.[J].Northern Horticulture,2018(3):92-100.
[13] GUAN R,ZHAO Y P,ZHANG H,et al.Draft genome of the living fossil[J].GigaScience,2016,5(1):49.
[14] HUANG Y,MO Y J,CHEN P Y,et al.Identification of SET domain-containing proteins inand their response to high temperature stress[J].Scientific Reports,6(1):32729.
[15] PI B Y,HE X H,RUAN Y,et al.Genome-wide analysis and stress-responsive expression of CCCH zinc finger family genes in[J].BMC Plant Biology,2018,18:373.
[16] ZHANG H,JIN J J,JIN L F,et al.Identification and analysis of the chloride channel gene family members in tobacco()[J].Gene,2018,676:56-64.
[17] WANG R Q,ZHAO P,KONG N N,et al.Genome-wide identification and characterization of the potato bHLH transcription factor family[J].Genes,2018,9(1):54.
[18] ROY S W,GILBERT W.Complex early genes[J]. PNAS,2005,102(6):1986-1991.
[19] WILLIAM ROY S,GILBERT W.The evolution of spliceosomal introns:patterns,puzzles and progress[J]. Nature Reviews Genetics,2006,7(3):211-221.
[20] ROY S W,PENNY D.A very high fraction of unique intron positions in the intron-rich diatomindicates widespread intron gain[J]. Molecular Biology and Evolution,2007,24(7):1447-1457.
[21] SAJEEVAN R S,NATARAJA K N,SHIVASHANKARA K S,et al.Expression of Arabidopsis SHN1 in Indian mulberry(L.) increases leaf surface wax content and reduces post-harvest water loss[J].Frontiers in Plant Science,2017,8:418.
[22] MCGRATH K C,DOMBRECHT B,MANNERS J M,et al.Repressor- and activator-type ethylene response factors functioning in jasmonate signaling and disease resistance identified via a genome-wide screen of Arabidopsis transcription factor gene expression[J].Plant Physiology,2005,139(2):949-959.
[23] OÑATE-SÁNCHEZ L,ANDERSON J P,YOUNG J,et al.AtERF14,a member of the ERF family of transcription factors,plays a nonredundant role in plant defense[J].Plant Physiology,2007,143(1):400-409.
Bioinformatics and expression analysis of ERF transcription factor family in
ZHANG Bin1,2, TANG Mansheng1,2*
(1.College of Chemistry and Bioengineering, Hunan University of Science and Engineering, Yongzhou, Hunan 425199, China; 2.Hunan Provincial Engineering Research Center for, Yongzhou, Hunan 425199, China)
Based on thegenome database, a total of 112 ERF transcription factor family members were obtained through blast, identification and screening, and bioinformatics and expression pattern analysis were performed. The results showed that 112 ERF members were classified into 10 groups on the phylogenetic tree. Gene structure analysis showed that only a small number of genes contain 1-4 introns. Gene expression analysis showed that 39genes were highly expressed in stamens, and 20 genes were highly expressed in seedlings, and 40 genes were highly expressed in embryos. Some genes have the same expression pattern in stamens, embryos and seedlings, suggesting that they may have similar functions.
;gene family; bioinformatics; gene structure; gene expression
Q943.2;S792.95
A
1007-1032(2020)05-0519-08
张斌,唐满生.银杏ERF转录因子家族的全基因组学鉴定及表达模式分析[J].湖南农业大学学报(自然科学版),2020,46(5):519-526.
ZHANG B, TANG M S. Bioinformatics and expression analysis of ERF transcription factor family in[J]. Journal of Hunan Agricultural University(Natural Sciences), 2020, 46(5): 519-526.
http://xb.hunau.edu.cn

2019-11-04
2019-12-23
湖南省自然科学基金(2019JJ50201);湖南科技学院重点项目(17XKY012)
张斌(1981—),男,湖南永州人,博士,讲师,主要从事植物发育生物学研究,zhangbin27104@163.com;*通信作者,唐满生,高级实验师,主要从事环境污染防治研究,manshengtang@sina.com
责任编辑:毛友纯
英文编辑:柳正
