基于线粒体基因标记的中西太平洋鲣群体遗传学分析
2020-11-10曹洋铭王丛丛
曹洋铭,王丛丛,2,3,徐 豪,刘 洋,2,3
(1.上海海洋大学海洋科学学院,上海 201306;2.大洋渔业资源可持续开发省部共建教育部重点实验室,上海 201306;3.国家远洋渔业工程技术研究中心,上海 201306)
鲣(Katsuwonus pelamis),属 鲈 形 目(Perciformes),鲭 亚 目(Scombroidei),鲭 科(Scombridae),鲣属,是一种高度洄游的物种,广泛分布于赤道至温带地区[1],是世界金枪鱼渔业重要的捕捞对象之一,其中中西太平洋金枪鱼渔场是我国捕捞鲣最为重要的渔场。鲣作为世界上总累计产量最高的金枪鱼,目前其年产量仍呈现上升趋势[2]。当前基于种群资源的评估中鲣虽然未出现过度捕捞,但捕捞量已经接近完全开发状态[3-4]。事实上,中西太平洋、大西洋的大眼金枪鱼(Thunnus obesus)以及印度洋的黄鳍金枪鱼(T.albacores)都正处在不同程度的过度捕捞状态。为了避免鲣因不合理的开发而出现过度捕捞现象,对其资源群体进行准确划分和评估至关重要。近年来除了利用生物学特征来识别和区分鲣群体外,最常用的方法是群体遗传研究,其中分子标记被广泛地应用于海洋生物的群体遗传学研究[5-6]。
物种的遗传多样性和种群遗传结构反映了物种对不同环境条件的适应性和可持续开发的潜力[7],进行鱼类遗传多样性及种群遗传结构的研究对渔业资源有效管理和充分开发利用及保护极为重要。目前,有关中西太平洋鲣遗传多样性的研究较少,仅有陈莹[8]等对中西太平洋相距较近的区域间鲣遗传多样性进行了研究。同时单一的基因标记已经无法满足物种遗传多样性的研究,为此本实验选取进化速率适中的线粒体细胞色素氧化酶Ⅰ(COⅠ)[9]基因和细胞色素b基因(Cytb)[10]这两种分子标记来分析中西太平洋鲣的群体遗传结构,以期为鲣渔业资源的管理和可持续开发利用提供科学依据。
1 材料与方法
1.1 样本采集和DNA提取
鲣样本采自于中国中水集团远洋股份有限公司,样本采集区域、数量与时间如表1所示,采集的样本取其背部肌肉,并利用标准酚氯仿抽提法提取基因组DNA[11],1%琼脂糖凝胶电泳检测,-20℃保存备用。
1.2 PCR扩增及序列测定
反应体系25μL,其中1μL的DNA模板、5XPS 5μL、PrimerSTAR MAX 0.5μL、dNTPs 2 μL、上下游引物各0.5μL、双蒸水16μL。PCR扩增引物序列根据软件Primer5.0设计,参考序列为NCBI鲣线粒体DNA全序列GI:40804664,由生工生物工程(上海)有限公司合成,具体引物序列如表2所示。
扩增Cytb与COⅠ序列的反应条件均为95℃2 min;95℃60 s;50℃30 s;72℃90s;30个循环;72℃5min。扩增后的PCR产物经琼脂糖凝胶电泳检测后,由生工生物工程(上海)有限公司进行序列测定。
1.3 数据处理与分析
Cytb与COⅠ基因测序后的序列在MEGA 7.0[12]软件中选用Clustal W进行序列比对。通过软件DnaSP6.12[13]分析核苷酸组成、单倍型数(H)、单倍型多样性指数(Hd)、核苷酸多样性指数(Pi)和基因流(Nm)。利用Network 4.0[14]软件制作基于两条基因序列的单倍型网络图,Arlepuin3.5.2.软件[15]中的AMOVA分析计算Fst值并评估种群的遗传多样性水平。采用Tajima’sD[16]、Fu’sF[17]中性检验和核苷酸不对称分布方法对种群历史动态进行分析。最后利用公式T=t×Tau/4kμ[17]计算种群的扩张时间(其中t为研究对象的生殖周期,Tau为扩张时间参数,k为本文中分析序列的碱基数,μ为对应基因序列每一代的进化速率)。
2 结果与分析
2.1 序列分析
经过ClustalW序列比对获得112条长度为1 059 bp的鲣Cytb基因序列,包含98个单倍型。其中88个单倍型分别只存在于3个群体中的某一个,所占总频率为0.897 9。在分析的Cytb基因序列中,检测到196个多态性位点,其中包含130个单一突变位点和66个简约信息位点。另外,C、T、A、G 4种碱基的比例分别为39.5%、24.6%、25.5%、10.4%。
鲣COⅠ基因序列分析长度为1 205 bp,在这106条COⅠ序列中共存在52个单倍型,其中31个单倍型分别只存在于3个群体中的某一个群体,所占总频率为0.596 2,远远低于Cytb序列。在COⅠ基因分析片段中,检测到62个多态性位点,其中包含26个单一突变位点和36个简约信息位点。另外,C、T、A、G 4种碱基的比例分别为35.2%、29.6%、28.3%、6.9%。鲣的Cytb和COⅠ基因序列中G的含量分别为10.4%、6.9%,核苷酸的组成明显偏离G。

表1 样本采集区域与数量Tab.1 Location and number of sam ples

表2 引物序列Tab.2 Primer sequences
2.2 遗传多样性分析
遗传多样性分析结果如表3所示,3个区域群体的单倍型多样性指数(Hd)均较高,但核苷酸多样性指数(Pi)均较低。在Cytb序列的分析中,Region2群体的Hd(0.995)略高于Region1(0.986)和Region3(0.993),Region2群体的Pi(0.012 0)略高于Region1(0.008 0)和Region3(0.008 0)。而在COⅠ的遗传多样性分析中,Region2群体的Hd(0.937)略低于Region1(0.953)和Region3(0.975),Region2群体的Pi(0.002 57)略低于Region1(0.002 74)和Region3(0.002 90)。
Fst值可用来估计亚种群间遗传分化,简而言之可用来衡量整个种群内亚种群之间的近交程度。如表4所示,在Cytb序列的分析中,Fst值均小于0.05,P值均大于0.05,且基因流Nm远大于1,则可认为3个群体间不存在遗传分化[18-19]。基于COⅠ序列的分析结果与Cytb序列的分析结果一致。
2.3 鲣历史种群动态
Tajima’sD和Fu’sFs中性检验在研究中被用来进行鲣的历史种群动态分析。如表5所示,基于Cytb基因序列的分析显示,所有群体的Tajima’sD和Fu’sFs均为负数,但Region 1区域群体的Tajima’sD值不存在显著差异(P>0.05),Fu’sFs值在所有区域均存在极显著性差异(P<0.01)。负的Fs值和差异显著的P值表示群体经历过扩张,从理论上说,Fu’sFs方法检测效果要比Tajima好,因此,可以认为3个区域群体在历史上经历了种群扩张事件。而基于COⅠ基因序列的分析结果显示,所有群体的Tajima’sD和Fu’sFs也均为负数,且在统计学上存在显著差异,两者结果一致。根据两段基因序列的核苷酸不配对分布分析显示(图1,图2),所有核苷酸错配分布曲线均呈现明显单峰,说明鲣群体在历史上经历了种群扩张事件,这与中性检验的结果相一致。
在种群扩张模型中对3个群体的平均Raggedness index进行计算,发现其P值均无显著性差异,表示这个数据适合于种群扩张模型。通过Arlepuin 3.5.2.2软件计算得到基于Cytb基因序列的鲣群体的Tau为6.682 93,Cytb基因的进化速率为(2%)/百万年[20-22],根据公式T=t×Tau/4kμ,进一步推算群体扩张时间,得到鲣群体扩张大概发生在7.88万年前。同样的,基于COⅠ基因序列计算得到的Tau为3.253 67,COⅠ基因的进化速率为(1%~3%)/百万年[23],推测鲣群体扩张大概发生在6.74万年~2.25万年前。

表3 基于Cytb和COⅠ序列的鲣遗传多样性参数Tab.3 Genetic diversity parameters of Cytb and COⅠgene sequences in Katsuwonus pelam is

表4 基于Cytb、COⅠ基因的鲣群体间的F st值(对角线下方)和基因流值(N m)(对角线上方)Tab.4 Population pairw ise F st values(below diagonal)and gene flow(N m)(above diagonal)of populations based on Cytb and COⅠgene sequences

表5 中性检验结果(包含P值)Tab.5 Results of neutrality tests(including associated P values)
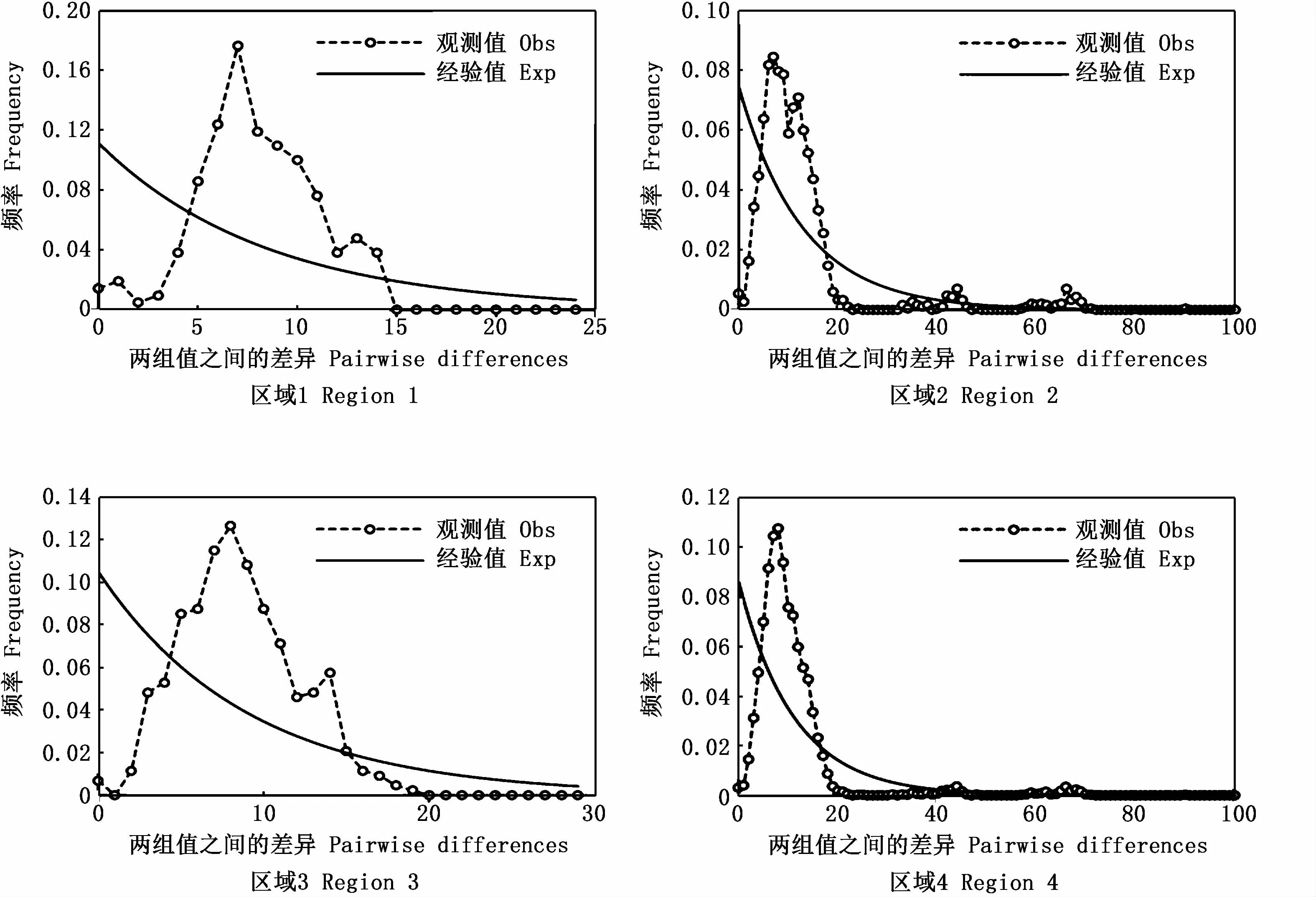
图1 基于Cytb基因的鲣群体核苷酸不配对分布Fig.1 M ismatch distribution graphs of populations based on Cytb gene sequences
2.4 鲣群体的系统进化关系
基于Cytb以及COⅠ基因单倍型序列利用Network绘制网络图(median-joining network,MJ网络图),如图3和图4所示。基于Cytb基因的MJ网络图,发现H61和H78是主要的核心单倍型,通过单一突变或多步突变相连,且3个区域单倍型间彼此相互关联,不存在与3个区域对应的独立单倍型,这也证明了3个区域群体间不存在显著的分化。在基于COⅠ基因的MJ网络图中,H1是唯一的核心单倍型,且网络图呈放射状分布,这与之前的种群扩张模式相一致,可验证种群扩张事件的发生。

图2 基于COⅠ基因的鲣群体核苷酸不配对分布Fig.2 M ismatch distribution graphs of populations based on COⅠgene sequences

图3 鲣Cytb基因单倍型的网络图Fig.3 M edian-joining network of Cytb gene hap lotypes of K.pelam is

图4 鲣COⅠ基因单倍型的网络图Fig.4 M edian-joining network of COⅠgene hap lotypes of K.pelam is
3 讨论
本研究中,两两群体间Fst分析结果表明,3个群体间不存在显著分化(Fst<0.05,P>0.05),存在频繁的基因交流(Nm>1),这与陈莹等[8]的研究结果一致(采样区域:164.80°E~172.44°E,7.71°S~3.53°N;使用线粒体DNA序列为Cytb和D-loop)。鲣是高度洄游的大洋性鱼类[24],运动能力强、活动范围广、有较为集中的产卵场,不同区域群体间有足够的机会进行基因交流,这可能是导致群体间的分化不明显的原因。FUJINO[25]的研究也表明,由于鲣随着海流的运动及其洄游行为,中西太平洋的鲣亚种群可能属于同一个产卵地洄游群体。基于Cytb以及COⅠ基因构建的MJ单倍型网络图中均出现了核心单倍型,其中基于COⅠ基因构建的MJ单倍型网络图表现为一个星状图谱,且仅有一个核心单倍型H1,其他单倍型均以H1单倍型为中心呈放射状分布,表明这3个群体可能来源于一个共同的祖先,证实了FUJINO[26]关于中西太平洋鲣为一个亚种群的研究结果。值得关注的是,基于Cytb以及COⅠ基因单倍型序列的网络图存在较大的差异,且基于两者的Hd和Pi值也在不同区域间显示出了明显的差异,这可能是由于不同基因片段进化速率不同导致的[18-21]。
Cytb和COⅠ基因序列中G的含量均明显低于其他3种碱基的含量,核苷酸的组成明显偏离G,可能与密码子位点的偏倚性和不均一性有关[27],这与硬骨鱼类核苷酸基本组成特征相符合[28]。遗传多样性分析结果显示,基于Cytb和COⅠ基因序列测得的单倍型多样性指数(Hd)都极高,而核苷酸多样性指数均较低,这可能是因为鲣群体在历史上存在迅速扩张的时期,单倍型多样性随着鲣数量的快速增加而变大,但核苷酸产生的变异没有足够的时间来积累,所以呈现高Hd低Pi的特征[7]。而基于两段基因序列的中性检验和核苷酸错配分布曲线也证实了鲣可能在末次冰期(约于7万年前开始,于1.15万年前完结)[29]经历快速的群体扩张事件。第四纪海平面变动的控制因素是冰川作用,随冰期-间冰期的交替,海平面脉动降升[30-31],本研究基于Cytb基因推算的鲣群体扩张时间(7.88万年前)与末次冰期初期的海平面上升(7万年前达到最高)时间吻合;随后海平面随着冰期与间冰期的交替不断升降并于3.5万年前再次达到最高[32],大致接近现在位置,而基于COⅠ基因推算的鲣群体扩张时间正发生在6.74万年~2.25万年前。由此,推测中西太平洋鲣种群的扩张极有可能与海平面的快速上升(海侵)有关。
4 小结
本研究结合两种分子标记对中西太平洋的鲣群体遗传结构进行研究,研究结果为鲣渔业资源的长期监测评估提供了重要依据。研究表明3个区域间鲣群体不存在显著的遗传分化,建议将中西太平洋不同区域的鲣群体划为一个种群进行管理,以便鲣资源更加合理的保护与开发利用。然而,由于Cytb和COⅠ序列作为线粒体的分子标记,相对于庞大的基因组而言无法反映鲣整体的遗传信息,对于准确检测群体遗传结构存在一定局限性。因此,中西太平洋是否存在相互独立的鲣群体仍存在不确定性,需要利用基因组覆盖面更广的分子标记对鲣群体遗传结构进行比较研究。此外,综合考虑遗传信息和海洋环境因素以及鲣生活史特征,将有助于在中西太平洋制定和实施有效的鲣渔业管理策略。
