花环肉质软珊瑚与短指软珊瑚转录组初步分析
2020-11-06李卫东王沛政
陆 昊,刘 敏,李卫东,王沛政
(1.海南热带海洋学院 生态环境学院,海南 三亚 572022;2.海南大学 海洋学院,海口 570228)
0 引言
软珊瑚(Alcyonacea)属于珊瑚虫纲的软珊瑚目,是珊瑚礁生态系统中重要组成部分[1].软珊瑚科下有肉芝软珊瑚属(SarcophytonLesson,1834)、豆荚软珊瑚属(Lobophytumvon Marenzeller,1886)和手指软珊瑚属(SinulariaMay,1898)等 18 个属,其中,肉芝软珊瑚属和豆荚软珊瑚属是软珊瑚科中比较大的属,在海南周边海域广泛分布.海南岛附近海域蕴藏着丰富的软珊瑚资源,其天然代谢产物具有非常好的生理活性,已成为人们研究的热点[2].近年来,由于环境因素的恶化,导致世界范围内珊瑚礁大面积白化和死亡[3].而珊瑚分子层面相关的研究起步较晚,李淑等[4]从细胞机制和光抑制机制2个方面综述了珊瑚白化的机制,但至今仍未有标准的解释.
随着高通量技术的发展,转录组(transcriptome)测序成为了解物种在特定条件下细胞或组织内的基因表达情况和生物学代谢途径调节更为有效的办法.2009年Meyer等[5]首次将高通量测序方法应用至珊瑚的研究,获得了大量的基因及相关注释.随后有越来越多的造礁石珊瑚转录组研究被报道,如Bellantuono等[6]报道了在高温胁迫下,鹿角珊瑚(Acroporamillepora)的70个基因出现表达异常并对其进行了注释.Moya等[7]利用改变CO2浓度研究海洋酸化对Acroporamillepora基因表达的影响,结果发现,大多数离子转运蛋白的表达不受酸化影响,但海洋酸化对一些钙化通路中蛋白有影响.刘金豆[8]的研究结果表明,在温度胁迫下,稀杯盔形珊瑚的一些基因表达出现差异,这为从基因层面解释珊瑚白化现象提供了一种新的技术手段.
近年来,关于珊瑚白化研究逐渐成为热点,大部分相关研究都集中在造礁石珊瑚.软珊瑚研究目前仅仅局限于活性物质的提取及共生微生物的分类等,软珊瑚基因组等相关研究较少.本研究选取海南省三亚市周边海域常见的软珊瑚种花环肉质软珊瑚(Sarcophytonehrenbergi)和短指软珊瑚(Sinulariaceramensis)作为研究对象.利用高通量测序技术对2种软珊瑚的转录组测序分析,比较2种软珊瑚的转录组特征和差异.旨在丰富我国南海软珊瑚的转录组信息,为珊瑚礁保护、软珊瑚白化、活性物质等研究提供理论依据.
1材料与方法
1.1 材料
花环肉芝软珊瑚(Sarcophytonehrenbergi)和短指软珊瑚(SinulariaceramensisVerseveldt)均采自三亚市西瑁岛( 18°15′N,109°22′E)的珊瑚礁区.于海南热带海洋学院珊瑚馆暂养1周,珊瑚缸水体量60 L,温度26 ℃,盐度35,pH 8.2,光暗比12 h︰12 h.待2种珊瑚所有个体触手伸展正常、恢复活力后,再进行后续实验.
1.2 RNA的提取
从2株珊瑚的冠部及茎部分别剪取2块质量约0.5 g的珊瑚组织,分别命名为P-1(花环肉芝软珊瑚冠部)、P-2(花环肉芝软珊瑚茎部)、S-1(短指珊瑚冠部)、S-2(短指珊瑚茎部),用RNAiso Plus试剂从每个样品中提取总RNA,经NanoDrop 2000c检测总RNA浓度和质量符合转录组建库标准后送往华大公司测序.
1.3 文库构建及测序
用带有OligodT的磁珠富集带有polyA尾巴的mRNA,并在得到的mRNA中加入适量打断试剂并在高温条件下使其片段化,以打断后的mRNA为模板合成一链cDNA,然后缓冲液、dNTPs、RNase H 和 DNA polymerase I合成二链cDNA[9]2,并使用试剂盒纯化回收、黏性末端修复、 cDNA 的3′末端加上碱基“A”并连接接头,然后进行片段大小选择,最后进行 PCR 扩增;构建好的文库用Agilent 2100 Bioanalyzer质检,合格后使用Illumina HiSeqTM2000进行测序.
1.4 数据处理
先过滤掉低质量、接头污染以及未知碱基N含量过高的reads得到clean reads,然后使用Trinity软件对clean reads进行组装得到Unigene,接着对得到的Unigene进行功能注释、SSR检测.最后,根据每个样品在All-Unigene的基础上计算表达量差异,完成聚类分析和功能富集分析.
2 结果与分析
2.1 花环肉芝软珊瑚和短指软珊瑚序列组装
花环肉芝软珊瑚和短指软珊瑚不同部位的4个样本测序后组装共获得130 587条Unigene(表1).Unigene是经过去冗余之后得到的基因序列,这条序列和其他的序列是非冗余的,也就是理论上其他序列都和此序列代表的不是同1个基因.花环肉芝软珊瑚冠部与茎部Unigene数目都超过80 000条,差异不大.短指珊瑚冠部Unigene数目远大于茎部,差异较为明显.2种不同珊瑚Unigene平均长度差异明显,花环肉芝软珊瑚的Unigene平均长度要略高于短指珊瑚.花环肉芝软珊瑚的N50(将所有 Unigene 从长到短排序,并依次累加长度.当累加片段长度达到总片段长度的50%时,对应片段的长度和数量,即为 Unigene N50 长度和数量)与GC含量都略大于短指珊瑚.从图1可以看出,Unigene的长度都≧200 nt.长度在200~299 nt的Unigene数目为25 420条,长度为300~399 nt的Unigene数目为13700条,而长度在2 900~2 999 nt的Unigene数目仅为643条.

表1 组装Unigene质量指标

图1 Unigene长度分布
2.2 花环肉芝软珊瑚和短指软珊瑚编码蛋白框CDS预测
花环肉芝软珊瑚和短指软珊瑚测序数据通过Blast比对SwissProt和Hmmscan,搜索Pfam蛋白同源序列,共得到86 002条CDS(编码序列,coding sequences)序列,占总Unigene的65.86%.不同长度CDS分布如图2所示,从图2可知,预测得到的CDS在300~399 nt之间所占比例最大,在200~299 nt之间所占比例最小.CDS大部分产物长度为200~1 000 bp范围,表明测序都是正确的.
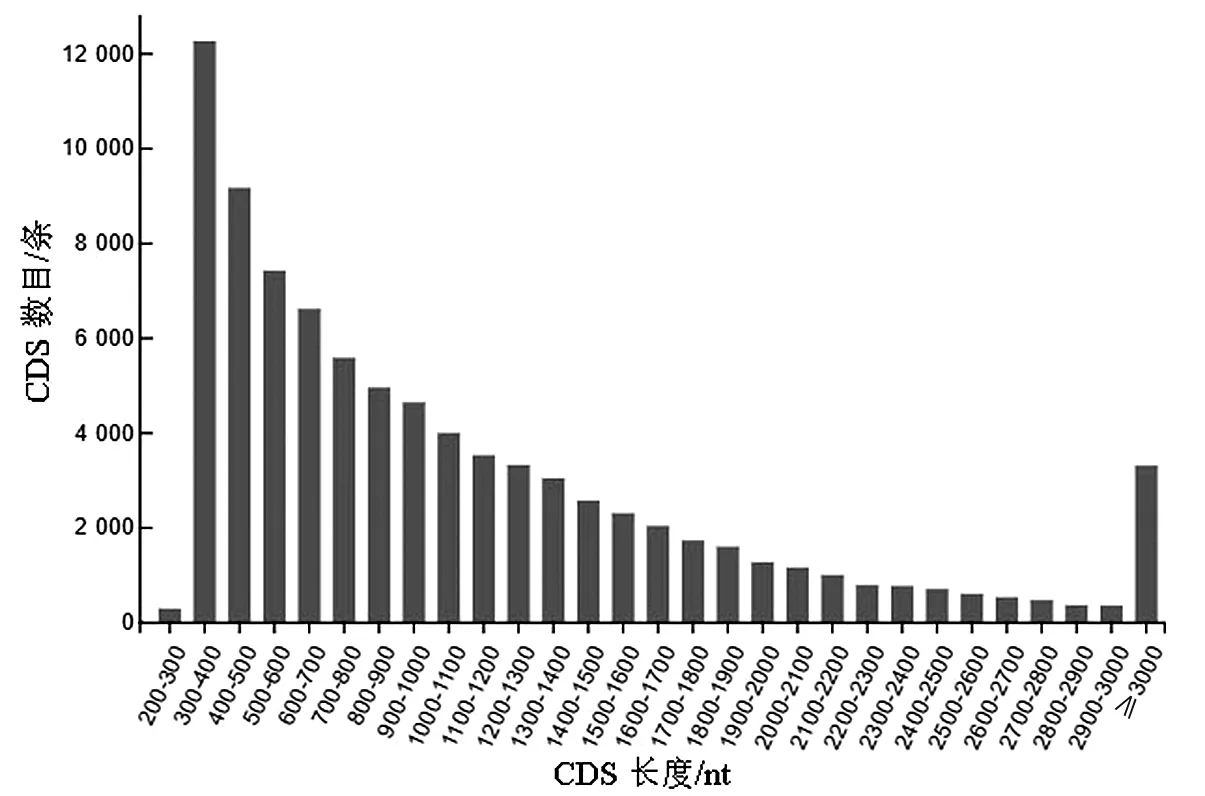
图2 CDS长度分布
2.3 花环肉芝软珊瑚和短指软珊瑚Nr注释物种分布
根据NR(Non-redundant,非冗余生物信息数据库)注释结果,统计花环肉芝软珊瑚和短指软珊瑚测序数据注释上不同物种比例(图3).从图3中可知,注释比例最高的为山地星珊瑚(Orbicellafaveolata),占所有注释结果的46.15%.注释结果比例前三占比分别为46.15%,17.83%(一种淡海葵Orbiceliafaveolata)和14.36%(形鹿角珊瑚Acroporadigitifera).另有6.48%的结果注释为另一种海葵(Nematostellavectensis). 结果显示,相关软珊瑚的基因信息几乎没有,说明花环肉芝软珊瑚和短指软珊瑚的相关研究极少.
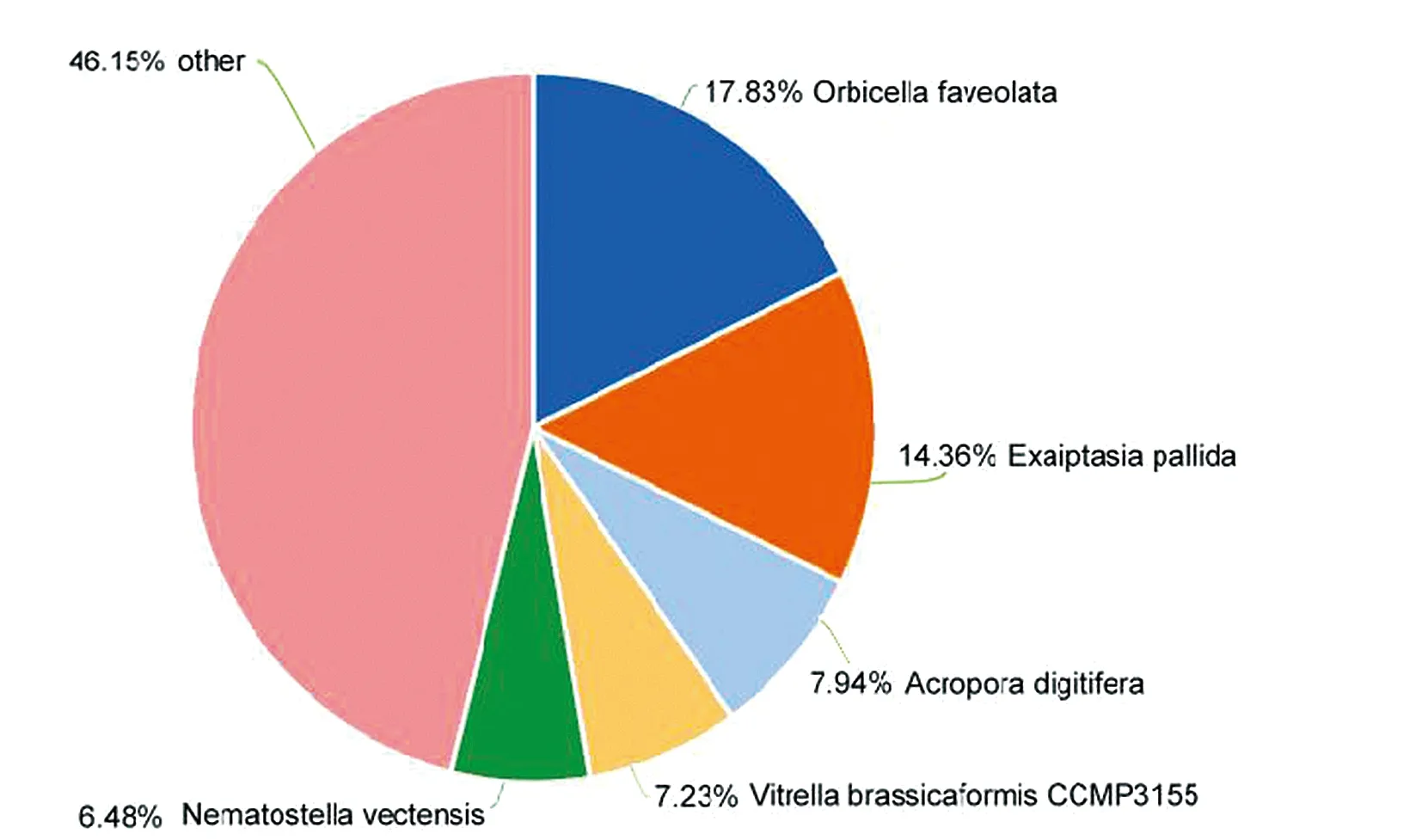
图3 NR数据库注释物种分布
2.4 花环肉芝软珊瑚和短指软珊瑚Unigene的功能分类
将所有花环肉芝软珊瑚和短指软珊瑚Unigene与NR、KOG(Clusters of orthologous groups for eukaryotic complete genomes,真核生物蛋白相邻类的聚簇)、KEGG(Kyoto encyclopedia of genes and genomes,京都基因与基因组百科全书)、Swissprot等基因数据库进行网上比对(表2),共获得花环肉芝软珊瑚和短指软珊瑚74 970条Unigene注释,占总Unigene数的57.41%.NR数据库比对注释获得的Unigene数最多,为74 019条,占总Unigene 数的56.68%.KOG数据库比对注释的Unigene 数最少,为52 318条,占总Unigene 数的40.06%.4个数据库比对结果均超过40%.以上结果表明,本研究测序的花环肉芝软珊瑚和短指软珊瑚数据约45%的基因功能注释未能比对上,表明所测序花环肉芝软珊瑚和短指软珊瑚数据中存在很多新的功能基因.

表2 Unigene功能数据库注释
花环肉芝软珊瑚和短指软珊瑚Unigene与KEGG数据库进行比对,获得56 822条注释Unigene,占总Unigene 数的43.51%.这些注释Unigene共分为6大类,分别为涉及到细胞组成过程,环境信息过程,遗传信息过程,人类疾病,新陈代谢和有机物合成过程等功能(图4).其中,注释为人类疾病的基因数最多为28 540条,占50.23%.这6个大类共分为44个分支,其中,注释为信号分支的基因最多为8 232条,占14.49%.这6大类的注释Unigene与KOG数据库进行对比,获得52 318条注释Unigene,占总Unigene 数的40.06%,共分为25类,其中:注释为细胞运动的基因最少,仅有155条,占0.3%.这可能与珊瑚附着生活运动较少有关.
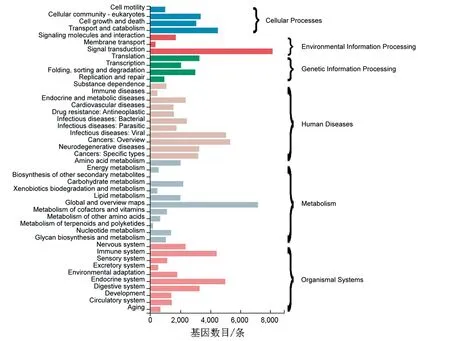
图4 Unigene KEGG功能分类
3 讨论
3.1 Unigene特征分析
本研究通过Illumina HiSeqTM2000对花环肉芝软珊瑚与短指珊瑚进行转录组测序,组装后共获得130 587条Unigene.这些Unigene通过比对4个数据库后,共得到74 970条注释基因,占总Unigene的百分比为 57.41%.出现大量未能注释的Unigene,与先前研究结果相似,这可能是由于珊瑚基因组信息的缺乏和二代测序的局限性所致[9]7.本研究中,组装后序列大小都在200~299 nt范围(25 420条),这些序列通过Blast比对SwissProt和Hmmscan搜索Pfam蛋白同源序列,仅有278条序列比对为CDS,这与魏利斌等[10]的研究结果相似(重头组装测序中序列长度较短的获得注释信息的可能性就越小).本研究结果还表明,短指珊瑚冠部Unigene数目远大于茎部,差异较为明显.其原因可能是短指珊瑚冠部细胞功能比其茎部细胞功能复杂,复杂的功能需要表达更多的功能蛋白去实现[11].Xin等[12]在牦牛转录组研究中发现,肺与肌肉之间存在显著差异,不同组织间基因表达差异可以很好解释在珊瑚不同组织间出现的表达差异.本研究中,花环肉芝软珊瑚的N50与GC含量都略大于短指珊瑚.花环肉芝软珊瑚的转录组的GC含量约49%,短指珊瑚GC含量约46%,花环肉芝软珊瑚的转录组GC含量明显大于短指珊瑚GC含量.王沛政等对花环肉芝软珊瑚与短指珊瑚进行全基因组测序研究,所获结果[13]表明:花环肉芝软珊瑚GC%含量37.96%,短指珊瑚基因组GC%含量37.09%,也呈现花环肉芝软珊瑚的基因组GC%含量高于短指珊瑚基因组GC%含量.通过以上比较发现,本研究的软珊瑚的转录组的GC含量远大于其基因组GC含量.
3.2 基因功能分析
花环肉芝软珊瑚与短指珊瑚进行转录组测序数据与NR,KOG,KEGG,Swissprot数据库进行比对共获得74 970条注释基因.在KEGG功能分类比对中,花环肉芝软珊瑚与短指珊瑚转录组中信号传导功能包含的Unigene 数目最多为8 232条,次级代谢产物生物合成的Unigene包含的数目最少,仅有45条.近年来,软珊瑚作为新型海洋药物的来源,其次级代谢产物受到关注,如王洪亮等[14]在珊瑚共生黄柄曲霉的代谢产物中发现4个细胞松弛素类化合物.随着更多的珊瑚基因组序列被挖掘,与刘金豆等[9]3先前关于稀杯盔形珊瑚转录组的研究相比,本研究组装了更多的Unigene,更多的Unigene获得了注释.本研究结果丰富了南海软珊瑚转录组数据库,可为从分子机制研究软珊瑚活性物质提供数据支持,也能为南海珊瑚礁的保护提供理论依据.
