稻曲病菌菌株GZ-14-07中携带的全病毒基因组特征研究
2020-11-02张婷婷滕丽蔡小姚龙慧李升愉刘红美
张婷婷 滕丽 蔡小姚 龙慧 李升愉 刘红美
(1. 贵州医科大学 生物与医学工程重点实验室,贵阳 550000;2. 贵州医科大学 基础医学院,贵阳 550000;3. 贵州医科大学 环境污染与
疾病监控省部共建教育部重点实验室,贵阳 550000)
水稻稻曲病(Rice false smut)称作伪黑穗病、绿黑穗病,因为它多发生在高产量收获的年份,所以又称为“丰收果”。水稻稻曲病是由于水稻花器官感染了稻曲病菌(有性态:Villosiclava virens;无性态:Ustilaginoidea virens)引起的真菌性病害[1]。感染了稻曲病菌的水稻,稻穗的空秕率增加,稻谷的千粒重降低,从而导致了水稻产量严重下降,稻米产量的损失率与稻曲球的病粒率呈显著性正相关[2]。自20世纪中叶以后,稻曲病在全世界主要稻区的发生无论在规模上还是严重程度方面均呈现出持续上升的趋势[3]。过去数十年来我国稻曲病菌导致的稻曲病的发生一直呈不断加重的趋势,已由一个次要病害逐步演变成为危害水稻生产的主要病害之一,在所有水稻种植区都已成为一种破坏性病害[4-5]。稻曲病菌侵染水稻小穗形成假黑穗球,产生大量的墨绿色或黑色的厚垣孢子可影响其他水稻米粒的光泽,造成巨大的产量损失[6-7]。此外,假黑穗球还含有黑粉菌毒素,此毒素是微管的抑制剂,可明显抑制动物的微管蛋白的组装,对人和牲畜都有毒害作用[8]。综上,稻曲病除了能使水稻严重减产,降低水稻稻米的品质,也对粮食食品安全造成了严重的威胁。
真菌病毒(Mycovirus或Fungal viruses)是一类宿主为真菌的病毒,广泛的存在于所有类群的真菌中。病毒基因组的核酸类型和病毒基因组的复制方式是真菌病毒分类的主要依据,目前所发现的绝大多数真菌病毒的基因组核酸是双链RNA(doublestranded RNA,dsRNA)。dsRNA病毒主要属于6个科,分别是:呼肠孤病毒科(Reoviridae)、全病毒科(Totiviridae)、双分病毒科(Paritiviridae)、产黄青霉病毒科(Chrysoviridae)、四分体病毒科(Quadriviridae)和巨型双节段病毒科(Megabirnaviridae)[9]。其中全病毒科具有无包膜结构的正二十面体球形颗粒的病毒粒体结构,直径约为40 nm。病毒基因组不分段,仅由一段4.6-6.7 kb的线性dsRNA分子组成,包含了两个开放阅读框(Open Reading Frame,ORF)分别编码衣壳蛋白(Capsid protein,CP)和依赖于RNA的RNA聚合酶(RNA-dependentRNA polymerase,RdRp)[9-10]。衣壳蛋白(CP)靠近5'端,而依赖于RNA的RNA聚合酶(RdRp)靠近3'端。全病毒科中一共有5个属,分别是:全病毒属(Totivirus),维多利亚属(Victorivirus),梨形鞭毛虫病毒属(Giardiavirus),滴虫病毒属(Trichomonasvirus)和 利 什 曼 原 虫 病 毒 属(Leishmaniavirus)[10]。其中能够侵染真菌的只有两个属,分别为:Totivirus和Victorivirus[11]。Totivirus主 要 侵 染Saccharomyces cerevisiae、Scheffersomyces segobiensis、Xanthophyllomyces dendrorhous等酵母和Ustilago maydis、Tuber aestivum等丝状真菌[12-14]。Victorivirus只寄生于丝状真菌中[15],而且在Victorivirus属的模式种维多利亚长螺孢190S病毒(Helminthosporimn victoriavirus 190S;HvV190S)基因组中,靠近5'端的衣壳蛋白(CP)和靠近3'端的依赖于RNA的RNA聚合酶(RdRp)之间存在一个AUGA的序列,能启动一种被称为翻译终止后又重新启动翻译(Termination and reinitiation)的机制将衣壳蛋白(CP)和依赖于RNA的RNA聚合酶(RdRp)融合成一个大的开放阅读框[16]。
2013年,首次报道了水稻稻曲病菌菌株中携带真菌病毒[17]。近几年来,越来越多关于稻曲病菌中携带真菌病毒的报道[17-25]。本研究中,我们报道了一株从稻曲病菌菌株GZ-14-07中分离的真菌病毒,病毒命名为Ustilaginoidea virensRNA virus 6(UvRV6)。在对UvRV6的全基因组序列测序和分析中,发现UvRV6属于全病毒科维多利亚属病毒的新成员。
1 材料与方法
1.1 材料
稻曲病菌菌株GZ-14-07于2014年从贵州省稻曲病菌侵染的水稻稻曲球中分离出。
1.2 方法
1.2.1 稻曲病菌菌株GZ-14-07的分离 将稻曲球切分成小块,用5%的次氯酸钠将新鲜的稻曲球进行表面消毒1 min左右,用无菌蒸馏水浸泡3次,随后用75%的酒精表面消毒l min左右,再用无菌蒸馏水浸泡3次,然后置于无菌离心管中震荡制备成稻曲病菌的厚垣孢子的悬浮液,并用无菌蒸馏水将厚垣孢子悬浮液稀释成10倍梯度的孢子液,用移液枪吸取l00 μL不同梯度的孢子液分别涂布在含有100 μg/mL头孢霉素的土豆葡萄糖培养基(Potato dextrose agar,PDA)上置于28℃的恒温培养箱中进行培养,逐日观察分离结果,待菌落长出后,挑取单个菌落。对所有成功分离获得的菌株进行编号,保存在-20℃冰箱中。
1.2.2 dsRNA的提取和纯化 挑取目标菌株GZ-14-07,在PDA培养基上培养7-10 d。将菌株GZ-14-07的菌丝体在液氮冷冻条件下用碾钵研磨成粉末状,装入无菌离心管中。每0.4 g样品中依次加入600 μL 2×GPS、600 μL苯酚(Tris-HCl 饱和,pH 8.0)、600 μL氯仿-异戊醇(24∶1)和138 μL 10% SDS,剧烈振荡。使之充分混匀,12 000 r/min 4℃离心10 min。然后取上清液约600 μL,加入0.04 g CF-11纤维素粉末和114 μL无水乙醇,混匀后4℃冰浴过夜。然后离心机12 000 r/min离心5 min后弃上清液,倒扣滤纸上吸干残液,移液器加入600 μL清洗缓冲液,混匀后静置1 min;重复洗脱3次后,加入640 μL 1× STE,混合摇匀,12 000 r/min离心8 min并吸取600 μL上清液至新的无菌离心管中,加入1/10体积的3 mol/L NaAc,加入1倍体积的异丙醇,充分混匀后于-20℃冰箱中静置2 h,12 000 r/min 4℃离心30 min,去除上清液,加入500 μL 75%乙醇,振荡无菌离心管,将沉淀弹起,清洗沉淀,12 000 r/min 4℃离心5 min后弃去上清液,用移液枪吸去管底的多余液体,风干沉淀10 min,加入20 μL DEPC-H2O,沉淀溶解完全得到菌株GZ-14-07中的真菌病毒。用DNase I(RNase free,TaKaRa,Dalian,China)和S1核酸酶(TaKaRa,Dalian,China)处理样品,去除样品中的DNA和单链RNA(single-stranded RNA,ssRNA)污染。样品在1%琼脂糖凝胶中电泳,用0.1 μg/mL溴化乙锭染色,紫外下观察UvRV6的条带特征。
1.2.3 cDNA合成和分子克隆 将纯化的dsRNA样品作为模板,采用随机引物扩增法获得了UvRV6的随机序列[26]。病毒末端克隆的方法参考Potgieter等采用的方法[27],首先在dsRNA的两末端加上一段已知序列的接头片段,然后将加上接头的dsRNA进行反转录合成cDNA第一条链,最后用与该序列互补的一段寡核苷酸作为引物,另一引物根据已测定序列设计,进行逆转录-聚合酶链反应(Reverse transcription-polymerase chain reaction,RT-PCR)获取dsRNA末端的序列。将扩增的cDNA产物与pMD18-T载体(TaKaRa,Dalian,China)进行连接,并热激转化大肠杆菌DH5α,鉴定阳性克隆,进行测序分析。
1.2.4 序列拼接和系统进化分析 得到的序列被组装并上传GenBank数据库中,获取的登录号为KX171636。序列拼接、分析和比对使用DNAMAN 7.0和COBALT web 服务器(https://www.ncbi.nlm.nih.gov/tools/cobalt/cobalt.cgi?link_loc=BlastHomeLink);序列同源性搜索使用NCBI网站的在线BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)。系统进化树的构建使用的是邻接法(Neighbor-Joining,NJ)。NJ树的计算使用MEGA6.0软件[28],采用默认参数设置,重复抽样次数为1 000。用DotKnot程序预测RNA假结[29]。
2 结果
2.1 UvRV6的条带类型
水稻稻曲病菌株GZ-14-07中提取的真菌病毒,经DNase I和S1核酸酶处理,在1%琼脂糖凝胶电泳检测可见到在6 kb附近有一条单一的明显的dsRNA条带(图1),命名为Ustilaginoidea virensRNA virus 6(UvRV6)。
2.2 UvRV6序列分析
UvRV6全长5 043个碱基(图2),G+C含量为48.8%。通过随机引物扩增法得到UvRV6的随机片段(图2中1-15),经过DNAMAN 7.0软件进行序列拼接,发现并不能完全拼接出一条完整的全基因组序列。随后,根据已获取的基因组序列设计特异性的搭桥引物A-F,通过PCR克隆出未获得的基因组序列,最后通过DNAMAN 7.0软件拼接得到一条完整的UvRV6基因组序列。

图1 菌株GZ-14-07的1%琼脂糖凝胶电泳检测图
2.3 UvRV6的全基因组序列
UvRV6含有两个开放阅读框(ORF1和ORF2)(图3)。ORF1起始于第222位碱基,终止于第2 505位的UGA密码子,全长2 280个碱基,编码760个氨基酸(79.6 kD)。ORF2起始于第2 502位的AUG密码子,终止于第4 991位碱基,全长2 385个碱基,编码830个氨基酸(91.5 kD)。5'和3'末端非翻译区分别长222个碱基和52 个碱基。通过BlastP比对分析显示ORF1与全病毒科维多利亚属病毒的衣壳蛋白(CP)有很高的相似性,尤其是与Ustilaginoidea virensRNA virus 3(UvRv3)的衣壳蛋白(CP)的同源性达到73%;ORF2与全病毒科维多利亚属病毒的依赖于RNA的RNA聚合酶(RdRp)有显著的相似性,跟UvRV3的依赖于RNA的RNA聚合酶(RdRp)同源性高达75%。因此,UvRV6的ORF1编码病毒的衣壳蛋白(CP),ORF2编码病毒的依赖于RNA的RNA聚合酶(RdRp),UvRV6是全病毒科维多利亚属中的一员。四核苷酸(AUGA)将ORF1和ORF2连接起来,它包含ORF1的终止密码子(UGA)和ORF2的起始密码子(AUG),在全病毒科维多利亚属中这4个核苷酸(AUGA),能启动一种叫翻译终止后又重新启动翻译(Termination and reinitiation)的机制使ORF1和ORF2被融合成一个大的开放阅读框(ORF)(图3)。
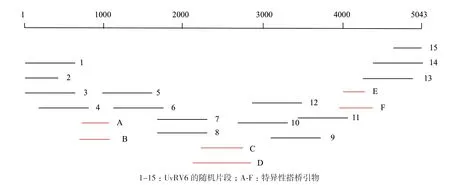
图2 UvRV6的克隆策略示意图
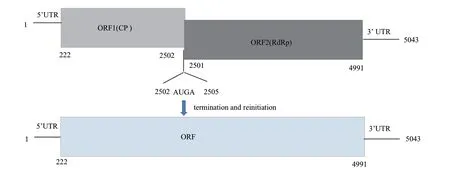
图3 UvRV6的翻译终止后又重新启动翻译机制
2.4 系统进化树构建
为了构建UvRV6的系统进化树,我们选择了21株全病毒科成员,包括4株Totivirus、9株Victorivirus、3株Leishmaniavirus、3株Trichomonasvirus和2株Giardiavirus。基于选择的21株全病毒科成员 和UvRV6的RdRp区 域,运 用MEGA6.0软 件中的邻接法构建系统进化树。从系统进化树得知,UvRV6跟其他的全病毒科(Totiviridae)维多利亚属(Victorivirus)的病毒成员汇集在一起,属于全病毒科维多利亚属的新成员。尤其跟Ustilaginoidea virensRNA virus 3(UvRV3)的亲缘关系最近(图4)。
2.5 UvRV6和相关病毒在病毒大小及其他结构方面的比较分析

图4 UvRV6的系统进化树

表1 UvRV6和相关病毒在病毒大小和其他结构方面的比较分析
选择6个已报道的从U.virens中分离的全病毒科维多利亚属的真菌病毒,在大小及其他结构方面的一些特征与UvRV6进行比较(表1)。与其他6个真菌病毒相比,UvRV6显示出一些异同点。由表1可知UvRV6的基因组全长5 043个碱基,其他成员的基因组都在5 000 个碱基左右,基因组的长度相差不大;UvRV6的两个ORF框的重叠区域是四核苷酸(AUGA),而其他成员的ORF框重叠区域是五核苷酸(UAAUG),显示出UvRV6和这6个全病毒科维多利亚属的真菌病毒中都存在翻译终止后又重新启动翻译(Termination and reinitiation)的机制,但是它们的重叠区域不同;通过对UvRV6和维多利亚属的这6个真菌病毒分析,发现这些真菌病毒的5'末端和3'末端分别有5个核苷酸是较保守的序列,其中UvRV6的5'末端是GGGCU,U. virensRNA virus 5、U. virensRNA virus 1 strain Uv0901和U.virensRNA virus 1 strain JYH-ZT是UGAAA,U. virensRNA virus L和U. virensRNA virus 3是CGAAA,U.virensRNA virus 1 strain HNHS-1是UAAUG,UvRV6的5'末端与这几个病毒相比都是不同的;UvRV6的3'末端是CAUCG,U. virensRNA virus 5是UCAAA,U. virensRNA virus L是UUUUU,U. virensRNA virus 3是GCCAA,U. virensRNA virus 1 strain Uv0901和U. virensRNA virus 1 strain JYH-ZT是GGCAA,U.virensRNA virus 1 strain HNHS-1是ACAAA,UvRV6的3'末端与这几个病毒相比都是不同的,从UvRV6的ORF框重叠区域和5'末端,3'末端与其他病毒相比没有相同的地方,显示了UvRV6在基因组结构上的独特性。BlastP分析表明,UvRV6的衣壳蛋白(CP)与U. virensRNA virus 3的同源性最高为73%,与其他病毒的同源性为38%-39%;UvRV6的依赖于RNA的RNA聚合酶(RdRp)与U. virensRNA virus 3的同源性最高为75%,与其他病毒的同源性分别为31%-38%。另外,UvRV6和其他6个病毒都有合成H型假结或茎环的结构。
3 讨论
在很多病毒的基因表达过程中存在一种叫程序性核糖体移码(Programmed Ribosomal Frameshifting,PRF)的蛋白质翻译调节机制,常见的类型包括,-1程序性核糖体移码机制(-1PRF)和+1程序性核糖体移码机制(+1PRF)。-1PRF机制的产生需要两个主要的信号元件,一个是七聚体核糖体移码序列(XXX_YYY_Z,XXX=(AAA,TTT,CCC,GGG),YYY=(AAA,UUU),Z=(A,U,C));另一个是能刺激程序性-1PRF的RNA二级结构序列,即一个“茎环”或“假结”结构;目前研究表明,+1PRF机制的产生只需要一个六聚体核糖体移码序列元件(UUU_CGN,N=A,U,G,C)[32]。然而,在真菌病毒的全病毒科(Totiviridae)维多利亚属(Victorivirus)成员中,由连续的终止密码子和起始密码子序列(如AUGA,UAAUG)和上游一个“茎环”或“假结”结构构成的-1程序性核糖体移码机制,这种机制也被称为内部起始机制[9,33]。
在UvRV6的ORF1和ORF2之 间,我 们 发 现AUGA移码序列和上游茎环结构序列(小写字母表示形成茎的碱基对):(GAUGCggccAcagcCACUgcugg gccUGCGCCGCAAGUAUGA)的存在。另外,UvRV6的BlastP比对结果,表明UvRV6中ORF1与全病毒科维多利亚属病毒的衣壳蛋白(CP)非常相似,其中,跟Ustilaginoidea virensRNA virus 3(UvRv3)的衣壳蛋白(CP)有73% 的同源性。Phomopsis vexansRNA virus(PvRV)的衣壳蛋白(CP)有66%的同源性。ORF2也与全病毒科的依赖于RNA的RNA聚合酶(RdRp)有显著的相似性,包括跟UvRV3的依赖于RNA的RNA聚合酶(RdRp)同源性为75%。Phomopsis vexansRNA virus(PvRV)的依赖于RNA的RNA聚合酶(RdRp)同源性为51%和Nigrospora oryzaevictorivirus 1(NoV1)的依赖于RNA的RNA聚合酶(RdRp)同源性为50%。从UvRV6的ORF1和ORF2跟全病毒科维多利亚属的衣壳蛋白(CP)和依赖于RNA的RNA聚合酶(RdRp)的相似性来看,可以推测出UvRV6属于全病毒科维多利亚属中的新成员。新的真菌病毒的发现,以及获取真菌病毒的全基因组序列并对其进行分类学研究,能丰富真菌病毒资源库,并提升对真菌病毒分类和进化方面的认识。
4 结论
UvRV6属于全病毒科维多利亚属的新成员,全长5,043个碱基,含有两个开放阅读框(ORF1和ORF2)。ORF1 长2,280个碱基,760个氨基酸(79.6 kD),编码衣壳蛋白(CP);ORF2 长2 385个碱基,830个氨基酸(91.5 kD),编码依赖于RNA的RNA聚合酶(RdRp)。UvRV6的ORF1和ORF2之间存在由AUGA移码序列和上游茎环结构序列构成的-1程序性核糖体移码信号元件。
