基于线粒体基因组序列的鞘翅目肉食亚目水生类群系统发育分析
2020-10-15田天,袁缓,陈斌
田 天, 袁 缓, 陈 斌
(重庆师范大学昆虫与分子生物学研究所, 媒介昆虫重庆市重点实验室, 重庆 401331)
肉食亚目(Adephaga)水生类群属鞘翅目(Coleoptera),是一类具有水生习性的肉食性真正水生甲虫,其中很多种类是重要的环境监测生物;还有一些类群的行为活动比较特殊,可以为化学防御及机器人运动等研究提供重要参考(Romey, 1995; Romey and Wallace, 2007; 习欠云和王殉章, 2010; Biltonetal., 2019)。根据Bouchard等(2017)的统计,全世界已描述的肉食亚目水生类群有218属5 480余种;肉食亚目水生类群共包括8个科,即豉甲科(Gyrinidae)、沼梭甲科(Haliplidae)、伪龙虱科(Noteridae)、瀑甲科(Meruidae)、龙虱科(Dytiscidae)、水甲科(Hygrobiidae)、两栖甲科(Amphizoidae)和壁甲科(Aspidytidae)(Bouchardetal., 2011, 2017)。肉食亚目水生类群已被广泛研究,但是其系统发育关系仍没有得到统一的结论。2002年之前对该类群的研究主要是利用不同形态学特征进行分析,Baehr, Kavanaugh和Beutel等多位分类学家先后根据不同的形态学特征提出了差异较大的多种系统发育假说(习欠云和王殉章, 2010)。自Ribera等(2002)使用18S rRNA基因作为分子标记对肉食亚目水生类群进行系统发育分析之后,Beutel, Balke及Xi 3位学者分别综合更多的形态学特征和分子标记数据对该类群进行了系统发育重建(Beuteletal., 2006, 2012; Balkeetal., 2008; Xietal., 2008)。这些综合更多数据的研究都支持豉甲科作为肉食亚目水生类群的基部类群,然而该类群其他科之间的系统发育关系仍没有得到统一的结论。
线粒体基因组是真核生物的细胞器基因组,具有母系遗传、基因组小、组成结构稳定等特点,被广泛应用于比较基因组学、系统发育分析、分子鉴定等研究领域(孙铮等, 2010)。线粒体基因组的13个蛋白质编码基因(protein-coding genes, PCGs)已被广泛证实可以用来解决昆虫纲中不同阶元的系统发育问题(Cameron, 2014)。近年来,许多学者利用线粒体基因组数据对鞘翅目不同阶元和类群进行过系统发育分析,但很少有研究对肉食亚目水生类群进行专门探究(Yuanetal., 2016; Lopez-Lopez and Vogler, 2017; Nieetal., 2018, 2019; Biltonetal., 2019)。GenBank数据库已公布的肉食亚目水生类群数据中(截止2019年9月30日),共有17个物种的线粒体基因组测序较完整,覆盖了肉食亚目水生类群的8个科。在本研究中,我们测定和分析了圆鞘隐盾豉甲Dineutusmellyi和齿缘龙虱Eretessticticus的线粒体全基因组序列,并利用肉食亚目水生类群8科19种的线粒体基因组序列进行了比较分析和系统发育构建,系统地讨论了肉食亚目水生类群的系统发育和进化关系。
1 材料与方法
1.1 线粒体基因组测序及GenBank数据下载
新测序的2种鞘翅目昆虫标本均侵泡在无水乙醇中,并保存于重庆师范大学昆虫与分子生物学研究所的-20℃冰箱中。齿缘龙虱E.sticticus于2017年7月采集自重庆市城口县大巴山;圆鞘隐盾豉甲D.mellyi于2018年10月采集自福建省武夷山。选择这两种昆虫的胸部和足的肌肉组织提取基因组总DNA,并送至深圳市惠通生物科技有限公司构建350 bp的小片段文库和进行高通量测序。采用Illumina HiSeq X Ten测序技术进行PE150测序,测序结果下机后经过质控分别得到16.47 Gb(齿缘龙虱)和18.48 Gb(圆鞘隐盾豉甲)的clean reads。之后,按如下步骤进行从头组装:(1)使用SPAdes 3.0.0将clean reads进行从头组装得到contigs(Bankevichetal., 2012)。(2)基于序列特征,将来源不同的contigs进行分类筛选。将抽取的线粒体序列合并成单独的fasta文件。(3)利用PRICE(paired-read iterative contig extension)进行迭代延伸,直至延伸的序列稳定,长度不再发生变化为止,最终得到完整的线粒体基因组全序列。
目前,GenBank数据库中已公布了线粒体基因序列的肉食亚目水生类群共有2 950余种,但是其中大部分物种都只测序了少数几个基因,只有14个物种的线粒体基因组数据是测序完整的。除此以外,Noteridae sp.,Meruphyllisae和Gyrinidae sp.这3个关键类群物种的已测序线粒体基因序列中仅缺少nad2基因,本研究选取这3个物种的其他12个PCGs序列用于本研究。因此,本研究主要是对19个肉食亚目水生类群线粒体基因组的PCGs序列进行比较分析,同时选用2个步甲科物种Damastermirabilissimus和Abaxparallelepipedus作为外群进行该类群的系统发育重建分析。本研究涉及到的物种及线粒体基因组信息见表1。
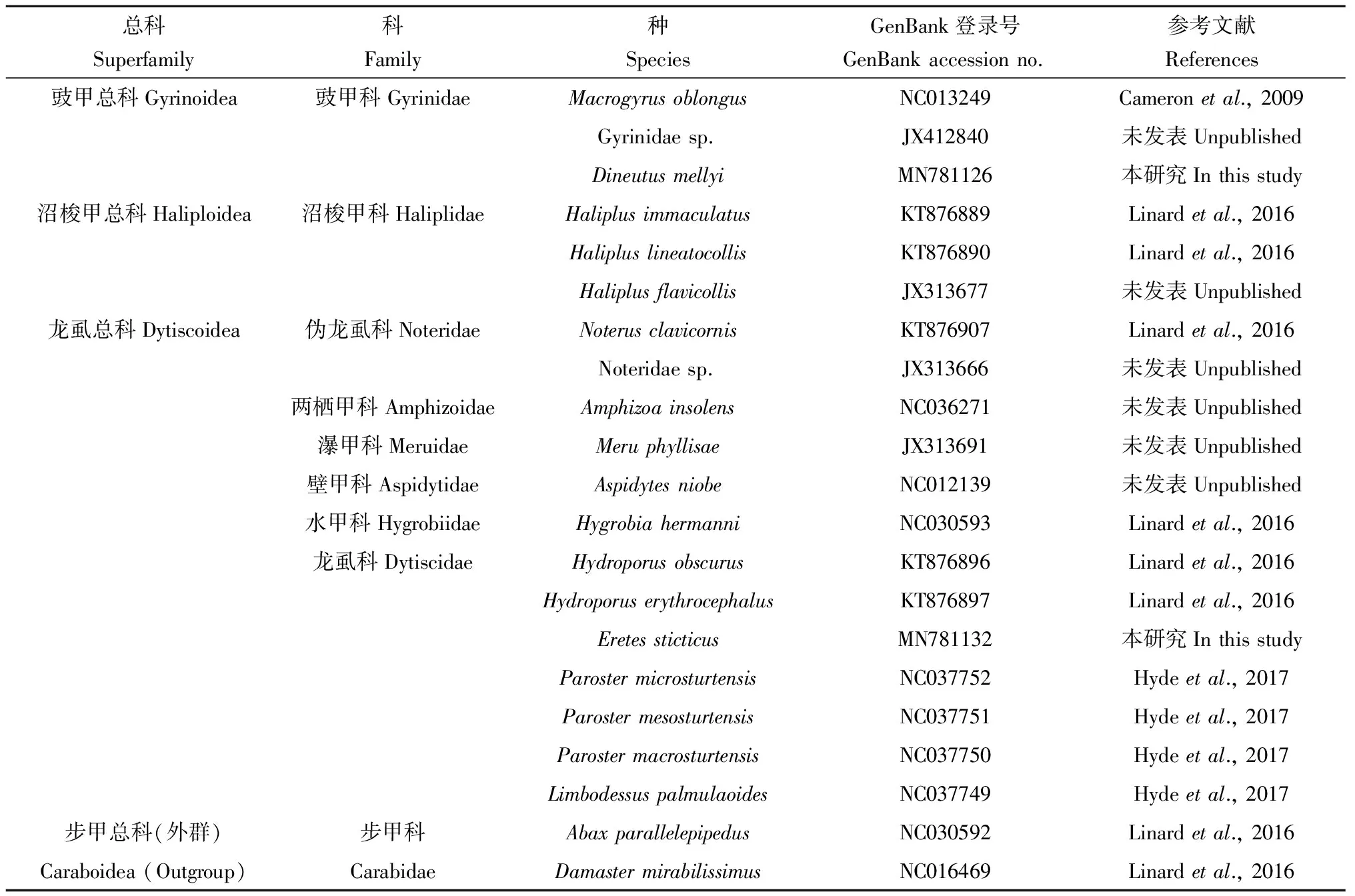
表1 肉食亚目水生类群19个物种的线粒体基因组信息
1.2 基因注释和比较基因组分析
先使用Mitos(http:∥mitos.bioinf.uni-leipzig.de/index.py)对圆鞘隐盾豉甲和齿缘龙虱的线粒体基因组全序列进行初步注释(Berntetal., 2013)。之后,将PCGs和rRNA基因通过与近缘种线粒体基因组的注释结果进行比较,并在Geneious v4.8.5中对初步注释结果进行校正(Kearseetal., 2012)。tRNA基因使用tRNAscan-SE 2.0进行校正和二级结构的预测分析(Lowe and Eddy, 1997)。注释完成后使用OGDRAW1.3.1绘制圆鞘隐盾豉甲和齿缘龙虱的线粒体基因组结构图(Lohseetal., 2007)。13个PCGs的AT含量使用MEGA 6.0进行计算(Tamuraetal., 2013);密码子使用次数及相对同义密码子使用频率(relative synonymous codon usage, RSCU)使用CodonW v.1.4.4进行计算(Peden, 2000);为了分析13个PCGs在肉食亚目水生类群进化过程中的基因突变模式是否存在差异,分别计算了13个PCGs的非同义突变率(Ka)、同义突变率(Ks)以及非同义突变率与同义突变率的比值(Ka/Ks)。Ka/Ks值是基因进化过程中选择压力的简单量度,值的大小分别表示:中性选择(Ka/Ks=1)、阴性或纯化选择(Ka/Ks<1)、阳性选择(Ka/Ks>1)。Ka/Ks越接近1,说明该基因的选择压力越小(Hurst, 2002; Tomasco and Lessa, 2011)。13个PCGs在进化过程中受到的选择压力通过DnaSP 5.10.01计算Ka和Ks值进行分析(Librado and Rozas, 2009)。
1.3 系统发育关系分析
13个PCGs的多重序列比对在TranslatorX Server(http:∥translatorx.co.uk/)上完成:使用MAFFT对13个PCGs分别进行多重序列比对,并使用Gblocks删除空位及模糊位点。单个基因的多重序列比对结果使用SequenceMatrix串联在一起(Vaidyaetal., 2011),最终得到13个PCGs的氨基酸串联序列和核苷酸串联序列两个数据集。使用PartitionFinder2计算每个数据集的最佳分区及其相应的进化模型(Lanfearetal., 2017)。
系统发育建树在CIPRES Science Gateway(https:∥cushion3.sdsc.edu/portal2/)在线平台上完成。我们利用最大似然法(maximum likelihood, ML)和贝叶斯推断法(Bayesian inference, BI)两种方法进行系统发育分析:使用IQ-TREE 1.6.10进行最大似然法建树,分支节点的可靠性采用1 000次ultrafast bootstrap进行评估;使用MrBayes 3.2.7a进行贝叶斯推断法建树,参数设置为:4条独立的马尔可夫链(Markov chains)同时运行800万代(generation)。每运行1 000代取样一次,当ESS(estimated sample size)>100且PSRF(potential scale reduction factor)接近1.0时,即认为两个分析过程趋于稳定状态,舍弃25%的老化样本, 剩余样本用来构建50%一致树,并计算出每个节点的贝叶斯后验概率值(posterior probability, PP)。
基于不同数据集进行系统发育分析的结果不完全一致,根据建树结果我们利用tree-puzzel 5.2软件中的四簇似然映射(four-cluster likelihood mapping, FcLM)分析进一步进行了系统发育树拓朴结构检验(Schmidtetal., 2002)。
2 结果
2.1 圆鞘隐盾豉甲和齿缘龙虱线粒体基因组
如图1所示,圆鞘隐盾豉甲和齿缘龙虱的线粒体基因组全长分别为16 123 bp和16 196 bp,都是由37个基因(13个蛋白质编码基因、2个rRNA基因、22个tRNA基因)和一个D-loop区(控制区)组成的环状DNA分子。基因排列顺序与已测定的肉食亚目水生类群物种完全一致,没有发现基因重排。与大部分昆虫一样,除trnS1缺少DHU臂外,剩下21个tRNA基因均能形成经典的三叶草结构,包括氨基酸接受臂、DHU臂、TψC臂和反密码子臂4个臂和1个可变环(孙铮等, 2010; Sunetal., 2019)。
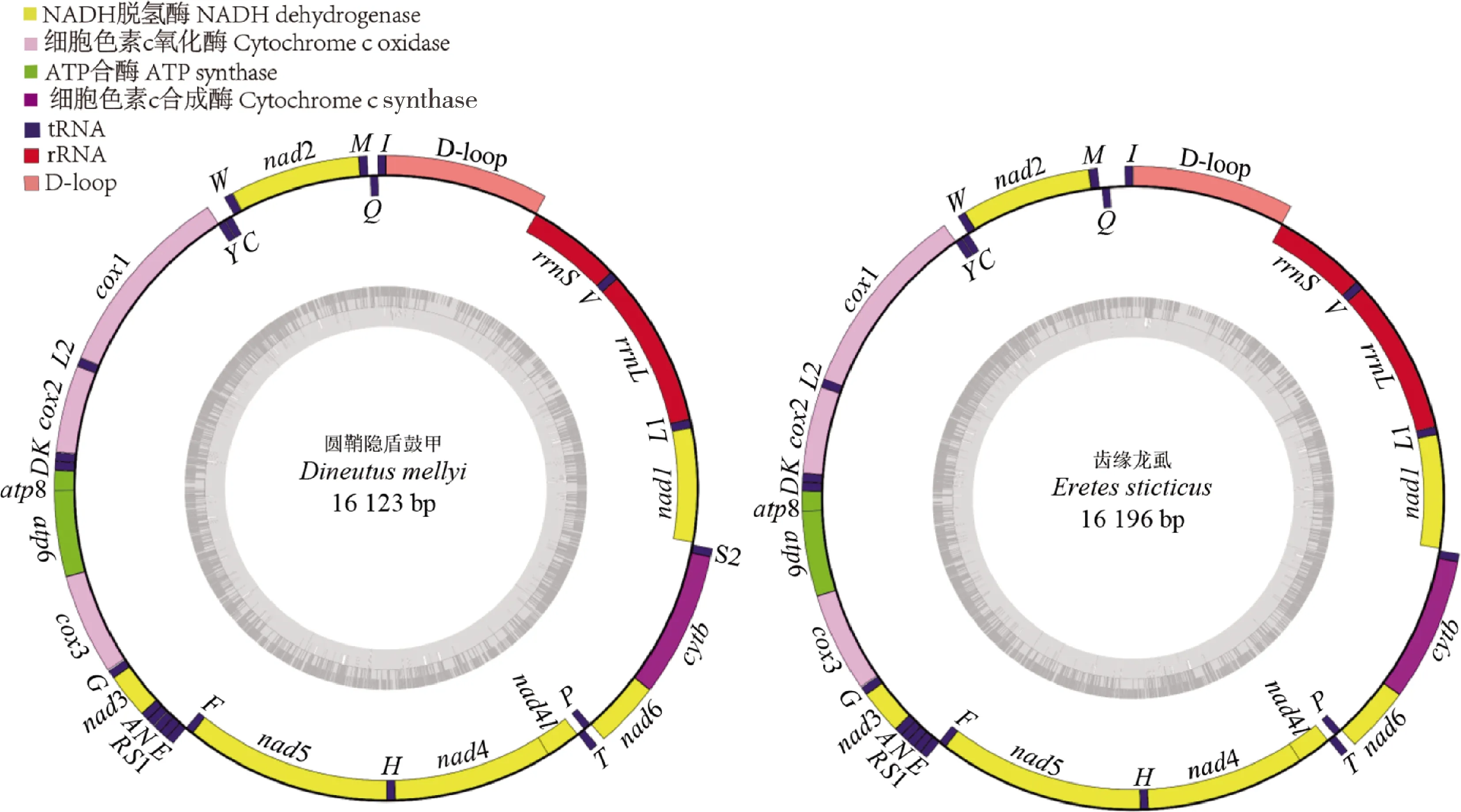
图1 圆鞘隐盾豉甲和齿缘龙虱的线粒体基因组结构
如表2所示,圆鞘隐盾豉甲线粒体基因组的37个基因排列比较紧凑,仅有一些较小的基因间隔区(1~22 bp)。13个PCGs的序列总长度为11 178 bp(去除终止密码子),除nad1基因使用TTG作为起始密码子外,其余12个PCGs的起始密码子均为ATN。除nad5基因和nad4基因具有不完整终止密码子T外,其余PCGs的终止密码子均为典型的TAA/TAG。rrnL基因和rrnS基因的大小分别是1 317 bp和785 bp;非编码的D-loop区位于rrnS和trnI之间,长度为1 289 bp。
如表2所示,齿缘龙虱的线粒体基因组除了一些较小的基因间隔区(1~16 bp)外,在trnI基因和trnQ基因之间还存在一个长度为125 bp的非编码区。13个PCGs的序列总长度为11 181 bp(去除终止密码子),除nad1基因使用TTG作为起始密码子外,其余12个PCGs的起始密码子均为ATN。cox2,nad3,nad5和nad4 4个基因的终止密码子为不完整终止密码子T,其余PCGs的终止密码子均为典型的TAA/TAG。rrnL基因和rrnS基因的大小分别是1 310 bp和785 bp;非编码的D-loop区位于rrnS和trnI之间,长度为1 286 bp。
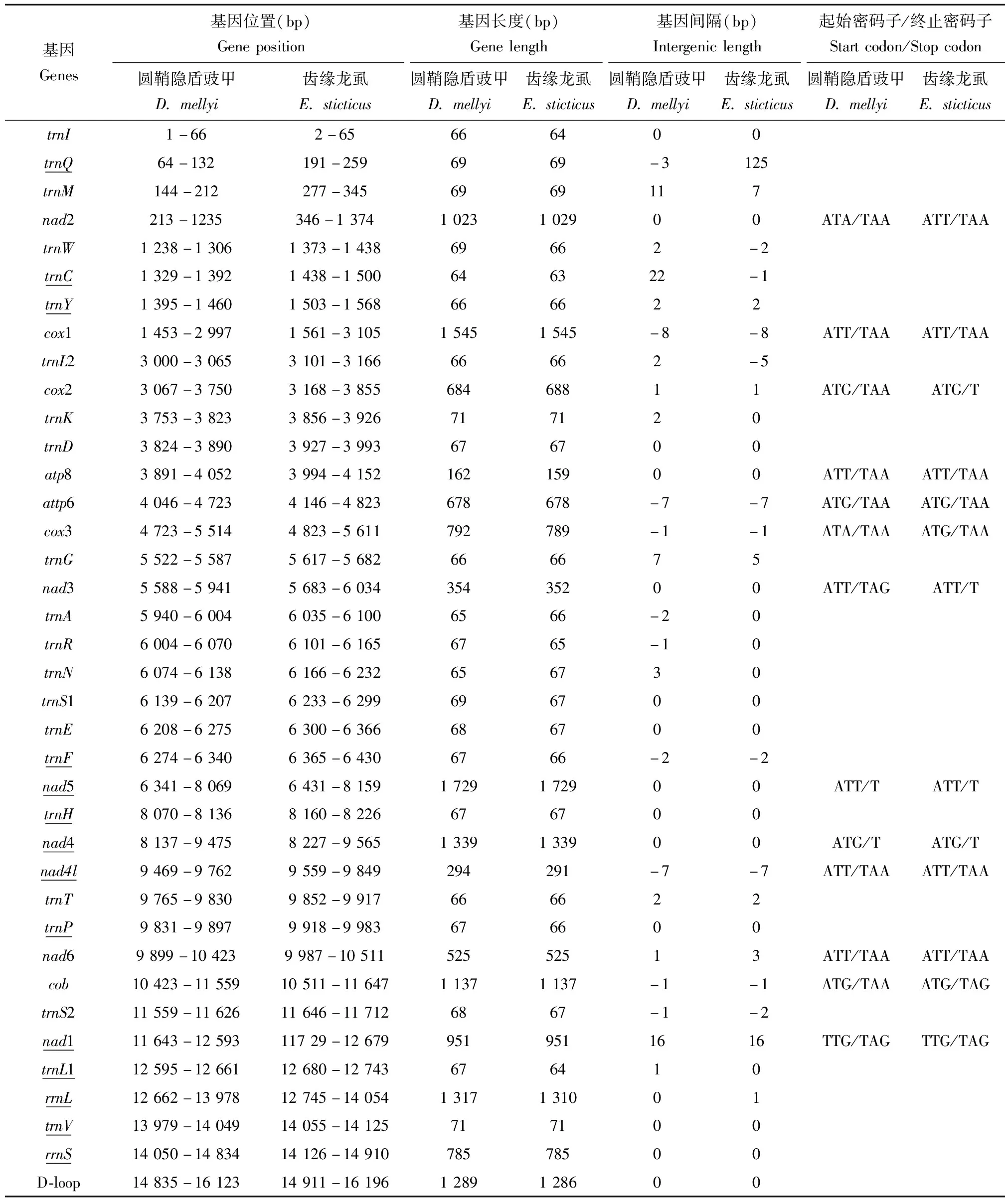
表2 圆鞘隐盾豉甲和齿缘龙虱的线粒体基因组注释
2.2 肉食亚目水生类群线粒体基因组PCGs的碱基偏好性和密码子使用
与其他已测序鞘翅目昆虫相似,肉食亚目水生类群线粒体基因组的PCGs在碱基使用上具有明显的A+T偏向性。沼梭甲科3个物种的AT含量相对较高分别为78.81%, 79.03%和79.13%。龙虱科Limbodessus属和Paroster属4个物种的AT含量相对较低,分别为73.84%, 72.23%, 74.25%和74.73%;而Hydroporus属和Eretes属3个物种的AT含量则相对较高,分别为78.51%, 77.44%和77.85%。线粒体蛋白质编码基因不同编码位点的AT含量存在较大差异,所有物种第3位点的AT含量均明显高于第1位点和第2位点。肉食亚目水生类群线粒体基因组的高AT含量,同样在蛋白质编码基因的密码子使用上也有所体现。如图2所示,从全部肉食亚目水生类群来看,62种密码子在其线粒体基因组中都有被使用;但是其中富含GC的12种密码子(ACG, AGG, AGC, CGC, CGG, CTG, GCG, GGC, GTC, GTG, TCG和TGC)在少数的不同物种中并没有被使用。此外,以G/C结尾的31种密码子在19个肉食亚目水生类群中的平均使用次数全部低于40(1~37.36),并且这些密码子的RSCU值都明显小于1(最高0.74);而以A/T结尾的31种密码子的平均使用次数几乎都高于40,并且RSCU值也几乎都大于1。尤其是完全由A和T组成的6种密码子(TAT, AAT, ATA, TTT, ATT和TTA)的使用次数明显偏高,在肉食亚目水生类群线粒体基因组中的平均使用次数高达144.42~412.21。
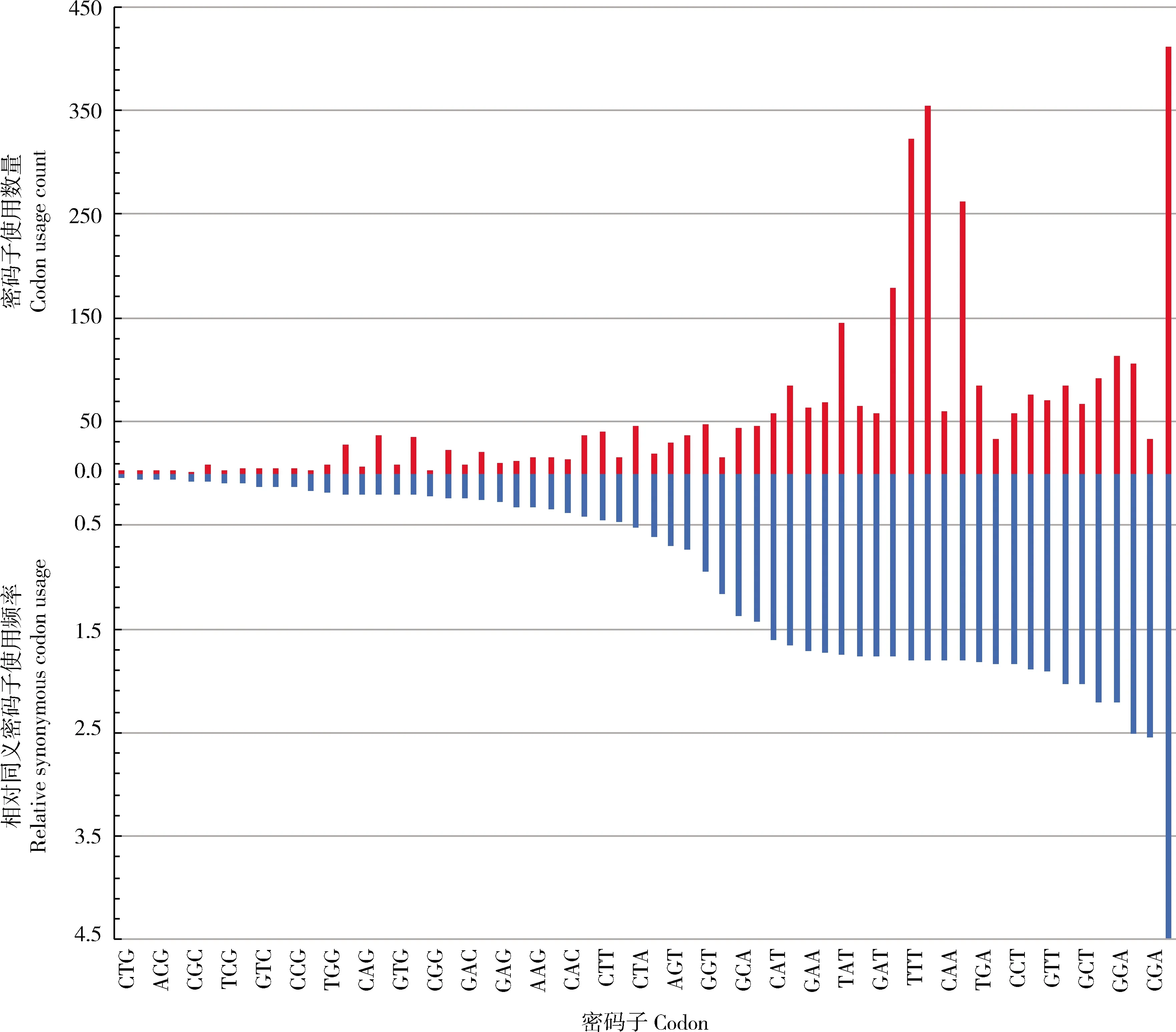
图2 肉食亚目水生类群线粒体基因组PCGs的密码子使用数量和相对同义密码子使用频率
2.3 PCGs在肉食亚目水生类群中的进化模式
通过比较分析13个PCGs的Ka, Ks以及Ka/Ks值的大小,我们发现线粒体基因组的13个PCGs在肉食亚目水生类群的进化过程中都受到了纯化选择。如图3所示,在肉食亚目水生类群线粒体基因组的13个PCGs中:cox1基因的Ka值最低(0.064),atp8基因的Ka值最高(0.284);nad4l基因的Ks值最低(0.458),cox3基因的Ks值最高(1.068)。虽然13个PCGs的Ka值和Ks值存在一些差异,但是所有13个PCGs的Ka/Ks值都显著小于1,atp8基因的Ka/Ks值最高为0.567,cox1基因的Ka/Ks值最低为0.075。
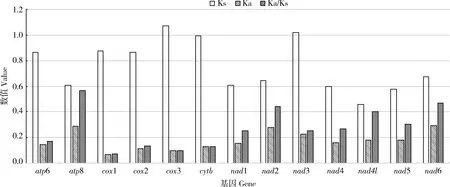
图3 13个蛋白质编码基因(PCGs)在肉食亚目水生类群中的进化速率
2.4 肉食亚目水生类群的系统发育关系
通过将线粒体基因组13个PCGs的多重序列比对结果进行串联,得到氨基酸序列数据集(3 693 bp)和核苷酸序列数据集(11 079 bp)。基于这两个数据集构建19个肉食亚目水生类群物种的最大似然树和贝叶斯树,总共得到了4个系统发育树。如图4和5所示,尽管这4个系统发育树的拓扑结构有所差异,但是它们都共同支持:瀑甲科与伪龙虱科互为姐妹群,两栖甲科与龙虱科构成姐妹群。
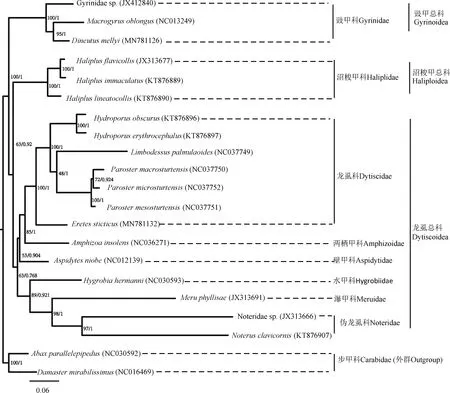
图4 基于线粒体蛋白质编码基因(PCGs)的氨基酸序列推断的肉食亚目水生类群的系统发育关系
如图4所示,基于氨基酸数据集得到的最大似然树和贝叶斯树的拓扑结构完全一致,仅在分支的支持率上存在轻微差异。基于氨基酸序列的建树结果将龙虱总科强烈恢复为一个单系类群,支持豉甲总科位于系统发育树的基部,沼梭甲总科与龙虱总科构成姐妹群。将龙虱总科分为2个分支:两栖甲科与龙虱科构成姐妹群,并一起与壁甲科构成一个分支;瀑甲科与伪龙虱科构成姐妹群,并一起与水甲科构成一个分支。如图5所示,基于核苷酸序列得到的最大似然树和贝叶斯树则不完全一致,在龙虱科内不同属之间的亲缘关系存在轻微差异:最大似然树支持Hydroporus属和Paroster属的亲缘关系更接近;贝叶斯树则与基于氨基酸序列的系统发育结果一致,支持Limbodessus属和Paroster属的亲缘关系更接近。基于核苷酸序列的系统发育结果将龙虱总科作为一个并系类群:支持(伪龙虱科+瀑甲科)与豉甲科构成姐妹群,并与沼梭甲科一起构成一个分支;将龙虱总科剩余类群单独构成一个分支:龙虱科与两栖甲科构成姐妹群,壁甲科与水甲科构成姐妹群。
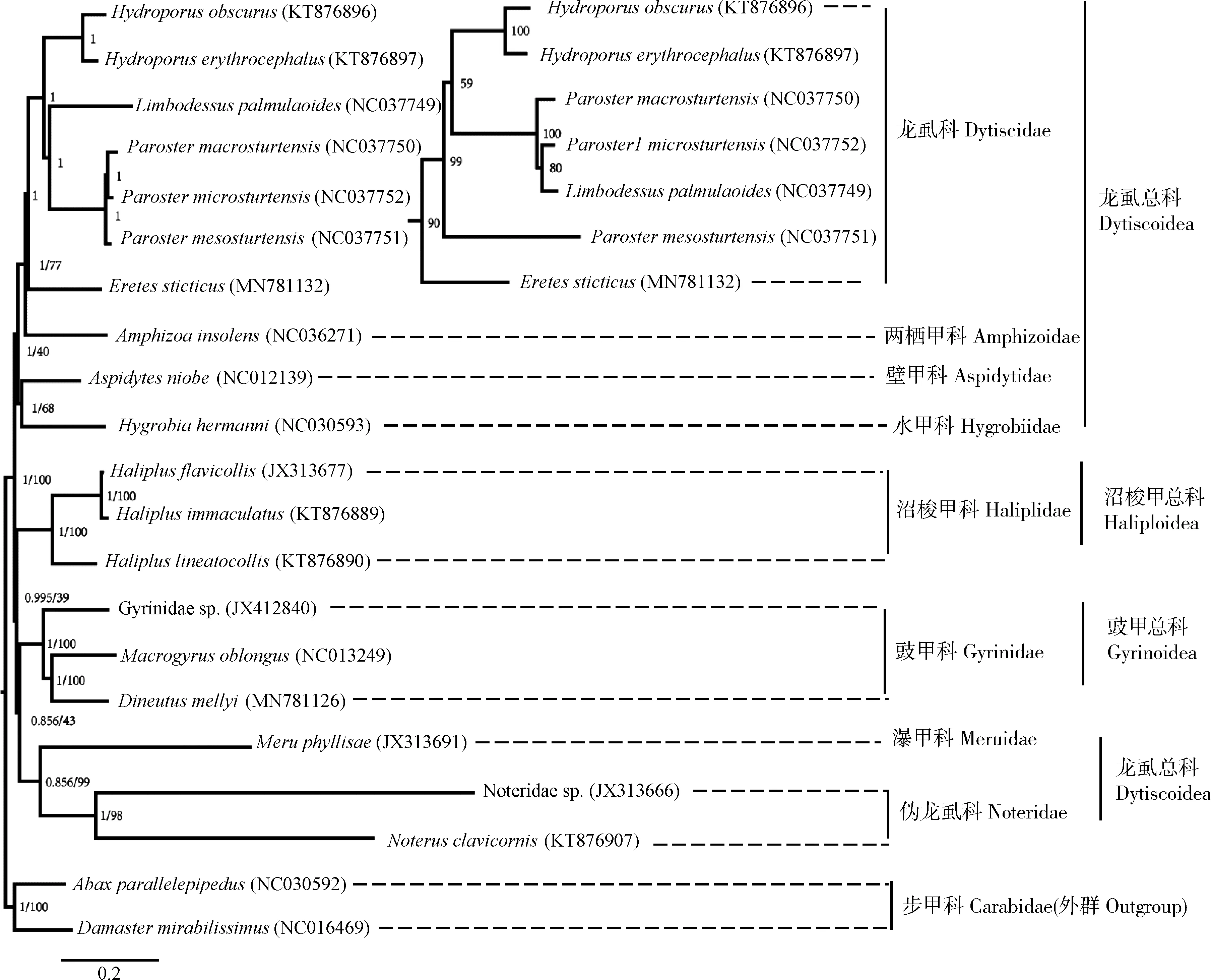
图5 基于线粒体蛋白质编码基因(PCGs)的核苷酸序列推断的肉食亚目水生类群的系统发育关系
2.5 系统发育树拓扑结构的FcLM分析
基于氨基酸序列和核苷酸序列的系统发育结果的差异主要表现在(伪龙虱科+瀑甲科)的系统发育位置发生了改变,这种变化也直接干扰了龙虱总科的单系性。利用FcLM分析可以对不同数据集支持不一致拓扑的问题进行解决,该方法需要指定4个单系类群,并对可能的四重奏拓扑结构(quartet topologies)的支持值进行估算(Zhangetal., 2019; Nieetal., 2020)。在本次FcLM分析中,我们将肉食亚目水生类群分为4个分支:(伪龙虱科+瀑甲科)单独作为一个分支;龙虱总科剩余类群作为一个单独的分支;豉甲科作为一个分支;沼梭甲科作为一个分支。我们基于氨基酸序列和核苷酸序列分别进行了FcLM分析,来进一步评估(伪龙虱科+瀑甲科)的系统发育位置。如图6所示,基于FcLM分析的结果都强烈支持(伪龙虱科+瀑甲科)与龙虱总科剩余类群构成姐妹群,氨基酸数据集和核苷酸数据集对于这种拓扑结构的支持值分别为66%和54%;而对于(伪龙虱科+瀑甲科)与豉甲科构成姐妹群的支持度则相对较低,氨基酸数据集和核苷酸数据集的支持值分别仅有25%和28%。所以,FcLM分析的结果与基于氨基酸序列的建树结果一致,这也进一步证实了龙虱总科的单系性。

图6 基于线粒体PCGs氨基酸序列和核苷酸序列的肉食亚目水生类群的FcLM分析
3 讨论
3.1 线粒体基因组特征
圆鞘隐盾豉甲和齿缘龙虱的线粒体基因组全长分别为16 123 bp和16 196 bp,在基因排序、碱基组成、密码子使用及tRNA基因二级结构等特征上与已测的肉食亚目水生类群线粒体基因组高度相似。本研究利用19个肉食亚目水生类群线粒体基因组的PCGs序列进行碱基偏好性分析,AT含量和62种密码子使用的统计结果都表明线粒体基因组PCGs偏向于使用碱基A+T。其碱基使用情况与大部分鞘翅目昆虫的研究结果一致,PCGs的3个编码位点都偏向于更多地使用碱基A+T;并且第3位点的AT含量明显高于第1和2位点(Yuanetal., 2016)。虽然蛋白质编码基因不同编码位点在碱基使用上存在明显差异,但是肉食亚目水生类群8个科的物种在碱基使用上并没有显著差异。
与其他昆虫中的研究结果一致,肉食亚目水生类群线粒体基因组PCGs的进化模式分析结果表明13个PCGs在进化过程中都受到纯化选择。在线粒体基因组的进化中纯化选择是最主要的力量,纯化选择有利于去除有害突变,即对氨基酸影响较小(Tomasco and Lessa, 2011)。为了维持功能需求,cox1基因的氨基酸序列经历了强大的功能约束和强烈的进化压力。尽管atp8基因经历了弱的进化压力和功能约束,但代谢约束的这种放松可能使线粒体基因组中积累更多的突变(Zsurkaetal., 2010; Lietal., 2012; Yangetal., 2018)。这些结构上和进化上相对保守的特点表明其可以作为研究肉食亚目水生类群进化关系的重要分子标记。
3.2 肉食亚目水生类群系统发育
基于氨基酸序列和核苷酸序列的建树结果不完全一致,但是通过FcLM分析发现基于氨基酸数据集的建树结果更加可靠。所以,基于线粒体基因组PCGs的氨基酸序列的肉食亚目水生类群系统发育重建结果为:(豉甲科Gyrinidae+(沼梭甲科Haliplidae+((壁甲科Aspidytidae+(两栖甲科Amphizoidae+龙虱科Dytiscidae))+(水甲科Hygrobiidae+(瀑甲科Meruidae+伪龙虱科Noteridae)))));豉甲科位于肉食亚目水生类群的基部,沼梭甲科与龙虱总科构成姐妹群。这些结果既支持了Bell(1966)最早提出的龙虱总科所有类群共同完成一次独立水生习性入侵的理论,也进一步支持了Arndt和Beutel(1995)早期提出的“豉甲科、沼梭甲科、龙虱总科3个类群独立水生习性入侵过程”的假说。伪龙虱科与瀑甲科构成姐妹群,这与Beutel等(2006)基于形态学以及Balke等(2008)基于分子数据的研究结果一致。此外,我们的结果将龙虱总科分在2支之内:两栖甲科与龙虱科构成姐妹群,并一起与壁甲科构成一个分支;瀑甲科与伪龙虱科构成姐妹群,并一起与水甲科构成一个分支。这与习欠云等基于cox1序列的研究结果(Xietal., 2008)基本一致。
蛋白质编码基因的进化模式分析表明氨基酸序列相对保守;基于氨基酸序列的建树结果也十分稳定;FcLM分析结果也表明使用氨基酸数据集的建树结果更加可靠。这些都表明利用线粒体基因组对肉食亚目水生类群进行系统发育重建时,氨基酸序列是更好的选择。另外,本研究中使用数据有限,部分科只包含了1个代表种,龙虱总科内部各科之间的亲缘关系还需更广泛的物种取样来验证。
