基于生物信息学分析丙肝相关肾病潜在生物标志物①
2020-09-29陈志敏林丹华莆田市莆田学院附属医院肾内科莆田351100
陈志敏 林丹华 (莆田市莆田学院附属医院肾内科,莆田 351100)
慢性丙型肝炎病毒(hepatitis C virus,HCV)感染是导致慢性肝病的主要原因之一,HCV全球患病率约为1%~2%,HCV活动后若未及时治疗,常进展为慢性肝炎、肝硬化甚至肝细胞癌,并可能造成相应的肝外并发症[1]。其中,肾脏受累最为常见,可表现为蛋白尿、肾小球肾炎、冷球蛋白血症(mixed cryoglobulinemia,MC)和慢性肾脏病(chronic kidney disease,CKD)等。目前认为HCV感染能够增加非透析CKD患者终末期肾病(end stage renal disease,ESRD)风险和病死率及已经进入血液透析患者病死率,并可导致肾移植受者的移植肾损伤,进而导致肾病患者的预后进一步恶化,这些病变的发病机制可能与肾小球中免疫复合物的沉积有关,然而现有研究仍未明确HCV相关性肾病的发病机制[2,3]。本研究利用生物信息学工具对GEO(gene expression omnibus)公共数据库中的HCV基因表达谱芯片和CKD基因表达谱芯片进行数据分析,筛选出各自的差异表达基因(differentially expressed genes,DEGs),并与人类基因综合数据库GeneCards中HCV和CKD疾病相关基因取交集,获得共表达差异基因(co-expressed DEGs,co-DEGs)。随后使用STRING数据库、CTD数据库及AMIGO数据库进行相关的生物信息学分析及注释。最后通过mirDIP数据库、miRDB数据库及TargetScan数据库预测co-DEGs的靶标miRNA。为进一步深入研究HCV和CKD相关基因的生物学功能及可能参与HCV相关肾病发生发展的分子机制奠定基础。
1 资料与方法
1.1资料
1.1.1Affymetrix芯片数据 NCBI-GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)[4]下载2个芯片数据集GSE20948和GSE15072。GSE20948采用的芯片平台是 GPL570 (Affymetrix Human Genome U133 Plus 2.0 Array),包括 14 例HCV感染样本和 14 例对照样本。GSE15072采用的芯片平台是GPL570和GPL96(Affymetrix Human Genome U133A Array),共包括8名健康对照者的血液样本和26名CKD患者的血液样本。选取GPL96平台上的8名健康对照者的血液样本为对照组,9例CKDⅡ~Ⅲ期患者的血液样本和12例进入血液透析的尿毒症患者血液样本为试验组,分析差异基因分析。
1.1.2GeneCards数据库获取CKD与HCV相关基因 在GeneCards数据库(https://www.genecards.org/)中以“chronic kidney disease”、“chronic hepatitis c”为关键词检索,获得CKD、HCV疾病相关基因。
1.2方法
1.2.1数据预处理及差异基因分析 利用R语言limma包对原始芯片数据进行归一化处理并筛选DEGs,应用贝叶斯检验方法进行多重检验校正,筛选符合条件的DEGs[5]。GSE20948数据集设置筛选条件为|log2 FC|>1,校正后P<0.05 的基因则为DEGs。GSE15072数据集设置筛选条件为|log2 FC|>1.5,校正后P<0.05,确定DEGs。对2个数据集筛选得到的DEGs与GeneCards数据库中CKD与HCV疾病相关基因通过韦恩图(Venn Diagram)取交集,获得co-DEGs。
1.2.2差异表达基因的GO和KEGG富集分析 利用R语言clusterProfiler包对筛选得到的两组DEGs行GO(gene notology)富集分析和KEGG(Kyoto encyclopedia of genes and genomes)通路分析,以P<0.05为显著性基因富集临界值,对差异显著的基因进行功能注释,并分析两组差异基因参与的生物学过程及通路。随后单独对co-DEGs行KEGG通路分析,获得co-DEGs主要富集通路。
1.2.3差异表达基因蛋白互作网络构建及关键节点基因筛选 将筛选出的DEGs列表分别上传至蛋白质互作关系数据库(STRING)通过对基因编码蛋白之间相互作用的可能性打分,评价相互作用可能性的大小[6]。分别构建DEGs编码蛋白相互作用网络(protein-protein interaction,PPI),筛选条件为代表中度相互作用的可能性阈值(结合分值>0.4)。将STRING数据库构建出的PPI互作网络结果导入Cytoscape(3.2.1版本)软件[7]中进行可视化分析,运用Cytoscape中cytoHubba插件和Degree算法绘制由前30个基因组成的PPI图,并选取排名前5名的基因为关键节点基因(hub-gene)。
1.2.4Co-DEGs基因产物与疾病的联系 利用比较毒理基因组学数据库(the comparative toxicogenom-ics database,CTD,http://ctdbase.org/)分析co-DEGs的基因产物与消化系统和泌尿系统疾病之间的联系[8]。取消化系统疾病中推断分值(inference score)排名前3名的疾病和泌尿系统疾病中推断分值排名前4的疾病为与基因产物可能有关联的疾病。
1.2.5Co-DEGs在AmiGO中的功能注释 在AmiGO(http://amigo.geneontology.org/amigo/)[9]数据库中检索co-DEGs,获得4个DEGs各自排名前5位的功能注释条目。
1.2.6Co-DEGs的靶标miRNA预测及相关通路分析 在mirDIP数据库(http://ophid.utoro nto.ca/mirDIP)、miRDB数据库 (http://mirdb.org/)及TargetScan数据库 (v7.1;http://www.targe tscan.org/vert_71/)中检索GLRX、NFIL3、PFKFB3和KLF10,对获得的miRNA取交集,得到在3个数据库都预测到的miRNA。随后将交叉miRNA输入miEAA数据库(https://ccb-compute2.cs.uni-saarland.de/mieaa_tool/),选用ORA(over-representation analysis)法,设定阈值为2、P<0.05,获得miRNA的主要富集通路。
2 结果
2.1DEGs的筛选 HCV数据集GSE20948共筛选出122个DEGs,其中包括77个下调基因和45个上调基因。CKD数据集GSE15072共筛选出235个差异基因,其中包括45个下调基因和190个上调基因。GeneCards数据库中以“chronic kidney disease”为关键词检索,共得到11 797个CKD相关基因,以“chronic hepatitis c”为关键词检索,共得到6 597个HCV相关基因,用韦恩图对四组基因取交集后,得到4个co-DEGs:GLRX、NFIL3、PFKFB3、KLF10(图1)。
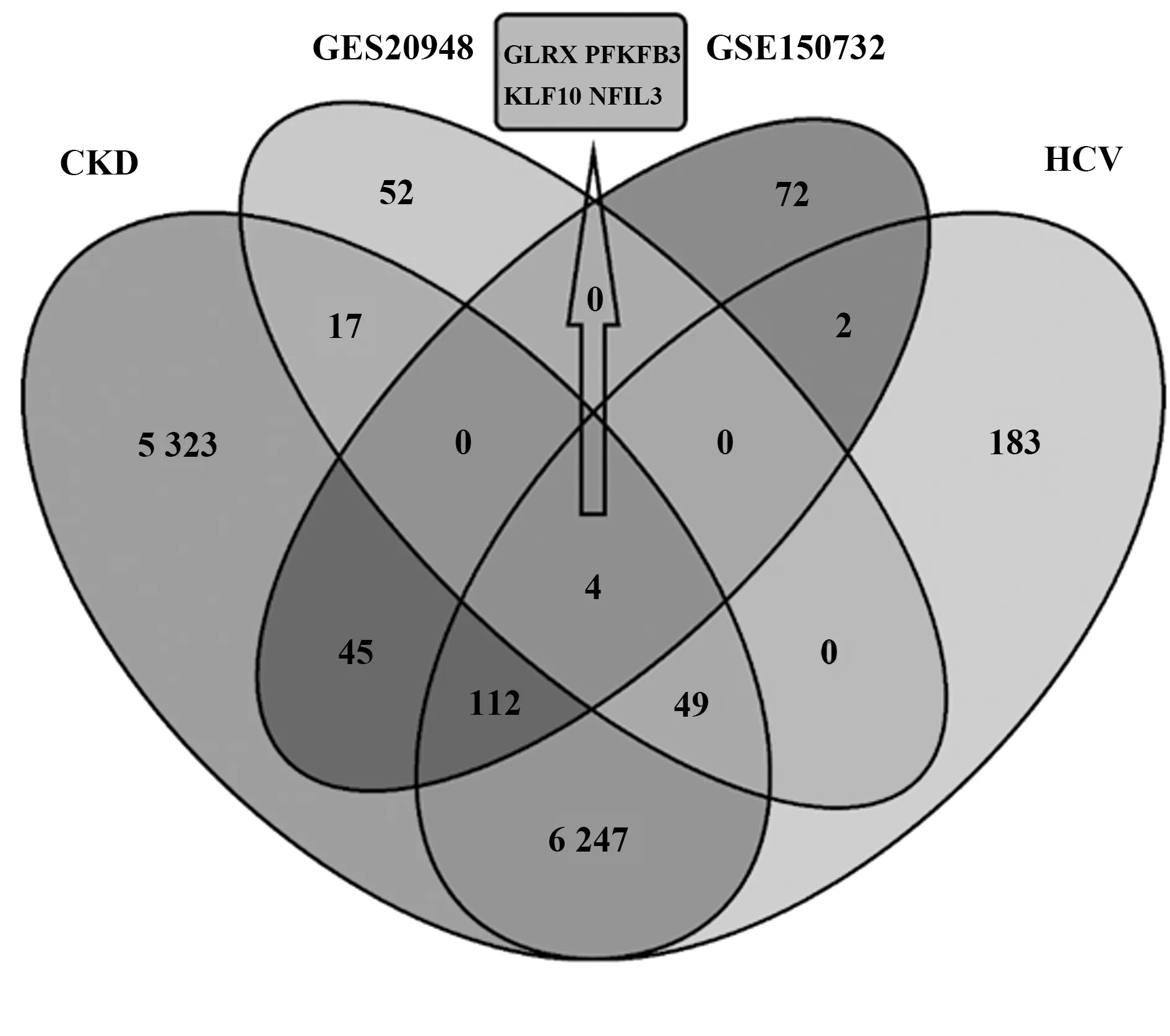
图1 Genecards数据库中CKD、HCV相关基因与HCV数据集(GSE20948)、CKD数据集(GSE15072)DEGs交集
2.2DEGs的GO富集分析和 KEGG 信号通路分析 GO富集分析显示HCV相关DEGs主要参与的分子生物学过程包括胺代谢过程、内源性细胞凋亡信号通路、氨基酸运输、氧化还原过程等;CKD相关DEGs主要参与的分子生物学过程包括核转录mRNA分解代谢过程、病毒转录、氢离子跨膜转运等。KEGG 通路分析显示HCV的DEGs主要参与抗生素的生物合成通路、甘氨酸,丝氨酸和苏氨酸代谢、氨基酸的生物合成及代谢途径等;CKD的DEGs主要参与核糖体、氧化磷酸化、帕金森病、非酒精性脂肪性肝病(NAFLD)通路等(图2)。Co-DEGs的KEGG通路分析显示,共表达基因主要富集于果糖和甘露糖代谢通路、HIF-1信号通路、AMPK信号通路,本研究选取HIF-1信号通路进行展示说明(图3)。
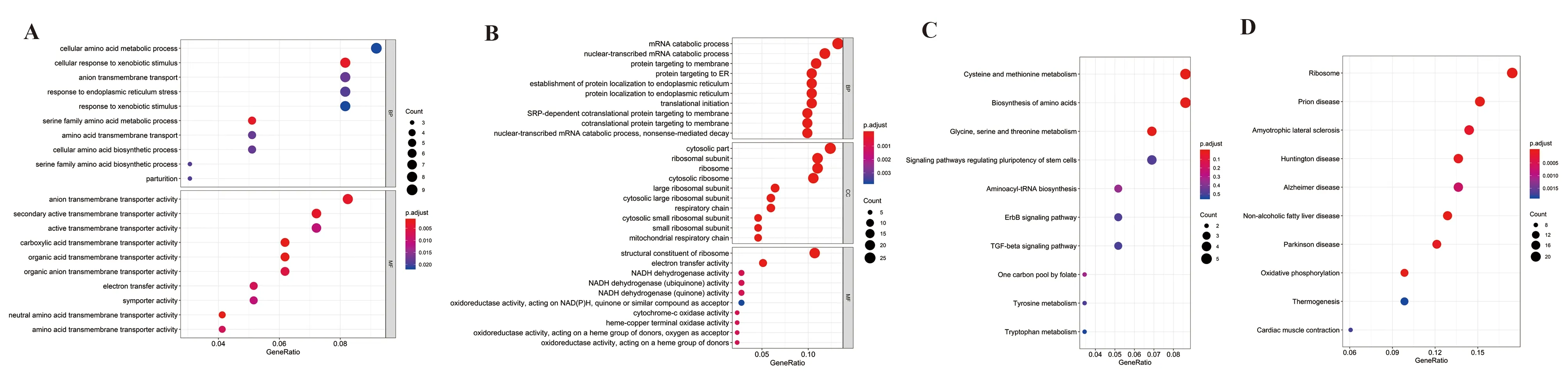
图2 DEGs的GO富集分析和KEGG-pathway富集分析

图3 Co-DEGs富集的信号通路之HIF-1信号通路
2.3差异基因PPI网络分析 通过STRING在线工具和Cytoscape软件对HCV的112个DEGs和CKD的235个DEGs进行PPI分析。在Cytoscape软件中绘制cytoHubba插件中degree排名前30的基因的编码蛋白互作网络图(图4)。HCV数据集的关键节点基因有ASNS(asparagine synthetase,degree=16)、MYC(MYC proto-oncogene protein,degree=12)、PSAT1(phosphoserine aminotransferase 1,degree=11)、ATF3(activating transcription factor 3,degree=10)和PHGDH(phosphoglycerate dehydrogenase,degree=8)。CKD数据集的关键节点基因有SNRPG(small nuclear ribonucleoprotein polypeptide G,degree=49)、COX7C(cytochrome C oxidase subunit 7C,degree=45)、RPS15A(ribosomal protein S15a,degree=43)、RPS17(ribosomal protein S17,degree=42)、RPS24(ribosomal protein S24,degree=42),这些关键节点基因在各自的PPI互作网络图中处于关键位置,与多个基因互相联系。

图4 DEGs的蛋白互作网络
2.4Co-DEGs基因产物与消化和泌尿系统疾病的联系 CTD数据库分析显示co-DEGs的基因产物在消化系统中与化学和药物诱导的肝损害疾病、肝肿大、脂肪肝、肝肿瘤等疾病有关。在泌尿系统中与急性肾损伤、蛋白尿、少精症等疾病有关(图5)。

图5 CTD数据库中共表达基因与消化和泌尿系统疾病的联系
2.5Co-DEGs的AmiGO功能注释 AmiGO数据库对co-DEGs的功能注释提示co-DEGs的主要功能与RNA聚合酶Ⅱ对转录的调控及糖代谢途径有关(表1)。

表1 AmiGO数据库中co-DEGs的功能注释
2.6Co-DEGs的靶标miRNA预测及相关通路分析 mirDIP数据库共得到153个潜在miRNA靶标,miRDB数据库共得到118个潜在miRNA靶标,TargetScan数据库共得到15个潜在miRNA靶标,对3者取交集后共得到10个交叉miRNA:hsa-miR-489-3p、hsa-let-7f-5p、hsa-let-7c-5p、hsa-let-7g-5p、hsa-let-7b-5p、hsa-miR-98-5p、hsa-let-7i-5p、hsa-let-7a-5p、hsa-let-7d-5p、hsa-let-7e-5p。将10个miRNA输入miEAA数据库后将结果按P值排序,结果显示其排名前10的主要富集的通路功能为细胞因子与炎症反应、哮喘、IL-5信号传导途径、IL-6信号传导途径、Jak STAT信号通路、催乳素信号通路、IL-2信号传导途径、嘧啶代谢、基础转录因子和炎症反应途径(表2)。
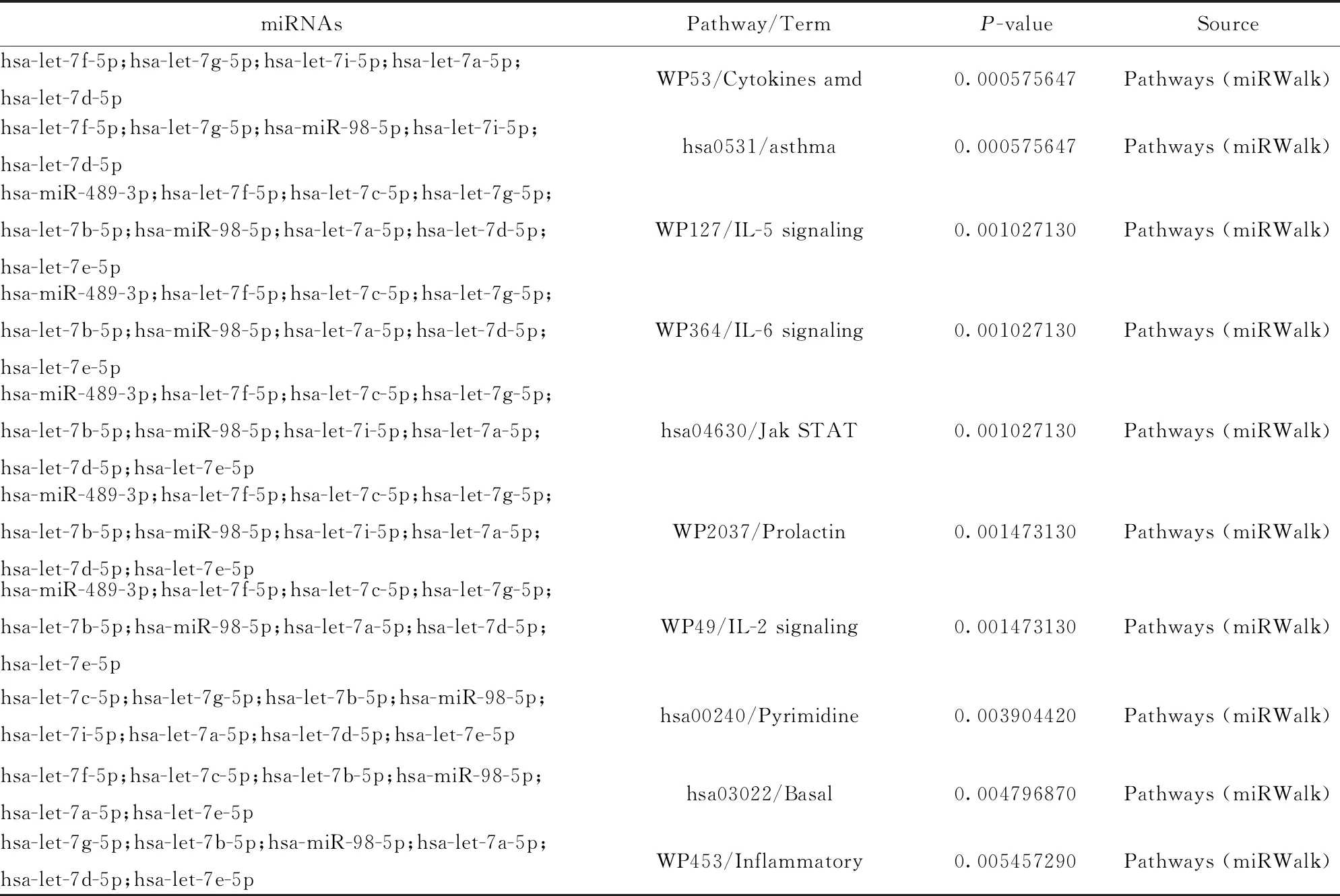
表2 miEAA数据库中靶向miRNA通路功能
3 讨论
在HCV感染的自然病程中,40%~74%的患者会至少出现1种肝外表现,常表现为混合型冷球蛋白血症,累及肾脏,多表现出膜增生性肾小球肾炎(MPGN),其临床表现为无症状性血尿、蛋白尿,约20%患者表现为肾病综合征;20%~30%的患者表现为肾炎综合征;约15%可逐渐进展为终末期慢性肾衰竭,需要透析治疗,其发病机制尚不完全明确[10-12]。有研究认为Ⅱ型及Ⅲ型冷球蛋白血症通过形成IgM-IgG 免疫复合物与补体C1q结合,C1q与内皮细胞受体结合,可激活补体系统进而导致血管炎,但具体机制及相关编码基因尚未可知[13]。
在co-DEGs中GLRX基因编码的蛋白质是谷氧还蛋白家族成员,其对抗氧化防御系统有重要作用。研究表明,由GLRX控制的GSH加合物参与细胞生长、细胞凋亡、血管生成和炎症等多种生物过程,GLRX缺乏会加速高脂肪诱导的非酒精性脂肪肝并进展为脂肪性肝炎,表现为肝损伤和炎症[14-16]。此外,Yang等[17]研究表明,GSH可为肾小管上皮细胞提供抗尿酸肾病的抗氧化保护。KLF10基因编码的蛋白质是转录抑制因子,大部分作用和基因靶向是通过TGF-β信号介导的,该蛋白在各种癌细胞(包括胰腺癌,白血病和骨质疏松症)中表现出抗增殖作用并诱导细胞凋亡[18]。肝脏具有显著的再生能力,TGF-β/Smad通路也会参与肝脏再生[19]。特异性靶向髓样TGF-β1可促进急性肾损伤后肾纤维化进展,是肾纤维化的主要驱动因素。但也有研究认为,不加选择地完全阻断TGF-β功能不足以减少纤维化,并可能在某些病理环境中加重疾病[20]。 Kowalewska等[21]研究表明,HCV继发冷球蛋白血症后引起炎症反应,可分泌血小板源生长因子PDGF及生长因子TGF-β等细胞因子以促肾小球固有细胞增殖及细胞外基质分泌,此外,还可分泌趋化因子、趋化单核-巨噬细胞等炎症细胞聚集于肾小球加重损害。基于KLF10基因的生物学功能,提示该基因可能涉及HCV相关肾病的发病机制。
NFIL3基因编码的蛋白质是转录调节因子,其参与的关键生物途径中NF-κB通路是先天性和适应性免疫的关键调节通路,目前研究表明,NF-κB 通路与AKI的发病密切相关。肾小管上皮细胞缺血后,NF-κB通路的活化会加剧肾小管损伤并加剧适应不良状态的炎症反应[22]。另外,单克隆IgMκ可与肾小球系膜中的纤连蛋白结合,形成更易于沉积在肾小球中的免疫复合物,这可能是HCV继发的Ⅱ型冷球蛋白血症导致肾炎的关键机制之一[14]。PFKFB3基因编码的蛋白质属于双功能蛋白质家族,其参与果糖-2,6-二磷酸的合成和降解,是控制真核生物糖酵解的调节分子。该蛋白也是细胞周期进展和预防细胞凋亡中的必需蛋白。Ding等[23]研究发现,糖酵解抑制可减少TGF-β1诱导的肌成纤维细胞活化并延缓肾纤维化进展。此外,本究显示co-DEGs主要富集的通路中最显著的是果糖和甘露糖代谢通路,提示HCV相关肾病可能与糖代谢有关,PFKFB3可能是其发生发展的关键基因。
在对co-DEGs预测的靶标miRNA富集的通路分析中发现,其靶向的miRNA主要涉及的信号通路多与炎症反应有关,说明co-DEGs介导的各种反应最后引起导致了生物体慢性炎症的发生从而影响多生理系统。
目前对于HCV相关肾病的研究尚有欠缺,本文通过筛选GEO数据库中两组不同疾病的芯片表达谱获得各自的DEGs,并与大型基因数据库中CKD和HCV疾病相关基因进行交叉验证,并进一步分析二者的共表达基因,找到了可能与HCV相关肾病致病相关的关键基因及重要通路,关键基因或可成为诊断该病的潜在生物标志。但受条件所限,未能获得相关样本行进一步实验验证。本研究从另一条思路探讨在HCV相关肾炎发生过程中可能的作用机制,为临床上继发性疾病机制研究提供新思路,也为未来进一步深入研究HCV相关肾病的发病机制提供了理论基础。