昆明犬PPARGC1A基因编码区克隆及生物信息学分析
2020-08-18万九生邓卫东黎立光
李 静,万九生,邓卫东,陈 超*,岳 锐,黎立光
(1.公安部昆明警犬基地,昆明 650201;2.公安部警犬技术重点实验室,昆明 650201;3.云南农业大学动物科技学院,昆明 650201)
过氧化物酶体增殖激活受体共激活剂也称为PGC1A(PPARGC1A基因),是由Puigserver 等作为新的转录共激活剂核受体的PPARG(过氧化物酶体增殖的激活受体),由脂肪细胞的主要调节器分化而被检测到[1]。PPARGC1A做为核受体在转录适应性产热链接中起着关键作用。适应性产热作用在体重调节和预防肥胖中起着重要的作用[2-3]。自适应产热至少包括三个可分离的过程:特异性解偶联蛋白(UCP)的表达、呼吸系统线粒体酶的表达链和线粒体的生物发生。PPARGC1A已经被证明可以诱导转录UCP1、UCP2和UCP的mRNA,其启动子解偶联蛋白中含有一个识别位点PPARG 以及THRB[4]。这两种受体都被激活与PPARGC1A相互作用[5]。过氧化物酶体增殖激活受体α(PPARA)是编码线粒体脂肪酸β-氧化酶的基因转录控制中的关键蛋白,PPARA的调节途径也与肥胖和糖尿病有关[6]。在肌肉细胞中,PPARGC1A诱导SLC2A4的表达(溶质载体家族2 成员4)[7],小鼠和人PPARGC1A的表达是通过β-肾上腺素受体途径的冷暴露诱导的[8-9]。人PPARGC1A活性的调节是通过与阻遏物的MAPK(促分裂原活化蛋白激酶)敏感相互作用来控制的[10]。PPARGC1AmRNA已在高能量需求且富含脂肪的组织中发现,如心脏、骨骼肌、褐色脂肪、肾脏、肝脏和大脑[11-13]。考虑到PPARGC1A蛋白的多功能该基因可视为肉品质性状的功能候选基因[3]。
目前国内外主要PPARGC1A基因针对的研究多集中在人、猪、牛上,犬上研究的较少。鉴于此,本研究以昆明犬为研究对象,采用RT-PCR技术通过对昆明犬PPARGC1A基因克隆的分析,确定PPARGC1A的序列,进行生物信息学分析,为警犬的肌肉形成机理和产能等影响提供理论依据。
1 材料与方法
1.1 样品采集
以公安部昆明警犬基地昆明犬为研究对象。试验采集于。犬只麻醉后,选择1岁昆明犬臀大肌取一小块组织块于1.5 mL 冻存管中,立即置于液氮中,随后于-80 ℃冰箱保存备用,用于RNA 的提取。
1.2 主要试剂和仪器
1.2.1 试验器材
Eppendorf 各量程移液器、湘仪L550 低速大型离心机、湘仪H1650-W 小型高速离心机、君意JY04S-3C 凝胶成像仪、君意JY02 紫外透视仪、君意JY300C 电泳仪、ABI 2720 PCR 仪、ABI 3730xl遗传分析仪、博日HB-T2-D 金属浴加热仪,天平、漩涡震荡仪、水平震荡仪,枪头,96孔反应板等。
1.2.2 试验试剂
I- 5 2X High- Fidelity Master Mix(Tsingke;TP001-1)、Tsingke Agarose、DNA 凝胶回收试剂盒、DL2000 Marker、pClone007 Versatile Simple Vector Kit、琼脂糖、蛋白胨、酵母粉、TBE、EB、无水乙醇、BDT 原液、buffer(BDT 稀释用)、甜菜碱、灭菌高纯水、样品、稀释过的引物、无菌高纯水、Magical Buffer、Ferrite Bead。
1.3 引物设计与合成
参 考GenBank(https://www.ncbi.nlm.nih.gov/genbank/)中PPARGC1A基因序列(登录号:XM_014112658.2)cDNA 引物,用Premier 5 设计出扩增引物1对。由擎科生物合成,引物序列见表1。
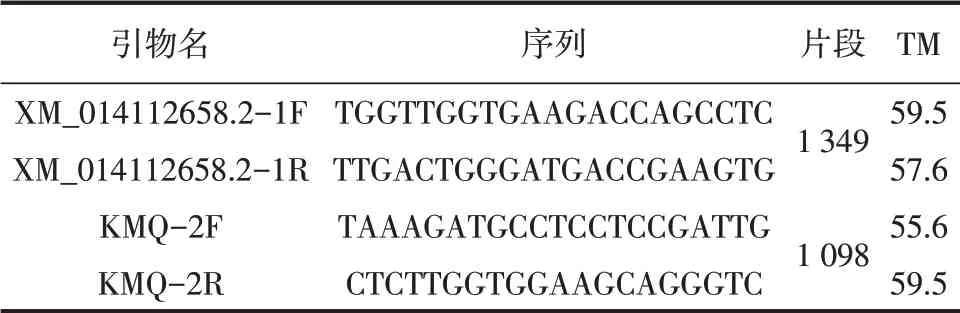
表1 引物序列
1.4 总RNA的提取
取适量液氮研磨成粉的样品到1.5 mL eppen⁃dorf管,加入1 mL Trizol试剂,混匀;按TRIzol:氯仿5:1 的比例加入氯仿,剧烈震荡15 s,室温静置3 min;4 ℃,12 000 r·min-1,离心15 min;吸取上清至另1新的1.5 mL eppendorf管,加入等体积异丙醇,混匀后室温静置20 min;4 ℃,12 000 r·min-1离心10 min 后,去上清;加入至少1 mL 4 ℃预冷的75%乙醇,洗涤沉淀;4 ℃,10 000 r·min-1离心5 min,弃上清;4 ℃,10 000 r·min-1再次离心5 min,吸去残液,室温干燥(不需完全干燥);加入20 μL RNase-free水,至完全溶解,取1 μL进行电泳检测。
1.5 反转录获得cDNA产物
以擎科金牌逆转率试剂盒Goldenstar RT6 cD⁃NA Synthesis Kit反转录扩增,按表2组分进行加样。

表2 PCR反应体系
以上加样混匀后按以下程序进行反转录:
25 ℃,10 min
50 ℃,30 min
85 ℃,5 min
反转录产物置于冰上或冷藏备用。
1.6 PCR扩增
PCR 反应体系为25.0 μL,其中含DNA 模板1 μL、酶mix25 μL、正反向引物(10 μmol·L-1)各2 μL。PCR扩增条件:98 ℃预变性2 min,98 ℃变性10 s,退火10 s,72 ℃延伸10 s,30 个循环,72 ℃后延伸5 min。
1.7 PCR产物切胶纯化步骤
试验准备:核对切胶记录与样品板中是否相符、有无特殊注意事项;确保Buffer W1、Buffer W2中加入了无水乙醇。
操作步骤:补水加loading buffer:按照(样品:6×Loading buffer=5:1)加入10 μL 6×Loading buffer。
点纯化胶:将样品点入事先准备好的1%的纯化胶中。
电泳:电泳仪电压设定160 V接上接头正负极恒压电泳40~60 min,开始电泳后要观察纯化胶槽正负极有气泡冒出后,定时。
采集图像:将胶块放入凝胶成像仪器中采集图像。
切胶:在长波365 nm 紫外透射仪下,用手术刀切下目的条带,切取的胶块质量应<3 g,将其放入对应的板孔号中。
溶胶: 纯化板配平4 000 r·min-1离心1 min,加入500 μL Buffer GL,盖上封口膜,65 ℃水浴12~15 min。
加磁珠:检查每孔胶块是否完全溶解,若没有完全溶解再次65 ℃水浴3 min,揭开封口膜,用连续加液器每孔加入10 μL 混匀的磁珠,盖上硅胶垫,漩涡震荡30 s,转入水平震荡仪600~800 r·min-1震荡5 min。
纯化:
①将96 孔板卡入磁力架中,磁吸30 s,将磁力架和样品正反轻微颠倒3次,再次静置磁吸1 min;
②弃废液,吸水纸上轻磕,用50~1 200 μL 8道电动移液器向每孔移取500 μL Buffer W1,盖上硅胶垫漩涡震荡30 s,将96 孔板卡入磁力架中,磁吸30 s,将磁力架和样品正反轻微颠倒3次,再次静置磁吸1 min;
③弃废液,吸水纸上轻磕,用50~1 200 μL 8道电动移液器向每孔移取500 μL Buffer W2 盖上硅胶垫漩涡震荡30 s,将96 孔板卡入磁力架中,磁吸30 s,将磁力架和样品正反轻微颠倒3次,再次静置磁吸1 min;
④弃废液,吸水纸上轻磕,倒离心至600 r·min-1。
洗脱:取下磁力架,加入35 μL 的Eluent(已65 ℃水浴加热),盖上封口膜,放入水平震荡仪上600~800 r·min-1,震荡5 min,震荡完成后离心至1 000 r·min-1,将96孔板卡入磁力架中,磁吸1 min。
点鉴定胶:2 μL样品+6 μL 0.5×溴酚蓝混合后点入0.8%的鉴定胶中,300 V电泳12 min。
采集图像:将鉴定胶放入凝胶成像仪中采集图像。
鉴定:对照纯化前后胶图,根据PCR 定量标准在PCR 记录表上标注每孔模板浓度并稀释至指定浓度。
1.8 克隆及测序
连入载体:连接反应体系:其中含有5×pClone007 VS mix2 μL,PCR 产物8 μL,ddH2O 补至10 μL。
连接反应:室温(22~30 ℃)反应1~5 min。
转化:
①使用擎科TreliefTM5α Chemically Competent Cell进行快速转化。
②取100 μL 冰上融化的TreliefTM5α Chemical⁃ly Competent Cell 感 受 态 细 胞, 加 入10 μL pClone007 VS连接产物,轻轻吹打搅拌混匀后冰上静置5 min。
③42 ℃水浴热激45 s,迅速移至冰浴中,静置2 min。
④涂布平板:向离心管中加入500 μL 无抗性LB 培养液,无需复苏,根据实验要求直接吸取不同体积均匀涂布到含Amp 抗生素的LB 平板上,37 ℃培养箱倒置培养过夜。
⑤挑取单克隆:按正常处理平板的步骤挑取单克隆,摇菌提质粒。
扩增鉴定后挑选出含有正确目的基因的菌液,送往擎科生物公司进行测序。
1.9 生物信息学分析
用ContigExpress拼接测序结果。
用拼接好的结果在NCBI 里比对查找出Homo sapiens人、Mus musculus小家鼠、Mus caroli卡氏小鼠、Sus scrofa欧亚野猪、Bos mutus野牦牛、Bos taurus牛、Canis lupus familiaris家犬、vulpes赤狐PPARGC1A基因序列。
将查找到的序列用BioEdit 进行格式转换,并用clustalx对测序结果进行比对。
将比对结果用MEGA的Neighbor Joining模式构建系统发育树,并对树进行编辑优化。
将所有序列用MegAlign进行同源性比对。
多序列比对分析利用BioEdit 软件进行,开放阅读框分析通过在线分析工具ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)实现。
利用NCBI 服务器上的Conserved Domains 的CD-Search工具(https://www.ncbi.nlm.nih.gov/Struc⁃ture/cdd/ wrpsb.cgi)获取蛋白的保守结构域。蛋白的理化性质分析(相对分子质量、理论pI值、氨基酸组成及半衰期等)通过在线软件ExPASy-Prot⁃Param 工具(https://web.expasy.org/protparam/)预测。疏水作用是蛋白质折叠的主要驱动力,蛋白的疏水性分析通过ExPASy-ProtScale 工具(https://web.expasy.org/protscale/)进行。信号肽分析通过在线分析工具Signal P 4.0(http://www.cbs.dtu.dk/services/SignalP/)进行。亚细胞定位与蛋白质功能存在重要的联系,亚细胞定位分析通过在线分析工具PSORT II(https://psort.hgc.jp/form2.html)进行。使用TMHMM 软 件(http://www.cbs.dtu.dk/services/TMHMM-2.0/)对蛋白进行跨膜预测。利用SOPMA程序(https://npsa-prabi.ibcp.fr/)预测的IGF-1蛋白的二级结构;利用瑞士生物信息研究院(Swiss Institute of Bioinformatics,SIB)的专业蛋白分析系统(Expert Protein Analysis System,ExPASy)服务器(https://swissmodel.expasy.org/)预测蛋白质的3D结构。
2 结果与分析
2.1 昆明犬PPARGC1A基因的克隆
提取昆明犬肌肉组织总RNA,并反转构成cDNA,经过PCR 鉴定(1%琼脂糖电泳,150 V、100 mA 、45 min)电泳观察发现,片段大小约2 246 bp(见图1),与预期结果一致,说明引物特异性良好,可以进行下一步研究。
2.2 昆明犬PPARGC1A 基因的同源性比对及系统进化树构建
测序结果显示,昆明犬PPARGC1A基因cds 区全长2 246 bp,共编码个724 个氨基酸。运用MegAlign 对昆明犬和其他物种进行同源性比对,结果与家犬(登录号:XM_014112658.2)、赤狐(登录号:XM_025997319.1)、野牦牛(登录号:XM_005897079.2)、牛(登录号:NM_177945.3)、人(登录号:NM_013261.5)、家鼠(登录号:NM_008904.2)、 卡 氏 小 鼠( 登 录 号: XM_021162167.2)、 欧 亚 野 猪(登 录 号: NM_213963.2)、同源性分别为100%、99.6%、95%、95%、95.4%、91.7%、91.6%、95%(见图2)。
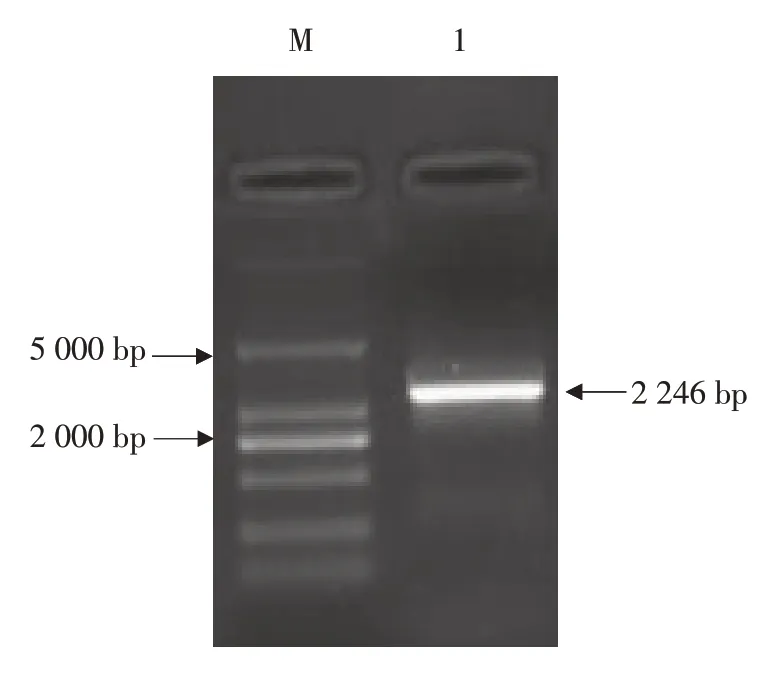
图1 昆明犬PPARGC1A基因PCR扩增产物电泳
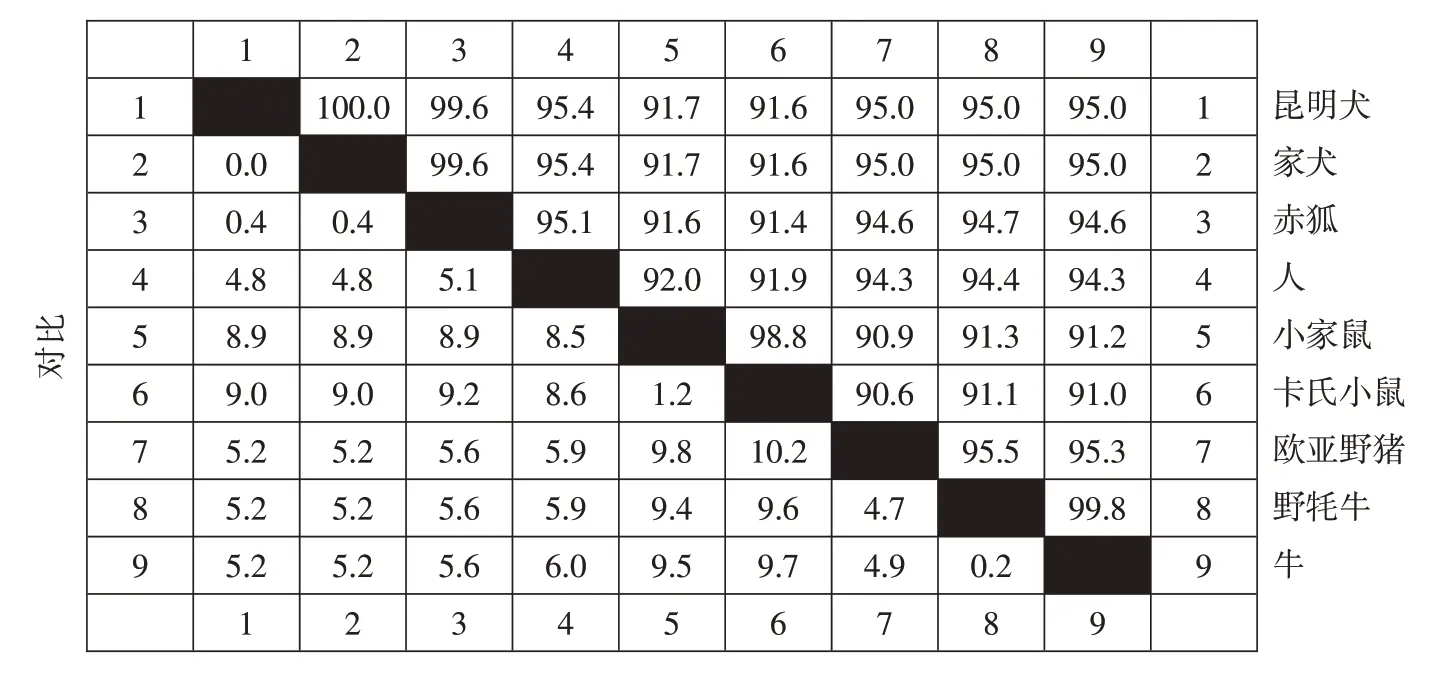
图2 昆明犬PPARGC1A基因与其他物种同源性比对
构建系统发生树结果,与家犬、赤狐聚为1支,与其他养脊椎动物相分离,表明其在进化上与犬类的亲缘关系较近。与猪、牛、人关系次之,与小鼠关系最远,该基因在哺乳动物中较为保守(见图3)。
2.3 PPARGC1A基因编码氨基酸理化特性的分析
根据在线软件ExPASy-ProtParam 预测结果显示,PPARGC1A基因编码蛋白的分子质量80 971.82 ku,理论等电点pI 为11.20。带负电荷的残基总数(Asp + Glu):30 个,带正电荷的残基总数(Arg +Lys):100 个,表示这个蛋白可能带正电荷。分子式为C3549H5835N1069O997S48,原子总数:11 498。序列的N 端是W(Trp),预计半衰期:2.8 h(哺乳动物网织红细胞,体外)>3 min(酵母,体内)>2 min(大肠杆菌,体内)。不稳定性指数(Ⅱ)为59.16,这表明PPARGC1A基因编码蛋白质分类为不稳定的。脂肪指数81.60,平均亲水系数为(GRAVY):-0.336,表明此蛋白亲水性差,脂溶性强。在组成昆明犬PPARGC1A基因编码20 种氨基酸中,亮氨酸Leu(L)占比最高为11.3%。
2.4 昆明犬PPARGC1A蛋白疏水性分析
PPARGC1A蛋白的疏水性分析结果见图4。
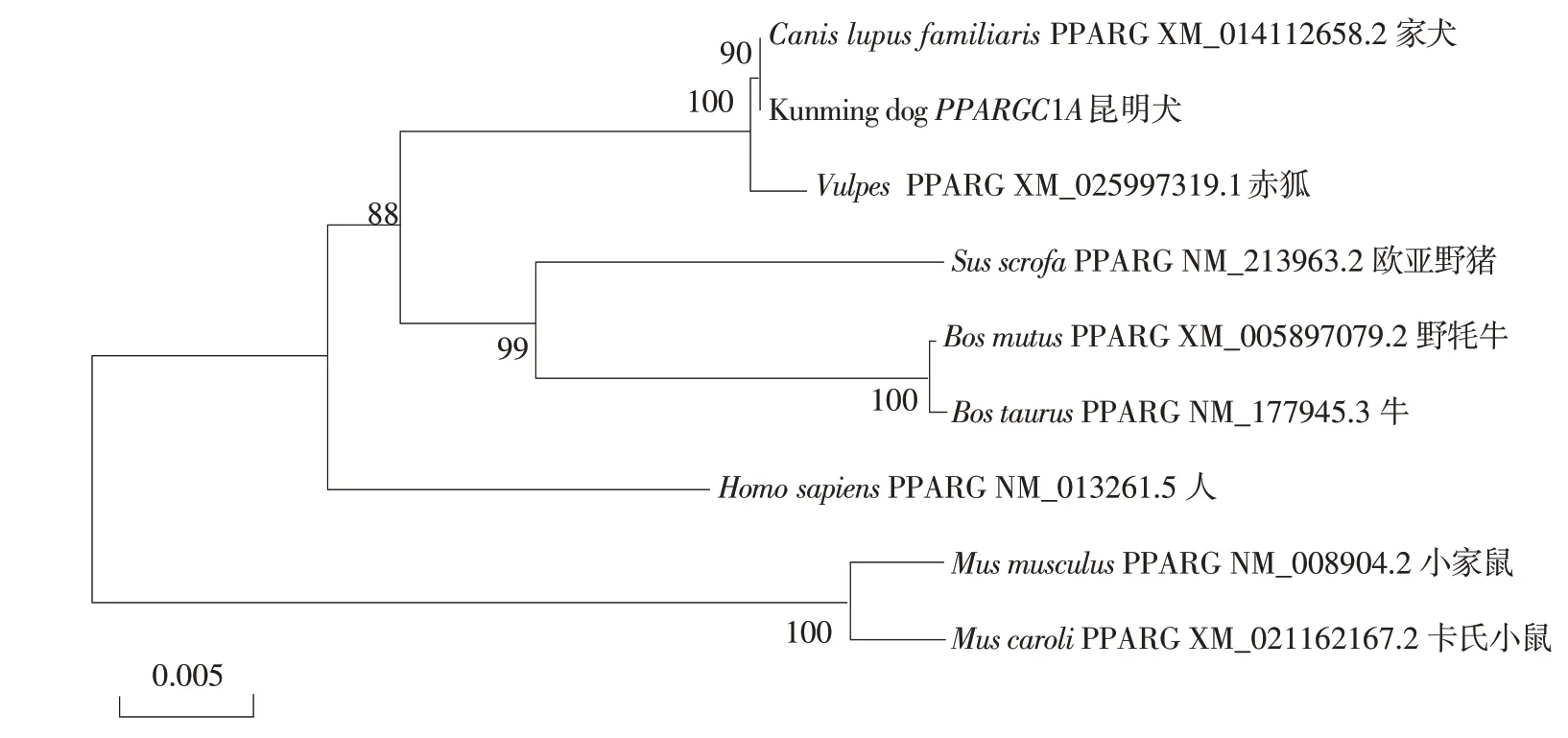
图3 昆明犬PPARGC1A基因与其他动物的系统发生树分析

表3 昆明犬PPARGC1A基因编码氨基酸组成分析
图4 中分值>0 表示氨基酸疏水,<0 表示氨基酸亲水,氨基酸分值越高疏水性越强,分值越低亲水性越强,昆明犬PPARGC1A蛋白在第683氨基酸处具有疏水最大值3.15636,在第627 氨基酸处具有疏水最小值-2.956。
2.5 昆明犬PPARGC1A蛋白亚细胞结构定位
昆明犬PPARGC1A 蛋白亚细胞结构定位见表4。

表4 昆明犬PPARGC1A蛋白亚细胞结构
2.6 昆明犬PPARGC1A 蛋白跨膜信号肽和跨膜结构预测
使用Signal P 4.0(http://www.cbs.dtu.dk/servic⁃es/SignalP/)在线预测,D=0.45是区分是否是信号肽的标志,昆明犬PPARGC1A 蛋白预测D 值=0.629,因此是一个分泌性蛋白,预测的信号肽剪切位点在第23和24位氨基酸之间(见图5)。
利用在线TMHMM-2.0分析发现PPARGC1A蛋白存在1 个跨膜结构(见图6)。跨膜位置处于第666 和第688 位氨基酸处,前665 位氨基酸处于膜外,第689至724位氨基酸处于膜内。
2.7 昆明犬PPARGC1A蛋白二级结构和三级结构
由图7可知,利用SOPMA程序预测PPARGC1A蛋白质的二级结构,共724 个AA 中含α 螺旋(Hh)203AA(28.04%),延伸链(Ee)120个AA(20.99%),β转角(Tt)49 个AA(6.77%)和无规则卷曲(Cc)352个AA(48.62%),昆明犬PPARGC1A蛋白的三级结构预测如图8,全局模型质量估计值为0.64,序列相似性为65.73%,认为所建立的三维结构模型较为准确。
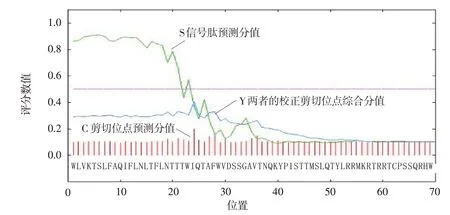
图5 昆明犬PPARGC1A蛋白的信号肽预测

图6 昆明犬PPARGC1A蛋白跨膜结构预测
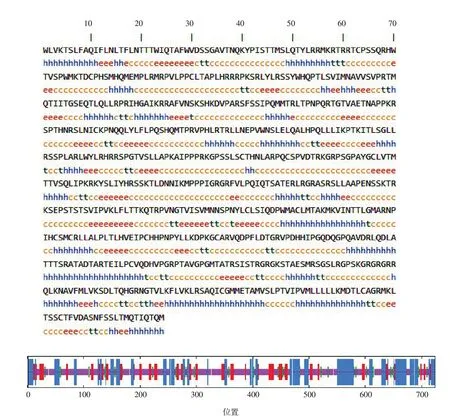
图7 利用SOPMA程序预测的昆明犬PPARGC1A蛋白的二级结构
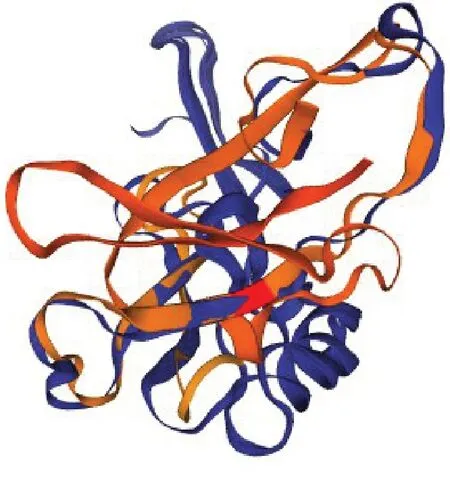
图8 预测昆明犬PPARGC1A蛋白的三级结构
3 讨 论
过氧化物酶体增殖激活受体伽玛共激活剂1-alpha(PPARGC1A)是一个在肌肉和脂肪形成中起着关键脂质代谢作用的候选基因[14]。控制一个基因阵列的表达从而影响葡萄糖和脂肪酸代谢[15]。该基因大量存在于肌肉、棕色脂肪和大脑以及有助于脂肪生成和脂肪细胞分化组织[16-18]。身体的肥胖是由于食物摄入和能量消耗之间的不平衡造成的。适应性生热响应、低温和过度进食可能导致肥胖。Puigserver P 将小鼠持续暴露于低温环境,增强适应性生热响应(BAT)和Pgc1 mRNA的表达。小鼠白色脂肪细胞中Pgc1的表达诱导UCP1基因转录激活表达UCP1,UCP 基因家族的成员仅在BAT 中使用,并通过耗散跨内线粒体膜的电化学梯度来进行ATP 合成,产生热量[19]。对啮齿动物的研究表明,体内脂肪储存可以调节[20]。成年人类没有明确定义的BAT 沉积物,但UCP1 mRNA 表达在白色脂肪组织中也可以检测到,表明棕色脂肪细胞散布在白色脂肪细胞中。
为进一步了解PPARGC1A 在UCP1 和其他线粒体组织特异性表达中的作用蛋白质及其影响犬的肌肉形成机理和产能等,本研究获得了昆明犬的PPARGC1A 基因cDNA 序列。昆明犬PPARGC1A 基因全长2 246 bp,共编码个724 个氨基酸。与参考序列家犬(登录号:XM_014112658.2)同源性为100%。与赤狐(登录号:XM_025997319.1)、野牦牛(登录号:XM_005897079.2)、牛(登录号:NM_177945.3)、人(登录号:NM_013261.5)、家鼠(登录号:NM_008904.2)、卡氏小鼠(登录号:XM_021162167.2)、欧亚野猪(登录号:NM_213963.2)、同源性分别为99.6%、95%、95%、95.4%、91.7%、91.6%、95%。昆明犬PPARGC1A基因编码蛋白的分子质量为80 971.82 ku,理论等电点pI 为11.20。人的PPARGC1A 基因跨越67 kb 的区域,由13个外显子组成,并编码一种91-ku蛋白质,与鼠具有94%的氨基酸同源性。 人类中已转录的mRNA 种类来自无TATA 启动子的序列分别为6.4和5.3 kb,被映射到染色体4p15.1[14]。猪PPARGC1A基 因(GenBank NO. NW_003540975.1 和 NM_213963.1)定位于8 号染色体以下位置的p2.1-p2.3,包含13 个外显子,其90 bp 为50-UTR。编码区大小为2 388 bp,编码796 个氨基酸[21-22]。猪PPARGC1A 基因编码的氨基酸序列与人类,大鼠,小鼠的同源性也较高。该基因在上述物种中高度同源性物种强烈暗示着该基因编码不同的生物体作为适应热原中的核心蛋白[23-25]。平均亲水系数(GRAVY)为-0.336,表明此蛋白亲水性差,脂溶性强。在组成昆明犬PPARGC1A 基因编码20 种氨基酸中,亮氨酸Leu(L)占比最高,比例为11.3%。本研究还利用生物信息学分析工具对昆明犬PPARGC1A 蛋白质二级结构、三级结构进行预测分析,含α 螺旋、延伸链、β 转角和无规则卷曲。这些结果为进一步研究该基因的结构和功能及用于昆明犬分子育种打下良好的基础。
4 结 论
本研究利用分子克隆技术获得了昆明犬PPARGC1A基因的编码区序列并进行序列分析,以期为进一步研究昆明PPARGC1A基因的表达调控及其编码蛋白质的结构对昆明犬的重要功能奠定一定的基础。
