基于纳米磷脂盘包被CD44v6的特异性多肽筛选
2020-08-12卢桂芳刘亚萍任牡丹卢新兰和水祥
张 丹,卢桂芳,冯 云,刘亚萍,赵 倩,任牡丹,卢新兰,和水祥
(西安交通大学第一附属医院消化内科,西安 710061;*通讯作者,E-mail:dyyyjxk@xjtu.edu.cn)
筛选肿瘤标志物的特异性配体是实现肿瘤靶向诊断以及治疗的重要措施。目前针对肿瘤分子影像的显像方法如PETCT/SPETCT、光学成像、MR和超声等平台的研究已获得重大的突破,但开发获取高灵敏性、特异性、亲和力的分子探针,是这项研究的难点与重点[1,2]。CD44是比较重要的肿瘤标志物,针对该蛋白的靶向药物开发已有报道。CD44v6是CD44中肿瘤特异性、灵敏性均较高的结构域。曾有针对此结构域的单克隆抗体药物进入了临床试验阶段,但因致死性副反应被中断试验[3]。相对于抗体类配体,多肽配体具有更小的分子量,因此免疫原性及毒性也相对较低[1],因此本研究致力于开发此类配体。而目前国内外尚无针对CD44v6的配体多肽报道。
噬菌体展示肽库是获得特异性亲和多肽的高通量筛选方法。本课题组前期使用噬菌体多肽展示技术分别筛选了重组蛋白、细胞、癌组织,结果表明利用细胞、组织筛选的多肽药物具体靶点分子不易明确,蛋白筛选的过程中其高级结构及活性形式难以保持,影响探针的亲和力及特异性[4-7]。而近来出现的纳米磷脂盘技术可以在体外模拟膜蛋白在细胞膜的磷脂双分子层结构中的活性结构[8]。本研究对CD44蛋白进行Nanodisc包被,改良传统噬菌体展示肽库筛选方法,筛选并验证了CD44v6特异性配体多肽序列。
1 材料与方法
1.1 细胞系及主要试剂
人胃癌SGC-7901细胞(中国科学院上海细胞库),人胚肾HEK-293细胞(美国菌种保藏中心)。真核来源的CD44v3-v10重组蛋白(OriGene,美国),噬菌体展示12肽库(New England Biolabs,美国),His-MSP蛋白(Cube Biotech,德国),抗CD44v6单克隆抗体(北京鼎国昌盛公司),抗M13噬菌体抗衣壳蛋白多克隆抗体(Santa Cruz,美国),HRP标记兔抗山羊抗体(北京博奥森公司),TMB显色试剂盒(北京博奥森公司)。
1.2 CD44v3-v10蛋白的Nanodiscs组装及检测
将137 μl的100 mmol/L胆酸钠溶液(溶于20 mmol/L Tris,pH 7.4)与4.65 mg二肉豆蔻酰磷脂酰胆碱(DMPC)混合,37 ℃摇动孵育30 min。将His-MSP蛋白溶于ddH2O中(2 mg/ml,W/V),CD44v3-v10蛋白(2 mg/ml,W/V)溶于缓冲液20 mmol/L Tris,100 mmol/L NaCl(pH 7.4),与等体积100 mmol/L胆酸钠混合。500 μl混合物与DMPC溶液混合,4 ℃、37 ℃交替孵育,每次20 min,共孵育2 h。使用3-8 kD孔径透析管4 ℃透析48 h,期间更换透析液4次。
采用分子排阻色谱鉴定,色谱柱为SuperdexTM200 Increase 10/300 GL分子排阻预装柱(GE,美国),流动相为50 mmol/L磷酸盐缓冲液含0.15 mol/L NaCl(pH 7.0),流速0.5 ml/min,上样量25 μl。
采用SDS-PAGE检测,25 μl样品与5 μl 6×SDS-PAGE上样缓冲液混合,煮沸5 min,加入10%分离胶与5%浓缩胶所制胶板,SDS-PAGE分离蛋白,考马斯亮蓝染色。
1.3 噬菌体展示肽库筛选
阴性筛选:1% BSA(W/V)溶于0.84% NaHCO3(W/V,pH 8.4),4 ℃过夜,包被至酶标板,3% BSA(W/V)封阻满孔2 h。将10 μl噬菌体展示肽库使用TBS(10 mmol/L Tris,pH 7.4)稀释至100 μl,加入阴性筛选孔,37 ℃摇动孵育2 h。
阳性筛选:将阴性筛选后未结合噬菌体上清与100 μl Nanodiscs包被CD44v3-v10蛋白混合,加入TBS溶1% BSA(W/V)以及Ni-NTA磁珠(Cube Biotech,德国),37 ℃摇动孵育2 h。去除未结合噬菌体上清,0.1% TBST(Tween-20 0.1%,W/V)洗涤磁珠6次。磁珠使用单克隆抗CD44v6抗体(15 mg/ml)竞争性洗脱30 min,收集洗脱液。
滴定:将待滴定文库使用LB液体培养基分别稀释10-6,10-7,10-8,10-9,10-10稀释度,分别加入培养至对数前期E.coli ER2738菌液中,混匀后加入3 ml顶层琼脂糖中,迅速混匀倒至LB/IPTG/Xgal固体培养基平板,37 ℃培养过夜,选择含有102数量级蓝色噬菌斑的平板计数,噬菌体滴度=(噬菌斑数目×稀释倍数)pfu/10 μl。计算回收率:回收率=回收噬菌体/投入噬菌体。
扩增:将阳性筛选后全部洗脱液加至20 ml对数期E.coliER2738菌液中,37 ℃、225 r/min振荡培养4.5 h,将培养物离心,每毫升上清中加入200 μl PEG/NaCl沉淀噬菌体,TBS重悬,重复上述沉淀-重悬过程,加入200 μl TBS/0.02%NaN3(W/V)噬菌体保存液,是为次级库。对次级库进行滴定。
后续筛选:重复以上过程进行后续筛选,每轮投入噬菌体为上轮筛选所得次级库,洗涤步骤中TBST根据筛选轮次分别为0.2%,0.3%及0.5%,第四轮筛选洗脱液滴定后不再扩增。
1.4 噬菌体测序
分别从第4轮筛选洗脱物滴定平板随机挑取30个噬菌体单克隆,分别加入3 ml对数期E.coliER2738菌液,37 ℃、225 r/min振荡培养4.5 h,将培养物10 000 r/min离心10 min,收取上清,命名为噬菌体单克隆贮液。在贮液500 μl中加入200 μl PEG/NaCl,4 ℃沉淀噬菌体,去除上清,使用碘化物缓冲液(10 mmol/L Tris,1 mmol/L EDTA,4 mol/L NaI)重悬,加入250 μl无水乙醇沉淀洗涤DNA,短时间真空干燥,使用30 μl TE缓冲液(10 mmol/L Tris,1 mmol/L EDTA)重悬沉淀。使用-96 gⅢ测序引物(5′-HOCCC TCA TAG TTA GCG TAA CG-3′)行全自动测序(上海生工公司,中国),根据噬菌体多肽展示文库附带使用手册,读取及翻译噬菌体测序结果。
1.5 噬菌体ELISA验证
取噬菌体单克隆贮液,常规扩增、纯化、滴定。阳性孔采用CD44v3-v10重组蛋白(1 μg/孔)加入100 μl 0.84% NaHCO3(W/V,pH 8.4),阴性孔采用3%(W/V) BSA,4 ℃过夜包被酶标板,3% BSA封阻满孔2 h。分别加入待测克隆以及随机对照克隆(1011pfu/孔),37 ℃孵育1 h,常规洗涤。加入0.1% TBST稀释的山羊抗M13噬菌体衣壳蛋白多克隆抗体(稀释度1 ∶1 000,V/V),加入0.1% TBST稀释的HRP标记兔抗山羊抗体(稀释度1 ∶5 000,V/V),一二抗均37 ℃孵育1 h,常规洗涤。TMB显色试剂盒显色,2 mol/L H2SO4终止反应,酶标仪在波长450 nm处读取吸光度。
1.6 噬菌体洗脱试验
分别接种SGC-7901、HEK-293于6孔培养板,待细胞长满单层。4%多聚甲醛固定液4 ℃固定,3% BSA 37 ℃封闭30 min。每孔加入1011pfu待测噬菌体或随机对照噬菌体与细胞37 ℃孵育1 h,0.1% TBST洗板6次,加入1 ml甘氨酸-盐酸洗脱缓冲液(1.5%甘氨酸,0.1%BSA,W/V,pH 2.2),室温洗脱,150 μl中和缓冲液(12.1%Tris,W/V,pH 9.1)中和,洗脱物采用1.3所述方法滴定。
1.7 统计学分析
统计采用SPSS19.0(SPSS, US),计量数据表示为均数±标准差,组间差异比较采用t检验,P<0.05被视为差异具有统计学意义。
2 结果
2.1 CD44v3-v10蛋白的Nanodics组装
对包被后的产物进行SDS-PAGE验证可以发现,图1A展示了包被后的左侧泳道相较右侧出现新增的大分子量条带,提示已形成Nanodiscs化的CD44v3-v10蛋白。随后在分子排阻色谱的进一步鉴定中发现,有明确的产物峰形成(见图1B),在产物峰前较低的峰可能提示了一些非特异性聚体的形成所产生的更大直径纳米盘,或者CD44v3-v10蛋白分子之间胞外段的交联。在前期预试验中,通过对反应时间的控制,将非特异峰值控制于最低水平。

图1 CD44v3-v10蛋白的Nanodiscs组装检测Figure 1 Detection of CD44v3-v10 assembled Nanodiscs
2.2 针对Nanodiscs的噬菌体展示
整个筛选过程中包括阴性消减及阳性筛选,增加筛选效力。洗脱目标噬菌体时采用了单克隆抗体的竞争性洗脱,以保证所获得噬菌体的特异性。整个筛选重复4次用于富集阳性噬菌体,并逐步降低非特异结合噬菌体所占比例。前三轮的洗脱噬菌体在下一轮筛选前均进行扩增,一方面保证筛选的力度及有效性,一方面可以使数量多的噬菌体进行指数级别富集,提高阳性噬菌体的比例。每轮筛选需滴定后计算回收率(产出噬菌体/投入噬菌体)。图2A为第4轮筛选后102级别噬菌体滴定结果,图2B提示了四轮筛选的回收率变化,可见随着筛选进行,回收率逐渐升高,四轮分别为10-7,10-5,10-4,10-3级别,提示阳性噬菌体所占比例逐渐增多以及目标噬菌体的富集效应。
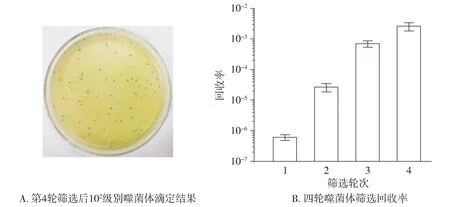
图2 噬菌体展示的滴定及筛选回收率Figure 2 Phage titer and recovery rates of phage display
第4轮筛选结束后,对随机的30个噬菌体克隆进行测序分析。根据噬菌体随机十二肽展示文库手册中的方法对测序结果分析,寻找限制性内切酶位点KpnⅠ以及EgalⅠ的酶切位点GGTACC和CGGCCG,从而获得插入DNA序列,再利用M13噬菌体专属密码子表进行翻译,最终获得噬菌体所展示的12肽序列。表1展示了所挑选30个噬菌体所携带的多肽序列。30个噬菌体克隆共携带6条多肽序列,分别命名为:NP-1(HNTPSVRHFYKQ)、NP-2(HNTYVTSFHRNY)、NP-3(WQKPSHIPFNAS)、NP-4(QTALYSKPGPPV)、NP-5(ELYYHNTDIESQ)、NP-6(YHWEAYSTTPIS)。6条序列重复中重复的噬菌体克隆分别为8次、12次、4次、3次、2次、1次。重复次数更多的序列体现出筛选过程中的富集效应,相较重复少的序列亲和力可能更好,因此也是后续验证的重点。经比对发现NP-1和NP-2、NP-2与NP-5分别包含有一个重复的三肽基序HNT,可能是多肽结合于靶蛋白的关键氨基酸位点。

表1 噬菌体展示多肽序列及重复情况Table 1 Peptide sequence and repetition frequency of phage display
2.3 亲和配体序列的选择
噬菌体ELISA是用于鉴定噬菌体单克隆的常用方法。为排除结合于Nanodiscs其他成分的噬菌体,选择固相包被较高量的CD44v3-v10蛋白作为噬菌体结合的靶点。6个待测噬菌体克隆、阴性对照随机序列克隆(unrelated random phage,Urps)以及空白对照噬菌体溶剂TBS,分别结合于靶蛋白包被孔以及BSA阴性对照孔。评价标准包括噬菌体与靶蛋白结合的吸光度绝对值,还包括噬菌体与靶蛋白、BSA吸光度的比值,分别代表了噬菌体结合的亲和性及特异性。图3A展示了ELISA验证待测噬菌体及对照噬菌体针对靶蛋白及对照蛋白的检测结果,可见噬菌体与靶蛋白结合的吸光度绝对值自高到低顺序为NP-2、NP-5、NP-1、NP-2、NP-4、NP-3、NP-6,靶蛋白、BSA吸光度的比值自高到低顺序与此一致。因此选择亲和性及特异性表现均最好的NP-2、NP-5、NP-1三个序列做进一步的鉴定。
由于本研究以获取结合于CD44v天然活性形式的配体为主要目的,因此在使用固相包被蛋白确定了特异性噬菌体待测克隆后,继续选择了CD44v6不同表达情况的人细胞做进一步结合验证。图3B为噬菌体洗脱试验结果。可以发现NP-2、NP-5、NP-1三个克隆均与CD44v6阳性细胞SGC-7901结合更多,结合噬菌体计数高于其与CD44v6阴性细胞HEK-293的结合噬菌体数(P<0.05),也高于无关随机对照克隆在SGC-7901细胞上的结合数量(P<0.05)。3个克隆中,NP-2、NP-1的结合噬菌体为107数量级,而NP-5仅为105数量级。此外,NP-2与阴性细胞结合噬菌体仅为105数量级,而NP-1与阴性细胞结合噬菌体为106数量级。从结合力、选择性两方面综合考虑,NP-2为此次筛选获得的最优序列。

Urps:随机无关克隆;TBS:空白对照图3 待选噬菌体克隆的鉴定Figure 3 Identification of canditate phages
3 讨论
CD44是多种重要的肿瘤标记物,现有报道支持了其与肿瘤的相关性。CD44作为细胞黏附分子,可以促进细胞与细胞外基质的黏附,调节细胞运动功能,调节细胞伪足形成以及其迁移运动[9]。CD44的不同剪切表达以及与透明质酸等配体的结合在肿瘤的发生发展中起到了重要的作用[10]。CD44与肿瘤的关系主要包括促进肿瘤细胞增殖、抑制凋亡、以及促进肿瘤侵袭迁移等方面[11-14]。
在已知药物的靶点蛋白中,大多数为膜蛋白,因其定位于细胞膜性结构,避免了细胞外分泌蛋白对探针定位的干扰,而且以胞外段为靶点也无需探针具备穿膜特性。CD44分子是跨膜糖蛋白,包含有胞外段,跨膜段以及胞内段3个部分,胞外段所占分子量最大,是理想的药物靶点。但CD44胞外段存在多种剪切表达结构,构成多种转录变异体。其中包含有变异型外显子编码区的CD44分子在普通细胞及肿瘤细胞间具有最明显的表达差异,具有最好的肿瘤特异性[15]。其中, CD44v6的肿瘤灵敏性及特异性最为理想,因此也成为靶向药物研发的热点,其单克隆抗体比伐珠单抗(bivatuzumab)在头颈部肿瘤的治疗中已进入临床试验阶段,但却因抗体药物引发的致死性毒副反应被终止研究[3]。
传统的常用的靶向药物包括抗体类、小分子类。但抗体分子量大,免疫原性过高,成本及运输储存条件制约性过大,小分子类易于获取,但缺乏抗体分子多样性所带来的高特异性和高亲和力[16]。多肽类药物是近年研究较多的靶向药物种类,有着结构多样、特异性好、代谢快、免疫原性低、价格低廉的多种优势[16]。
噬菌体展示肽库是获得特异性亲和多肽的高通量筛选方法。该技术的靶点可以是蛋白、核酸等分子,也可以是细胞、离体或者活体组织[17]。现有报道中,使用细胞、组织筛选的多肽药物具体的分子靶点多不明确[4,18]。由于细胞、组织表面成分复杂,后续可能需要免疫共沉淀、蛋白组学研究寻找可能的分子靶点,试验难度相对大,影响因素也比较多[19]。使用重组蛋白,在筛选过程中进行固相包被会损害大分子蛋白的高级结构,难以实现分子在体内的状态,为日后的应用带来限制。
CD44是膜蛋白,只有在双层膜及类脂环境中才能保证活性状态,体外难以模拟,而在细胞中表达量往往较低,不足以实现严格的消减筛选[20]。为了实现对活性膜蛋白的研究,近年出现了磷脂纳米盘(Nanodisc)技术。它是通过在体外模拟细胞膜的磷脂双分子层结构,使用高密度脂蛋白中的ApoA-1蛋白改造形成膜脚手架蛋白(MSP),该蛋白可以包裹磷脂双分子形成类膜结构,靶点膜蛋白可以组装于这种类膜结构上以获得其活性形式[8]。
膜蛋白的Nanodics组装包括3个要点,首先是膜蛋白的获取,然后是蛋白、磷脂、骨架蛋白的包被比例及条件,最后是对包被结果进行验证。由于膜蛋白的分离纯化较为复杂,难以获得单一蛋白,因此拟以重组蛋白作为靶蛋白。CD44v3-v10 (NP 001001389.1) 蛋白是已知天然人源性CD44中,包含全部肿瘤相关结构域的异构体,其cDNA也可以实现常规文库的钓取。在重组蛋白的载体选择中,优先选取了人源性细胞作为表达来源,因其可提供最接近于自然状态下的CD44形态及高级结构,提高后续筛选配体在实际应用中的亲和力及特异性。
Nanodisc通过MSP蛋白包裹磷脂双分子形成类膜结构,靶点膜蛋白可以组装于这种类膜结构上以获得其活性形式。其中膜蛋白与MSP蛋白的比值最为重要。为避免多个膜蛋白结合于一个Nanodiscs或膜蛋白分子之间发生非特异性结合,本研究采取过量MSP蛋白,这也是大多数研究采取的方法[8]。而MSP蛋白与磷脂的投入比相对固定。本研究采用了商品化的Nanodiscs 试剂盒,可提供既定的MSP及磷脂投放量。
噬菌体展示可以实现对固相及液相靶蛋白的筛选,固相包被相对需要的靶蛋白量较多,且不利于Nanodiscs中膜蛋白高级结构的充分展示,因此本研究选择了液相筛选方法。为实现目标噬菌体的收集,靶蛋白包被时采用了His-tag-MSP蛋白,筛选时可利用Ni-NTA 磁珠进行特异性吸附。在每轮筛选前,噬菌体随机肽库需要与BSA进行非特异性结合,以消减筛选的形式减少非特异性吸附的产生。阳性筛选过程通过逐渐增加强度的洗涤除去亲和力不足的噬菌体。在获取结合噬菌体时采取CD44v6单克隆抗体的竞争性洗脱。以保证获取噬菌体均结合于该抗原决定簇。
在对CD44v3-v10蛋白包被后,使用了SDS-PAGE及分子排阻色谱双重鉴定。两种验证均提示大量产物形成,因此可以考虑进行下一步的筛选。在噬菌体展示筛选过程中,可以发现第1轮回收率仅10-7级别,有较强的筛选力度,而后回收率逐轮上升,第4轮上升至10-3级别,提示了有效噬菌体在每轮次级库中的比例逐渐升高。对获取克隆的测序发现,待选噬菌体出现了比较明显的富集现象,30个克隆仅携带6条序列,重复最高的序列占所有克隆的40%。其中两条序列还出现了相同的三肽基序,证明筛选的有效性,而这个三肽基序则可能是整条多肽结合的关键位点。
为了排除结合于Nanodisc其他成分的噬菌体,噬菌体ELISA检验中仅使用靶蛋白而非组装好的Nanodisc。检验发现,阳性克隆结合于靶蛋白明显高于对照蛋白,也高于对照克隆,证明所筛选的阳性克隆对靶蛋白的特异性。而经过综合考虑每个克隆与靶蛋白结合的OD值,以及与对照蛋白结合的情况,对待选噬菌体进行了初筛,排除部分结合较低或特异性差的噬菌体。继而待测克隆还需结合于细胞水平表达的靶蛋白,以确定噬菌体与靶蛋白活性形式的结合力及特异性。噬菌体细胞洗脱试验也证实了几个待测克隆对靶蛋白肿瘤细胞以及靶蛋白阴性细胞的结合。NP-2噬菌体因其结合力及结合特异性被选择为最佳序列。后续工作会将筛选获得的多肽序列进一步合成探针,检验其体内外与靶蛋白、靶细胞的结合,为CD44v6阳性肿瘤的靶向诊断及靶向治疗提供方法和临床前期基础。
