基于Cytb和D-Loop的日本囊对虾遗传多样性分析
2020-07-28杜景豪王伟峰陈秀荔侯春秀王焕岭
杜景豪,王伟峰,陈秀荔,侯春秀,王焕岭
( 1.华中农业大学 水产学院,湖北 武汉 430070; 2.广西壮族自治区水产科学研究院,广西水产遗传育种与健康养殖重点实验室,广西 南宁 530021 )
日本囊对虾(Marsupenaeusjaponicus)属甲壳纲、十足目、对虾科、囊对虾属。日本囊对虾的分布极其广泛,在我国主要分布在长江口以南沿海,其具有耐低温、生长快、营养高等特点,在我国对虾养殖中具有较高的经济地位[1]。近年来,捕捞量的增加、养殖规模的扩大以及水域生态环境的恶化等,导致其自然资源不断衰退。而遗传变异水平与生物的生长速度、抗病能力等生产性状密切相关[2]。因此开展日本囊对虾的遗传多样性分析对其种质资源保护与合理开发利用具有重要的意义。
线粒体DNA(mtDNA)是一种核外遗传物质,由于具有分子量小、结构简单、进化速度快、母系遗传及无组织特异性等特点[3],而成为评估群体遗传和生物进化的有效标记[4]。Tsoi等[5-6]曾利用mtDNA和微卫星标记研究西太平洋日本囊对虾,发现其可以分成2个变种,并且都具有很高的遗传多样性。郭慧等[7-8]利用微卫星标记对我国沿海3个日本囊对虾群体遗传多样性进行了研究,发现不同地理群体间具有不同程度的分化。
而动物线粒体基因组中含细胞色素氧化酶3个亚基基因 (COⅠ、COⅡ、COⅢ)、细胞色素b(Cytb)、2个ATP酶亚基因(ATPase6、ATPase8)和7个NADH还原酶复合体亚基因共13个结构蛋白基因[9]。其中由于Cytb基因的进化速度适中,诸多学者选择其作为遗传多样性分析的标记,用于分析种群母系起源、系统进化、遗传结构和遗传多样性等[10-13]。另外线粒体DNA控制区(D-Loop)处于非编码区,富含A-T碱基,进化速度较快,是揭示种群遗传结构常用的分子标记[14-15]。近年来,研究发现利用D-Loop序列进行遗传多样性和系统进化研究信息更加全面,结果更为准确[16-17]。因此,笔者基于Cytb和D-Loop序列,对我国日本囊对虾遗传多样性展开研究,为日本囊对虾种质资源保护及合理利用和杂交育种的群体选择提供科学依据。
1 材料与方法
1.1 试验材料
试验所用日本囊对虾于2018年8月—2019年3月分别采自浙江舟山、福建福州、福建厦门、广西北海、广东湛江5个近海水域,每个群体约20尾。所有样品均取对虾腹部第六腹节肌肉,于-20 ℃无水乙醇中保存。
1.2 基因组DNA的提取
取肌肉100~150 mg,采取SDS法[18]提取基因组DNA,使用超微量紫外分光光度计测定DNA质量浓度并稀释到150 ng/μL,1%琼脂糖凝胶电泳检测DNA,于4 ℃保存。
1.3 PCR扩增
根据美国国立生物技术信息中心中日本囊对虾线粒体的基因组序列(序列号:AP006346)设计引物用于扩增Cytb基因和D-Loop区。其中扩增Cytb的引物序列为:Cb-L 5′-GGTCCTTTACG CTTATCATC-3′和Cb-R 5′-GGGTGAGTGGGTTAATAATG-3′;扩增D-Loop的引物序列为:Dp-L 5′-ATTAGCACTAGGTACTGAGA-3′和Dp-R 5′-GGTAAGGCTTGACTTACATA-3′;由武汉天一辉远生物科技有限公司合成引物。PCR反应体系为20 μL,其中双蒸水15.2 μL,10×PCR Buffer 2.0 μL,DNA模板1.0 μL,左右引物各0.6 μL,dNTPs 0.3 μL,TaqDNA聚合酶0.3 μL。PCR扩增反应程序为:94 ℃预变性5 min,94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸75 s,共32个循环,最后72 ℃终延伸7 min。1%琼脂糖凝胶电泳检测PCR产物,送至武汉天一辉远生物科技有限公司进行双向测序。
1.4 数据分析
使用Seqman软件对比测序结果,并辅以人工校正;DnaSP[19]软件统计多态位点数、单倍型数、单倍型多态性、核苷酸多态性、多态简约信息位点数、Fu和Li′sD检验参数、Tajima′sD检验参数;MEGA[20]软件用于分析序列的平均碱基组成、转换/颠换比率R、群体内和群体间的遗传距离以及系统发育树[21]的构建;Arlequin 3.1[22]软件分析群体间及群体内的遗传变异和核苷酸错配分布情况。
2 结 果
2.1 碱基组成
测序结果经过对比和校对后,分别选取长度1000 bp和900 bp的Cytb和D-Loop区用于后续分析。分析结果显示,5个群体中Cytb和D-Loop序列的碱基颠换比值R(TS/TV)分别为6.267和2.694,突变以碱基的颠换为主。4种碱基在所得序列中平均含量A为24.77%和37.22%、T为35.88%和45.32%、C为24.07%和8.68%、G为15.28%和8.78%,其中A+T含量为60.65%和82.54%,显著高于C+G含量(表1),D-Loop序列较Cytb序列碱基组成更具偏倚性。
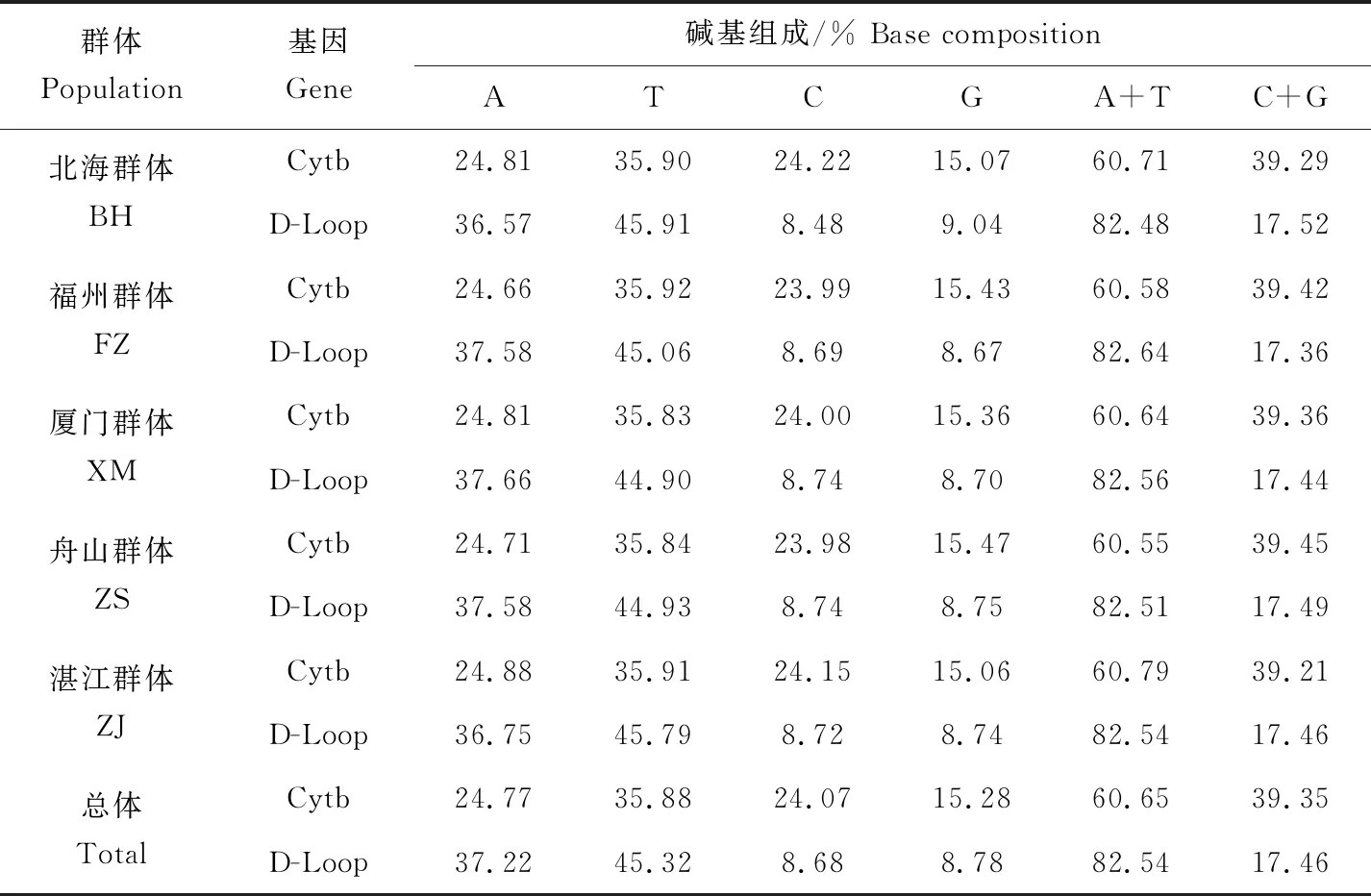
表1 日本囊对虾5个群体Cytb和D-Loop序列碱基组成
2.2 遗传多样性和遗传结构
基于Cytb序列在5个群体中检测到76个简约信息位点,共定义了55种单倍型,其中Hap-30、Hap-36数量最多,均为10个,占样本总数的10.1%;Hap-6数量为9个,占样本总数9.1%;Hap-50、Hap-53数量均为4个,占样本总数的4.1%;Hap-21、Hap-54数量均为3个,占样本总数的3.1%;Hap-2、Hap-9、Hap-19、Hap-29、Hap-35、Hap-47、Hap-49、Hap-51数量均为2个,占样本总数的2.1%;其他单倍型均只有1个(表2)。单倍型多态性为0.969,核苷酸多态性为0.033。群体内遗传距离为0.004~0.022,群体间遗传距离为0.007~0.068,其中遗传距离最大是舟山群体与北海群体,为0.068(表3)。基于D-Loop序列分析,在5个群体中发现了75个简约信息位点,共定义了78种单倍型,其中Hap-22数量为4个,占样本总数的4%;Hap-7、Hap-65数量均为3个,占样本总数的3%;Hap-2、Hap-6、Hap-19、Hap-26、Hap-30、Hap-38、Hap-42和Hap-49数量均为2个,占样本总数的2%;其余单倍型均为1个。单倍型多态性为0.995,核苷酸多态性为0.036。群体内遗传距离为0.005~0.017,群体间遗传距离为0.012~0.075,其中遗传距离最大是舟山群体与北海群体(0.075)(表4)。D-Loop序列中单倍型数、单倍型多态性及核苷酸多态性均高于Cytb序列,这表明D-Loop序列具有更高的遗传多样性,这可能与其进化速度较快有关。

表4 基于D-Loop序列的群体内及群体间遗传距离

表3 基于Cytb序列的群体内及群体间遗传距离
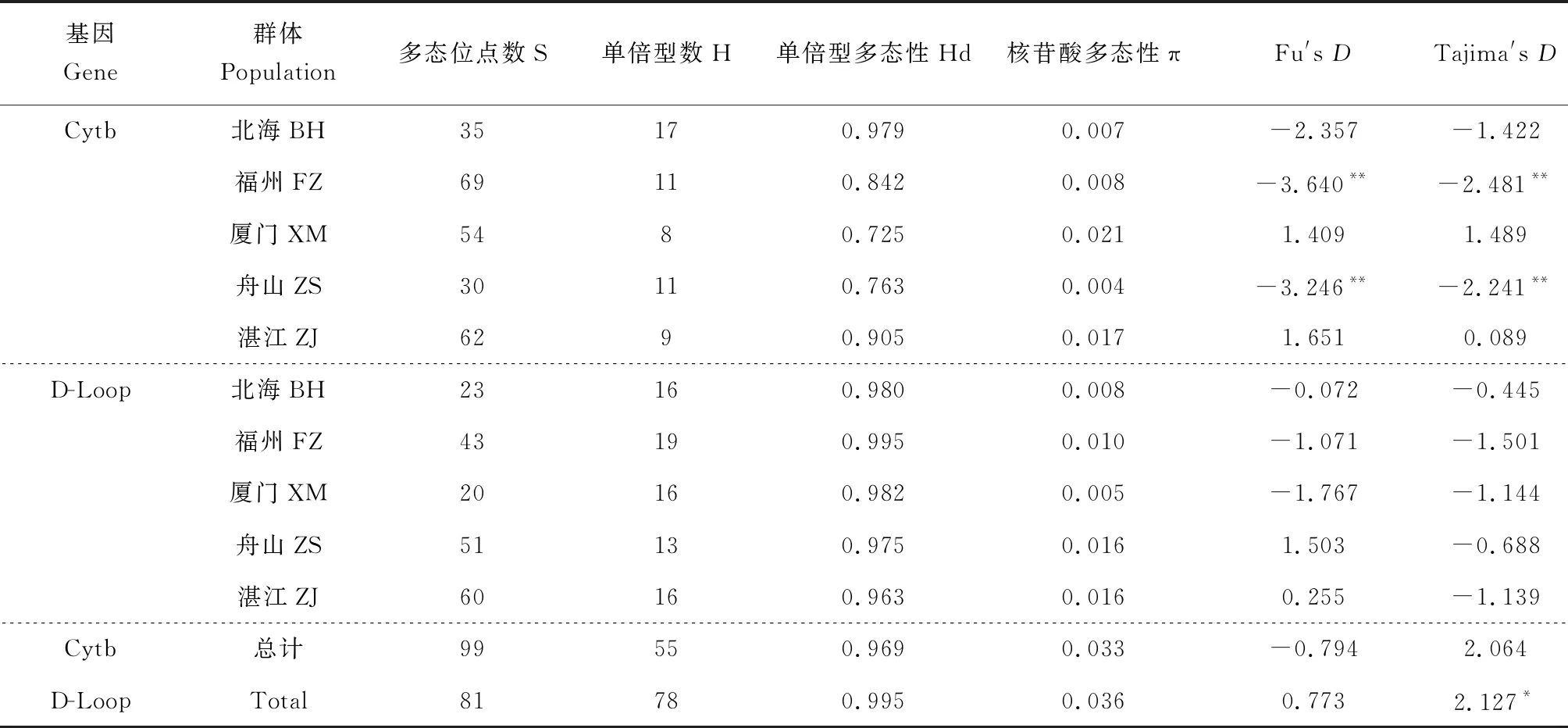
表2 群体遗传多样性参数
分子方差分析结果显示,基于Cytb和D-Loop序列获得的群体内变异贡献率分别为29.99%和25.61%,群体间变异贡献率达到70.01%和74.39%,可见群体间变异是变异的主要来源(表5)。遗传分化指数(FST)常用于表示群体间的遗传关系及分化程度,值越大表示群体分化程度越大[23]。两者分化指数分别为0.7001和0.7439,表明群体间遗传分化程度明显。

表5 群体分子方差分析
2.3 单倍型系统发育树
基于单倍型分别构建系统发育进化树,结果显示(图1),不同群体间单倍型分化比较明显,主要分化成两个单系,即北海群体和湛江群体聚成一支,其他3个群体聚成另一支,同一单系间又有不同程度的分化,说明日本囊对虾5个群体间存在比较明显的地理谱系,种群间分化具有明显的地域性。

图1 基于Cytb(a)和D-Loop(b)序列单倍型系统发育树
2.4 种群历史动态
中性检验结果见表2,基于Cytb序列发现,北海、福州和舟山3个群体Fu′D值和Tajima′D值为负值,而其他2个群体Fu′D值和Tajima′D值为正值,表明北海、福州和舟山3个群体近期发生了种群扩张,但总体未明显偏离中性突变,种群存在平衡选择,近期未出现明显扩张。基于D-Loop序列研究发现,5个群体Tajima′D值均为负值,总体 Tajima′D值为2.127,具有显著性差异,表明各群体之间存在定向选择,总体存在平衡选择。而基于Cytb和D-Loop序列中性检验值在5个群体内具有差异性,可能是由于不同群体所处环境不同且D-Loop区更容易发生碱基突变。日本囊对虾5个群体Cytb和D-Loop区核苷酸错配分布见图2,两者成多峰分布,表明整个种群保持相对稳定,近期未曾经历过瓶颈效应和明显扩张。
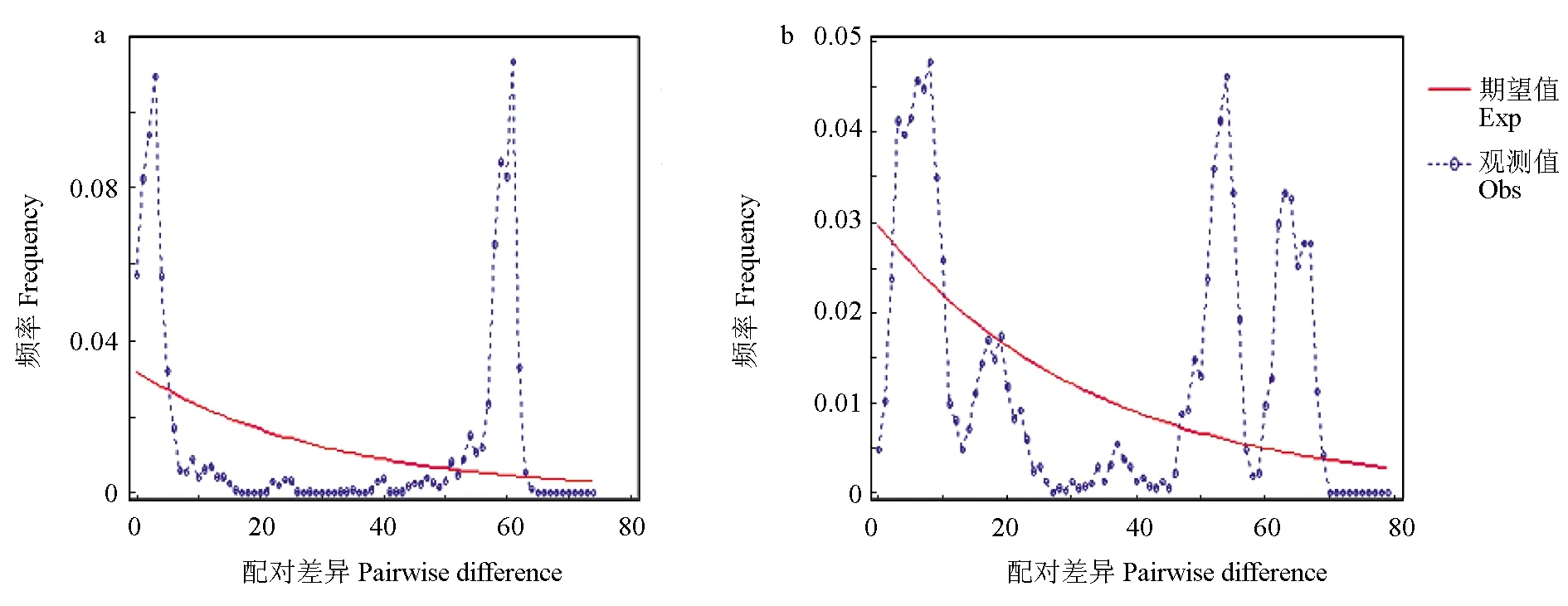
图2 群体Cytb(a)和D-Loop(b)核苷酸错配分布
3 讨 论
3.1 序列碱基组成分析
本研究基于线粒体Cytb和D-Loop序列对我国沿海5个日本囊对虾群体的遗传多样性进行了分析,发现4种碱基出现明显的偏随机分布,即A+T含量显著高于C+G含量,符合节肢动物mtDNA碱基组成中G+C含量相对缺乏,A+T含量高的普遍现象[24]。曾凡荣等[25]基于日本囊对虾线粒体Cytb的研究发现,A+T含量高达59.4%,与本研究相关结果基本相符。利用颠换比值R,可以估计序列的饱和度,R>5表示序列未达到饱和,一般来说分歧时间越长、亲缘关系越远的群体间碱基发生颠换的频率越高,即R值越小,由此可初步判断群体间的进化关系[26]。Cytb和D-Loop区碱基颠换比值R分别为5.970和2.694,说明Cytb突变未达到饱和而D-Loop区突变可能已达到饱和,两者均产生了一定程度的进化。
3.2 群体遗传多样性与遗传分化分析
遗传多样性即基因多样性,存在于生物个体内、单个物种内以及物种间,是生物适应和进化的基础,种内的遗传多样性与该物种对环境的适应能力呈正向线性关系[27]。物种遗传多样性的分布不仅受到现阶段演化力量(如迁移)的影响, 还受到历史的影响,生活环境的不连续性和种群数量变动的不稳定,均会造成种群间的分化[28]。刘若余等[29]研究表明,线粒体DNA的单倍型多态性和核苷酸多态性是衡量群体遗传多样性和遗传分化的重要指标,两者数值越大,群体遗传多样性越高。本研究从Cytb和D-Loop区两个角度分析发现,两者的单倍型多态性分别为0.969和0.995,核苷酸多态性为0.033和0.036,这说明我国沿海5个日本囊对虾群体遗传多样性处于较高的水平。Tzeng等[30]研究日本囊对虾线粒体控制区发现,东亚地区日本囊对虾具有很高的遗传多样性,与本研究的结果基本相符。
从遗传距离来看,物种内的遗传距离通常小于10%[31]。大于6%的群体间会有明显的亚种或者种的分化[32-33],不论是从Cytb或D-Loop来衡量,北海群体和湛江群体与其他3个群体遗传距离均较远,接近亚种的分化水平,可能由于不同群体间地理位置相距较远,阻碍了群体间基因交流。基于Cytb和D-Loop序列分子方差分析显示,群体间变异是总变异的主要来源。而曾凡荣等[25]基于Cytb基因的研究也发现,日本囊对虾群体间遗传变异占69.9%。据溯祖理论[34],Cytb中单倍型Hap-30和Hap-36数量最多可能为祖先类型,而D-Loop区单倍型Hap-22可能为祖先类型。D-Loop区单倍型分析发现,5个群体间共享单倍型较少,群体分化十分明显,这与Cytb基因分析结果有所不同,可能是由于D-Loop区受到的选择压力小,进化速度较快,而Cytb是编码蛋白质的功能基因,受到的选择压力较大,进而导致Cytb和D-Loop的变异存在不均一性[35]。基于Cytb和D-Loop序列单倍型进化树发现,各群体特有单倍型基本聚在一起,部分存在交叉的现象,可能是群体间的部分个体在自然生长过程中遗传背景存在交叉[36-37]。基于Cytb和D-Loop序列分析发现,D-Loop进化速度要高于Cytb,遗传多样性更加丰富,相对更加接近分子钟假说。因此,D-Loop比Cytb更加适合于研究日本囊对虾群体遗传多样性的分子标记。
由种群的错配分布图可见,种群呈多峰分布,表明种群近期未曾经历过瓶颈效应和明显扩张[38],保持相对稳定的状态,自然选择将突变保持在平衡水平。此外,日本囊对虾产卵洄游并不是特别明显,仅随个体生长由近岸向远岸迁移,这样地理距离相距较近的群体更容易发生基因交流。气候急剧变化会引起群体数量突然减少,可能导致种群遗传多样性差异,而洋流、迁徙和人类活动会促进不同群体间的基因交流而减少分化[39-40]。
本研究通过日本囊对虾不同地理种群的遗传多样性分析发现,我国日本囊对虾群体具有较高的遗传多样性,野生资源状况良好,为日本囊对虾的种质资源保护工作提供科学依据,也为杂交育种的群体选择提供参考资料。
