赤眼鳟β-肌动蛋白基因克隆、组织表达与稳定性分析
2020-07-18陈蕾陈淑颐周刚黄宇宏李伟王静安肖调义刘巧林
陈蕾, 陈淑颐, 周刚, 黄宇宏, 李伟, 王静安, 肖调义, 刘巧林
赤眼鳟-肌动蛋白基因克隆、组织表达与稳定性分析
陈蕾, 陈淑颐, 周刚, 黄宇宏, 李伟, 王静安, 肖调义, 刘巧林
(湖南农业大学 湖南省特色水产资源利用工程技术研究中心, 湖南 长沙, 410128)
为探究赤眼鳟()-肌动蛋白(actin)基因的结构和表达特性, 本文采用RT-PCR技术和RACE法, 从赤眼鳟肝脏中获得了赤眼鳟actin cDNA全长序列(GenBank: MT119965), 该基因cDNA全长共1 816 bp, 由94 bp的5’端非编码区, 594 bp的3’端非编码区和1 128 bp的开放阅读框(95-1222)组成, 阅读框共编码375个氨基酸。通过序列比对和系统进化树的构建, 赤眼鳟actin与鲤科鱼类聚为一支且与鲮鱼、鲫鱼、黄颡鱼等多个物种的actin氨基酸同源性都为99%。荧光定量(qPCR)表达分析显示,actin在赤眼鳟肌肉、肠、脾脏、皮肤、血液、鳃、体肾、头肾、心脏和肝脏十个组织中都有表达, 但不同组织的表达量存在差异。利用Best keeper软件对赤眼鳟体内的延伸因子1(elongation factor 1,)和actin基因进行稳定性对比, 发现actin基因稳定性低于EF1基因。以上结果为赤眼鳟的分子系统进化研究提供证据, 也为赤眼鳟功能基因表达研究提供参考依据。
赤眼鳟;-肌动蛋白; 基因克隆; 组织表达; 稳定性
肌动蛋白在真核细胞内广泛存在, 是细胞骨架和肌小节的主要组成成分, 几乎参与了细胞所有的生命活动[1], 在细胞结构、细胞运动、细胞分裂等生命活动中都发挥了重要作用[2–3]。根据等电点的不同, 肌动蛋白可分为、、3类[4]。目前针对水产动物-肌动蛋白(actin)基因的研究较多, 如三角帆蚌()[5]、牡蛎()[6]和草鱼()[7]等。多种物种actin基因研究表明,actin氨基酸编码区具有高度保守性。大鳞副泥鳅()actin氨基酸序列与脊椎动物的相似性在98%以上, 与其他动物的相似性不低于97%[8]松江鲈()actin基因序列与其他物种的同源性在79.58%~97.16%[9]。同时, 因actin基因具有表达数量高且稳定的特点[10–11], 所以在荧光定量PCR技术中常采用actin基因作为参照, 如草鱼[12]、皱纹盘鲍()[13]和达氏鲟()[14]等。但随着对actin基因的深入研究, 发现存在表达量不稳定的情况, 如Sarmiento等[15]发现广温性的鲤()的actin基因在夏季和冬季的表达量发生了变化。因此近些年来为了筛选出最稳定的内参基因, 有大量研究关于几个常用内参基因在不同生物体内的稳定性, 如GAPDH(3-磷酸甘油醛脱氢酶)、EF1(延伸因子1)以及actin等。大部分研究表明, 无论是在植物体内[16], 还是动物体内[17]actin都是较为可靠的内参基因。
赤眼鳟属硬骨鱼纲、鲤形目、鲤科、雅罗鱼亚科、赤眼鳟属, 俗称野草鱼、红眼鱼[18], 具有生长快、适应性强、经济价值高等优点。目前已针对赤眼鳟开展了生物学特性[19]、资源现状[20]、免疫特性[21]、基因挖掘[22]等功能多方面的研究, 但没有赤眼鳟actin基因cDNA克隆和表达分析, 更无内参基因筛选的相关报道。
本研究通过RT-PCR和RACE技术, 从赤眼鳟肝脏中克隆出actin基因cDNA全长序列, 对其进行氨基酸理化性质和同源性分析, 并采用actin基因为外源基因, 构建系统进化树, 探究赤眼鳟与其他物种的进化关系。通过qPCR技术分析赤眼鳟actin基因在10个组织的表达, 并与生物体内常用内参EF1基因做稳定性对比。为利用actin基因作为内参基因分析赤眼鳟的基因表达研究提供序列依据, 也为赤眼鳟的分子系统进化研究提供参考。
1 材料与方法
1.1 实验动物与样品采集
湖南浏阳市乌龙渔场采5尾5月龄健康赤眼鳟, 丁香酚(国药, 上海)麻醉后解剖采集赤眼鳟肝脏、肌肉、肠、脾脏、皮肤、血液、鳃、体肾、头肾和心脏, 剪成100 mg左右大小分装样品管中, 液氮速冻,-80 ℃保存备用。
1.2 研究方法
1.2.1 赤眼鳟组织总RNA抽提和cDNA合成
按照TaKaRaMiniBEST Universal RNA Extraction (TaKaRa, China)试剂盒说明书分别提取肝脏、肌肉、肠、脾脏、皮肤、血液、鳃、体肾、头肾和心脏总RNA。按Clontech公司SMARTTMRACE cDNA Amplification Kit的操作步骤将肝脏总RNA合成 5’-RACE- Ready cDNA和3’-RACE- Ready cDNA, 备用。取上述10个组织总RNA分别按照RevertAid First Strand cDNA Synthesis Kit(Fermentas, 美国)试剂盒说明书合成荧光定量分析用cDNA模板, 备用。
1.2.2 赤眼鳟基因克隆
根据NCBI中公布的草鱼(, GenBank: AAA49197.1)和鲫鱼(, GenBank: AB039726.2)的核苷酸序列设计保守区域PCR扩增引物(表1), 并送往上海生工生物工程公司合成。扩增β肌动蛋白基因cDNA核心片段, 反应体系(总体积50 μL): 赤眼鳟肝脏cDNA 2 μL、0.5 μL TaKaRa ExTaq 酶(TaKaRa, 大连)、5 μL 10×Ex Taq缓冲液、2 μL dNTP(10 mmol/L each)、正反引物(10 μmol)各1 μL和38.5 μL 灭菌超纯水。扩增产物直接送上海生工测序。根据克隆得到的赤眼鳟actin的cDNA 核心片段, 设计5’和3’的特异性引物(表1, 上海生工合成), 按照TaKaRa RACE试剂盒说明书进行扩增, 获得基因cDNA 5’端序列和3’端序列。

表1 本实验中用的引物序列
1.2.3 序列分析
运用ExPASy Proteomics Server-ProtParam软件分析赤眼鳟-肌动蛋白基因氨基酸理化性质。利用SOSUI工具和SignalP 3.0分别对-肌动蛋白氨基酸序列进行了信号肽预测。通过软件TMHMM-2.0对-肌动蛋白进行跨膜结构预测。通过MEGA-X软件的邻接法, 设置重复值为1000构建系统进化树。
1.2.4 赤眼鳟actin基因的组织表达
以EF1为内参基因, 对赤眼鳟actin基因进行组织表达分析。根据NCBI中鱼类EF1基因序列和扩增赤眼鳟actin基因序列设计荧光定量引物(表1), 送往上海生工公司合成。用SYBR Green I 染液(TAKARA,China)在CFX96 荧光定PCR仪(Bio–Rad, USA)测得赤眼鳟actin基因在肌肉、肠、皮肤、脾脏、体肾、血液、头肾、心脏、鳃和肝脏的表达量, 共进行5次生物重复, 每个生物重复进行3次技术重复, qPCR 反应体系参照金生振等的文献报道[23]。应用Bio-Rad CFX Manager软件,采用2–ΔΔCt法计算相对表达量, 并用SPSS 19.0软件进行单因素方差分析(One-WayANOVA), 显著性水平为<0.05。
1.2.5 内参基因稳定性分析
利用Excel计算出1.2.4步骤所获得的3次技术重复所得值的几何平均数, 即为CP值, 将CP值输入Best Keeper软件的“CP date input”区域。当CP值输入完毕后, Best Keeper程序将会自动计算好的相关系数()、标准偏差(SD)和变异系数(CV)等数据, 最后根据所得结果推断基因表达稳定性。
2 结果与分析
2.1 赤眼鳟β-actin基因序列分析
赤眼鳟actin基因cDNA全长共1 816 bp(GenBank: MT119965), 开放阅读框(95-1222)1 128 bp, 编码375个氨基酸, 5’端非编码区为94 bp, 3’端非编码区为594 bp, 推算的分子量约为41.75 kDa, 理论等电点5.30。3’端非编码区还含有1个“AATAAA”的mRNA加尾信号(图1)。
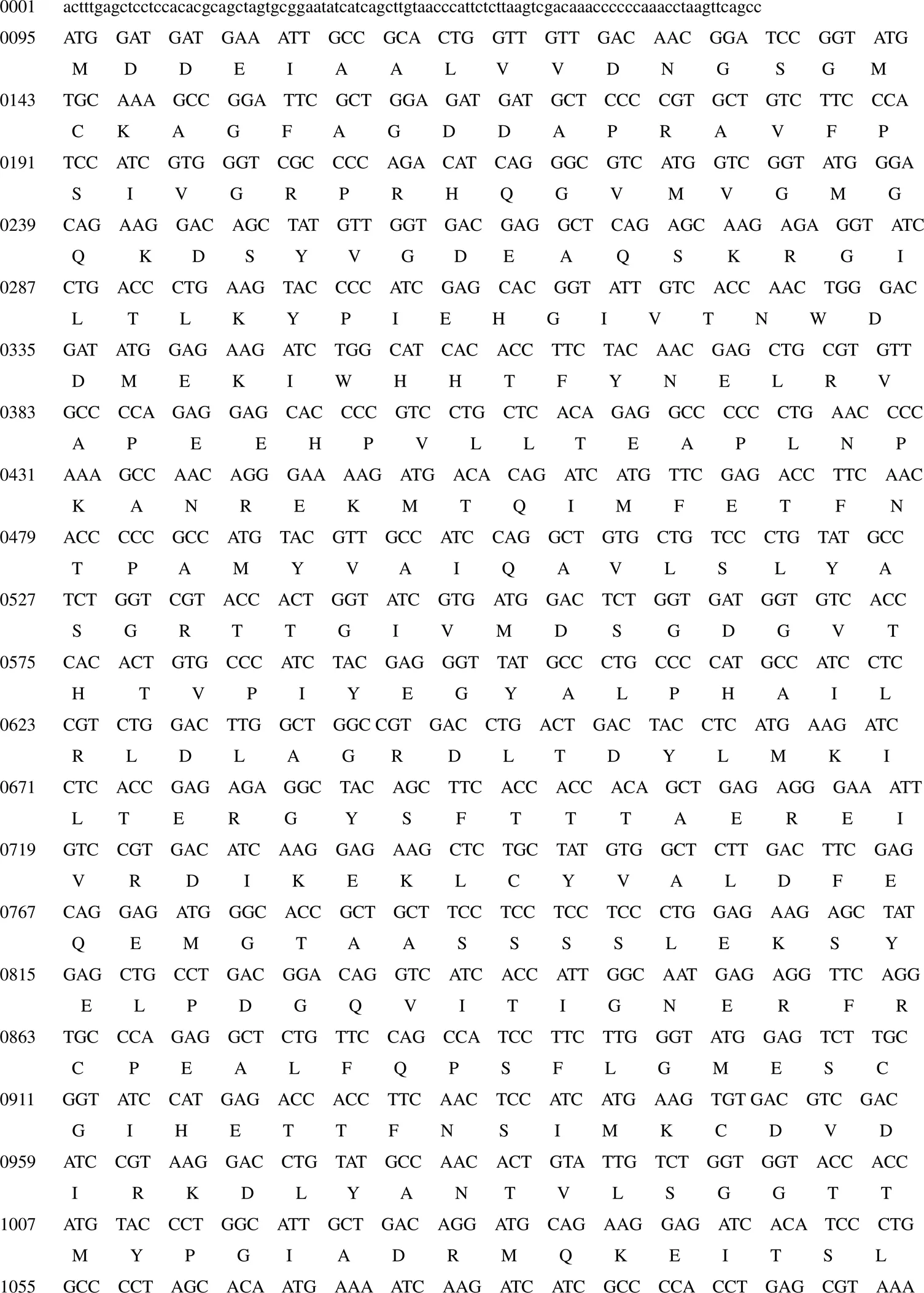
注: 小写字母: 非翻译区; 大写字母: 编码区; 下划线: 多聚腺苷酸加尾信号(AATAAA); *: 终止密码子。
2.2 β-actin氨基酸序列同源性分析和进化树的构建
将赤眼鳟与多个物种的actin氨基酸序列通过NCBI中Protein Blast比对发现, 赤眼鳟与黄颡鱼(, AJE63408.1)、鲫鱼(, BAA92339.2)以及尖头鱥(, AAF63689.1)等的同源性高达99%。通过MEGA-X的邻接法构建系统进化树(图2),actin和分别聚为两大支。本研究克隆的赤眼鳟actin与鲤科鱼类的actin聚为一支, 鲈形目的罗非鱼与石鲷(, ACR56337.1)聚为一支, 软体动物的蜗牛(, AXI69344.1)和三角帆蚌(, ADG266 59.1)聚为一支, 哺乳类大嘴乌鸦(, BAJ07872.1)、家麻雀(, AAR00510.1)和绿猴(, BAA20266.1)聚为一支, 表明本研究克隆所得actin基因分类地位与物种进化地位一致。

图2 NJ系统进化树
2.3 β-actin蛋白的理化性质分析
运用ExPASy Proteomics Server-ProtParam在线服务软件分析赤眼鳟actin氨基酸理化性质表明, 分子式为C1850H2903N491O562S23; 不稳定系数为35.25小于40, 表明该蛋白是稳定的, 总平均亲水性为-0.213, 脂肪系数为81.41。亲水性氨基酸残基包括C、N、Q、S、T和Y, 共105个, 占氨基酸含量的27.9%, 疏水性氨基酸残基共186个, 占氨基酸含量的49.6%, 包括A 、F、G、I、L、M、P、V和W, 由此可得actin蛋白为疏水蛋白。
2.4 β-actin蛋白结构分析
经信号肽预测和蛋白质分析表明该蛋白为无信号肽、非跨膜蛋白。二级结构预测结果表明该蛋白由9个-螺旋和15个片层结构组成-螺旋和-片层结构规则不易变形, 可作为蛋白质骨架起稳定作用[24–25]。对赤眼鳟actin氨基酸序列进行同源三级结构建模,模型构建的氨基酸范围从第2个氨基酸到第375个氨基酸, 其建模参考模板为原鸡肌动蛋白, 目标蛋白与参考蛋白的序列比对同源性为99.20%, 模型评估=0.99 , 模型中可见典型的-螺旋结构(图3)。
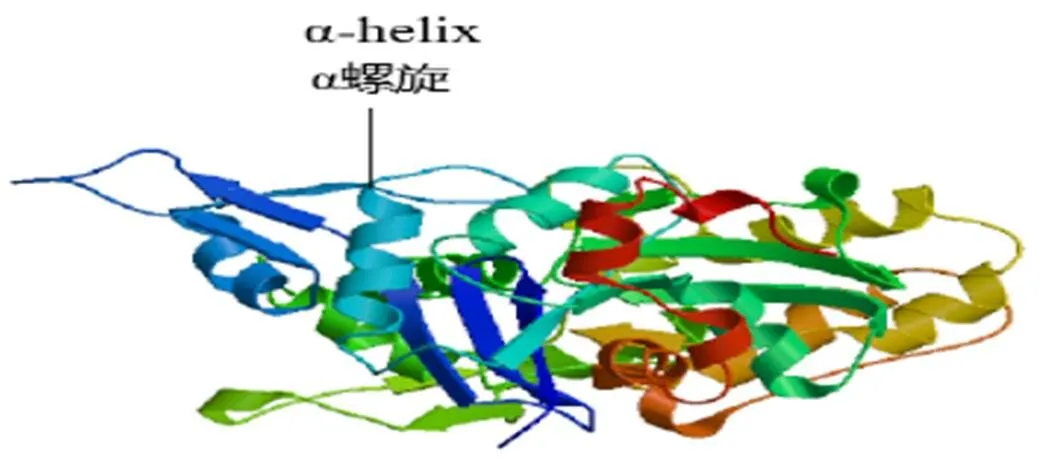
图3 SWISS-MODEL预测赤眼鳟β-肌动蛋白同源三级结构模型
2.5 赤眼鳟β-actin基因组织表达差异
以赤眼鳟EF1基因为参照, 利用qPCR技术测得赤眼鳟actin基因在肌肉、肠、脾脏、皮肤、血液、鳃、体肾、头肾、心脏和肝脏组织中都有表达(图4)。赤眼鳟actin基因在肌肉和肠组织中相对表达量较高, 且显著高于其他8个组织; 肝脏中表达量最低, 且显著低于其他9个组织。

图4 赤眼鳟β-actin基因的组织表达
注: 字母相同为差异不显著, 字母不同为差异显著。
2.6 β-actin的稳定性分析
利用Best Keeper软件计算得到actin与EF1两个基因的CP最小值(min)、CP最大值(max)、相关系数()、标准偏差(SD)和变异系数(CV),actin的三个数值均高于EF1, 相关系数两者相差不大, 但EF1的标准偏差和变异系数均显著小于actin(表2)。根据Best Keeper软件稳定性的判定标准[26], 综合可得, EF1的稳定性高于actin, EF1更适合作为赤眼鳟的内参基因。

表2 Best keeper软件稳定性分析数据
3 讨论
本实验通过RT-PCR技术和RACE法克隆得到赤眼鳟actin基因cDNA序列, 该基因cDNA全长共1 816 bp, 由长94 bp的5’端非编码区, 长594 bp的3’端非编码区和长1 128 bp的开放阅读框(95-1222)组成, 阅读框共编码375个氨基酸。与NCBI上登陆的鲢鱼(, AAG17452.1)、绿猴 (, BAA20266.1)、泥鳅(, AAG48576.1)和红原鸡(, AAA98527.1)等的actin氨基酸序列对比时发现,actin的阅读框编码的氨基酸均在375个左右, 与胡松年等[27]在多种真核生物中综合分析所得出结论一致。
近年来有关actin基因的研究表明, 该基因在不同生物体内组织表达存在差异。一方面, 作为公认的内参基因,actin基因在一些物种中的组织表达水平是一致的, 如宽体沙鳅()的actin基因在检测的11个组织都有表达, 且表达量一致[28]; 三角帆蚌的actin基因在检测的8个组织的表达量基本一致, 较稳定[5]。另一方面actin基因在不同组织内表达量存在明显差异, 如大鳞副泥鳅()[8]actin基因在卵巢内有较高的表达, 肠表达量较低;actin基因在拟穴青蟹肌肉组织中的表达量最高, 在结缔组织中的表达量最低[29]。本研究赤眼鳟actin基因在各组织中均有表达, 但各组织内的表达存在差异, 在肌肉、肠中相对表达量较高。肠的主要组成结构是平滑肌, 肌肉组织更是由大量的肌细胞组成, 因此actin基因可能在赤眼鳟体内主要发挥支持细胞运动的功能。
实时荧光定量PCR技术(qPCR)因其定量准确、实时监测以及自动化高等优点而被广泛运用于生物基因表达分析、医学诊断和环境监测等方面[30]。actin基因作为管家基因的一员, 在生物体内表达广泛且稳定, 具高度保守性, 在许多水产动物的分子生物学研究中都用作参照基因, 如鳜鱼()[31]等。本研究采用常用的稳定性分析软件Bestkeeper对赤眼鳟EF1基因和赤眼鳟actin基因进行了稳定性分析, 得出了CV、SD和等数据,actin基因的相关系数虽然较EF1基因高, 但标准偏差和变异系数均远大于EF1基因, 综合得actin基因的稳定性不如EF1基因。在本次检测中赤眼鳟体内的两个基因的标准偏差均大于1, 说明两个基因均不稳定, 作为内参基因时可能存在一定误差。本研究的actin基因和EF1基因的稳定性分析具有一定的限制性, 可能在不同生物体内, 以及不同的外界刺激和生理条件下稳定性会发生改变, 因此赤眼鳟的最佳内参基因的筛选存在一定难度。
4 结论
本研究首次克隆得赤眼鳟actin基因cDNA全长, 并探究了其序列特征及其表达特点。该基因全长共1 816 bp; 且氨基酸同源性分析发现鲤科鱼类亲缘关系近, 与哺乳动物亲缘关系较远; 在肌肉、肠组织内表达量最高; 表达稳定性不如赤眼鳟EF1基因, 因此EF1基因更适合作为赤眼鳟的内参基因。以上结果为筛选赤眼鳟其他基因量化、分子研究的内参基因提供了actin基因和EF1基因的基础数据。
[1] Welch M D, Holtzman D A, Drubin D G. The yeast actin cytoskeleton [J]. Curr Opin Cell Biol, 1994, 6(1): 110–119.
[2] Rubenstein P A. The functional importance of multiple actin isoforms [J]. BioEssays, 1990, 12(7): 309–315.
[3] Cadoret J P, Debon R, Cornudella L, et al. Transient expression assays with the proximal promoter of a newly characterized actin gene from the oyster Crassostrea gigas [J]. FEBS Lett, 1999, 460(1): 81–85.
[4] Vandekerckhove J, Weber K. Mammalian cytoplasmic actins are the products of at least two genes and differ in primary structure in at least 25 identified positions from skeletal muscle actins [J]. Proc Natl Acad Sci U S A, 1978, 75(3): 1 106–1 110.
[5] 袁一鸣, 李家乐, 汪桂玲, 等. 三角帆蚌β-肌动蛋白基因的cDNA全长克隆及表达分析[J]. 水产学报, 2010, 34(6): 871–880.
[6] 林群, 梁旭方, 王琳, 等. 近江牡蛎等7种养殖鱼虾贝类参照基因β-肌动蛋白cDNA序列的克隆与比较分析[J]. 生态毒理学报, 2008(3): 256–261.
[7] 周瑞雪, 蒙涛, 赵发兰, 等. 草鱼MYH mRNA表达量分析中采用的内参基因稳定性比较[J]. 基因组学与应用生物学, 2009, 28(5): 896–900.
[8] 刘士力, 王雨辰, 张德华, 等. 大鳞副泥鳅β-肌动蛋白基因的cDNA全长克隆及表达分析[J]. 中国农学通报, 2013, 29(35): 96–101.
[9] 薛茂云, 胡承俊, 张营, 等. 松江鲈β-肌动蛋白基因全长cDNA的克隆和序列分析(英文)[J]. Agricultural Science & Technology, 2010, 11(6): 121–124.
[10] 张殿昌, 江世贵, 邵艳卿, 等. 鲮β-肌动蛋白基因的分子克隆及其作为分子内标的可靠性分析[J]. 中国水产科学, 2006(5): 708–713.
[11] Zhong H, Simons J W. Direct comparison of GAPDH, beta-actin, cyclophilin, and 28S rRNA as internal standards for quantifying RNA levels under hypoxia [J]. Biochem Biophys Res Commun, 1999, 259(3): 523–526.
[12] 王欣欣, 孙宝剑, 昌鸣先, 等. 草鱼免疫球蛋白M重链基因的克隆及表达[J]. 水产学报, 2008(1):13–20.
[13] 丁鉴锋, 张国范, 李莉. 皱纹盘鲍一种丝氨酸蛋白酶抑制因子克隆与表达分析[J]. 水产科学, 2011, 30(12): 731–738.
[14] 龙治海, 陈虎, 汪斌, 等. 达氏鲟内参基因β-actin、GAPDH和EF1-α的克隆及表达稳定性[J]. 农业生物技术学报, 2018, 26(11): 1 846–1 855.
[15] Sarmiento J, Leal S, Quezada C, et al. Environmental acclimatization of the carp modulates the transcription of beta-actin [J]. J Cell Biochem, 2000, 80(2): 223–228.
[16] 赖呈纯, 潘红, 张静, 等. 葡萄不同摘心处理下qRT-PCR内参基因的筛选与验证[J]. 江西农业大学学报, 2019, 41(5): 890–900.
[17] 王彦平, 朱荣生, 王怀中, 等. 大蒲莲猪不同发育阶段组织RT-qPCR分析中适宜内参基因筛选[J]. 华北农学报, 2018, 33(S1): 45–53.
[18] 杨明生, 陈金安, 黄孝湘, 等. 赤眼鳟繁殖生物学研究[J]. 淡水渔业, 2005(3): 38–40.
[19] 刘巧林, 肖调义, 刘敏, 等. 赤眼鳟生物学研究进展[J]. 水产科学, 2012, 31(11): 687–691.
[20] 李策, 李新辉, 李跃飞, 等. 基于单位补充量模型的西江赤眼鳟种群资源利用现状评价[J]. 中国水产科学, 2019, 26(1): 151–160.
[21] 陈开健, 王静安, 刘巧林, 等. 赤眼鳟TRAF6基因的cDNA克隆及其应对GCRV的免疫表达特性[J]. 湖南农业大学学报(自然科学版), 2016, 42(6): 641–646.
[22] Wang R, Li Y, Zhou Z, et al. Involvement of interferon regulatory factor 3 from the barbel chub Squaliobarbus curriculus in the immune response against grass carp reovirus [J]. Gene, 2018, 648: 5–11.
[23] 金生振, 王荣华, 周智愚, 等. 赤眼鳟Toll样受体9基因的cDNA全长克隆及表达特性分析[J]. 湖南农业大学学报(自然科学版), 2017(3): 304–309.
[24] 吕凤林, 朱锡华. 人C5aR(CD88)序列结构分析及其B细胞表位预测[J]. 免疫学杂志, 1998(03): 15–18.
[25] 宋建勋, 朱锡华, 陈克敏. 人Fas抗原表位预测[J]. 免疫学杂志, 1999(1): 18–20.
[26] 吴建阳, 何冰, 杜玉洁, 等. 利用geNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法[J]. 现代农业科技, 2017(5): 278–281.
[27] 胡松年, 阎隆飞. 肌动蛋白与真核生物的进化[J]. 动物学报, 1999(4): 440–443.
[28] 覃川杰, 陈立侨, 岳兴建, 等. 宽体沙鳅(Botia reevesae)β-肌动蛋白基因的cDNA克隆与表达分析[J]. 海洋与湖沼, 2013, 44(2): 396–402.
[29] 徐真, 马洪雨, 马春艳, 等. 拟穴青蟹β-肌动蛋白基因的克隆、组织表达及作为内参的可靠性分析[J]. 生物技术通报, 2013(8): 105–112.
[30] 梁子英, 刘芳. 实时荧光定量PCR技术及其应用研究进展[J]. 现代农业科技, 2020(6): 1–3.
[31] 端金霞. 鳜鱼等5种淡水鱼β-肌动蛋白基因克隆与结构功能分析研究[D]. 广州: 暨南大学, 2006.
Cloning, expression and stability analysis ofgene in squaliobarbus curriculus
Chen Lei, Chen Shuyi, Zhou Gang, Huang Yuhong, Li Wei, Wang Jing’an, Xiao Tiaoyi, Liu Qiaolin
(Hunan Engineering Technology Research Center of Featured Aquatic Resources Utilization, Hunan Agricultural University, Changsha 410128, China)
To investigate the structure and expression characteristics of beta-actin (actin) gene in,the full length sequence ofcDNA is cloned from the liver by the way of RT-PCR and RACE (GenBank: MT119965). The full length is 1816 bp, which consists of a 94 bp 5’ untranslated region (UTR), a 594 bp 3’ UTR and an 1128 bp open reading frame (95-1222). And there are 375 amino acids in the translated protein. The phylogenetic tree shows thatactin ofgathers into one branch with Cyprinidae. And sequence analysis reveals that the homology ofactin amino acids with,andand other species is 99%, which suggestesactin gene is highly conserved in fish. The expression analysis of qPCR shows thatactin is expressed in 10 tissues including muscle, intestine, spleen, skin, blood, gill, body kidney, head kidney, heart and liver, but the expression level ofactin is different among these tissues. The stability of the EF1 (elongation factor 1) andactin inis compared by the Best Keeper software. EF1 gene is more stable.The above research provides evidence for the evolution of the molecular system and a reference for the study of the gene expression of.
;actin; gene clone; tissue expression; stability
Q 785
A
1672–6146(2020)03–0036–07
10.3969/j.issn.1672–6146.2020.03.007
刘巧林, qiaolinliu2017@hunau.edu.cn。
2020–5–10
国家自然科学基金青年项目(31402289); 湖南省自然科学基金青年项目(2018JJ3221); 湖南农业大学双一流建设项目(SYL201802020)。
(责任编校: 郭冬生)
