RNA甲基化转移酶在肺腺癌中的表达及意义
2020-07-04赵琦相绿竹彭瑞王晔牟晓峰
赵琦 相绿竹 彭瑞 王晔 牟晓峰
[摘要]目的 比較人肺腺癌细胞系A549和正常人肺上皮细胞系BEAS-2b、肺腺癌和癌旁组织中RNA甲基化转移酶的表达。方法 分别采用实时荧光定量聚合酶链反应(RT-qPCR)和蛋白免疫印迹(Western blot)方法检测A549和BEAS-2b细胞中RNA甲基化转移酶mRNA及蛋白的表达水平;采用免疫组化法(IHC)检测肺腺癌与癌旁组织中RNA甲基化转移酶的表达。结果RT-qPCR检测结果显示,与BEAS-2b细胞相比,A549细胞中甲基化转移酶样蛋白3(METTL3)mRNA的表达水平显著上升(t=5.708,P<0.05),而甲基化转移酶样蛋白14(METTL14)和肾母细胞瘤1相关蛋白(WTAP)mRNA的表达水平显著下降(t=24.080、9.207,P<0.05)。Wes-tern blot检测显示,与BEAS-2b细胞相比,A549细胞中METTL3、METTL14蛋白的表达水平均显著上升(t=6.502、6.869,P<0.05),而WTAP蛋白的表达水平显著下降(t=8.822,P<0.05)。IHC检测显示,METTL3、METTL14定位于细胞核内,肺腺癌组织中二者的表达水平较癌旁组织显著升高(t=3.055、4.339,P<0.05);WTAP定位于细胞核和细胞质内,肺腺癌组织中其表达水平较癌旁组织显著下降(t=3.611,P<0.05)。结论A549细胞和肺腺癌组织中RNA甲基化转移酶的表达与BEAS-2b细胞及癌旁组织相比有明显差异,提示可能存在某些依赖于N6-甲基腺嘌呤(m6A)修饰的机制影响肺腺癌的发生发展。
[关键词] 肺肿瘤;腺癌;tRNA甲基转移酶类
[中图分类号] R734.2[文献标志码] A[文章编号] 2096-5532(2020)04-0394-05
doi:10.11712/jms.2096-5532.2020.56.069[HT]
[网络出版] http://kns.cnki.net/kcms/detail/37.1517.R.20200417.1001.019.html;2020-04-18 16:17
EXPRESSION AND SIGNIFICANCE OF RNA METHYLTRANSFERASE IN LUNG ADENOCARCINOMA
ZHAO Qi, XIANG Lüzhu, PENG Rui, WANG Ye, MU Xiaofeng
(Department of Medicine, Qingdao University, Qingdao 266071, China)
[ABSTRACT]Objective To compare the expression of RNA methyltransferase in human lung adenocarcinoma cell line A549 and normal human lung epithelial cell line BEAS-2b, as well as in lung adenocarcinoma and adjacent tissues. Methods Real-time quantitative polymerase chain reaction (RT-qPCR) and Western blot were used to determine the expression levels of RNA methyltransferase mRNA and protein, respectively, in A549 and BEAS-2b cells. Immunohistochemistry (IHC) was used to localize and determine the expression of RNA methyltransferase in lung adenocarcinoma and adjacent tissues. Results RT-qPCR results showed that compared with BEAS-2b cells, A549 cells had a significantly higher mRNA expression level of methyltransferase-like protein 3 (METTL3) (t=5.708,P<0.05) and significantly lower mRNA expression levels of methyltransferase-like protein 14 (METTL14) and Wilms tumor 1-associating protein (WTAP) (t=24.080,9.207;P<0.05). Western blot results showed that compared with BEAS-2b cells, A549 cells had significantly higher protein expression levels of METTL3 and METTL14 (t=6.502,6.869;P<0.05) and a significantly lower protein expression level of WTAP (t=8.822,P<0.05). IHC results showed that METTL3 and METTL14 were localized in the nucleus, and their expression in lung adenocarcinoma tissues was signi-
ficantly higher than that in adjacent tissues (t=3.055,4.339;P<0.05); WTAP was localized in the nucleus and cytoplasm, and its expression in lung adenocarcinoma tissues was significantly lower than that in adjacent tissues (t=3.611,P<0.05). Conclusion Compared with BEAS-2b cells and adjacent tissues, A549 cells and lung adenocarcinoma tissues have significantly different RNA methyltransferase expression, suggesting that there may be some mechanisms that rely on N6-methyladenosine modification affec-ting the development and progression of lung adenocarcinoma.
[KEY WORDS] lung neoplasms; adenocarcinoma; tRNA methyltransferases
中国的癌症发病率和死亡率逐年上升,据统计,2015年中国约有429.2万例癌症新发病例和281.4万例癌症死亡病例,肺癌是其中最为常见的一种癌症,也是癌症死亡的首要原因[1]。肺腺癌占所有肺癌的80%左右,且大多数肺腺癌病人在确诊时已处于晚期阶段,病人术后5年生存率较低[2]。目前,肺腺癌治疗方式有手术、放化疗以及靶向治疗等,靶向治疗以其精准、高效的特点在癌症治疗中起到了越来越大的作用[3]。因此,发现并筛选新的肺腺癌有效的预后标志物和潜在的治疗靶点是目前研究的重要临床课题之一。
N6-甲基腺嘌呤(m6A)修饰是真核细胞mRNA及非编码RNA中最为丰富的表观遗传学修饰[4-5],而且该修饰过程是动态、可逆的。目前普遍认为,m6A修饰受3种蛋白调控,包括“读写器”、“消除器”及“阅读器”。其中,“读写器”即“writers”,指RNA甲基化转移酶,主要包括甲基化转移酶样蛋白3(METTL3)、甲基化转移酶样蛋白14(METTL14)以及肾母细胞瘤1相关蛋白(WTAP),它们的主要作用为促进RNA的m6A修饰过程[6]。METTL3与METTL14具有43%的同源性,两者常以复合体的形式发挥作用,其中METTL3主要作为催化核心,而METTL14主要作为RNA结合平台发挥作用[7]。WTAP可与METTL3-METTL14复合物相互作用,影响细胞内m6A的沉积[8]。既往研究发现,RNA甲基化转移酶与肿瘤的發生发展密切相关,如神经胶质瘤、胃癌、结直肠癌、乳癌、肝癌、膀胱癌等[9-14]。但这3种RNA甲基化转移酶在肺癌中的作用机制还不十分清楚。因此,本研究采用多种方法,比较人肺腺癌细胞及正常人肺上皮细胞、人肺腺癌组织和癌旁组织中RNA甲基化转移酶的表达差异,为探究m6A甲基化在肺癌发生中的作用机制提供实验基础。
1 材料和方法
1.1 实验材料
1.1.1 细胞与组织 人肺腺癌细胞株A549和人正常肺上皮细胞株BEAS-2b均购自上海歌凡生物科技有限公司。10对肺腺癌与癌旁组织(距离癌组织2 cm)标本均来自青岛市中心医院病理科,取材自2016—2018年青岛市中心医院的肺癌手术病人。10例病人中,男6例,女4例;病人年龄54~64岁,平均58.7岁;肿瘤位于右肺6例,位于左肺4例;分化程度为中高分化3例,低分化7例;肿瘤最大径为2~10 cm,平均4.7 cm;TNM分期为1A期2例,1B期1例,2B期1例,3A期5例,3B期1例。
1.1.2 主要试剂与仪器 胎牛血清、RPMI 1640及DMEM培养基均购自美国Sigma公司;扩增用ETTL3、METTL14、WTAP引物以及Trizol、反转录试剂盒均购自英潍捷基(上海)贸易有限公司;[LL]实时荧光定量聚合酶链反应(RT-qPCR)检测试剂盒(SYBRSelect Master Mix)购自美国ABI公司;细胞裂解液RIPA、BCA蛋白检测试剂盒、磷酸盐缓冲液(PBS)及牛血清清蛋白(BSA)、二氨基联苯胺(DAB)试剂盒、苏木精染液均购自北京索莱宝科技有限公司;METTL3、METTL14、WTAP、GAPDH一抗和辣根过氧化物酶标记IgG二抗购自美国Abcam公司。荧光定量PCR仪购自美国ABI公司;电泳仪购自美国伯乐公司;光学显微镜购自德国蔡司公司;多功能分子成像系统购自美国Azure Biosystems公司。
1.2 实验方法
1.2.1 细胞培养 A549、BEAS-2b细胞分别在含有体积分数0.10胎牛血清和体积分数0.01青霉素-链霉素的RPMI 1640、DMEM培养液中培养。将处于指数生长期的细胞制成细胞悬液,以每孔6×105个细胞的密度接种于6孔板中,待细胞融合度约为90%时分别用Trizol和RIPA裂解液收取细胞。
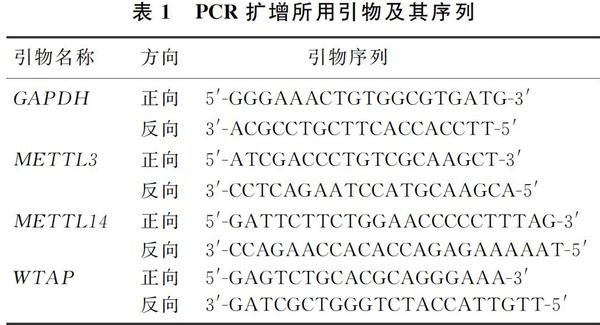
1.2.2 RT-qPCR检测RNA甲基化转移酶mRNA的表达 将Trizol法提取的A549、BEAS-2b细胞总RNA反转录为cDNA。以cDNA作为模板,分别应用METTL3、METTL14、WTAP特异性引物进行扩增,以GAPDH为内参。反应体系10 μL,包括:1∶14稀释的cDNA模板4.6 μL,上、下游引物各0.2 μL,Master Mix 5.0 μL。每个样本设3个复孔。反应条件:95 ℃热启动10 min;95 ℃、15 s,60 ℃、1 min(收集荧光信号),40个循环。反应完成后根据Ct值计算目的基因的相对表达量。PCR扩增所用引物及其序列见表1。
1.2.3 蛋白免疫印迹(Western blot)检测RNA甲基化转移酶蛋白的表达 已处理的细胞加入RIPA裂解液和蛋白酶抑制剂提取总蛋白,采用BCA法测定蛋白浓度。在120 g/L的聚丙烯酰胺凝胶中电泳后,于冰上转至PVDF膜上。使用50 g/L脱脂奶粉室温封闭1 h,加入兔抗人或鼠抗人的METTL3、METTL14、WTAP一抗(稀释度均为1∶1 000),4 ℃孵育过夜。加入辣根过氧化物酶标记的羊抗兔或羊抗鼠二抗(稀释度均为1∶3 000),再加入GAPDH直标抗体(稀释度为1∶5 000),室温孵育1 h,洗膜后显影成像。以GAPDH为内参照,计算METTL3、METTL14、WTAP的相对表达量。
1.2.4 免疫组化法(IHC)检测肺腺癌及癌旁组织中RNA甲基化转移酶的表达 将10对肺腺癌与癌旁组织石蜡切片脱蜡至水,抗原修复,放入体积分数0.03的过氧化氢溶液中室温避光孵育25 min,应用PBS(pH值7.4)洗涤。以30 g/L BSA室温封闭30 min,加按比例配好的一抗4 ℃孵育过夜;洗涤后加入与一抗相应种属的二抗室温孵育50 min;DAB显色,苏木精复染细胞核。放入乙醇及二甲苯中脱水晾干,中性树胶封片。光学显微镜下观察,进行图像采集分析。以细胞核深棕色为强阳性,棕黄色为中度阳性,浅黄色为弱阳性,蓝色为阴性。对每个组织点识别分析出强阳性、中度阳性、弱阳性及阴性的细胞数量、百分比,进行H-score评分。H-score=∑(PI×I),其中PI为阳性细胞数占切片中所有细胞数的百分比,I为着色强度。
1.3 统计学分析
采用Graph Pad Prism 5软件自带的统计学分析模块进行统计学处理。计量资料以[AKx-D]±s的形式表示,两组之间比较采用t检验,以P<0.05为差异有统计学意义。
2 结果
2.1 RNA甲基化转移酶mRNA在BEAS-2b以及A549细胞中的表达比较
RT-qPCR检测结果显示,与正常肺上皮细胞BEAS-2b相比较,肺腺癌细胞A549中METTL3 mRNA的表达水平显著上升(t=5.708,P<0.05),METTL14和WTAP mRNA的表達水平显著下降(t=24.080、9.207,P<0.05)。见表2。
2.2 RNA甲基化转移酶蛋白在BEAS-2b及A549细胞中的表达比较
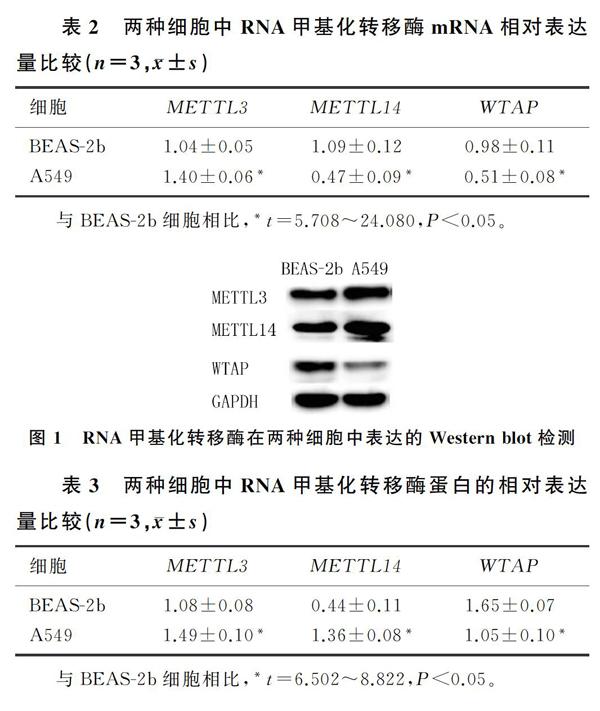
Western blot检测结果显示,与正常肺上皮细胞BEAS-2b相比,肺腺癌细胞A549中METTL3、METTL14蛋白的表达水平均显著上升(t=6.502、6.869,P<0.05),而WTAP蛋白的表达水平则显著下降(t=8.822,P<0.05),差异均有统计学意义。见图1、表3。
2.3 RNA甲基化转移酶在人肺腺癌及癌旁组织中的表达比较
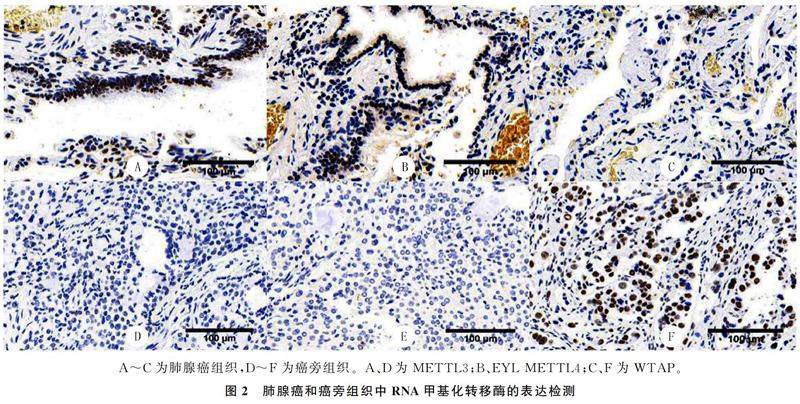
IHC检测显示,METTL3、METTL14定位于细胞核内,肺腺癌组织中二者的表达水平较癌旁组织显著升高(t=3.055、4.339,P<0.05);WTAP定位于细胞核和细胞质内,肺腺癌组织中其表达水平较癌旁组织下降,差异有统计学意义(t=3.611,P<0.05)。见图2、表4。
3 讨论
已有研究结果表明,RNA的m6A修饰在多种生物学过程中发挥着重要作用,包括干细胞的自我更新和分化、组织发育、热休克反应、昼夜节律控制、DNA损伤修复等[15-17]。除此之外,m6A修饰还可以影响mRNA的稳定性、剪接、转运、定位、翻译以及微小RNA(miRNA)加工和RNA与蛋白质间的相互作用[18-22]。RNA甲基化转移酶作为重要的m6A修饰蛋白在多种肿瘤的发生发展中起着重要作用。已有研究证实,RNA甲基化转移酶可以作为“促癌基因”发挥促进肿瘤细胞增殖和转移的作用,例如,METTL3在胃癌表达明显升高,可促进mym型锌指蛋白1(ZMYM1)mRNA的m6A修饰,增强其稳定性,进而促进ZMYM1相关复合物对E-cadherin的转录抑制,从而促进胃癌细胞上皮间质转化和转移[10];WTAP在急性髓细胞白血病(AML)表达上调,发挥着类似“原癌基因”的作用[23];WTAP在膀胱癌组织表达明显上升,有望成为潜在的治疗靶点[24]。此外,还有研究认为,RNA甲基化转移酶在部分癌症中发挥“抑癌基因”的作用,例如,上调METTL3能明显抑制肾细胞癌细胞增殖、迁移和侵袭等,并诱导G0/G1期阻滞,这可能是通过PI3K-Akt-mTOR通路来实现的[25];而METTL14在肝癌中通过与DGCR8相互作用,能上调microRNA-126的表达,从而抑制肿瘤细胞转移[13]。
RNA甲基化转移酶在肺腺癌中的作用尚不明确,因此本研究比较了人肺腺癌细胞和正常人肺上皮细胞、人肺腺癌组织和癌旁组织中RNA甲基化转移酶的表达变化。结果显示,与正常人肺上皮细胞及癌旁组织相比,人肺腺癌细胞及肺腺癌组织中METTL3的mRNA及蛋白表达水平均显著上升;METTL14蛋白水平在肺腺癌组织中的表达同样显著升高,但mRNA表达水平下降。METTL14蛋白和mRNA表达不一致,分析原因可能为:mRNA虽表达下降,但因某种原因稳定性增加,导致降解减少,进一步导致蛋白表达增加;或者是METTL14蛋白降解减少,蛋白表达量增加对mRNA的表达产生阻遏,使mRNA表达减少。其具体机制尚不清楚,这是我们下一步探讨的重点问题之一。但作为酶类,METTL14发挥生物学作用主要在蛋白水平,因此我们认为蛋白水平的结果更有意义。另外,作为METTL3-METTL14复合体的重要的协同蛋白,不论在mRNA水平还是在蛋白水平,WTAP的表达均明显下降。WTAP表达下降的可能原因之一是,在肺腺癌中,其RNA m6A修饰并不主要是METTL3-METTL14复合体介导的,METTL3和METTL14可能单独作用介导RNA m6A的催化作用;另一原因可能为,在肺腺癌组织中存在另外一种METTL3-METTL14复合体的协同蛋白,该蛋白与WTAP相互竞争,共同协助复合体的催化作用,具体蛋白类别还需进一步研究。
从本文研究结果结合RNA m6A的广泛生物学功能来看,一定存在某些依赖于m6A修饰的机制影响着肺腺癌的生物学过程。需要进一步结合m6A高通量测序等一系列手段挖掘潜在的结合靶点,这也是我们下一步将要进行的研究工作。
[参考文献]
[1]CHEN W, ZHENG R, BAADE P D, et al. Cancer statistics in China[J]. A Cancer Journal for Clinicians, 2016,66(2):115-132.
[2]PAZ-ARES L, TAN E H, OBYRNE K, et al. Afatinib versus gefitinib in patients with EGFR mutation-positive advanced non-small-cell lung cancer: overall survival data from the phase Ⅱb LUX-Lung 7 trial[J]. Annals of Oncology, 2017,28(2):270-277.
[3]NUKAGA S, YASUDA H, TSUCHIHARA K, et al. Ampli-fication of EGFR wild-type alleles in non-small cell lung cancer cells confers acquired resistance to mutation-selective EGFR tyrosine kinase inhibitors[J]. Cancer Research, 2017,77(8):2078-2089.
[4]ROUNDTREE I A, EVANS M E, PAN T, et al. Dynamic RNA modifications in gene expression regulation[J]. Cell, 2017,169(7):1187-1200.
[5]LIN Xinyao, CHAI Guoshi, WU Yingmin, et al. RNA m6A methylation regulates the epithelial mesenchymal transition of cancer cells and translation of Snail[J]. Nature Communications, 2019,10(1):2065-2077.
[6]LAN Q, LIU P Y, HAASE J, et al. The critical role of RNA m6A methylation in cancer[J]. Cancer Research, 2019,79(7):1285-1292.
[7]BUJNICKI J M, FEDER M, RADLINSKA M, et al. Structure prediction and phylogenetic analysis of a functionally diverse family of proteins homologous to the MT-A70 subunit of the human mRNA: m6A methyltransferase[J]. Journal of Molecular Evolution, 2002,55(4):431-444.
[8]LIU Jianzhao, YUE Yanan, HAN Dali, et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation[J]. Nature Chemical Biology, 2014,10(2):93-95.
[9]CUI Qi, SHI Hailing, YE Peng, et al. m6A RNA methylation regulates the self-renewal and tumorigenesis of glioblastoma stem cells[J]. Cell Reports, 2017,18(11):2622-2634.
[10]YUE Ben, SONG Chenlong, YANG Linxi, et al. METTL3-mediated N6-methyladenosine modification is critical for epithelial-mesenchymal transition and metastasis of gastric cancer[J]. Molecular Cancer, 2019,18(1):142.
[11]PENG W, LI J, CHEN R, et al. Upregulated METTL3 promotes metastasis of colorectal cancer via miR-1246/SPRED2/MAPK signaling pathway[J]. Journal of Experimental & Cli-
nical Cancer Research: CR, 2019,38(1):393.
[12]LIU Liwen, LIU Xin, DONG Zihui, et al. N6-methyladenosine-related genomic targets are altered in breast cancer tissue and associated with poor survival[J]. Journal of Cancer, 2019,10(22):5447-5459.
[13]MA Jinzhao, YANG Fu, ZHOU Chuanchuan, et al. METTL14 suppresses the metastatic potential of hepatocellular carcinoma by modulating N6-methyladenosine-dependent primary MicroRNA processing[J]. Hepatology (Baltimore, Md.), 2017,65(2):529-543.
[14]HAN Jie, WANG Jingzi, YANG Xiao, et al. METTL3 promote tumor proliferation of bladder cancer by accelerating pri-miR221/222 maturation in m6A-dependent manner[J]. Mole-cular Cancer, 2019,18(1):110.
[15]XIANG Y, LAURENT B, HSU C H, et al. RNA m(6)A methylation regulates the ultraviolet-induced DNA damage response[J]. Nature, 2017,543(7646):573-576.
[16]WANG Y, LI Y, TOTH J I, et al. N6-methyladenosine modification destabilizes developmental regulators in embryonic stem cells[J]. Nature Cell Biology, 2014,16(2):191-198.
[17]WANG X, LU Z K, GOMEZ A, et al. N6-methyladenosine-dependent regulation of messenger RNA stability[J]. Nature, 2014,505(7481):117-120.
[18]WANG X, ZHAO B S, ROUNDTREE I A, et al. N(6)-methyladenosine modulates messenger RNA translation efficiency[J]. Cell, 2015,161(6):1388-1399.
[19]CHEN T, HAO Y J, ZHANG Y, et al. m(6)A RNA methy-
lation is regulated by microRNAs and promotes reprogramming to pluripotency[J]. Cell Stem Cell, 2015,16(3):289-301.
[20]LIU Nian, DAI Qing, ZHENG Guanqun, et al. N(6)-methyladenosine-dependent RNA structural switches regulate RNA-protein interactions[J]. Nature, 2015,518(7540):560-564.
[21]ALARCN C R, LEE H, GOODARZI H, et al. N6-methyladenosine marks primary microRNAs for processing[J]. Nature, 2015,519(7544):482-485.
[22]ZHOU Jun, WAN Ji, GAO Xiangwei, et al. Dynamic m(6)A mRNA methylation directs translational control of heat shock response[J]. Nature, 2015,526(7574):591-594.
[23]BANSAL H, YIHUA Q, IYER S P, et al. WTAP is a novel oncogenic protein in acute myeloid leukemia[J]. Leukemia, 2014,28(5):1171-1174.
[24]CHEN Lezhong, WANG Xinghuan. Relationship between the genetic expression of WTAP and bladder cancer and patient prognosis[J]. Oncology Letters, 2018,16(6):6966-6970.
[25]LI Xiao, TANG Jingyuan, HUANG Wen, et al. The M6A methyltransferase METTL3: acting as a tumor suppressor in renal cell carcinoma[J]. Oncotarget, 2017,8(56):96103-96116.
(本文編辑 马伟平)
[收稿日期]2019-09-04; [修订日期]2020-03-19
[基金项目]国家自然科学基金面上项目(81670822);青岛市医疗卫生B类重点学科(青卫科教字〔2019〕9号)
[第一作者]赵琦(1994-),女,硕士研究生。
[通信作者]牟晓峰(1970-),男,硕士,主任技师,硕士生导师。E-mail:muxiaofeng2005@126.com。
