基于16S rDNA 高通量测序技术分析水牛原料乳中细菌的多样性
2020-07-02谢芳李孟伟唐振华彭开屏彭丽娟梁辛杨承剑
谢芳,李孟伟,唐振华,彭开屏,彭丽娟,梁辛,杨承剑
(中国农业科学院广西水牛研究所,南宁530001)
0 引 言
我国水牛资源丰富,主要集中在广西、云南、贵州、四川等地,水牛具有较强的抗病和免疫力,迄今尚未有水牛疯牛病例报道[1]。水牛奶作为我国南方地区的一种特色优势乳品,几乎含有人体所需营养元素及微量元素,在民间享有“奶中之王”的美誉[2],水牛奶乳化特性好,胆固醇低,酪蛋白含量高,深加工优势明显。而作为生产乳品的基础,原料乳的品质备受关注,其微生物含量又是衡量乳品质量的关键指标,故了解原料乳中微生物多样性至关重要。
近年来,随着分子生物技术的发展,高通量测序技术作为“下一代”测序技术已迅速发展,研究者们已将高通量测序技术应用于各个领域,包括海洋、土壤、植物和肠道环境,而利用高通量测序对乳品进行研究的报道较少, 人们对原料乳中微生物的组成也没有很好的描述,然有关水牛乳微生物多样性的研究也尚未见报道[3],研究人员更多的是采用纯培养方法,根据菌落的生长形态和生理生化实验来确定其微生物的种类和数量。谢芳[4]等采用传统培养的方法,从三品杂交水牛乳中分离出一百多株乳酸菌,并对其进行了测序鉴定。由此对水牛乳中可培养乳酸菌的多样性有了初步了解,但仅通过这种传统培养技术去认识水牛乳中微生物的多样性是远远不够的。
因此,本研究将采用16S rDNA 高通量测序技术,分别对广西水牛研究所种畜场存栏的纯种印度摩拉水牛、巴基斯坦尼里/拉菲水牛以及三品杂水牛的原料乳进行分析,拟借助生物学方法对原料乳的序列数据进行统计,以期更全面解析水牛原料乳中细菌的多样性,比较不同品种水牛乳中微生物群落结构及其组成差异,旨为后续乳品加工、乳品安全以及菌种发掘等提供详实的微生物学背景资料。
1 材料与方法
1.1 样本采集
原料乳样本采集自广西水牛研究所水牛种畜场,分别采自纯种印度摩拉水牛、纯种巴基斯坦尼里-拉菲水牛及水牛所培育的三品杂水牛三个水牛品种,每个品种采集三头水牛原料乳等体积混合样本,分别编号为 N1(三品杂)、N2(摩拉)、N3(尼里·拉菲),采样时间为刚挤完奶10 min 左右,经无菌离心管密封好后于置于4 ℃冰上迅速带回实验室,置于-20 ℃冰箱冷藏备用。
1.2 样本总DNA 提取和PCR 扩增
取50 mL 水牛乳样品,以12 000 r/min 高速冷冻离心30 min,用灭菌脱脂棉去掉乳脂层,弃上清液,收集底部沉淀[5],按E.Z.N.A.®soil 试剂盒(Omega Bio-tek,Norcross, GA, U.S.)说明书进行总DNA 抽提,DNA 浓度和纯度利用NanoDrop2000 进行检测,利用1%琼脂糖凝胶电泳检测DNA 提取质量;为避免序列太长影响测序,同时兼顾扩增特异性,选用338F(5-ACTCCTACGGGAGGCAGCAG-3)和806R(5-GGACTACHVGGGTWTCTAAT-3)引物对V3-V4 可变区进行PCR 扩增,扩增程序为:95 ℃ 预变性 3 min,27 个循环(95 ℃ 变性30 s,55 ℃ 退火30 s,72 ℃ 延伸 30 s),最后 72 ℃ 延伸 10 min(PCR 仪:ABI GeneAmp®9700 型)。扩增体系为 20 uL,4 uL 5*FastPfu 缓冲液,2 uL 2.5 mmol/L dNTPs,0.8 uL引物(5 umol/L),0.4 uLFastPfu 聚合酶;10ng DNA 模板。
1.3 Illumina Miseq 测序
使用2%琼脂糖凝胶回收PCR 产物,用AxyPrep DNA Gel Extraction Kit (AxygenBiosciences, Union City, CA, USA) 进行纯化,Tris-HCl 洗脱,2%琼脂糖电泳检测。利用QuantiFluor™-ST (Promega, USA)进行检测定量。根据Illumina MiSeq 平台(Illumina,SanDiego,USA)标准操作规程,将纯化后的扩增片段构建PE 2*300 文库。文库构建步骤:(1)连接“Y”字形接头。(2)使用磁珠筛选去除接头自连片段。(3)利用PCR 扩增进行文库模板的富。(4)氢氧化钠变性,产生单链DNA 片段。利用Illumina 公司的Miseq PE300 平台进行测序(上海美吉生物医药科技有限公司)。原始数据上传至NCBI 数据库进行比对分析。
1.4 数据处理
采用Trimmomatic 软件处理原始测序数据,使用FLASH 软件进行拼接:(1)设置50 bp 的窗口,如果窗口内的平均质量值低于20,从窗口前端位置截去该碱基后端所有序列,之后再去除质控后长度低于50 bp的序列。(2)根据重叠碱基overlap 将两端序列进行拼接,拼接时overlap 之间的最大错配率为0.2,长度需大于10 bp。(3)根据序列首尾两端的barcode 和引物将序列拆分至每个样本,barcode 需精确匹配,引物允许2 个碱基的错配,去除存在模糊碱基的序列。使用的UPARSE 软件(version 7.1 http://drive5.com/uparse/)根据97%的相似度对序列进行OTU 聚类,并在聚类过程中去除单序列和嵌合体。利用RDP classifier(http://rdp.cme.msu.edu/) 对每条序列进行物种分类注释,比对Silva 数据库(SSU123),设置比对阈值为70%。
2 结果与分析
2.1 测序数据与聚类分析
通过对3 份水牛乳样本进行细菌16S rDNA 高通量测序,共得到优化序列 139356 条,平均长度426.404 bp,具体测序数据见表1、图1和图2。
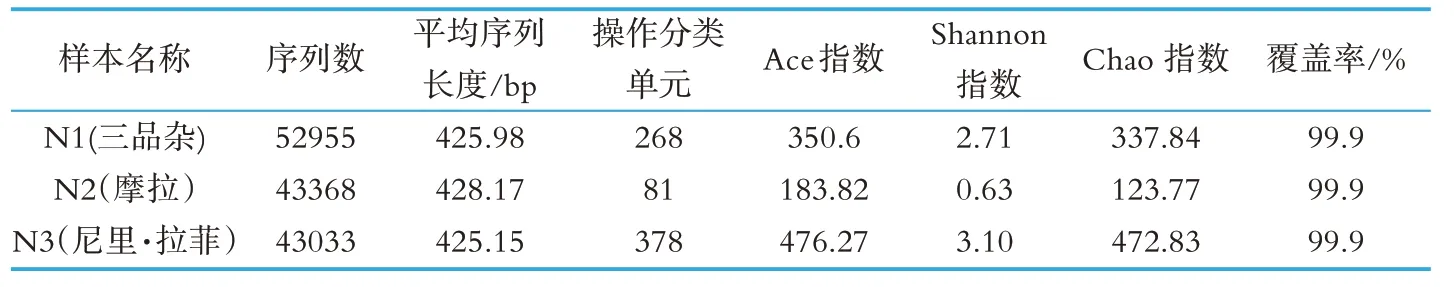
表1 样本测序数据分析
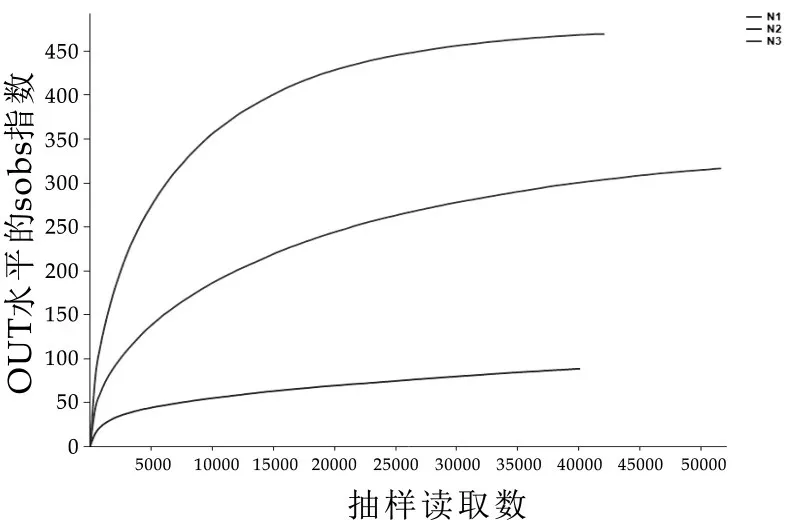
图1 Sobs稀释性曲线
对序列数据量进行随机抽取,统计抽取样本的序列数和OUT 数[6],绘制样本稀释性曲线(Rarefaction curve),如图1 显示,3 样本的曲线都逐渐趋于平缓,说明增加测序量可能会发现新的OUT,但图2 中3 样本Shannon 曲线明显趋于平坦,表明样本大部分微生物已产生,更大的测序量不会引起物种多样性的显著增长[7],故本研究现有数据量的分析结果准确可靠。Shannon 指数曲线(图2)也同时表明,样本N3 的微生物群落比N1和N2更具多样化。
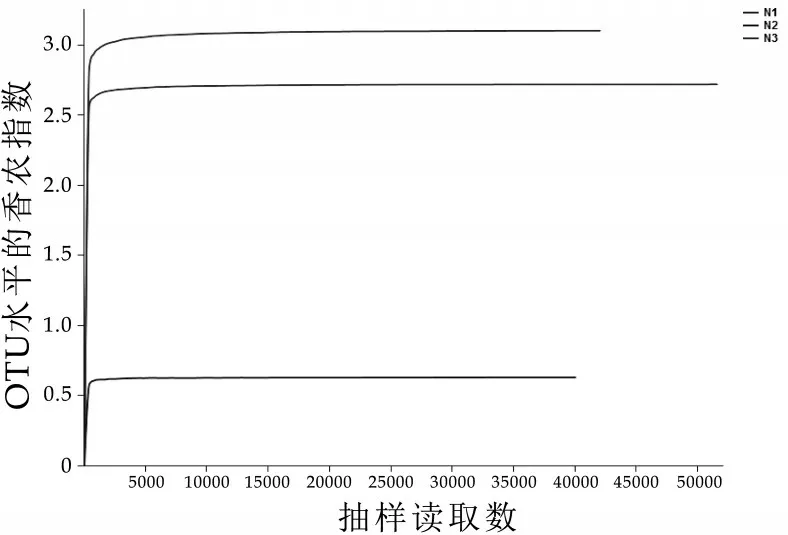
图2 Shannon 曲线
选取属水平的OTU 列表,(三品种的水牛乳是在属水平下,对其OUT 进行分类)在相似性为97%的水平下对聚类结果进行分析,绘制OTU 韦恩图。从图3 中可以看出,3 品种水牛乳样本共得到OTU 为540,其中 N1(三品杂)、N2(摩拉)、N3(尼里·拉菲)各自包含的 OTU 数目分别为 297、88 和 469,三品种所共有的OTU 数目为 66,N1、N2、N3 所特有的 OTU 数目分别为54、14 和224,其余OTU 为三样本间两两共有的物种数目,其中,N1(三品杂)与N3(尼里·拉菲)共有的OTU 较多,为174,说明这两个品种的水牛乳样本共有的核心微生物较多,物种组成较相似,而N3(尼里·拉菲)所特有的OTU 最多,为224,这预示着N3(尼里·拉菲)品种拥有比N1(三品杂)、N2(摩拉)更丰富的细菌微生物种类。

图3 OTU分布韦恩图
2.2 物种组成与丰度分析
以OTU 的物种分类结果为依据,通过菌落饼图(图4)在属水平对3 样本中的细菌种类和相对丰度进行统计分析。本文自定义相对丰度>8%的菌群为优势菌群。相对丰度<0.1%的菌不统计。由图4 可知,N1(三品杂)和N3(尼里·拉菲)中的微生物种类变化不大,但各种细菌所占百分比有所变化,二者共有的细菌为假单胞菌属(Pseudomonas)、肠球菌属(Enterococcus)、大肠埃希菌属-志贺菌(Escherichia-Shigella)、链球菌属(Streptococcus)、放线菌属(Actinomyces)、库克菌属(Kocuria)、不动杆菌属(Acinetobacter)、棒状杆菌属(Corynebacterium)、肠杆菌属(Enterobacter)、乳酸杆菌属(Lactobacillus),其中N1(三品杂.SP)的优势菌为肠球菌属 34.3%(Enterococcus)、假单胞菌属 13.7%(Pseudomonas)、大肠埃希菌属-志贺菌8.1%(Escherichia-Shigella),N2(摩拉.ML)中主要以籍伏氏菌属88.3%(Vogesella)为主,占比88.3%其次是黄杆菌属(Flavobacterium)4.95%和硫氧化湖沉积杆菌(Limnobacter)2.84%,N3(尼里·拉菲)中主要以假单胞菌属52.8%(Pseudomonas)为优势菌,另有4.93%的棒状杆菌属(Corynebacterium)、4.86%不动杆菌属(Acinetobacter)及18.8%未分类的其它细菌。
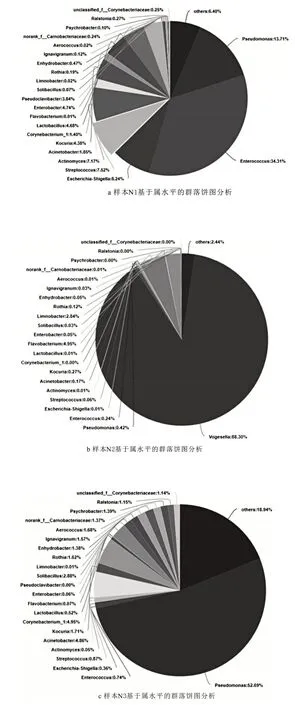
图4 a(N1)、b(N2)、c(N3)为样本基于属水平的群落组成
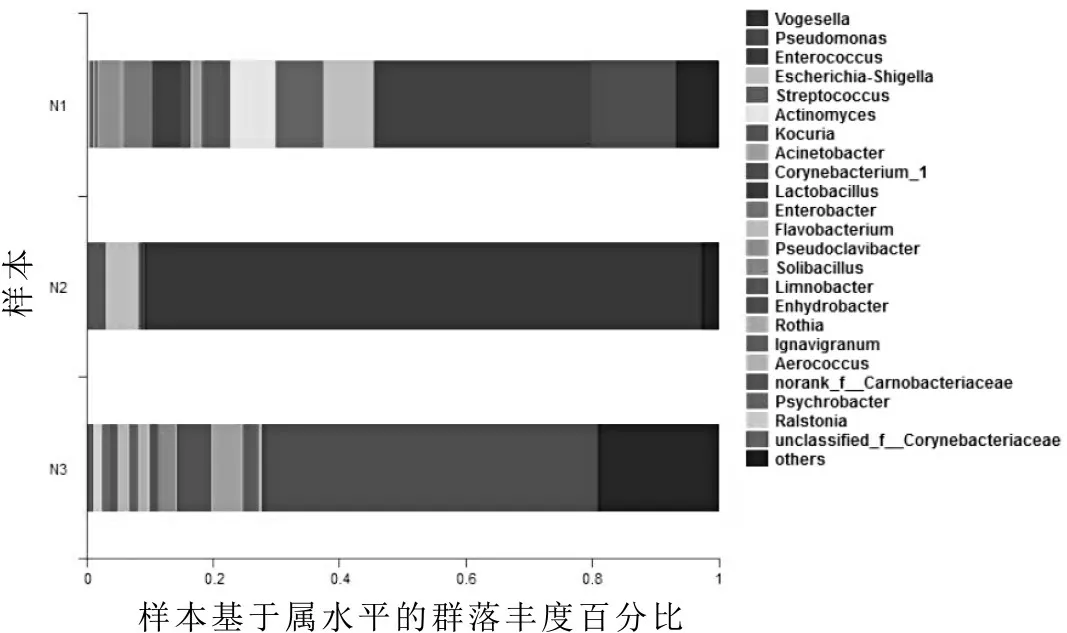
图5 样本基于属水平的群落丰度分布
图5 通过柱形图从属水平描述N1(三品杂)、N2(摩拉)、N3(尼里·拉菲)三样本中微生物菌群结构的组成,不同颜色的柱子代表不同的物种,柱子的长短代表该物种所占比例大小,由图5 可知,三品杂、摩拉、尼里·拉菲三个水牛乳样本中主要微生物菌群分布差异明显,N1(三品杂) 、N2(摩拉) 、N3(尼里·拉菲)的优势菌群分别为肠球菌属(Enterococcus)、籍伏氏菌属(Vogesella)和假单胞菌属(Pseudomonas)。三样本共鉴定出有21 个共有的细菌属,它们分别是籍伏氏菌属(Vogesella)、假单胞菌属(Pseudomonas)、肠球菌属(Enterococcus)、大肠埃希菌属-志贺菌(Escherichia-Shigella)、链球菌属(Streptococcus)、放线菌属(Actinomyces)、库克菌属(Kocuria)、不动杆菌属(Acinetobacter)、棒状杆菌属(Corynebacterium)、乳酸杆菌(Lactobacillus)、肠杆菌属(Enterobacter)、黄杆菌属(flavobacterium)、粪便假棍状杆菌属(pseudoclavibacter)、土壤芽胞杆菌属(Solibacillus)、硫氧化湖沉积杆菌(Limnobacter)、水栖菌属(Enhydrobacter)、罗斯氏菌属(Rothia)、肉杆菌属(Ignavigranum)、气球菌属(Aerococcus)、嗜冷杆菌(Psychrobacter)、雷尔氏菌属(Ralstonia),其中N3(尼里·拉菲)的细菌种类最多,覆盖15个属,这预示着尼里·拉菲品种的水牛乳中微生物多样性最丰富,三样本细菌丰度大小依次为N3(尼里·拉菲)>N1(三品杂)>N2(摩拉)。
2.3 样本间菌群比较分析
为研究不同样本物种组成结构的相似性和差异关系,可对样本距离矩阵进行聚类分析,构建样本层级聚类树。样本聚类树可以直观地反映样本间菌群的相似度[8]。3 个不同品种水牛乳样本基于属水平OTU 的聚类树见图6,树枝长度代表样本间的距离,分支宽度表示样本间距离的远近,相似度越高,距离越小[9]。由图6 可知,3 原料乳样本聚类成两个群,N1(三品杂)与N3(尼里·拉菲)聚成一类,相似度较高,N2(摩拉)与 N1(三品杂)和 N3(尼里·拉菲)的相似度差别较大。很显然,不同品种的水牛乳菌群组成结构不同,差异明显。

图6 样本层级聚类分析
基于OUT 的分类结果,在种分类水平对N1(三品杂)、N2(摩拉)、N3(尼里·拉菲)3 样本进行比较分析,其共有或独有的细菌数量见表2,合并小于1%的数值区域,发现在其共有的65 个种层面的菌群中,占比大于1%的细菌有8 个,其分布情况见图7,不同颜色表示不同的物种,饼面积表示该物种数目占物种总数目的百分比。由图7 可知,3 样本共有的核心菌落较多,并以假单胞菌属(Pseudomonas)和肠球菌属(Enterococcus)为主。N1(三品杂)与N3(尼里·拉菲)共有的菌落种类为210,占总数的48.8%,这说明N1(三品杂)与N3(尼里·拉菲)的菌落构成具有较高的相似性。N3(尼里·拉菲)特有的菌落最多,为163,占物种总数的37.9.%,说明N3(尼里·拉菲)特有的物种丰度最高。
样本通过降维分析后,在主成分上均有相对坐标点,样本点越接近,表明两样本物种组成越相似。由图8 可知,样本 N1、N2 和 N3 均分离较开,没有聚类现象,且主成分1(PC1)和主成分2(PC2)对样本差异性的贡献值分别达到了75.95%和24.05%,可以代表全部变量信息,说明这三个不同品种的水牛乳细菌群落结构存在显著差异。

表2 样本(种分类水平)共有或独有的物种数量
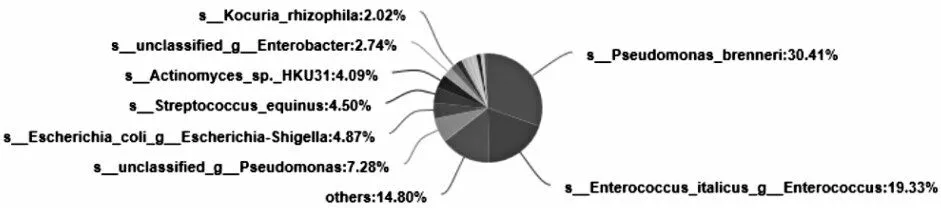
图7 样本基于种水平的物种占比
3 结 论
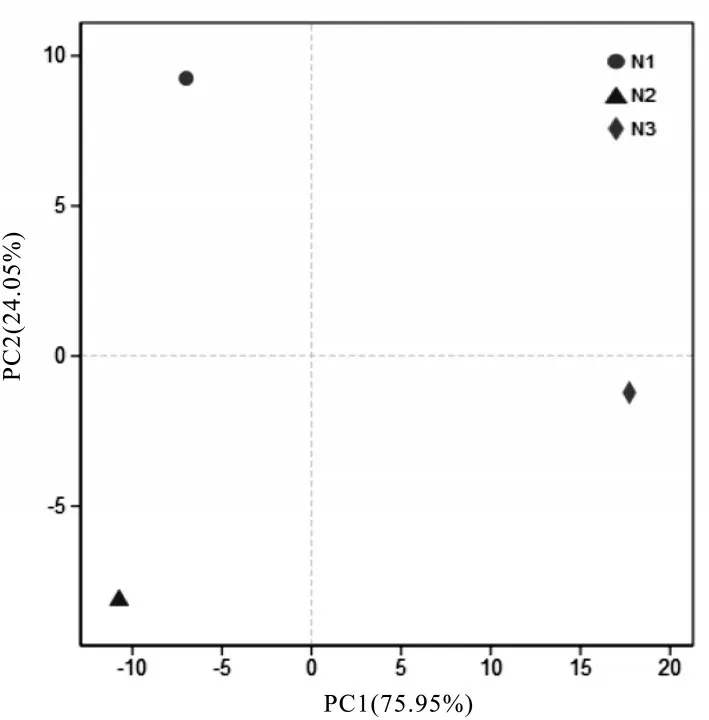
图8 样本基于属水平的主成分分析
本研究利用高通量测序,首次对N1(三品杂) 、N2(摩拉) 、N3(尼里·拉菲)3 品种的水牛原料乳细菌多样性进行解析。结果发现3 样本所含微生物共覆盖314个属430 个种,呈现出较丰富的多样性。其中假单胞菌属(Pseudomonas)、肠球菌属(Enterococcus)、大肠埃希菌属-志贺菌(Escherichia-Shigella)、链球菌属(Streptococcus)放线菌属(Actinomyces)、库克菌属(Kocuria)、不动杆菌属(Acinetobacter)为3 样本所共有且占比较大的细菌属,N1 、N2、N3 优势菌群分别为肠球菌属(Enterococcus)、籍伏氏菌属(Vogesella)和假单胞菌(Pseudomonas)。3 样本中均检出了病原性大肠埃希菌属-志贺菌(Escherichia-Shigella)。相较传统鉴定,高通量测序更全面和准确地反映了水牛原料乳中菌群的多样性,为后续研究者评估水牛乳中菌群对水牛乳品质的影响提供了技术支持。
