基于16S rRNA高通量测序技术分析安格斯牛瘤胃微生物多样性和功能预测的研究
2020-06-16王思珍胡宗福牛化欣
吴 琼, 王思珍*, 张 适, 尤 欢, 胡宗福, 牛化欣*
(1.内蒙古民族大学 动物科学技术学院,内蒙古 通辽 028000;2.通辽市家畜繁育指导站,内蒙古 通辽 028000)
反刍动物瘤胃内具有丰富的和复杂的微生物群体,其微生物群落不仅受到日粮、遗传种类、生长发育阶段、生存环境等方面的影响,还与宿主的消化吸收、营养代谢、生长性能、免疫功能和宿主的健康状况息息相关,认知瘤胃微生物数量和组成对建立宿主功能具有重要作用[1-5]。然而,瘤胃是一个严格厌氧的环境,在种类繁多、数量巨大的瘤胃微生物中绝大多数是不可培养的,利用传统的培养技术和常规的克隆-测序分子生物等方法来探索瘤胃微生物多样性及分析其功能是很难实现的,因此,阐明和揭示反刍动物瘤胃微生物基因资源的研究受到技术和方法的限制[6-7]。随着高通量测序技术的发展和在不同研究方向的广泛应用,宏基因组学科通过高通量测序的方法可全面探索微生物多样性和系统发生,揭示代谢通路和功能分析的研究[8-10]。近年来,国内外已有许多应用宏基因组学方法研究牛瘤胃微生物的报道,Jami等[11]对1日龄至2.5年龄段牛瘤胃细菌群落变化研究结果发现,出生后随着年龄的增长需氧和兼性厌氧菌减少,厌氧菌增多。De等[12]利用16S rRNA-焦磷酸测序方法分析了巴西食用牛(Nelore steer)消化道不同部位微生物多样性的变化。周熊艳等[13]研究了云南大额牛瘤胃微生物多样性。张慧敏等[14]利用宏基因组学技术系统探讨了海子水牛瘤胃内的微生物组成及木质纤维素降解酶系。以上报道主要研究了不同牛瘤胃微生物的菌落组成,但对其功能特征分析还处于探索阶段[15]。安格斯牛(Angus)原产于苏格兰北部的阿伯丁和安格斯等地区,具有良好的肉用性能,养殖范围分布广,是世界上专门化肉牛品种中的典型品种之一[16-17]。近年来,我国不同地区也从不同国家引进多批次的安格斯牛,对其遗传性能、生长性能、肉质肉品等指标进行研究。然而,对安格斯牛瘤胃微生物多样性及其功能分析的研究鲜见报道。本研究利用16S rRNA高通量测序技术对安格斯牛瘤胃微生物多样性及其功能进行研究和分析,为探索安格斯牛瘤胃生境微生物提供基础,也为瘤胃微生物功能的探索和进一步挖掘与某些重要营养生理功能密切相关的瘤胃微生物功能提供参考。
1 材料与方法
1.1 材料
试验牛取自内蒙古通辽谷润肉业有限公司,选取6头22月龄左右的安格斯牛,分2组为an1和an2,平均体重550 kg。实验牛进行标准化饲养与管理,每天饲喂2次,自由采食,饲粮组成及营养水平见表1[18]。
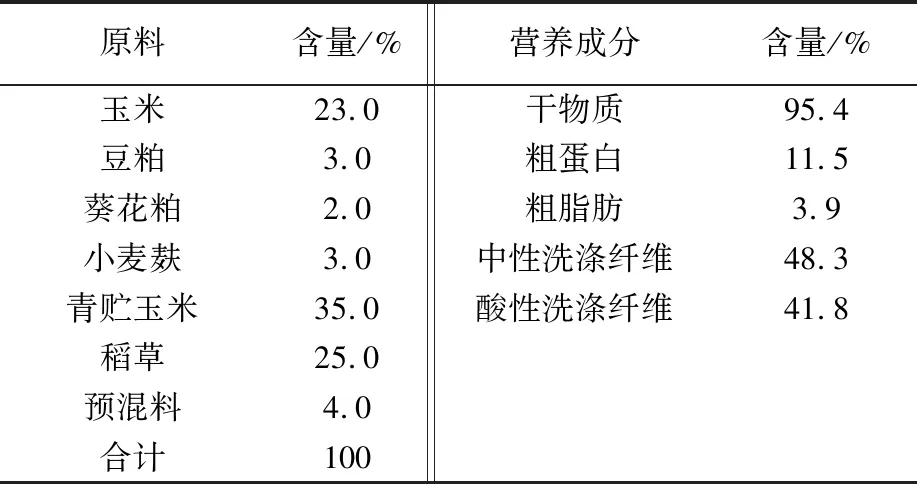
表1 饲粮组成及营养水平(干基)
屠宰后立刻采集瘤胃液样品,经4层灭菌纱布过滤后放入50 mL冻存管中,按照1∶ 1∶ 1的比例将每组的瘤胃液混合均匀,迅速置于液氮中,-80 ℃保存,备用。
1.2 方法
1.2.1 瘤胃细菌DNA的提取、16S rRNA基因扩增及测序 按照FastDNA® SPIN Kit for Soil (MP Biomedicals,Santa Ana,CA,美国)试剂盒的使用说明,抽提瘤胃液中的细菌基因组DNA。用紫外分光光度计检测基因组DNA浓度及纯度,1%琼脂糖凝胶电泳检测DNA完整性。使用细菌16S rRNA基因V3~V4区通用引物338F(ACTCCTACGGGAGGCAGCA)及806R(GGACTACHVGGGTWTCTAAT)[19],在引物两端设计12 bp的条形码用于识别不同样品序列,对以上提取的DNA进行序列扩增,PCR反应体系参考胡宗福等[20]方法。 PCR扩增产物经纯化后,使用NEB Next® UltraTMDNA Library Prep Kit for Illumina (New England Biolabs Inc., Ipswich,127 MA, 美国)建立扩增序列文库,之后采用Illumina Miseq测序平台(Illumina Corporation,San Diego,美国) 进行克隆文库的高通量测序,测序公司为上海美吉生物医药科技有限责任公司(上海,中国)。
1.2.2 生物信息学分析 序列拼接与质控、操作分类单元(OTU)聚类及生物信息学在http://www.i-sanger.com/云平台按胡宗福等[20]方法进行操作和分析。
1.2.3 16S功能预测分析 为探讨瘤胃微生物群落在其体内发挥的功能,使用PICRUSt[21]方法对16S RNA扩增子数据库进行了宏基因组功能组成预测,该方法将高通量测序所得的16S RNA扩增子数据库与Greengenes数据库进行比对,获得OUT对应物种的功能信息,并根据最新的Kyoto Encyclopedia of Genes and Genomes (KEGG)数据库信息,对菌落做出功能组成的预测。
2 结果与分析
2.1 安格斯牛样本序列信息、序列过滤与质控
通过序列拼接及质控,2个测序样本共获得了108 378条有效序列,平均长度441 bp,经过优化处理后,共得到86 298条优化序列,这些优化序列将用于后续的OTU聚类及分类学分析(表2)。
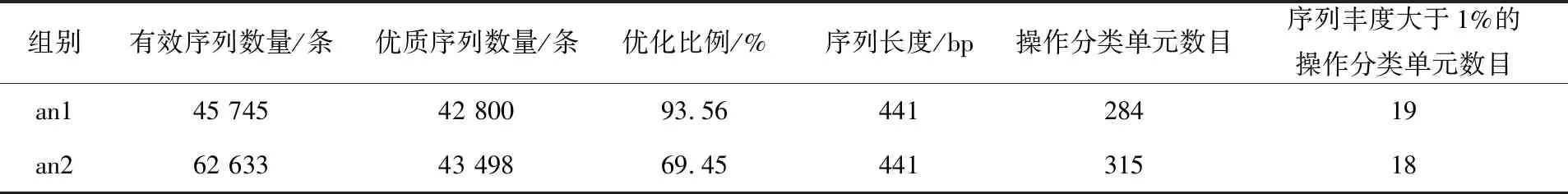
表2 样本序列信息
2.2 微生物群落的Alpha多样性分析
表3为两组安格斯牛的Alpha多样性指数。Alpha多样性用来表示样本内微生物群落的多样性,其中Chao、Ace指数反映菌群丰度,其值与菌群丰度正相关;而Simpson和Shannon指数反映菌群多样性,Shannon值与菌群多样性正相关,Simpson值与菌群多样性负相关。结果表明,两组牛菌群数量及组成相似,菌群丰度较大,多样性指数也处于较高的水平。
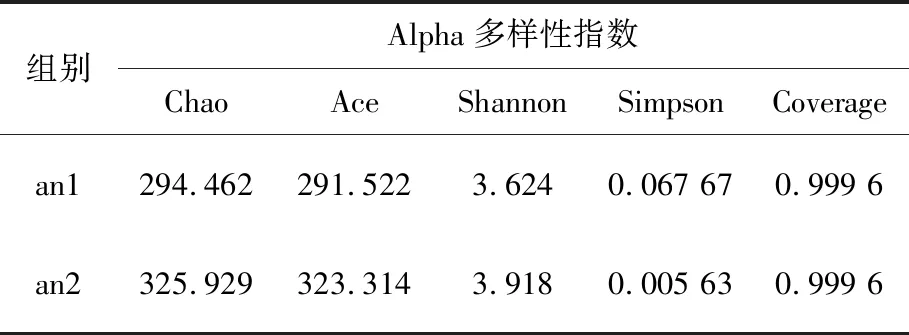
表3 不同样本的Alpha多样性指数
2.3 基于门和属水平的微生物群落结构分析
经过序列相似性大于97%水平上的OTU聚类,共获得了346个OTU,隶属于13个门,21个纲,24个目,40个科,123个属。图1A所示,丰度大于1%的门有拟杆菌门(Bacteroidetes,43.16%)、厚壁菌门(Firmicutes,36.29%)、变形菌门(Proteobacteria,14.50%)、纤维杆菌门(Fibrobacteres,1.53%)、互养菌门(Synergistetes和其他1.86%),前三者所占比例极大。在属水平上,丰度大于1%的菌属有16个,依次为普雷沃氏菌属_7 (Prevotella_7,29.28%)、琥珀酸弧菌科_UCG-001 (Succinivibrionaceae_UCG-001,11.30%)、琥珀酸菌属(Succiniclasticum,11.10%)、普雷沃氏菌属_1 (Prevotella_1,6.65%)、瘤胃球菌属_1(Ruminococcus_1,5.17%)、琥珀酸弧菌属(Succinivibrio,2.75%)、普雷沃氏菌科_UCG-001(Prevotellaceae_UCG-001,2.45%)、罗氏菌属(Roseburia,2.34%)。
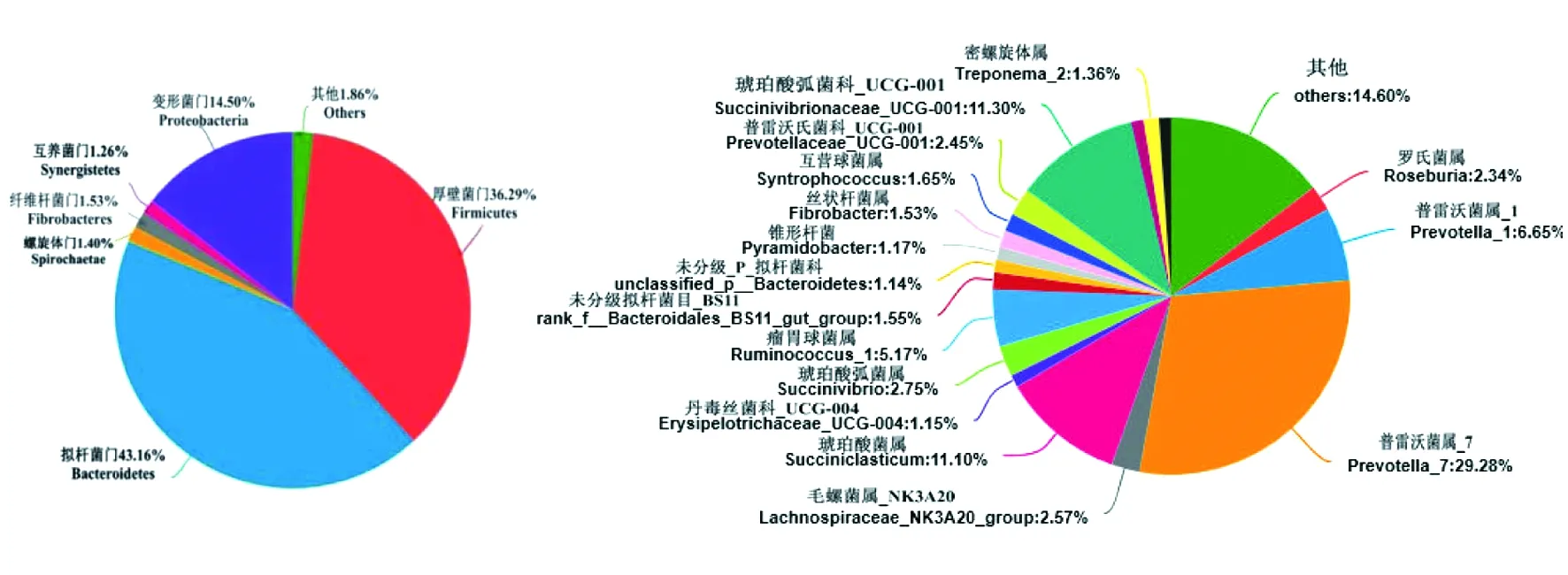
图1 基于门水平(A)和属水平(B)瘤胃细菌多样性Fig.1 Microbial community structure of Angus cattle rumen on the phylum(A) and genus level(B)
由图2可以看出,拟杆菌门由进化相近的普雷沃氏菌科等6种组成。相反,厚壁菌门所包含的科比较分散,由氨基酸球菌科、瘤胃球菌科、毛螺菌科、丹毒丝菌科、韦荣氏菌科等7种组成。变形菌门由两个进化相对较远的琥珀酸菌科和红椿菌科组成,其中前者丰度占绝对优势。纤维杆菌门由两个进化较远的纤维杆菌科和未分级组成。
2.4 功能预测分析
2.4.1 COG 数据库比对结果 基于PICRUSt分析平台的16S功能预测将获得的OTU丰度表与Greengene数据库比对,获得相应的COG及KO功能信息及其丰度。分析发现牛瘤胃菌群功能主要集中于氨基酸运输、代谢、通用功能预测、碳水化合物转运及代谢和细胞壁/膜/包膜生物合成等方面,由此可预测可能含有丰富的蛋白分解、转运及代谢酶相关基因,也含有大量的纤维素和木质素降解酶基因(图3)。
2.4.2 KEGG分析结果 图4 KEGG一级和二级代谢通路显示,基因依次富集在新陈代谢49%、遗传信息处理22%、环境信息处理12%、细胞过程3%、人类疾病0.8%、生物体系统0.7%,还有未分类功能基因14%。二级代谢通路表明,基因主要富集在新陈代谢中氨基酸代谢10%和碳水化合物代谢10%,还有遗传信息处理中的复制与修复10%、环境信息处理中膜转运10%。
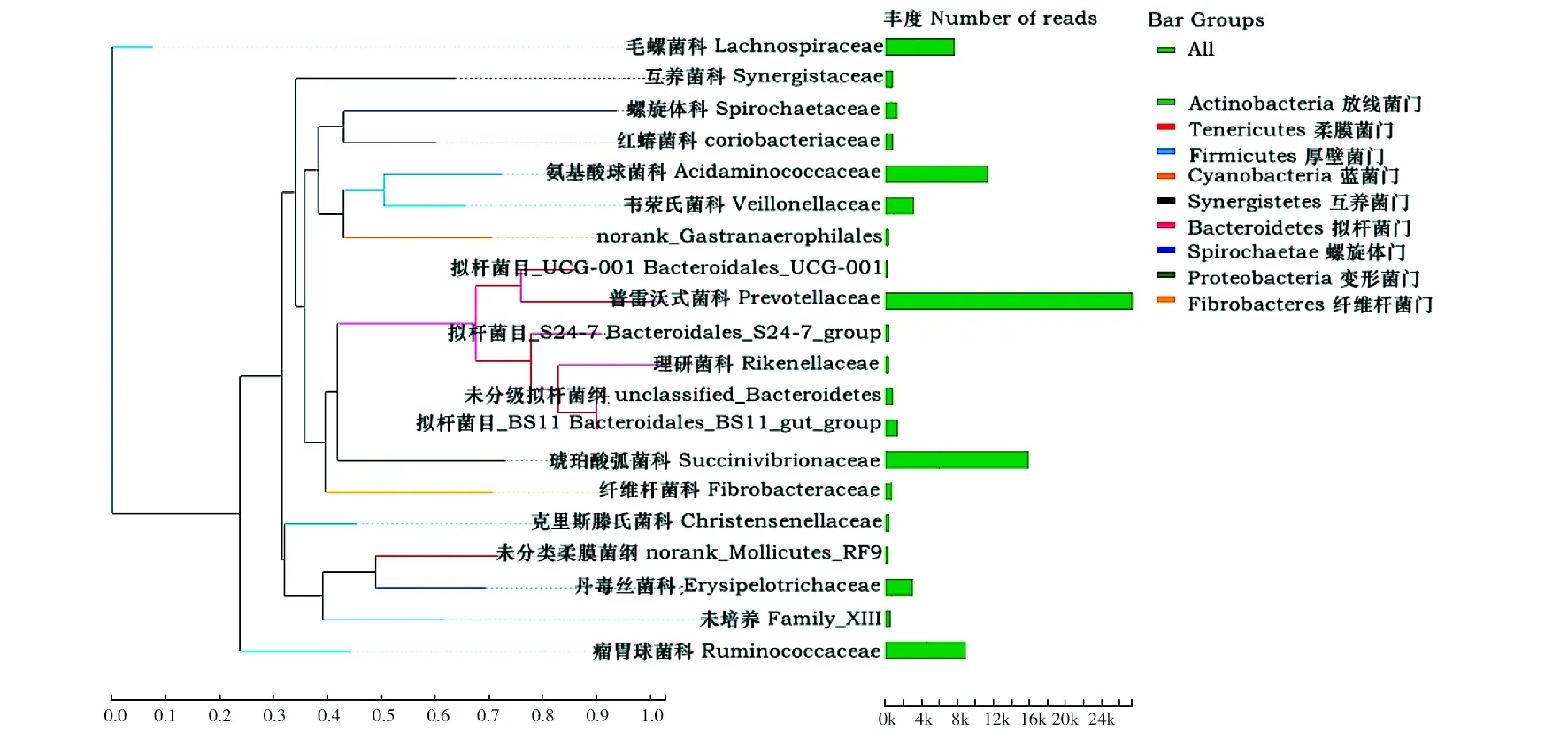
图2 基于科水平安格斯牛瘤胃微生物系统进化树Fig.2 Microbial biological phylogenetic tree of Angus cattle rumen on the family level
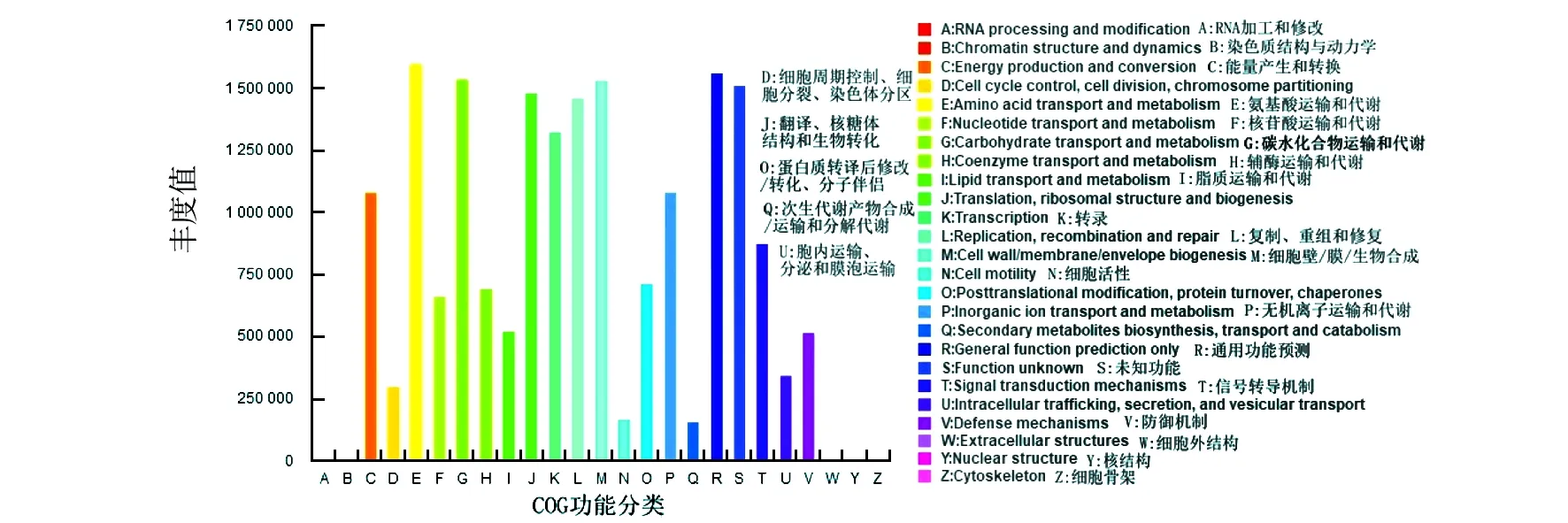
图3 COG 分类Fig.3 COG classification
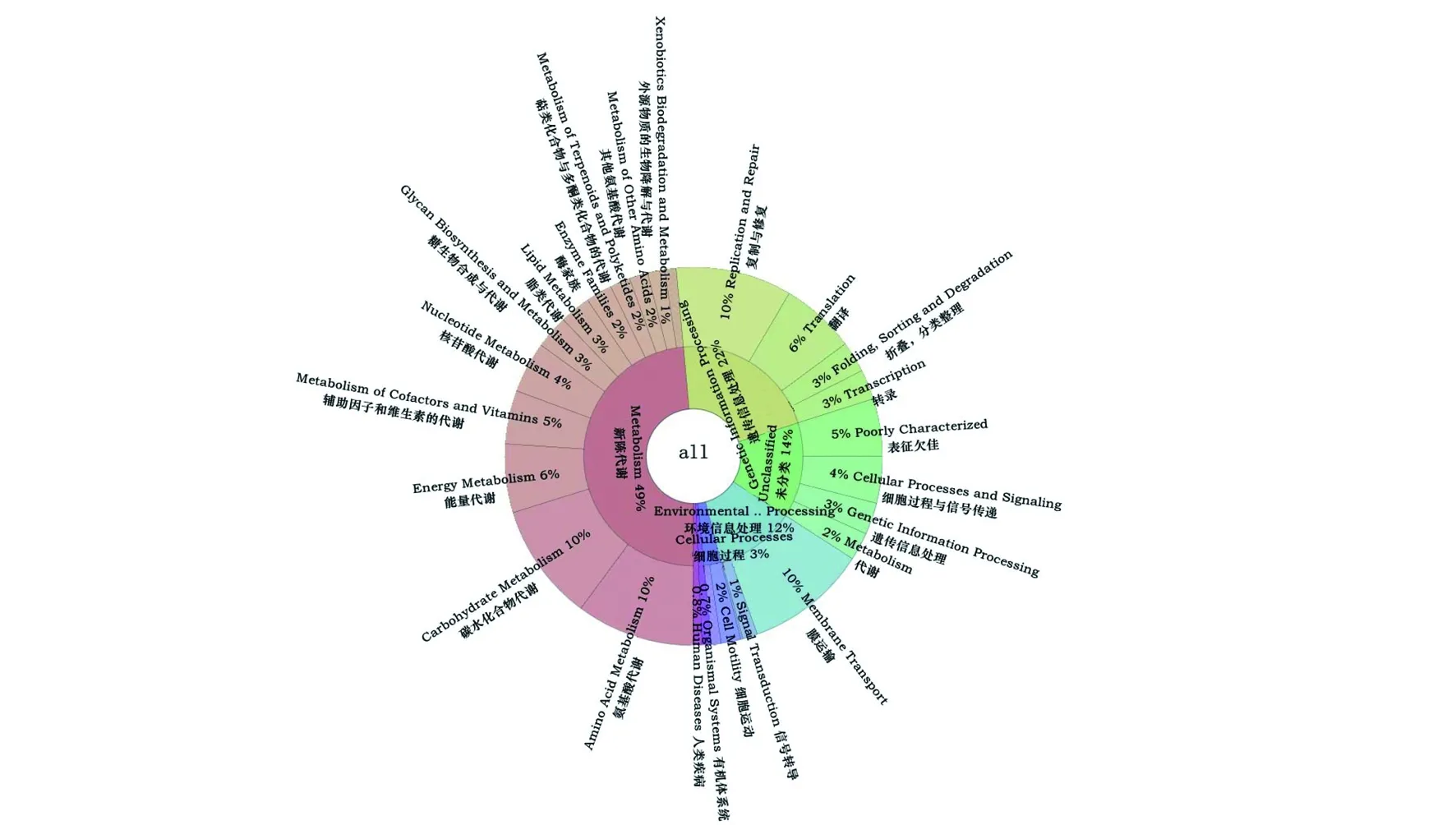
图4 KEGG一级和二级代谢通路Fig.4 KEGG of category level 1 and 2
2.4.3 碳水化合物酶(CAZy)注释 根据蛋白质结构域中氨基酸序列的相似性,将不同物种来源的碳水化合物活性酶分为糖苷水解酶(Glycoside Hydrolases GHs)、糖基转移酶(Glycosyl Transferases,GTs)、多糖裂合酶(Polysaccharide Lyases,PLs)、碳水化合物酯酶(Carbohydrate Esterases,CEs) ,辅助氧化还原酶(Auxiliary Activities,AAs),碳水化合物结合模块(Carbohydrate-Binding Modules CBMs)等6大类蛋白质家族。图5显示预测的功能基因GH所占比例最高,其他依次为GT、CBE、CE、AA和PL。
2.4.4 短链脂肪酸(SCAA)生成酶 参与牛瘤胃甲酸、乙酸、丙酸和丁酸短链脂肪酸生产的细菌酶COG编号和基因丰度由见图6,产乙酸相关酶基因丰度最高,为33%(3种),其他依次为丁酸32%(4种)、丙酸23%(3种)和甲酸12%(1种)。
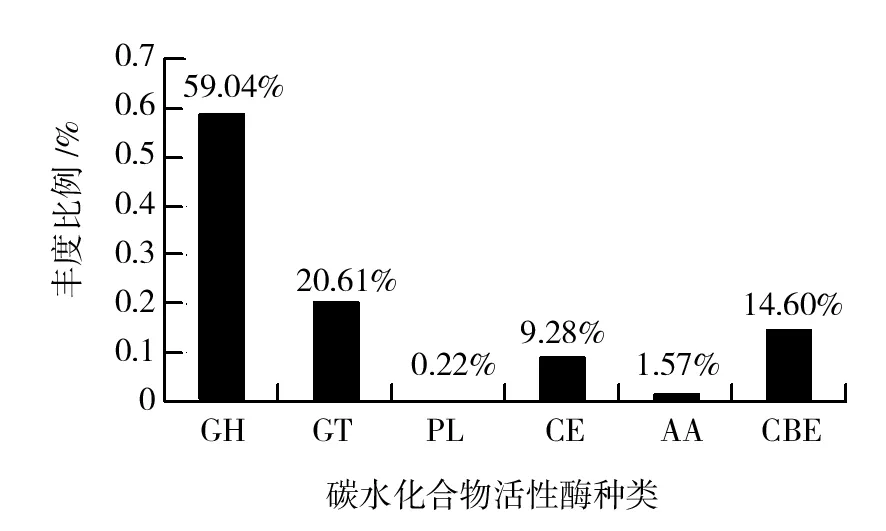
图5 CAZy 数据库比对结果Fig.5 Distribution of CAZyme encoding genes
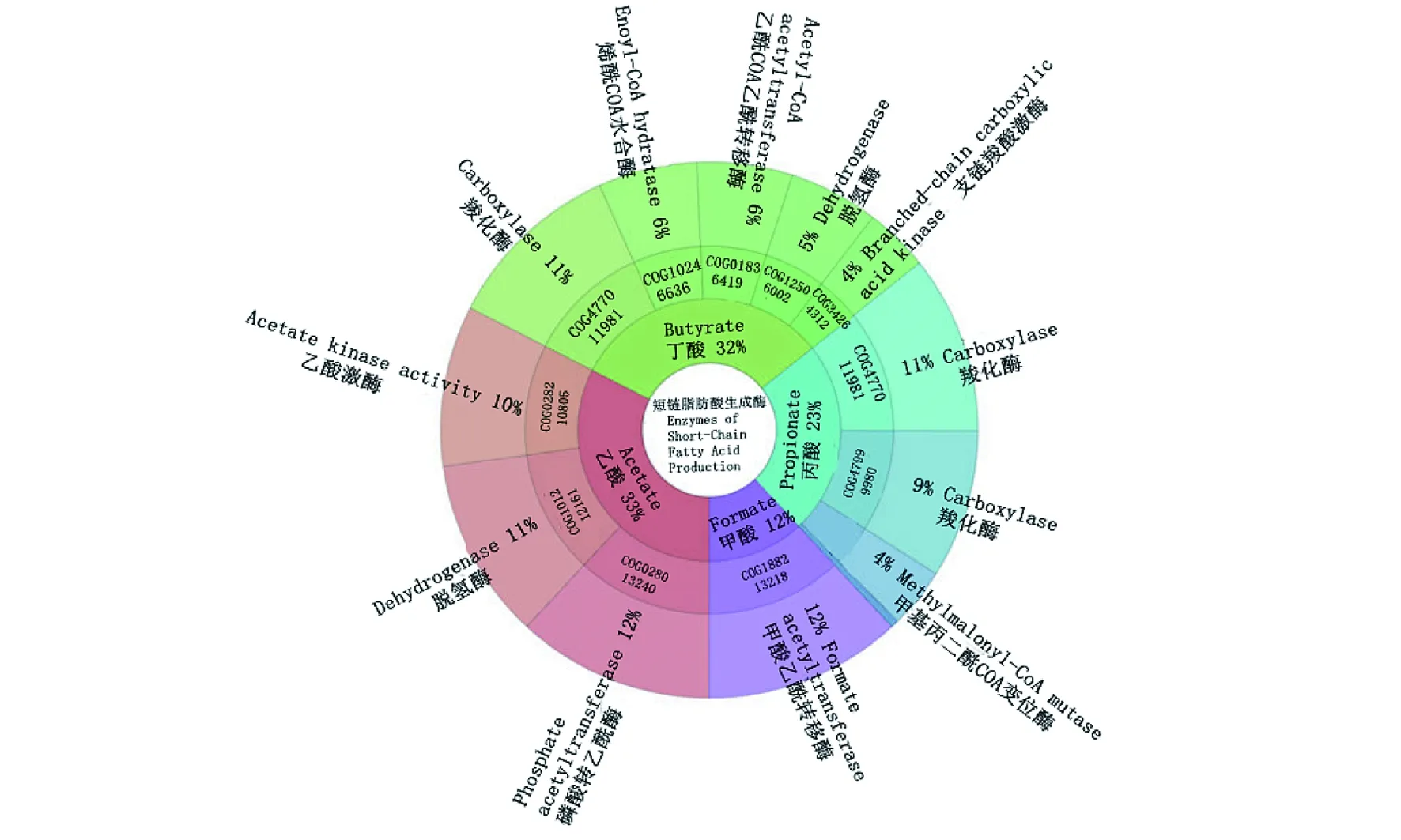
图6 短链脂肪酸生成酶COGFig.6 Enzymes of short-chain fatty acid production COG 图由内向外依次为第1环至第4环,第3环表示COG编号和基因丰度,第4环表示SCAA生成的相关酶类 From inside to outside, theFigure shows the first ring to the fourth ring, the third ring represents the COG number and gene abundance, and the fourth ring represents the enzymes related to SCAA production
3 讨 论
3.1 安格斯牛瘤胃菌群多样性
本研究通过高通量测序技术对安格斯牛瘤胃液菌群结构及组成进行检测,并基于原核16S rDNA高通量测序结果对菌群功能进行预测。安格斯牛瘤胃菌群基于门水平,优势菌群主要包括拟杆菌门(43.16%)、厚壁菌门(36.29%)和变形菌门(14.50%),与周熊艳等[13]研究的大额牛瘤胃优势菌群(拟杆菌门48%、厚壁菌门42%和变形菌门3%)组成有类似的结果。De等[12]利用16S rRNA-焦磷酸测序分析巴西食用牛消化道不同部位微生物多样性的变化。结果显示,在所有的样本中,与厚壁菌门和拟杆菌门有关的序列占优势,这与本研究有类似的结果。张慧敏等[14]分析的海子牛瘤胃的优势菌群为拟杆菌门占比55.3%,但厚壁菌门较安格斯牛和大额牛所占比例要少。其原因主要是受到遗传种类方面的影响。安格斯牛瘤胃优势菌群与诸多研究[22-23]反刍动物瘤胃中优势细菌为拟杆菌门和厚壁菌门的结果一致。根据科和属水平分类,安格斯牛瘤胃中隶属于拟杆菌门的普雷沃氏菌属(35.93%)占绝对优势,还含有2.45%的普雷沃氏菌科_UCG-001(未分到属水平),可降解非纤维素植物多糖如木聚糖,在果胶的利用和蛋白质的降解等方面发挥着重要的作用[24-25]。隶属于厚壁菌门、氨基酸球菌科的琥珀酸菌属占比11.10%,毛螺菌科的瘤胃球菌属占比5.17%。研究表明,以上菌群被认为是瘤胃中降解木质纤维素的主要微生物,可产出大量的纤维素酶[26-27]。另外,隶属于变形菌门的琥珀酸弧菌科_UCG-001占比11.30%,琥珀酸弧菌属占比2.75%。安格斯牛瘤胃细菌中占比最大的门为拟杆菌门、最大的科为普雷沃氏菌科、最大的属为普雷沃氏菌属。
3.2 安格斯牛瘤胃菌群功能预测
瘤胃微生物菌群是微生物共生联合体,它既是反刍动物中重要的蛋白质来源,也是反刍动物通过纤维发酵产生挥发性脂肪酸(VFA)的主要能量来源,因此瘤胃细菌是庞大的生物资源库,积极挖掘一些与重要营养生理功能密切相关的瘤胃菌群功能基因是非常重要的,如碳水化合物转运及代谢、氨基酸运输和代谢、VFA的生成等[9,28]。Jami等[11]对1日龄至2.5年龄段牛瘤胃细菌群落变化研究结果发现,出生后随着年龄的增长需氧和兼性厌氧菌减少,厌氧菌增多。本研究未做厌氧菌的分析,但对安格斯牛瘤胃细菌测序数据与COG数据库进行比对分析预测发现,功能集中在氨基酸运输及代谢、通用功能预测、碳水化合物转运及代谢、细胞壁/膜/包膜生物合成等方面,这与印度水牛[29]、海子水牛[14]的报道和中国西门塔尔牛[30]有些差异,后三者瘤胃菌群功能集中在碳水化合物转运及代谢的基因最多,而本研究中安格斯牛瘤胃菌群功能集中在氨基酸运输及代谢的基因最多,造成此差异的原因可能是宿主、日粮、养殖环境等多方面的因素。
Pitta等[31]基于16S rRNA测序技术,报道了为肉牛投喂低蛋白质高纤维的牧草能够转换高蛋白质高可溶性营养物的冬小麦时,普雷沃氏菌的比例由28%上升到56%,是瘤胃中比例最高的菌属,其与蛋白降解有关,与本研究结果类似,因此所预测功能也集中在与高蛋白日粮有关的氨基酸运输及代谢上。 KEGG一级代谢通路分析显示,基因最主要富集在新陈代谢上;二级代谢通路表明,基因主要富集在新陈代谢中氨基酸代谢和碳水化合物代谢上,这与本研究COG数据库所预测的氨基酸运输和代谢、碳水化合物转运及代谢功能一致。
经过KEGG代谢酶与CAZy数据库比对发现,本研究GH基因数量最多,GT次之,与海子水牛[14]、泽西奶牛[32]利用宏基因组预测的CAZy一致。瘤胃细菌可分泌半纤维素酶、淀粉酶、木聚糖酶、内切葡聚糖酶、葡糖苷酶和木聚糖乙酰酯酶等,构成复杂的瘤胃内纤维降解体系,能高效降解植物细胞壁中的纤维素和半纤维素,产生的乙酸、丙酸和丁酸等可为反刍动物机体提供能量[33]。碳原子数为1~6的有机脂肪酸称为SCFA,反刍动物短链脂肪酸(SCFA),也称挥发性脂肪酸(VFA),主要有瘤胃菌群所产生的SCFA生成酶发酵碳水化合物的最终产物,包括乙酸、丙酸、丁酸、戊酸、己酸,其中乙酸、丙酸和丁酸含量最高,是瘤胃内最重要的挥发性脂肪酸,约占SCFA的90%以上[34],为机体提供所需能量的60%~80%[35]。本研究安格斯牛在精养工厂化摄饲精料育肥条件下,产乙酸相关酶基因丰度最高,依次为丁酸、丙酸。Deusch等[32]研究表明,分泌产SCFA生成酶的瘤胃菌群分别为厚壁菌门中毛螺菌科、瘤胃球菌科、韦荣氏菌科等,其产物为丁酸;拟杆菌门中普雷沃氏菌科_15和厚壁菌门中韦荣氏菌科,其产物为丙酸;拟杆菌门中普雷沃氏菌科_9其产物为乙酸;厚壁菌门中毛螺菌科和变形菌门中琥珀酸弧菌科,其产物为甲酸。这与本研究检测到的菌群的种类和丰度类似。瘤胃菌群、内容物组成(摄食日粮组成)与瘤胃SCFA的生成有着非常密切的关系[36-37],因此,通过调控瘤胃菌群和摄食日粮组成是反刍动物合理和高效利用SCFA的重要途径之一。
安格斯牛瘤胃液通过Illumina Miseq测序平台共获得86 298条高质量优质序列,经分类学鉴定分属13个门,21个纲,24个目,40个科,123个属,拟杆菌门和厚壁菌门为优势菌群门,基于属的组成依次为普雷沃氏菌属_7、琥珀酸弧菌科_UCG-001、琥珀酸菌属、普雷沃氏菌属_1、瘤胃球菌属_1、琥珀酸弧菌属等。16S功能预测和COG、KEGG代谢通路、CAZymes注释数据库对比发现,功能集中在氨基酸运输和代谢、碳水化合物转运及代谢的相关基因上,说明可能含有丰富的蛋白分解、转运及代谢酶相关基因和大量的纤维素、木质素降解酶基因。产乙酸和丁酸相关酶基因丰度较高。
