转Pi9抗稻瘟病基因水稻株系的比较转录组分析
2020-05-30许赵蒙李利华高晓庆袁正杰李莘田旭丹王岚岚瞿绍洪
许赵蒙 李利华 高晓庆 袁正杰 李莘 田旭丹 王岚岚 瞿绍洪, *
转抗稻瘟病基因水稻株系的比较转录组分析
许赵蒙1, 2李利华2, 3高晓庆1袁正杰2李莘2田旭丹1, 2王岚岚2瞿绍洪2, *
(1浙江师范大学 化学与生命科学学院,浙江 金华 321004;2浙江省农业科学院 病毒学与生物技术研究所,杭州 310021;3湖南农业大学 植物保护学院,长沙 410128;*通信联系人,E-mail: squ@mail.zaas.ac.cn)
【】在转录水平上解析基因介导的稻瘟病抗性调控机理,为培育抗病水稻品种提供理论依据。向水稻品种日本晴(NPB)及其转抗稻瘟病基因株系(NPB/Pi9)接种稻瘟菌。分别于接种后0 h、12 h、24 h、36 h提取叶组织样品,选取12 503个水稻基因定制基因芯片,进行水稻基因转录组分析,并通过qRT-PCR对部分差异表达基因进行验证。NPB/Pi9在接种后12 h、24 h和36 h的基因表达量分别与其接种0 h表达量比较,共检测到7 754个差异表达基因;相应地,感病水稻NPB在以上时间点共检测到7 385个差异表达基因;在接种后36 h,NPB/Pi9的差异表达基因数目显著多于NPB。比较NPB/Pi9和NPB相同时间点的基因表达量,共获得4 065个差异表达基因,其中接种后36 h的差异表达基因显著多于接种后0 h、12 h或24 h。因此,NPB/Pi9的稻瘟病防御反应更强烈。对NPB/Pi9与NPB相同时间点的差异表达基因进行GO和KEGG分析,细胞外区域、植物对刺激应答、转录调控、氧化还原、离子结合、次生代谢和植物激素相关的GO分类在接种后呈显著富集,苯丙氨酸代谢、类黄酮生物合成和植物激素信号途径的KEGG通路在接种后显著富集。与效应分子触发的免疫反应(ETI)相关的水杨酸信号途径、几丁质酶,以及与病原相关分子模式触发的免疫反应(PTI)相关的胞外区域、对刺激的应答、木质素合成等,均在抗感水稻之间差异表达。而且PTI/ETI共有的WRKY转录因子、MAPK激酶、茉莉酸和乙烯信号途径等发生差异表达。综上所述,NPB/Pi9和NPB的差异表达模式与ETI和PTI相关,两者相互联系并在介导的稻瘟病抗性中发挥作用。与日本晴比较,抗病基因型NPB/Pi9对稻瘟病防御反应更强烈。转录因子、激酶、基因、几丁质酶、水杨酸、茉莉酸和乙烯信号途径,以及植物次生代谢在介导的稻瘟病抗病反应中发挥重要作用。
水稻稻瘟病;转录组;抗病基因;植物激素;次生代谢
水稻()是重要粮食作物之一。由水稻稻瘟病菌()引起的稻瘟病是水稻最严重的病害之一,是限制水稻高产稳产的主要因素[1]。水稻基因是目前水稻育种中广泛使用的广谱抗稻瘟病基因之一[2]。编码NBS-LRR抗病蛋白[3]。Pi9蛋白与稻瘟病菌无毒效应蛋白AVR-Pi9互作从而诱导水稻抗病反应[4]。基因是基因的重要成员,在效应分子触发的免疫反应(effector-triggered immunity, ETI)即病菌小种特异性抗病反应中起重要作用[5]。基因介导的ETI信号转导途径主要包括丝裂原激活的蛋白激酶信号途径、WRKY等转录因子启动的基因表达以及水杨酸(salicylic acid, SA)、茉莉酸(jasmonic acid, JA)和乙烯(ethylene, ET)等抗病激素的基因调控[6]。
宿主基因转录调节是植物防御反应的中心部分,而阐明基因表达差异的调控机制是理解植物抗病分子基础的关键[7]。Wei等[8]利用基因芯片技术研究基因介导的水稻免疫反应,发现WRKY转录因子在水稻抵抗稻瘟病的过程中发挥重要作用。Zhang等[9]对具有稻瘟病部分抗性的-RNAi转基因水稻株系进行转录组分析,解析了介导的水稻防御反应和信号转导途径。
明确水稻抗病感病基因型的基因表达差异,是解析抗病机理和培育抗病品种的基础。Li等[10]对水稻抗病品种地谷(含和抗稻瘟病基因)与感病品种丽江新团黑谷进行比较转录组分析,根据稻瘟病菌附着胞成熟之前地谷启动防御基因的表达,鉴定出水稻早期防御中发挥重要作用的受体激酶基因。Kitony[11]以水稻感病品种CO39及其回交转育抗稻瘟病基因的5个近等基因系为材料,进行抗感基因型比较转录组分析,发现表达基因的数量、功能、共表达模块等的差异导致供试水稻品种具有不同稻瘟病抗性水平。Meyers等对转日本晴水稻株系和未转化日本晴的稻瘟菌接种水稻样品进行比较转录组分析,建立了水稻与稻瘟菌分子互作的MPSS数据库(https://mpss.danforthcenter. org/mpss/rice/)。
本研究以日本晴水稻遗传背景的转基因抗稻瘟病株系和感病的日本晴为材料,利用基因芯片技术,对稻瘟病菌接种水稻样品进行比较转录组分析,并对部分差异基因进行验证,在转录水平上解析相关的抗病调控机理,为抗稻瘟病水稻育种提供理论依据。
1 材料与方法
1.1 水稻材料育苗
供试水稻材料为日本晴(NPB)及其转基因抗稻瘟病水稻株系(NPB/Pi9)。挑选籽粒饱满、无霉斑水稻种子,置于羊皮纸袋,37℃下浸种发芽5 d后移栽入盆,每盆种植抗感基因型两种材料各5~8株。在24℃~26℃、14 h光照/10 h黑暗、湿度85%的生长箱中生长10 d,用于稻瘟菌接种。
1.2 水稻稻瘟病菌喷雾接种和取样
水稻稻瘟病菌株KJ201在CM培养基上活化培养12 d,用0.02%吐温20孢子洗脱液制备孢子悬浮液,调整孢子浓度至5×105个/mL,对水稻植株进行喷雾接种。分别于接种后0 h、12 h、24 h、36 h用无菌剪刀剪取水稻叶片组织,液氮速冻后保存于–80℃超低温冰箱备用。
1.3 差异基因筛选及GO分类富集分析
从稻瘟菌侵染条件下水稻MPSS和SBS转录组数据(美国丹佛斯植物研究中心和俄亥俄州立大学,https://mpss.danforthcenter.org/mpss/rice/)一共选取 12 503个水稻基因,包括、类受体蛋白激酶、E3连接酶、转录因子、SNARE蛋白、钙依赖性激酶等抗病相关基因。委托上海伯豪生物技术公司定制基因芯片,检测抗病基因型NPB/Pi9和感病基因型NPB响应稻瘟菌侵染的实时转录谱。
根据基因芯片杂交检测信号,计算每个基因接种后0 h的相对表达量(记为0 hexp),以及接种后12 h、24 h和36 h的相对表达量(分别记为12hexp、24hexp和36hexp)。把每个水稻材料(NPB/Pi9或NPB)各接种时间点的基因表达量与其接种后0 h的表达量比较,计算表达量比值(12hexp/0hexp、24hexp/0hexp和36hexp/0hexp),以此作为每个材料的每个基因随时间变化发生差异表达的量值。计算抗感基因型NPB/Pi9与NPB中同一基因在相同时间点表达量的比值(0hexp-Pi9/0hexp-NPB、12hexp-Pi9/12hexp-NPB、24hexp-Pi9/24hexp-NPB和36hexp-Pi9/36hexp-NPB),作为抗感基因型在相同时间点表达差异的量值。将表达量比值不小于2或不大于0.5的基因认定为差异表达基因(differentially expressed genes,DEGs)。
利用Goatools在线软件(https://github.com/ tanghaibao/GOatools)对抗感基因型NPB/Pi9和NPB之间的差异表达基因进行GO分类富集分析。将值小于0.05的GO分类视为显著富集项。利用KOBAS在线软件(http://kobas.cbi.pku.edu.cn/kobas3)对以上抗感基因型之间的差异表达基因进行KEGG富集分析,将值小于0.05的KEGG通路定义为显著富集通路。
1.4 水稻RNA提取及qRT-PCR验证
参照Trizol总RNA提取试剂说明书,从稻瘟病菌侵染的水稻样品提取总RNA。用NanoDrop 2000分光光度计(Thermo公司)检测RNA质量和浓度。参照FastQuant反转录试剂盒说明书(TIANGEN公司)进行cDNA第1链合成。以EF-Tu(Os03g08020)为内参基因;使用NCBI引物设计工具(Primer3和Primer-BLAST)设计水稻基因RT-PCR引物(表1);使用SYBR Premix ExⅡ(2x)荧光染料法实时定量试剂盒(TaKaRa公司),ABI PRISM7900®HT实时荧光定量PCR检测系统(ABI公司)进行qRT-PCR。每个PCR设置3次重复。按照2方法定量分析qRT-PCR数据。
2 结果与分析
2.1 稻瘟菌诱导水稻基因表达芯片数据的分析
根据12 503个水稻基因的芯片检测数据,转抗稻瘟病基因株系(NPB/Pi9)在接种后12、24和36 h的表达量分别与其0 h的表达量比较,共检测到7 754个响应稻瘟菌侵染的差异表达基因,包括4 323个上调和3 743个下调差异表达基因(图1)。感病基因型日本晴(NPB)在接种后12 h、24 h和36 h共有7 385个差异表达基因,包括4 153个上调和3 588个下调差异表达基因。按照接种时间进程,NPB/Pi9和NPB的差异表达基因数目在接种后24 h达到高峰,在接种后36 h NPB/Pi9的差异表达基因数目略有下降,NPB的差异表达基因数目则显著下降(图2)。
比较抗病基因型NPB/Pi9和感病基因型NPB在相同时间点(接种后0 h、12 h、24 h或36 h)的基因表达量,共获得4 065个差异表达基因,其中接种后0 h、12 h、24 h的差异表达基因数目较少,接种后36 h的差异表达基因数目显著增加(图3)。在4个时间点均呈差异表达的差异表达基因有76个(图4),这些差异表达基因主要编码蛋白激酶、NBS-LRR抗病蛋白、转录因子、转运相关蛋白、E3连接酶等。
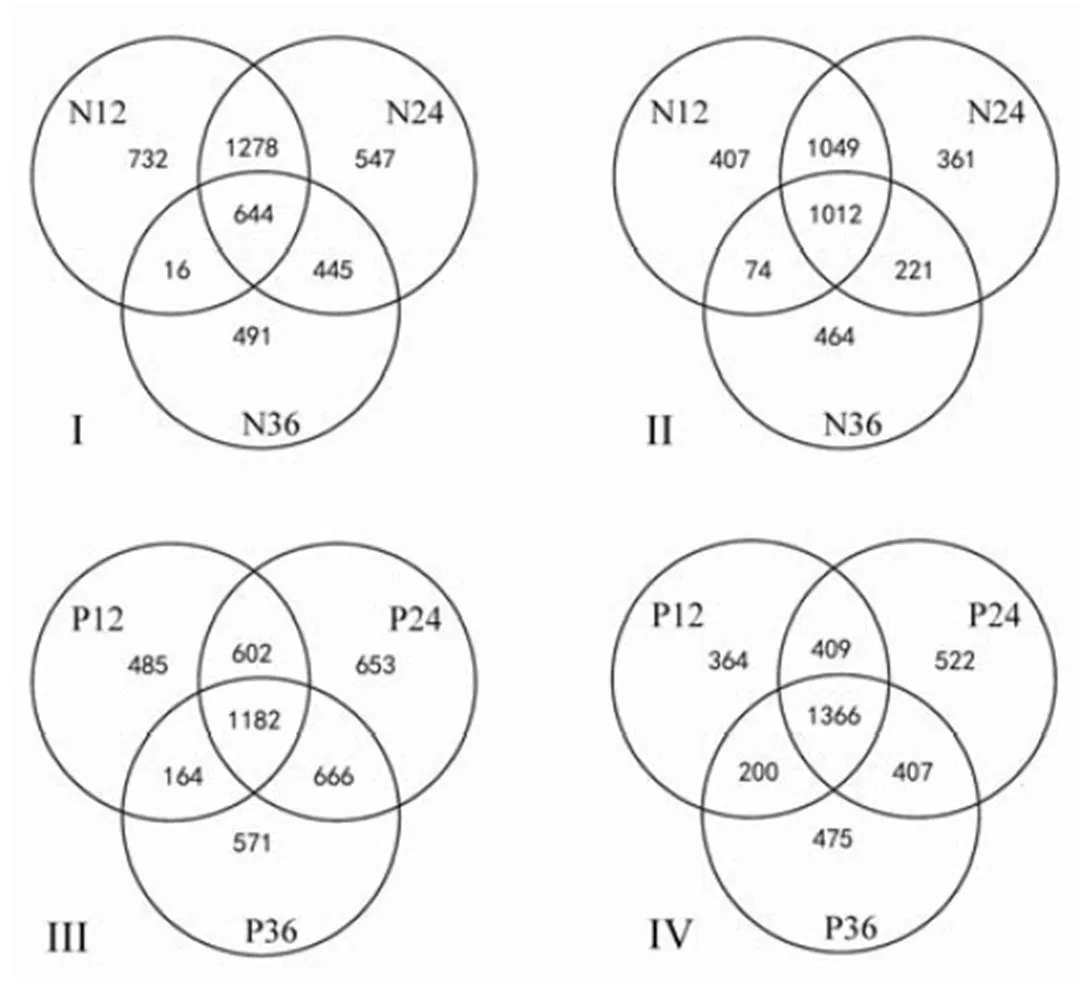
Ⅰ和Ⅲ表示上调表达基因;Ⅱ和Ⅳ表示下调表达基因;N12、N24和N36表示NPB在接种后12 h、24 h和36 h的差异表达基因数目;P12、P24和P36表示NPB/Pi9在接种后12 h、24 h和36 h的差异表达基因数目。
Fig. 1. Number of differentially expressed genes(DEGs) in NPB/Pi9 and NPB after inoculation.
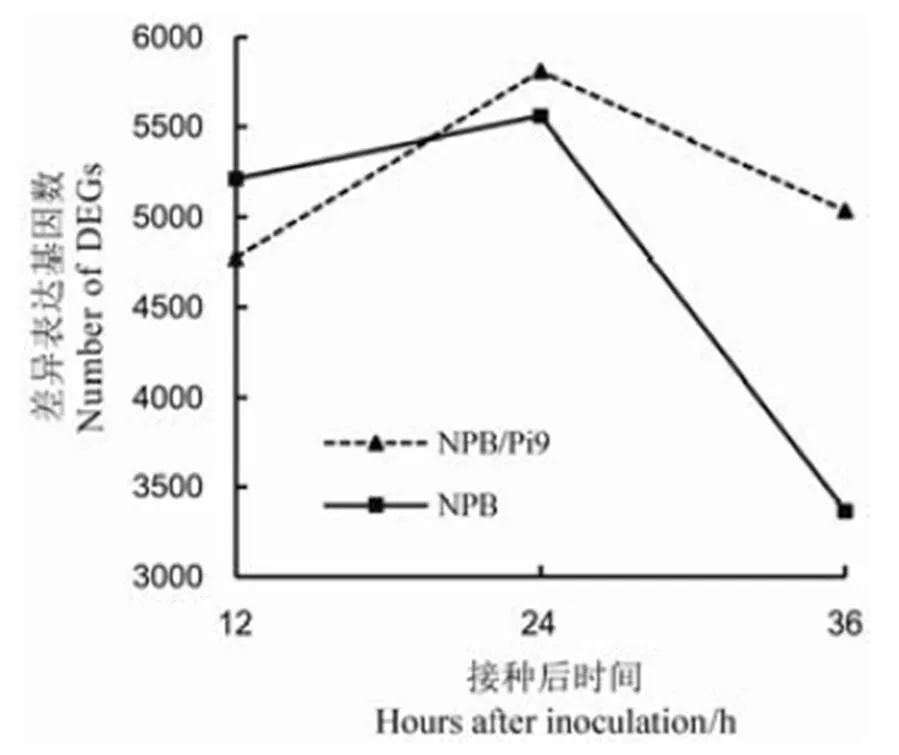
图2 NPB/Pi9和NPB在接种后的差异表达基因数的变化
Fig. 2. Changes of differentially expressed gene (DEG) number in NPB/Pi9 and NPB after inoculation.
2.2 水稻差异表达基因的GO和KEGG分析
利用Goatools在线软件对抗感水稻相同时间点的差异表达基因进行生物学途径(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function, MF)的GO分类富集分析。细胞组分GO分析结果表明,叶绿体或类囊体膜等光合组分的GO分类在接种后0 h及稻瘟病菌侵染的所有时间点显著富集;细胞外区域GO分类在接种后0 h、24、36 h呈显著富集(表2)。根据分子功能GO分析结果,转录调控因子活性、氧化还原酶活性、离子结合活性的GO分类在0 h及所有接种时间点显著富集。生物学过程GO分析结果显示,植物对刺激应答的GO分类在接种后0 h和12 h显著富集;光合作用以及次生代谢的GO分类分别在接种后12 h、24 h和36 h呈显著富集;植物激素相关的GO分类在接种后24 h和36 h显著富集。由此推测,在接种后不同时间,抗病水稻NPB/Pi9与感病水稻NPB的生物学途径、细胞组分和分子功能相关基因呈显著差异表达。
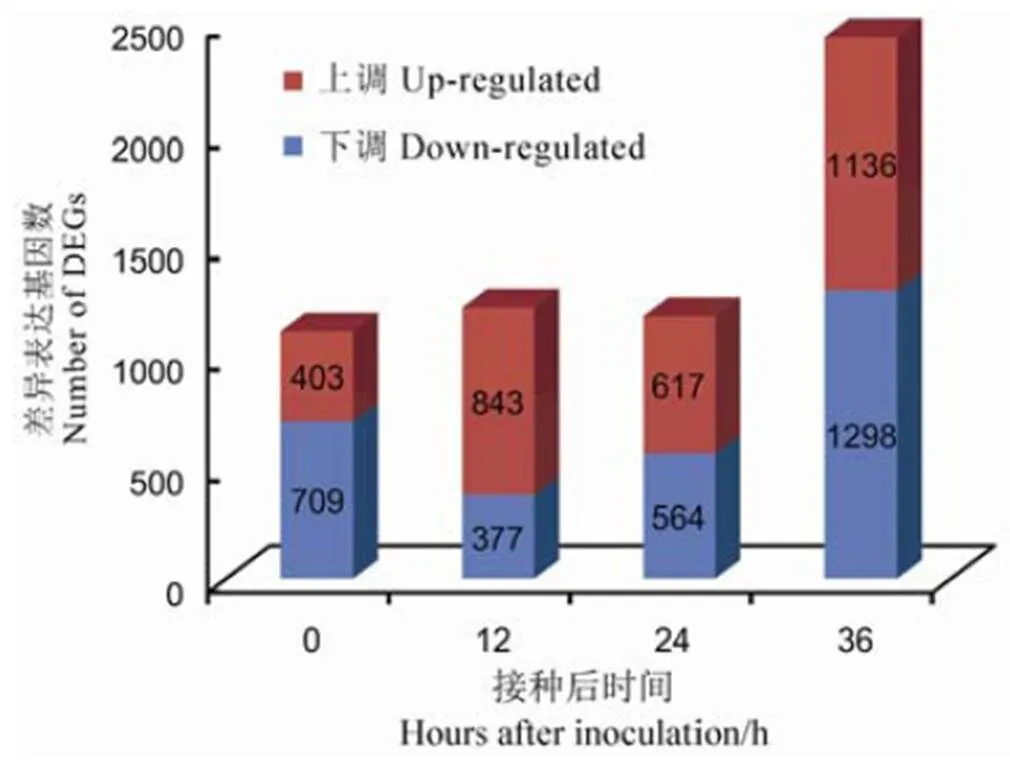
图3 不同接种时间点抗病基因型NPB/Pi9与感病基因型NPB之间的差异表达基因数
Fig. 3. Numbers of differentially expressed genes (DEGs) between the resistant genotype NPB/Pi9 and the susceptible genotype NPB after inoculation.
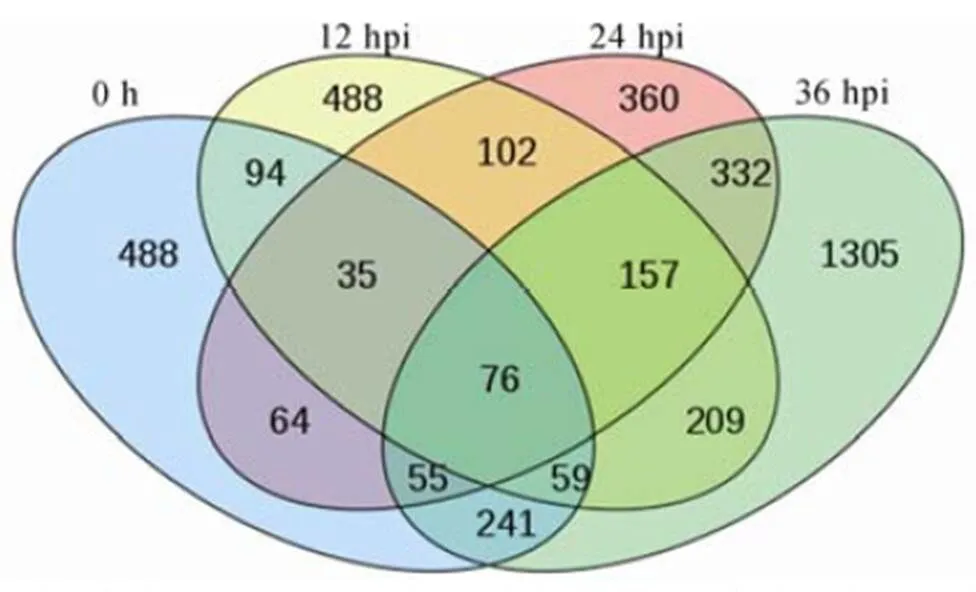
每一种颜色代表一组差异比较,数字代表不同实验独有或共有的基因数,重叠区是代表不同实验组共有的基因,非重叠区代表不同差异比较组独有的基因。0 h, 12 hpi, 24 hpi和36 hpi分别代表接种后0 h, 12 h, 24 h和36 h.
Fig. 4. Venn diagram of differentially expressed genes (DEGs) between the resistant genotype NPB/Pi9 and the susceptible genotype NPB.
利用KOBAS在线软件对抗感水稻的差异表达基因进行KEGG富集分析。在0 h未见任何KEGG通路呈显著富集。光合作用途径的KEGG通路在接种后12 h、24 h和36 h显著富集;苯丙烷合成途径KEGG通路在接种后24 h和36 h有显著富集;苯丙氨酸代谢、类黄酮生物合成和植物激素信号转导途径的KEGG通路在接种后36 h均有显著富集(表3)。在接种后24 h和36 h,抗感水稻的差异表达基因中分别有18个和42个差异表达基因参与苯丙烷生物合成;在接种后36 h,抗感水稻分别有30个差异表达基因参与苯丙氨酸代谢,9个差异表达基因参与类黄酮生物合成,40个差异表达基因参与植物激素信号转导。因此,NPB/Pi9和NPB的次生代谢以及植物激素信号转导途径在稻瘟病菌接种水稻之后基因表达差异显著。

表2 NPB/Pi9与NPB之间差异表达基因的GO富集分析结果
2.3 NBS-LRR抗病基因、多种激酶和转录因子参与NPB/Pi9的稻瘟病抗性
根据GO分类富集分析结果,在接种后12 h、24 h和36 h,抗感基因型之间多个RPM1和类抗病基因呈差异表达(表4)。WAK和DUF26等类受体蛋白激酶,钙依赖性蛋白激酶,丝裂原活化蛋白激酶,以及WRKY、MYB、bZIP、ERF/AP2、bHLH等转录因子在接种后12 h、24 h和36 h呈差异表达,并且相应差异表达基因的数目随接种时间进程而增多。
2.4 NPB/Pi9的稻瘟病抗性与植物激素信号转导途径相关
根据抗感基因型差异表达基因的GO和KEGG分析结果,植物激素水杨酸、茉莉酸和乙烯相关差异基因的分类呈显著富集。其中,水杨酸生物合成途径(GO: 0009697)和对水杨酸的反应(GO: 0009751)分别在接种后12 h和36 h有显著富集,相关差异基因主要是MYB转录因子和凝集素样激酶。

表3 稻瘟病菌接种后NPB/Pi9和NPB之间差异表达基因的KEGG富集分析结果
值0.05的通路被认定为显著富集。
KEGG pathways with correctedvalue 0.05 are regarded as significantly enriched.
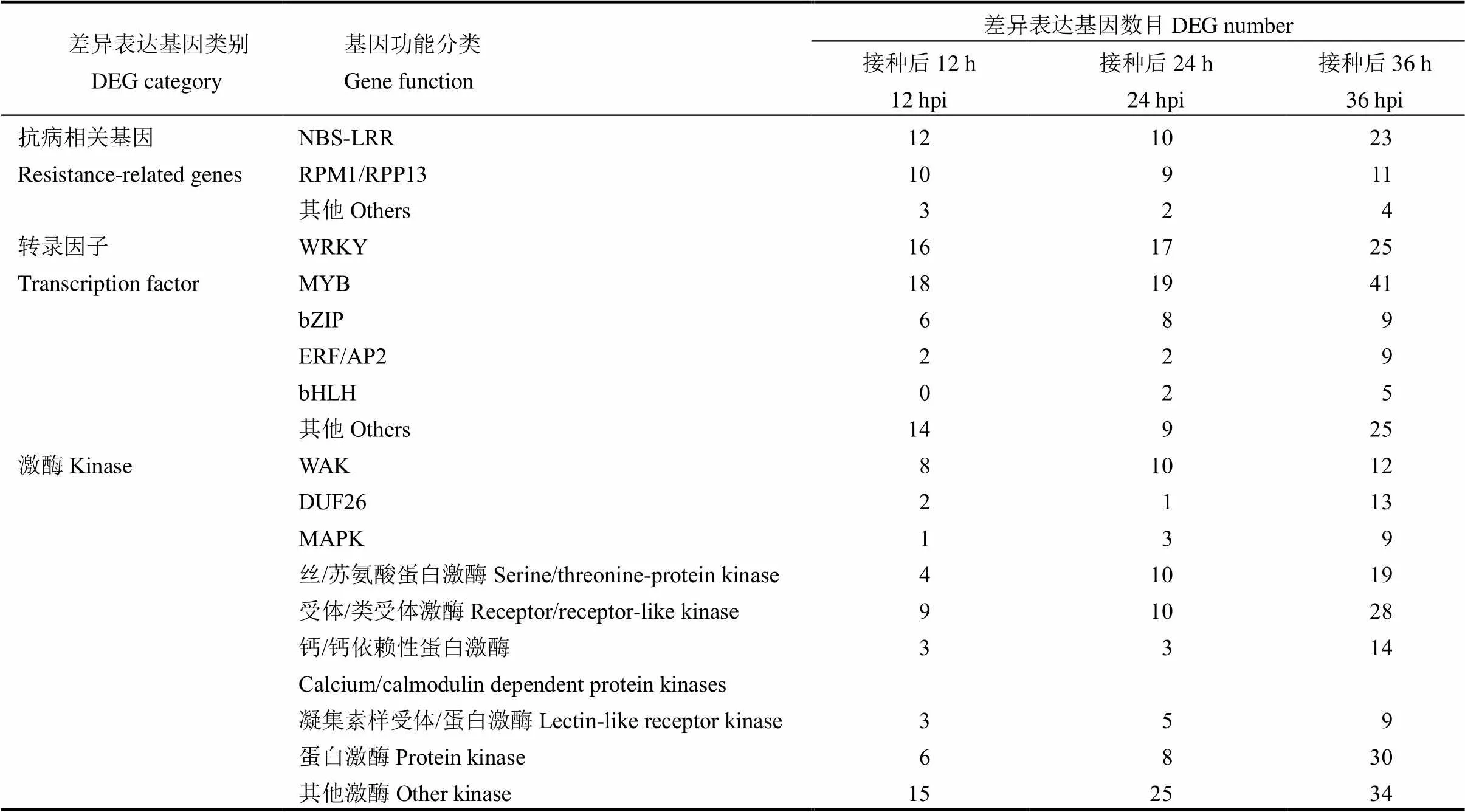
表4 稻瘟菌接种后NPB/Pi9和NPB之间的差异表达基因中与激酶和转录因子相关的基因
在抗感基因型NPB/Pi9和NPB之间与水杨酸信号转导相关的差异表达基因中,6个编码ZIM功能域蛋白的基因在接种后36 h下调表达,2个和3个ZIM基因分别在接种后12 h和24 h下调表达。
在抗感基因型之间与乙烯相关的差异表达基因中,分别有2个乙烯相关基因在接种后12 h和24 h呈差异表达,9个乙烯相关转录因子、2个ACC氧化酶基因(Os01g39860、Os09g27820)以及2个乙烯不敏感基因(Os04g38400和Os07g48630)在接种后36 h呈差异表达。因此,水杨酸、茉莉酸以及乙烯信号转导途径参与调控NPB/Pi9的稻瘟病抗性。
2.5 NPB/Pi9的稻瘟病抗性与植物次生代谢途径相关
苯丙烷生物合成是植物次生代谢的重要途径之一,与植保素、木质素和酚类物质等抗病化合物的合成相关[12]。根据KEGG通路富集结果,抗感基因型NPB/Pi9和NPB的苯丙烷生物合成、类黄酮代谢以及苯丙氨酸代谢相关基因呈显著差异表达,其差异表达基因主要是过氧化物酶前体。过氧化物酶前体可形成过氧化物酶体,后者可以调节活性氧,分解过氧化氢从而提高植物抗病性[13]。
苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL)是植物次生代谢的关键酶之一[14]。在抗感基因型的差异基因中检测到2个基因(Os05g35290,Os02g41680)显著上调表达,1个基因(Os04g43800.1)显著下调表达。肉桂酰辅酶A还原酶(cinnamoyl-CoA reductase,CCR)是木质素生物合成的第一个关键酶[15],苯丙烷生物合成途径的3个基因发生差异表达。因此,植物次生代谢途径参与调控介导的稻瘟病抗性。
2.6 NPB/Pi9的稻瘟病抗性与几丁质酶、硫酸盐转运蛋白相关
植物几丁质酶是一类病程相关蛋白,在植物抵抗病原真菌入侵方面具有重要作用[16]。水稻几丁质酶活性通常与广谱抗病性相关[17]。在接种后36 h,NPB/Pi9相对于NPB有3个几丁质酶(Os04g41620,Os06g51060,Os10g39680)显著上调表达。把NPB/Pi9在接种后24 h和36 h的基因表达量分别与其0 h的基因表达量比较,上述3个几丁质酶和另一几丁质酶(Os08g41100)显著上调表达。
抗感基因型差异表达基因的富集结果显示,硫酸盐转运蛋白参与离子转运和膜相关转运途径。在接种后36 h,NPB/Pi9相对于NPB有4个硫酸盐转运蛋白显著下调。把NPB/Pi9在接种后12 h、24 h、36 h的基因表达量与其0 h的比较,上述4个转运蛋白和另一硫酸盐转运蛋白(Os09g06499)显著下调。采用来源于NPB/Pi9和NPB的另一批稻瘟病接种样品,对部分几丁质酶基因和硫酸盐转运蛋白基因进行qRT-PCR验证(图5),各基因的表达趋势与其芯片数据(表5)基本一致,表明NPB/Pi9的稻瘟病抗性与几丁质酶、硫酸盐转运蛋白相关。
2.7 基因芯片数据的qRT-PCR验证
为了验证水稻基因芯片检测结果,从另一批稻瘟病接种试验取NPB/Pi9和NPB水稻样品,从转录因子、病程相关基因等不同功能类别挑选差异表达基因进行定量RT-PCR分析。根据芯片检测数据(表6)和qRT-PCR验证结果(图6),各差异表达基因表达量的变化趋势基本一致。
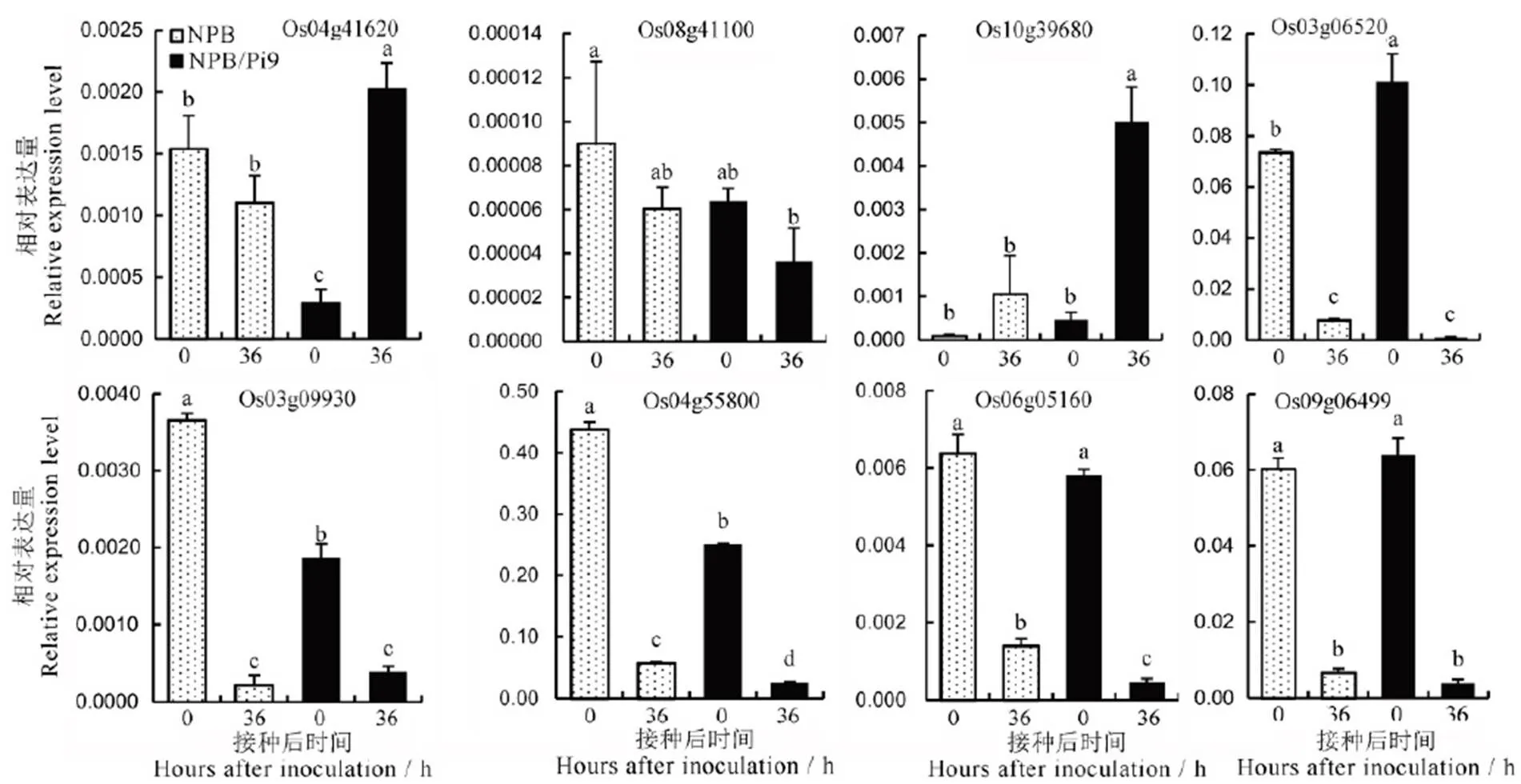
数据为平均数±标准差。相同小写字母表示处理间差异不显著(P<0.05)。表6同。
Fig. 5. qRT-PCR analysis of differentially expressed genes conferring chitinase and sulfate transporter after inoculation.
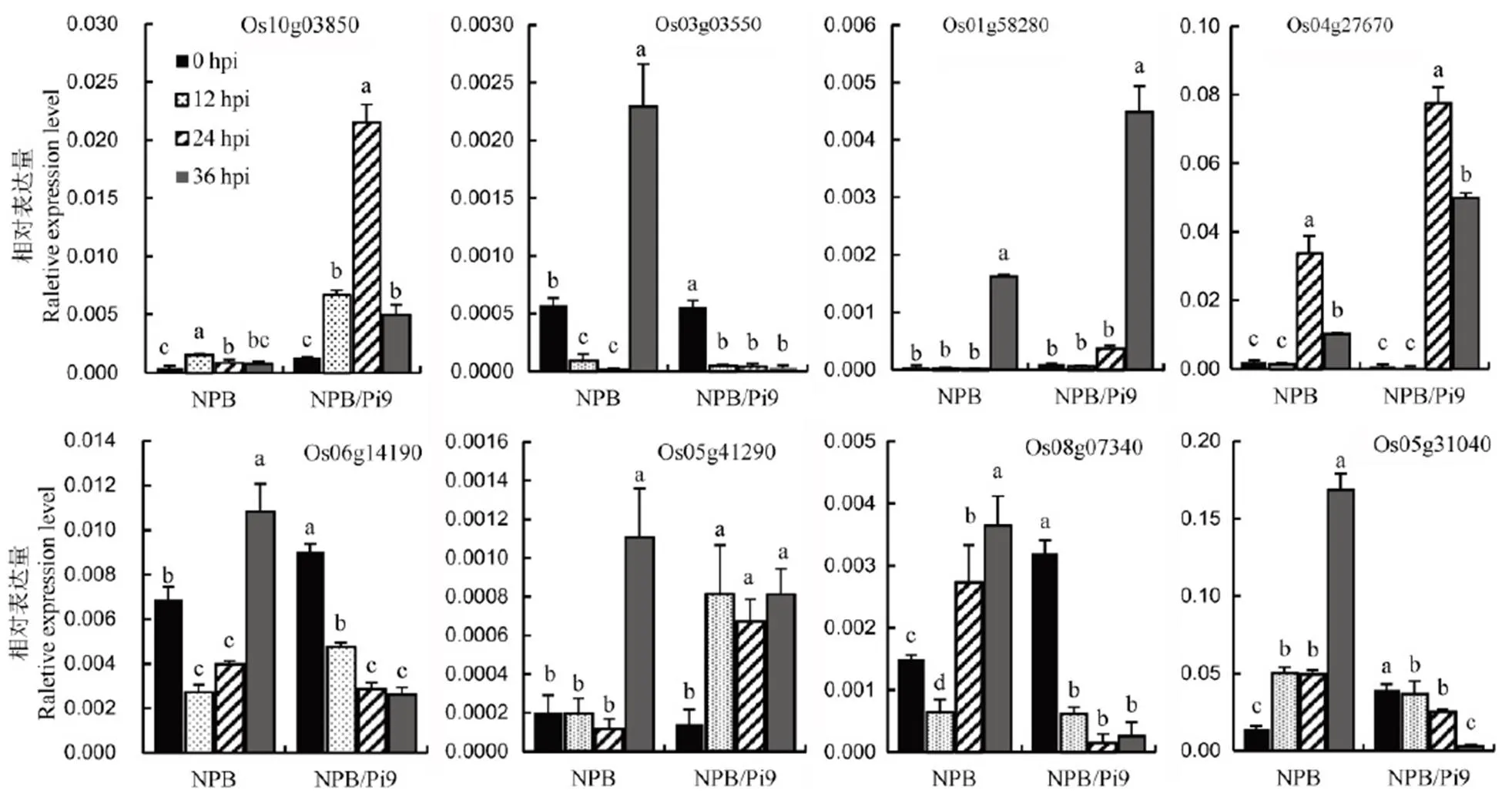
图6 NPB/Pi9和NPB接种后差异表达基因相对表达量的qRT-PCR分析
Fig. 6. qRT-PCR analysis of relative expression levels of differentially expressed genes at different hours after inoculation in NPB/Pi9 and NPB.
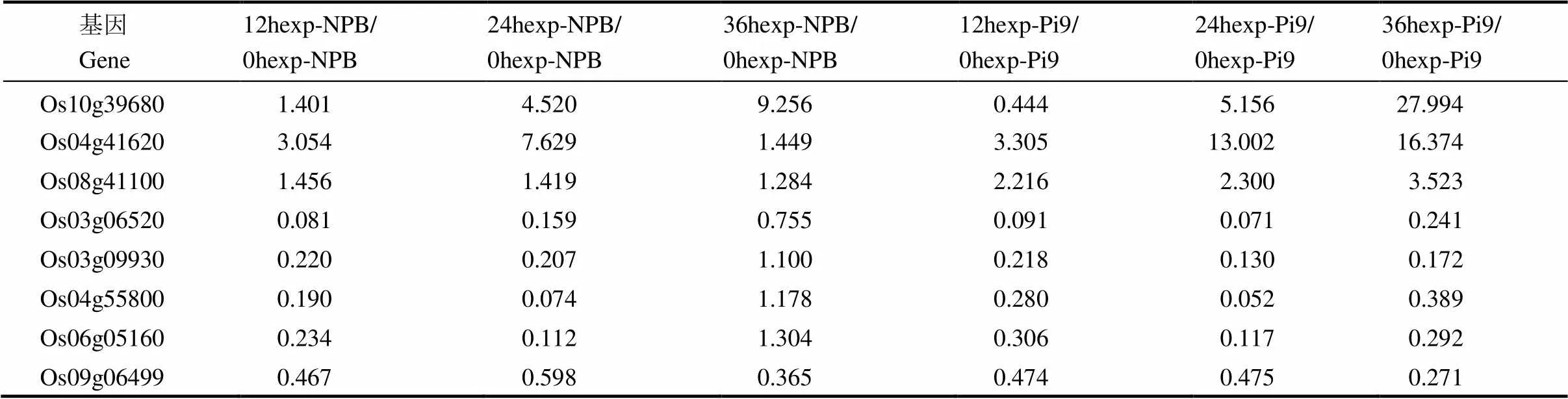
表5 NPB/Pi9和NPB水稻基因型中几丁质酶和硫酸盐转运蛋白的基因芯片检测数据
NPB(或NPB/Pi9)的每个水稻基因在接种后12 h、24 h和36 h的基因表达量的芯片检测量值,分别与该水稻基因型的相同基因在接种0 h的芯片检测量值相比,计算两个量值的比值。0hexp, 12hexp, 24hexp, 36hexp分别表示在接种0, 12, 24 和36 h后的基因相对表达量。
The gene-chip detection value of each rice gene in NPB or NPB/Pi9 at 12, 24 and 36 h after inoculation, respectively, was compared to that of the same gene in the same rice genotype at 0 h, by calculating the ratio of the value of 12 h (24 h or 36 h) after inoculation to the value of 0 h.
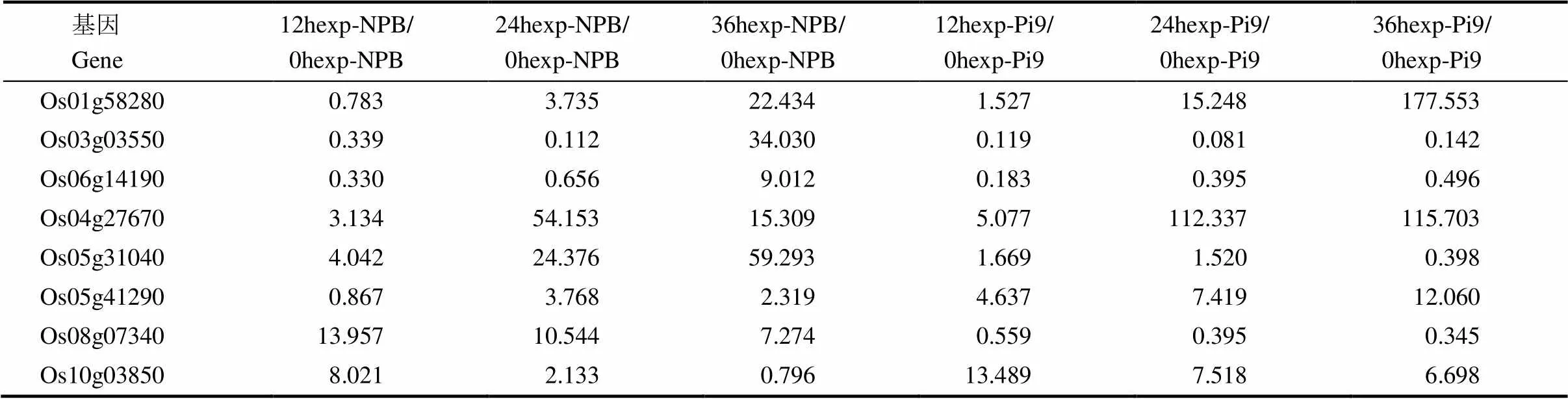
表6 NPB/Pi9和NPB水稻基因型中不同功能类别水稻基因的芯片检测数据
3 讨论
本研究采用遗传背景差异较小的抗病水稻与感病水稻进行比较转录组分析,NPB/Pi9和NPB的遗传差异仅在于转基因的有无。基于单基因差异的水稻遗传材料的比较转录组研究,更利于解析抗感水稻材料的基因表达差异,进而阐明抗病调控的分子机理。
NBS-LRR抗病基因在植物ETI免疫途径中起重要作用[5]。抗病水稻NPB/Pi9与感病水稻NPB有76个基因在接种后0 h、12 h、24 h和36 h呈差异表达,其中多个差异表达基因属于基因家族。一类的基因通常呈组成型表达[18],并且0 h和接种后各时间点抗感水稻的差异表达基因在生物学过程、分子功能、细胞组分方面均有GO分类显著富集,由此可以解释抗感水稻的差异表达基因在上述4个时间点呈差异表达。
根据水稻差异表达基因的GO分析结果,在接种后0 h,NPB/Pi9和NPB在生物学过程(包括对刺激的应答、氧化还原过程、转录过程),分子功能(包括离子结合活性、转录调控因子活性、氧化还原酶活性),细胞组分(包括细胞外区域、叶绿体组分、类囊体膜)方面分别发生显著差异表达,说明在稻瘟菌侵染之前,NPB/Pi9抗病水稻株系的转录调控就已发生显著改变,这种转录重编程现象的根源在于转基因的作用。Zhang等[9]对NPB遗传背景的水稻基因RNAi抑制表达转基因株系和NPB进行比较转录组分析,在接种后0 h,检测到两种水稻的多个抗病相关基因发生显著差异表达,说明基因抑制表达导致抗病相关基因的转录调控发生改变,进而导致上述RNAi株系和NPB在接种稻瘟病菌后分别呈抗病和感病表型。
抗感水稻光合作用过程和光合组分的GO分类在接种后12 h、24 h和36 h显著富集,并且抗感水稻光合作用KEGG通路在接种后12 h、24 h和36 h也显著富集,所以稻瘟菌接种导致两种水稻基因型的光合功能相关基因产生显著差异表达。这些可以理解为抗病水稻NPB/Pi9受病菌影响较小,感病水稻NPB因病菌侵染使得其基因表达发生显著变化。此外,在接种后0 h,抗感水稻的差异表达基因的GO分类还显著富集于叶绿体组分和类囊体膜,说明NPB/Pi9和NPB在稻瘟菌侵染之前就已存在光合组分相关基因的差异表达。在接种后0 h,NPB/Pi9和NPB之间发生离子结合活性、氧化还原酶活性、转录调控因子活性等分子功能的差异表达,可能影响其叶绿体、类囊体膜等细胞组分的功能,从而导致差异表达。
细胞外区域是水稻与稻瘟菌分子互作的主要场所[19]。在接种后24 h和36 h,抗感水稻的差异表达基因的GO分类显著富集在细胞外区域,说明稻瘟菌在侵染点与水稻细胞表面的分子互作异常活跃。抗感水稻的WAK、DUF26等多个类受体蛋白激酶在接种12 h、24 h和36 h分别呈显著差异表达。这些激酶属于跨膜蛋白,可能在PTI免疫中起到模式识别受体(PRR)[20]的作用。此外,在接种后0 h,抗感水稻的差异表达基因的GO分类也显著富集在细胞外区域,说明在稻瘟病菌侵染之前,就发生有胞外区域相关水稻基因的差异表达。根据生物学过程的GO分类富集结果,在接种后0 h,抗感水稻差异表达基因的GO分类在植物对刺激应答有显著富集。因此,NPB/Pi9和NPB的上述胞外相关基因、刺激应答相关基因在接种后0 h呈差异表达的结果,可以通过NPB/Pi9水稻株系中抗病调控途径发生转录重编程作用加以解释。
WRKY、MYB和NAC转录因子参与水稻对生物胁迫和非生物胁迫的反应以及抗病分子调控[21]。在接种后36 h,抗感基因型之间检测到25个WRKY转录因子发生差异表达,其中显著上调。过表达促进水稻植株体内茉莉酸积累,增强水稻稻瘟病抗性[22]。本研究检测到负调控JA信号转导途径的ZIM功能域蛋白基因,在NPB/Pi9中显著下调表达,说明在稻瘟菌侵染条件下转抗病株系的JA信号途径被激活。因此,基因的抗病功能可能与WRKY30的调控通路相关。
根据抗感基因型NPB/Pi9和NPB差异表达基因的分析结果,苯丙烷生物合成、苯丙氨酸代谢、类黄酮代谢等植物次生代谢途径参与调控介导的稻瘟病抗性。在接种后36 h,MYB转录因子呈显著差异表达,MYB转录因子调控苯丙烷的生物合成进而合成木质素、植保素等抗菌物质[12]。木质素合成的关键酶PAL也发生差异表达,表明木质素的积累在介导的稻瘟病抗性中具有重要作用。
一类的抗病基因参与ETI免疫途径[5]。植物R蛋白识别病菌分泌到植物细胞内的效应蛋白,激活ETI免疫反应,导致基因的激活和抗病激素的合成[19]。在稻瘟菌侵染后期,NPB/Pi9的水杨酸信号转导途径被激活,几丁质酶基因显著上调表达,这些为ETI免疫反应的典型结果。在另一方面,作为水稻与稻瘟菌互作主要场所的细胞外区域在接种后0 h,24 h和36 h发生差异基因GO分类显著富集,在接种后0 h和12 h发生植物对刺激应答的GO分类显著富集,并且WAK、DUF26蛋白激酶等跨膜蛋白在接种后12 h、24 h和36 h分别呈显著差异表达,可能参与水稻细胞和病菌的分子识别,因此NPB/Pi9和NPB差异表达还与PTI免疫途径相关。研究表明,ETI和PTI共用WRKY等转录因子的转录调控和MAPK级联信号转导等下游信号途径[23]。NPB/Pi9和NPB的WRKY转录因子、MAPK激酶、茉莉酸和乙烯信号途径在相同时间点呈差异表达。综上所述,抗感基因型的基因差异表达同时与ETI免疫途径和PTI免疫途径相关,ETI和PTI相互联系并在介导的稻瘟病抗性中发挥作用。
本研究选取不同功能类别的水稻差异表达基因进行qRT-PCR分析,对水稻基因芯片数据进行验证。qRT-PCR实验结果所显示的差异表达趋势与相应芯片数据基本一致。个别差异表达基因或个别接种时间点的qRT-PCR和芯片数据表现不完全一致的结果,可能源于qRT-PCR和基因芯片实验条件的波动,或由于水稻RNA样品来源于不同稻瘟病接种试验。
水稻抗稻瘟病基因是水稻杂交育种的重要抗源基因[2]。在转录水平上解析相关的水稻与稻瘟菌互作的分子机制以及水稻抗病调控途径,其研究结果丰富了植物抗病分子基础,为通过杂交育种方法改良水稻稻瘟病抗性以及通过基因编辑和转基因技术创制水稻抗病种质资源提供理论依据。
[1] Deng Y, Zhai K, Xie Z, Yang D, Zhu X, Liu J, Wang X, Qin P, Yang Y, Zhang G, Li Q, Zhang J, Wu S, Milazzo J, Mao B, Wang E, Xie H, Tharreau D, He Z.Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance[J]., 2017, 355(6328): 962-965.
[2] Jain P, Singh P K, Kapoor R, Khanna A, Solanke A U, Krishnan S G, Singh A K, Sharma V, Sharma T R. Understanding host-pathogen interactions with expression profiling of NILs carrying rice-blast resistancegene[J]., 2017, (8): 93-93.
[3] Liu J, Hu Y, Ning Y, Jiang N, Wu J, Jeon J, Xiao Y, Liu X, Dai N, Wang G. Genetic variation and evolution of theblast resistance locus in the AA genomespecies[J]., 2011, 54(5): 294-302.
[4] Wu J, Kou Y, Bao J, Li Y, Tang M, Zhu X, Ponaya A, Xiao G, Li J, Li C, Song M Y, Cumagun C J, Deng Q, Lu G, Jeon J S, Naqvi N I, Zhou B. Comparative genomics identifies theavirulence effector AvrPi9 that triggers-mediated blast resistance in rice[J]., 2015, 206(4): 1463-1475.
[5] Jones J D, Dang I J L. The plant immune system[J]., 2006, 444(7117): 323-329.
[6] Liu W, Liu J, Triplett L, Leach J E, Wang G L. Novel insights into rice innate immunity against bacterial and fungal pathogens[J]., 2014, 52(11): 213-241.
[7] Kou Y, Wang S. Broad-spectrum and durability: Understanding of quantitative disease resistance[J]., 2010, 13: 181-185.
[8] Wei T, Ou B, Li J, Zhao Y, Guo D, Zhu Y, Chen Z, Gu H, Li C, Qin G, Qu L J. Transcriptional profiling of rice early response toidentifiedas important regulators in rice blast resistance[J]., 2013, 8(3): e59720.
[9] Zhang Y, Zhao J, Li Y, Yuan Z, He H, Yang H, Qu H, Ma C, Qu S. Transcriptome analysis highlights defense and signaling pathways mediated by ricegene with partial resistance to[J]., 2016, 7: 1834.
[10] Li W, Liu Y, Wang J, He M, Zhou X, Yang C, Yuan C, Wang J, Chern M, Yin J, Chen W, Ma B, Wang Y, Qin P, Li S, Ronald P, Chen X. The durably resistant rice cultivar Digu activates defence gene expression before the full maturation ofappressorium[J]., 2015, 16: 973-986.
[11] Kitony J K. 水稻CO39品种及其5个近等基因系对稻瘟病感染应答的转录组分析[D]. 福州: 福建农林大学, 2016.
Kitony J K. Transcriptome analysis of rice cultivar CO39 and its five Near-Isogenic Lines in response to blast fungus infection[D]. Fuzhou: Fujian University of Agriculture and Forestry, 2016. (in Chinese with English abstract)
[12] 王玉, 杨雪, 杨蕊菁, 王玉霞, 杨飞霞, 夏鹏飞, 赵磊. 调控苯丙烷类生物合成的MYB类转录因子研究进展[J]. 安徽农业大学学报, 2019, 46(5): 859-864.
Wang Y, Yang X, Yang R Q, Wang Y X, Yang F X, Xia P F, Zhao L. Advances in research of MYB transcription factors in regulating phenylpropane biosynthesis[J]., 2019, 46(5): 859-864. (in Chinese with English abstract)
[13] 崔慧萍, 周薇, 郭长虹. 植物过氧化物酶体在活性氧信号网络中的作用[J]. 中国生物化学与分子生物学报, 2017, 33(3): 220-226. (in Chinese with English abstract)
Cui H P, Zhou W, Guo C H. The role of plant peroxisomes in ROS signaling network[J]., 2017, 33(3): 220-226. (in Chinese with English abstract)
[14] 杜欣谊. 苯丙氨酸解氨酶的研究进展[J]. 现代化农业, 2016(7): 24-26.
Du X Y. Research advances on phenylalanine ammonia-lyase gene[J]., 2016(7): 24-26. (in Chinese with English abstract)
[15] 李魏, 谭晓风, 陈鸿鹏. 植物肉桂酰辅酶A还原酶基因的结构功能及应用潜力[J]. 经济林研究, 2009, 27(1): 7-12.
Li W, Tan X F, Chen H P. Structural function and application potential of cinnamoyl-CoA reductase gene in plant[J]., 2009, 27(1): 7-12. (in Chinese with English abstract)
[16] Kasprzewska A. Plant chitinases-regulation and function[J]., 2003, 8(3): 809-824.
[17] 许明辉, 李成云, 李进斌, 谭学林, 田颖川, 陈正华, 唐祚舜, 田文忠. 转几丁质酶-葡聚糖酶基因水稻稻瘟病抗谱分析[J]. 中国水稻科学, 2003, 17(4): 307-310.
Xu M H, Li C Y, Li J B, Tan X L, Tian Y C, Chen Z H, Tang Z S, Tian W Z. Analysis on resistant spectrum to rice blast in transgenic rice lines with chitinase gene and β-1, 3-glucanase gene[J]., 2003, 17(4): 307-310. (in Chinese with English abstract)
[18] Qu S, Liu G, Zhou B, Bellizzi M, Zeng L, Dai L, Han B, Wang G L. The broad-spectrum blast resistance geneencodes a nucleotide-binding site-leucine-rich repeat protein and is a member of a multigene family in rice[J]., 2006, 172(3): 1901-1914.
[19] Liu W, Liu J, Ning Y, Ding B, Wang X, Wang Z, Wang G. Recent progress in understanding PAMP- and effector-triggered immunity against the rice blast fungus[J]., 2013, 6(3): 605-620.
[20] Zipfel C. Pattern-recognition receptors in plant innate immunity[J]., 2008, 20(1): 10-16.
[21] Ramamoorthy R, Jiang S Y, Kumar N, Venkatesh P N, Ramachandran S. A comprehensive transcriptional profiling of thegene family in rice under various abiotic and phytohormone treatment[J]., 2008, 49(6): 865-879.
[22] Peng X, Hu Y, Tang X, Zhou P, Deng X, Wang H, Guo Z. Constitutive expression of ricegene increases the endogenous jasmonic acid accumulation,gene expression and resistance to fungal pathogens in rice[J]., 2012, 236(5): 1485-1498.
[23] Pitzschke A, Schikora A, Hirt H. MAPK cascade signalling networks in plant defence[J]., 2009, 12(4): 421-426.
Comparative Transcriptome Analysis of Transgenic Rice Line Carrying the Rice Blast Resistance Gene
XU Zhaomeng1, 2, LI Lihua2, 3, GAO Xiaoqing1, YUAN Zhengjie2, LI Xin2, TIAN Xudan1, 2, WANG Lanlan2,QU Shaohong2, *
(1College of Chemistry and Life Sciences, Zhejiang Normal University, Jinhua 321004, China;2Institute of Virology and Biotechnology, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China;3College of Plant Protection, Hunan Agricultural University, Changsha 410128, China;*Corresponding author, E-mail:squ@mail.zaas.ac.cn)
【】The purpose of the study is to analyze the mechanism behindgene-mediated rice blast resistance at the transcriptional level, and to lay a theoretical basis for breeding disease-resistant rice cultivars. 【】Rice cultivar Nipponbare (NPB) and NPB-transgenic line carrying therice blast resistance gene (NPB/Pi9) were inoculated withLeaf tissues were sampled at 0 h, 12 h, 24 h, and 36 h after inoculation, respectively. Gene-chip transcriptome analysis of 12503 rice genes was performed using the rice samples, and the microarray data were verified by qRT-PCR of a set of differentially expressed genes (DEGs). 【】7754 DEGs were identified by comparing the gene expression levels in the resistant NPB/Pi9 line at 12 h, 24 h and 36 h after inoculation, respectively, to that at 0 h after inoculation. Accordingly, 7385 DEGs were identified in the susceptible rice NPB at the same time points. At 36 h after inoculation, the DEG number of NPB/Pi9 was significantly higher than that of NPB. 4065 DEGs were identified by comparing the gene expression levels in NPB/Pi9 and NPB at the same time points; there were significantly more DEGs at 36 h after inoculationthan at 0 h, 12 hor 24 h after inoculation. Therefore, NPB/Pi9 exhibited more intensive defense responses to. Gene Ontology (GO) and KEGG analyses were carried out on the DEGs between NPB/Pi9 and NPB at the same time points. The GO terms classifications related to extracellular regions, plant response to stimuli, transcriptional regulation, redox activity, ion binding activity, secondary metabolism and plant hormones were significantly enriched for the time points post inoculation. The KEGG pathways of phenylalanine metabolism, flavonoid biosynthesis and plant hormone signaling were enriched significantly at the time points post inoculation. Distinctive gene expression patterns were observed between NPB/Pi9 and NPB at the same time points for the genes of the effector-triggered immunity (ETI) related salicylic acid signaling pathway and chitinase, and the PAMP-triggered immunity (PTI) related extracellular regions, response to stimuli, and lignin biosynthesis. Moreover, distinctive gene expression patterns were observed between NPB/Pi9 and NPB at the same time points for the genes of WRKY transcription factors, MAPK kinases, jasmonate and ethylene signaling pathways that were utilized by both PTI and ETI. Based on all the results, the differential expression patterns between NPB/Pi9 and NPB are related to ETI and PTI, which are involved with each other and play important roles in the-mediated rice blast rice resistance. 【】 Compared with NPB, the resistant genotype NPB/Pi9 showed more intensive defense responses against. Transcription factors, kinases,genes, chitinases, salicylic acid-, jasmonic acid-, and ethylene-signaling pathways, and plant secondary metabolism play important roles ingene-mediated rice blast resistance.
rice blast; transcriptome; resistance genes; plant hormones; secondary metabolism
S435.111.4+1; S511.034
A
1001-7216(2020)03-0245-11
10.16819/j.1001-7216.2020.9126
2019-11-23;
2020-03-11。
国家自然科学基金面上项目(31272016);浙江省农业科学院省部共建国家重点实验室培育基地开放基金资助项目(2010DS700124-ZZ1906);浙江省农业科学院扶持学科建设2018年B类项目。
