褐牙鲆中miRNAs与甲状腺激素受体TRβ的相互作用研究
2020-05-25刘素萍付元帅张俊玲施志仪
刘素萍,付元帅,喻 杰,张俊玲,施志仪
( 上海海洋大学,农业农村部淡水水产种质资源重点实验室,上海水产养殖工程技术研究中心,水产科学国家级实验教学示范中心,上海 201306 )
MicroRNAs(miRNAs)是一类内源性非编码的、大小在20~25 nt的单链RNA分子,它们具有分布广泛的特点,从植物、线虫到人类的细胞中均有存在[1],microRNA的前体为pre-miRNA,长度约70~80 nt,其两个臂可能分别产生两种microRNA。命名规则为表达量较高的microRNA后面不加任何符号,而表达量较低的microRNA后接*号,例如mmu-miR-7和mmu-miR-7*。研究表明,miRNAs通过结合靶基因3′-非翻译区(3′-UTR)的作用方式,而得以在基因的转录后水平发挥其调控功能[2]。据推测miRNAs可能调控着动物基因组内约1/3以上基因的表达,从而参与众多生理活动的调控[3]。
褐牙鲆(Paralichthysolivaceus)属鲽形目,主要分布于中国、日本沿海,是目前我国重要的海水养殖鱼类。在褐牙鲆胚后时期,有从仔鱼向稚鱼转变的奇特现象,变态期间,眼睛逐渐迁移至身体同一侧,鱼体形态也由侧卧转变为平卧,生活方式也由浮游性转变为底栖性。研究表明,褐牙鲆的变态发育受甲状腺激素(TH)调控,而甲状腺激素主要依赖于与其受体(TRs)结合发挥生物学作用[4-5]。甲状腺激素受体可分为不同亚型,包括TRαA、TRαB和TRβ,三者在褐牙鲆变态期间表达升高,而在变态结束后则恢复到较低的水平,且TRβ在变态高峰期较TRαA、TRαB有更丰富的表达[6]。因而,甲状腺激素受体尤其是TRβ在褐牙鲆变态中发挥着至关重要的作用。
先前已建立了褐牙鲆变态不同时期的miRNAs和转录组文库,筛选出66个在褐牙鲆变态发育的不同阶段具有明显差异性表达的miRNAs[7]。为进一步阐述miRNAs在褐牙鲆变态发育中的作用,笔者利用miRBase、RNAhybrid、PicTarWEB INTERFACE和TargetScanFish 6.2等多种生物信息学软件预测上述差异miRNAs和褐牙鲆TRβ基因3′-UTR的结合情况,发现多个miRNAs分子可能作用于褐牙鲆甲状腺激素受体TRβ基因,因而从中选取pol-miR-125a、pol-miR-214、pol-miR-460-5p、pol-miR-138和pol-miR-125a*共5个miRNAs作为研究对象,利用3′-RACE技术克隆褐牙鲆TRβ基因的3′-UTR序列,并构建含TRβ 3′-UTR的双荧光素酶重组报告载体,利用双荧光素酶报告基因技术检测上述5个miRNAs与褐牙鲆TRβ之间是否存在真实的靶向关系,为后续深入研究甲状腺激素及相关miRNAs调控褐牙鲆变态发育的分子机制提供更多资料。
1 材料与方法
1.1 样品采集
褐牙鲆样品的采集在中国水产科学研究院北戴河中心实验站完成。取1龄半褐牙鲆成鱼,解剖后取脑、肌肉等不同组织样品,用DEPC水冲洗后,立即置于液氮速冻,-80 ℃保存备用。
1.2 主要试剂
Trizol Reagent(Invitrogen公司);Reverse Transcription System(Promega公司);3′-RACE试剂盒、Ex Taq DNA polymerase、pMD®19-T Vector、T4 DNALigase(TaKaRa公司);SacⅠ、XbaⅠ(NEB公司);DNA 凝胶回收试剂盒(天根生化科技有限公司);质粒小提取试剂盒(OMEGA公司);双荧光素酶报告基因检测试剂(翊圣生物科技有限公司);293T细胞;miRNA mimics(吉玛生物科技有限公司),是针对miRNA的成熟体设计并合成的小片段双链miRNA,以下简称为miRNA模拟物;胎牛血清、0.25% Trypsin-EDTA (1×) 胰蛋白酶、青霉素—链霉素溶液、成纤维生长因子、MEM 非必需氨基酸(GIBCO公司);FuGENE®HD转染试剂、pmirGLO载体(Promega公司)。
1.3 总RNA提取和反转录
上述褐牙鲆组织样品采用Trizol法提取总RNA,并将所提取的总RNA样品在NanoDrop 2000c上测定OD260/OD280值,其比值为1.8~2.0,且1%琼脂糖凝胶电泳显示28S和18S条带亮度清晰,说明提取的总RNA质量良好,可满足后续试验要求。然后利用反转录试剂盒,按照说明书的操作步骤将各总RNA样品反转录为cDNA。
1.4 TRβ 3′-UTR序列的克隆
根据美国国立生物技术信息中心TRβ已有序列,设计3′-RACE-outer 引物、3′-RACE-inner 引物,然后按照3′-RACE试剂盒使用说明进行操作,将扩增得到的RACE PCR产物进行胶回收、连接、转化和克隆测序后,得到褐牙鲆TRβ基因的3′-UTR cDNA序列。
1.5 靶向miRNAs预测
通过miRNA靶基因预测软件 miRBase(http:∥www.mirbase.org/),PicTarWEB INTERFACE(http:∥pictar.mdc-berlin.de/cgi-bin/PicTar_vertebrate.cgi),TargetScanFish 6.2(http:∥www.targetscan.org/fish_62/)和 computer-based RNAhybrid(http:∥bibiserv.techfak.uni-bielefeld.de/rnahybrid/submission. html)等预测与褐牙鲆TRβ基因3′-UTR相结合的miRNA分子。
1.6 重组报告载体的构建
根据已克隆的褐牙鲆TRβ 3′-UTR cDNA序列设计构建重组质粒所需目的片段的PCR引物,同时加入SacⅠ和XbaⅠ酶切位点(下划线部分):
3′-TRβSacIF:5′-TCTAGTTGTTTAAACGAGC TCCCAATGGATTCAAGTGAATGC-3′, 3′-TRβXbaIR: 5′-TGCCTGCAGGTCGACTCTAGAGGCAGGCA AGGGGGGGGTT-3′。
利用上述cDNA为模板,3′-TRβSacIF和3′-TRβXbaIR为PCR引物,进行PCR扩增。PCR反应体系包含1.0 μL cDNA、2.0 μL 10×Ex Taq Buffer、1.6 μL dNTP Mixture(2.5 mmol/L)、1.0 μL上游引物(10 μmol/L)、1.0 μL下游引物(10 μmol/L)、1.6 μL MgCl2(25 mmol/L)、0.2 μL ExTaq Ploymerase和11.6 μL ddH2O。PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s、58 ℃ 30 s、72 ℃ 1 min,35次循环;72 ℃ 10 min,4 ℃ 保持。将PCR产物进行胶回收、连接、转化和克隆测序后,抽提质粒DNA,并用NanoDrop 2000c检测含量和纯度。
用SacⅠ和XbaⅠ限制性内切酶对含有目的片段3′-UTR序列的质粒和pmirGLO双荧光素酶报告基因载体进行双酶切,将酶切反应液进行琼脂糖凝胶电泳分析,并将目的片段纯化回收,经含量和纯度测定后用于后续试验;用T4 DNA连接酶将上述酶切的目的片段和报告基因载体进行连接,然后将连接产物转化到DH5α感受态细胞,转化后随机挑取若干阳性克隆抽提重组质粒,并进一步利用双酶切和测序对重组质粒进行鉴定。
1.7 细胞转染试验
将293T细胞置于DMEM高糖培养基内在37 ℃、5%CO2恒温培养箱中进行培养;每隔1~2 d 更换一次培养基;当细胞长满时,用1×PBS清洗两次,0.25%的胰蛋白酶消化90 s,待多数细胞变圆即将脱壁时吸除消化液,加入新鲜培养基,以一传二模式进行传代培养。转染前将293T细胞接种于24孔板,每孔加入500 μL细胞,细胞汇合度达80%时进行转染;转染第一步为将细胞培养液换成50 μL不含抗生素的新鲜DMEM培养基。
用新鲜DMEM培养基将0.8 μg重组质粒、24 pmol miRNA模拟物、0.6 μL平衡至室温的FuGENE®HD分别稀释至25 μL,FuGENE®HD混合液总数与重组质粒、miRNA模拟物混合液总数相等,试验重复3次。随后将FuGENE®HD转染试剂/重组质粒/miRNA 模拟物混合液分别加入相应细胞培养孔中,吹吸混匀,将细胞放回培养箱培养6 h,用新鲜培养基替换细胞培养板中的培养液继续培养18~39 h。
1.8 双荧光素酶报告基因检测
将双荧光素酶报告检测试剂盒中试剂按说明书要求进行配置;吸除细胞培养液,用1×PBS清洗细胞后加入PLB细胞裂解液(200 μL/孔,24孔板),并于室温下摇晃裂解15 min,最终将细胞裂解液分别转移至检测管;在检测仪上选择相应程序,向检测离心管的细胞裂解液中加入 100 μL LAR Ⅱ吹吸混匀,置于发光检测仪中检测萤火虫荧光素酶活性,记录数值a;再向检测离心管中加入100 μL Stop & Glo®试剂,立即吹吸混匀,置于发光检测仪中检测海肾萤光素酶活性,记录数值b;计算a/b。用SigmaPlot 12.5 软件作图并通过单因素方差分析检验各组数据显著性差异,当P<0.05时表示有显著差异,P<0.01时表示有极显著差异。
2 结 果
2.1 TRβ 3′-UTR序列的克隆
采用3′-RACE技术从褐牙鲆成鱼各组织混合总RNA中成功扩增出一段长为437 bp的特异性条带(图1)。将该片段纯化回收后连接到pMD19-T克隆载体,挑选阳性克隆进行DNA测序,结果显示,获得的目的序列为褐牙鲆TRβ基因的3′-UTR cDNA序列。

图1 褐牙鲆TRβ 3′-UTR的克隆
2.2 miRNAs结合位点的预测
pol-miR-125a、pol-miR-138、pol-miR-214、pol-miR-460-5p和pol-miR-125a*与褐牙鲆TRβ基因3′-UTR的潜在结合位点见表1,5个miRNAs的第2~8位种子序列分别与TRβ 3′-UTR完全互补结合,表明褐牙鲆TRβ基因很可能是5个miRNAs的潜在靶基因。
2.3 重组报告载体的构建与鉴定
双酶切验证结果显示,重组质粒可以被酶切成与预期大小相符合的目的片段和pmirGLO报告载体片段(图2)。此外,测序结果也表明上述构建的双荧光素酶报告基因重组质粒是正确的,将其命名为pmirGLO-TRβ-3′-UTR。

表1 褐牙鲆miRNAs和TRβ 3′-UTR的结合位点

(续表1)

图2 重组质粒的双酶切验证
2.4 靶基因的验证结果
双荧光素酶检测结果(图3)显示,转染miR-125a模拟物后,荧光素酶活性明显低于阴性对照组,二者具有极显著差异(P<0.01);同样,与阴性对照组相比,转染pol-miR-138、pol-miR-214和pol-miR-460-5p模拟物后,其荧光素酶活性也都分别显著降低(P<0.05);但是转染miR-125a*模拟物组的荧光素酶活性与阴性对照组相比,无显著差异(P>0.05)(图3)。这些结果表明,pol-miR-125a、pol-miR-138、pol-miR-214和pol-miR-460-5p能在细胞水平直接调节TRβ基因表达,而pol-miR-125a*则不能直接调节TRβ基因的表达。
3 讨 论
本试验通过3′-RACE技术克隆得到褐牙鲆TRβ基因的3′-UTR序列,同时在参考文献[8]的方法后,通过采用PCR扩增技术将特异的限制性内切酶位点以及基因重组技术引入,从而得以成功构建褐牙鲆TRβ基因的双荧光素酶报告重组载体,其可作为多个miRNAs的靶标检测工具;同时本试验还在此基础上通过细胞转染操作技术以及双荧光素酶报告系统分析了5个miRNAs与潜在靶基因褐牙鲆TRβ基因间的作用关系,本试验结果最终证实,褐牙鲆TRβ能够在细胞水平对pol-miR-125a、pol-miR-138、pol-miR-214和pol-miR-460-5p进行直接调节,但并不能在细胞水平对pol-miR-125a*进行直接调节,表明褐牙鲆TRβ不是能够对pol-miR-125a*作用的真实靶基因。
3.1 报告基因载体的选择
首先,pmir-GLO载体是一款目前在众多关于靶基因的研究中使用较多的双荧光素酶报告载体。其试验原理为:利用荧光素作为底物来检测萤火虫荧光素酶的活性[9],在淬灭荧光反应后非常迅速地将腔肠素作为底物加入到试管中随后放至仪器上快速检测海肾荧光素酶的活性,将海肾荧光素作为内参可以用来减少内在因素的一些变化从而减少对试验所造成的影响。其次,本研究采用的miRNA 模拟物,具有能模拟细胞中内源性成熟的miRNA的高水平表达的功能,其直接优点为增强内源性miRNA的调控作用[10]。另外在操作方面,miRNA 模拟物由于只需用转染试剂将其包裹便可较大概率转染进入细胞,此操作由于不需要额外构建载体,所以具有简单、高效、灵敏、安全无毒等显著优势。据此,本试验利用以上两种工具成功构建了应用于miRNA靶标检测的pmirGLO-TRβ-3′-UTR重组报告质粒,并在细胞水平进行了靶基因验证。
3.2 miRNA在鱼类发育中的作用
通过已有的研究结果,已经发现众多miRNAs在褐牙鲆变态期间呈现出非常明显的差异性表达[7]。构建斑马鱼(Daniorerio)不同发育阶段的miRNA文库后,鉴定出了与斑马鱼生长发育相关的217 个miRNAs[11],其中有一部分的miRNAs功能已经被成功注释,如pol-miR-125b被发现与斑马鱼脑发育相关[12]以及pol-miR-126 和pol-miR-150 能够协同调控红细胞与巨噬细胞的发育状况[13],而pol-miR-143与斑马鱼心室的形成联系紧密[14],以及pol-miR-451能够对斑马鱼红细胞成熟程度进行相关调控[15]等。另外在青鳉(Oryziaslatipes)中,同样有许多miRNAs能够参与调控其生长发育,如pol-miR-202-5p与青鳉的生殖细胞特异相关,在青鳉的性别分化以及生殖细胞发育等方面发挥着非常重要的作用[16]。卢荣华等[17]综述了9种鱼类的miRNAs序列收录情况,多种miRNAs参与了鱼类的生长、发育及诸多生理过程。这些研究表明,miRNAs在鱼类生长、发育和变态中可能发挥极其重要的作用。
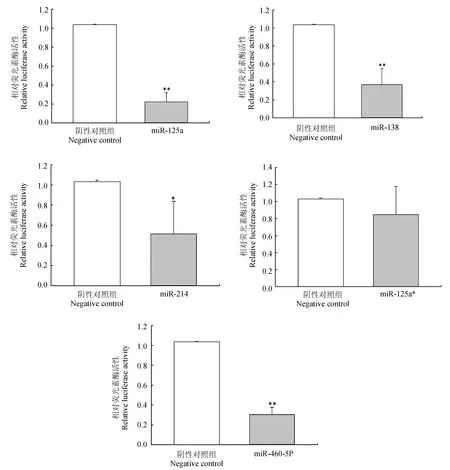
图3 转染各miRNAs模拟物后重组质粒荧光素酶的活性变化
3.3 miRNAs与TRβ基因的作用关系
以上信息表明,甲状腺激素及其受体基因在褐牙鲆变态中的发挥极其重要的作用,因此,本研究首先利用生物信息学方法对作用于褐牙鲆TRβ基因的miRNAs进行预测,综合考虑后从中挑选出了5个可能性最大的miRNAs,随后利用双荧光素酶报告基因技术分别检测了这5个miRNAs在细胞水平对TRβ基因表达的作用结果。通过文献查阅可知,这5个miRNAs在肿瘤发生、增殖和迁移中有较多的报道[18-20],但目前在鱼类中的研究较少。通过已有资料显示,pol-miRNA-214能够参与调控斑马鱼肌肉细胞的发育[21-22];pol-miRNA-138在斑马鱼心脏发育过程中, 能控制心房的发育,在敲除pol-miR-138后,可使斑马鱼出现心房心室发育异常以及心脏功能异常等现象[23]。谢彩霞等[24]研究报道,pol-miR-125b在褐牙鲆变态发育过程中有显著性表达,而pol-miR-125b与pol-miR-125a 高度同源,这一结果表明,pol-miR-125a 极有可能与褐牙鲆的变态发育存在某种联系。并且,这5个miRNAs均为先前筛选鉴定出来的在褐牙鲆变态期间存在差异表达的miRNAs范围之内,这些研究结果综合表明,以上5个miRNAs(pol-miR-125a、pol-miR-138、pol-miR-214、pol-miR-460-5p和pol-miR-125a*)在鱼类发育以及褐牙鲆变态中极有可能发挥着重要的作用,对以上所有资料信息进行综合考虑后,选择这5个miRNAs作为研究对象,并最终在细胞水平上成功验证了其与褐牙鲆TRβ基因之间的作用关系,为后续继续深入研究这些miRNAs在褐牙鲆变态及发育中的功能及作用机制奠定了理论基础。
4 结 论
本试验通过3′-RACE技术克隆得到褐牙鲆TRβ基因的3′-UTR序列,并利用PCR扩增、基因重组技术成功构建了应用于miRNAs靶标检测的pmirGLO-TRβ-3′-UTR重组报告载体;通过细胞转染和双荧光素酶报告基因技术分析了5个miRNAs与褐牙鲆TRβ基因间的作用关系,结果表明,褐牙鲆TRβ是pol-miR-125a、pol-miR-138、pol-miR-214和pol-miR-460-5p在细胞水平直接调节的靶基因,而不是pol-miR-125a*作用的真实靶基因。
