龙须菜四分孢子体发育过程的WGCNA分析❋
2020-04-24姜敏杰毕桂萁王津果张敬宇胡依依李晓东隋正红
姜敏杰, 毕桂萁, 王津果, 张敬宇, 胡依依, 李晓东, 刘 娟, 隋正红
(中国海洋大学海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003)
红藻龙须菜(Gracilariopsislemaneiformis)可提取琼胶,也可作为鲍鱼饵料[1-3],已经成为中国仅次于海带的第二大栽培海藻[4]。另外龙须菜兼具吸收氮磷,缓解富营养化的修复功能[5]。目前共有3个中国认定的良种:“981”[6],“2007”[7]和“鲁龙1号”[8]。对不同良种生理性状的研究发现“981”具备与其他良种不同的特性[6,9-12],其在四分孢子体成熟时放散孢子数量低,孢子畸形,即表现出低育的特性[10-12]。此前对龙须菜的繁殖缺乏分子层面的研究,981品系的低育特性为龙须菜的育性相关研究提供了良好材料。另外目前龙须菜的栽培产业仍以营养繁殖为主,低育性状也具有更高的产业价值。
近年来测序技术发展和成本降低使得组学分析手段更加多样化,转录组和表达谱数据也成为常见的原始分析数据之一。从高通量基因表达数据中筛选挖掘出所关注的基因信息成为转录组分析中必要的一环。加权基因共表达网络分析(Weighted Gene Co-Expression Network Analysis, WGCNA)侧重于基因之间的相关性以及基因表达与所研究性状之间的关联性[13],是一种高效、准确的生物信息学分析技术。2005 年,Zhang 和Horvath[14]最早提出WGCNA分析理论。2008 年,Langfelder和Horvath[15]在该研究方法的基础上开发了WGCNA的R软件包,其分析思路是定义基因共表达相关矩阵并确定基因网络形成的邻接函数,进而计算不同节点的相异系数并形成相异矩阵,根据相异矩阵进行层次聚类后依此将基因划分成以枢纽基因(Hub gene)为核心的基因共表达模块(Module),进一步将模块与性状特征相关联从而提取出目的信息[13]。这一技术被广泛应用于动植物性状的调控机制解析上,如对玉米花期基因[16]、胡杨异形叶发育[17]、苹果黄皮突变[18]、番茄果实成熟[19]以及雨生红球藻中虾青素的积累[20]等过程的相关分析中。
针对龙须菜的各类组学研究也在近几年相继展开。Zhou等[21]通过HiSeq 2000测序预测龙须菜基因组大小约为97 Mb,基因数约为3 490个以及GC%含量为48%。Hu等[22]在基因组survey结果的基础上开发了丰富的简单序列重复(SSR)标记,并用多态性SSR标记对龙须菜不同地理群体进行遗传多样性分析。Huang等[23]对龙须菜野生型和绿色突变型的转录组数据比对分析后认为藻红蛋白、藻红胆素和叶绿素光捕捉复合体的表达水平改变导致突变的形成。Sun等[24]在对981品系的全基因组分析中首次报道了与植物激素如生长素,水杨酸和茉莉酮酸等相关的信号通路。在之前针对龙须菜四分孢子体发育放散时期的生理研究中,认为四分孢子体的发育时期可划分成未成熟期(I期)即四分孢子囊未形成时期、成熟前期(II期)即四分孢子囊开始形成但未有四分孢子放散时期、成熟期(III)即四分孢子大量放散时期和成熟后期(IV期)即四分孢子基本放散留下大量孔洞时期[25]。对981品系的生理研究发现其在成熟期(III期)释放的孢子数量少且形态畸形[12],根据对四分孢子形成过程的认识推测981品系可能在II期经减数分裂形成四分孢子这一过程中出现异常。本文以I、II、IV期不同时期的藻体为材料,进行表达谱分析后结合WGCNA技术,探索龙须菜四分孢子体发育过程中的相关调控机制,并对981与鲁龙1号品系之间存在的差异进行基因表达层面上的探究。
1 材料与方法
1.1 材料培养
981品系和鲁龙1号品系于2014 年5 月采自青岛胶州湾养殖海区(36°08′N,120°17′E)。使用毛刷尽可能去除藻体表面污染,并用灭菌海水冲洗3遍。每个品系的一级分枝切成2 cm小藻段,在含有Pro 培养基[26]的200 mL无菌海水的250 mL三角瓶中放置5 段,分别将981品系和鲁龙1号品系置于其四分孢子放散的最佳条件下培养[27],每3 d更换一次新鲜培养基。每周在光镜下对藻段进行观察以确定四分孢子的形成和放散情况。样品收取的具体信息见表1,每个阶段样品设置3个生物学平行。
1.2 总RNA提取与文库构建
采用总RNA 提取试剂盒(天根生化科技有限公司,北京)提取RNA,使用RNase-Free DNase I(天根生化科技有限公司,北京)进行柱上DNA消化。提取RNA后进行琼脂糖凝胶电泳检测,并用Nanodrop 2000和Agilent 2100检测浓度和纯度。
使用NEBNext Ultra RNA Library Prep Kit for Illumina(NEB,USA)试剂盒按照说明书步骤进行18个RNA-seq文库的构建,建库完成后进行IlluminaHiSeq 2000 测序。文库的构建以及测序均由北京诺禾致源生物信息科技有限公司完成。
1.3 测序数据质控与分析
对原始测序序列(Raw reads)进行质量控制,过滤掉带接头以及低质量的序列得到clean reads。使用Hisat2[28]软件将clean reads与龙须菜Survey数据[21]比对,将比对成功的reads挑出后用SOAPdenovo-Trans进行拼接构建参考序列(Reference),拼接后过滤掉小于200 bp的contig。使用bowtie对每个样品的reads进行比对并使用RSEM统计样品中基因的read count数目,edgeR[29-30]进行差异基因分析,TMM算法对read count数换算成FPKM (fragments per kilobase of exon per million fragments mapped, 每千碱基外显子百万片段数)值来估算基因表达水平并进行矩阵标准化矫正。对差异表达基因的表达矩阵进行提取,聚类检验样品重复性。

表1 龙须菜表达谱分析的样品信息
1.4 加权基因共表达网络构建
利用R 软件的WGCNA软件包完成共表达网络构建。首先, 导入预处理的表达量表对数据进行过滤后构建表达矩阵,并对样品进行聚类分析;利用WGCNA 中的pickSoftThreshold函数计算软阈值β参数使得满足无尺度网络分布前提条件;选取R2达到0.9 时的阈值参数β[13],计算所有基因间的相异系数创建邻接矩阵,并用TOMsimilarity函数将邻接矩阵转换为拓扑矩阵后,取逆得到相异性矩阵并以此构建系统聚类树。设定每个模块最小基因数为30 个,利用cutreeDynamic动态剪枝法修剪聚类树识别初始共表达模块。随后用moduleEigengene函数计算每个模块的特征向量值ME (moduleEigengene),用mergeCloseModules函数选择相似性在100%以上的模块进行合并融合(cutHeight=0.0)。
1.5 特异性模块的鉴定及枢纽基因的筛选
重新计算各模块特征向量基因ME,利用cor函数量化模块ME与样本特征的相关系数并绘制热图。相关系数越高表示模块内基因与样本特征关联度越高,相应地,基因相对表达量越高。选择与样本特征具备较高相关系数的模块作为备选特异性模块。
利用Alldegrees1函数计算模块内基因间连接数,筛选模块内连接数前10%的基因为Hub基因。
1.6 模块的GO(Gene Ontology)富集分析和基因互作网络的构建
对模块内基因使用Cytoscape软件中的binGO功能进行GO富集分析,并用Cytoscape[31]软件输出网络图像。基于KEGG 生物学通路数据库(http://www.kegg.jp/),进行KEGG通路富集分析,并借助http://www.omicshare.com的在线工具作图。
2 结果与分析
2.1 WGCNA数据的预处理及去除离群样本
为了排除污染和尽可能的利用基因组信息,对测序数据向基因组survey拼接结果上进行RNA-seq的可变剪切mapping,将有能map到的RNA-seq数据进行分离。然后,利用分离出的981品系的RNA-seq reads进行de novo拼接,构建reference。拼接后,得到13 426条Unigene。并对转录组reference分别进行GO和KEGG代谢通路注释(见图1)。KEGG结果中注释到一级大类为新陈代谢(Metabolism)、遗传信息过程(Genetic information processing)、环境信息过程(Environmental information processing)、细胞过程(Celluar processing)、生物系统(Organismal systems)。注释到的基因数最多的二级分类为翻译(Translation)过程,有224个基因(见图1A)。GO结果中多数基因富集到代谢过程(Metabolic process)、细胞过程(Cellular process)、细胞(Cell)、细胞部分(Cell part)、催化活性(Catalytic activity)、结合(Binding)等Go term(见图1B)。后续样本定量分析与功能注释则采用此reference。
对18个样品的差异表达基因的表达矩阵进行提取后聚类。初步聚类分析后,为确保分析结果的严谨和可靠性,确定采纳具有较高相关性的样品,去除皮尔逊相关系数小于0.95的样品,每个取样阶段保留2个生物学平行。对样品之间相关性进行统计见图2,除981.IV组样品,相关性为0.89。其他样品相关性分别在0.95~0.98之间,显示相关性较好。样品间的聚类分析,显示重复性无问题。
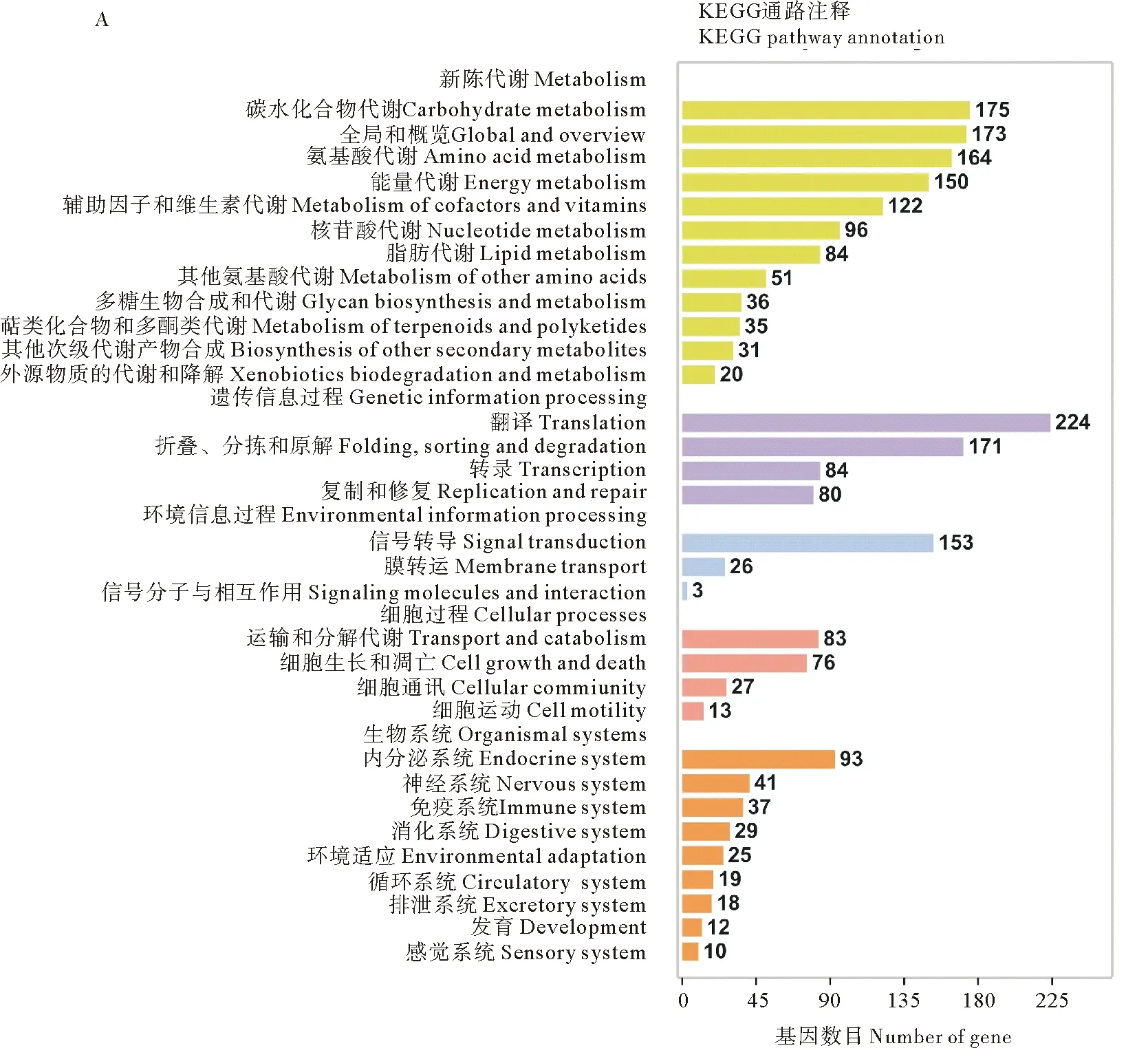
图1 龙须菜转录本KEGG代谢通路注释(A)与GO注释(B)二级分类统计
将构建好的样品FPKM表达矩阵导入WGCNA软件包,进行gsg数据过滤,共去除857个数据不良的基因,剩余12 569个基因进行后续分析。
2.2 基因共表达网络构建
设置软阈值β为7,计算所有基因间的相异系数创建邻接矩阵和相异性矩阵并以此构建层次聚类树(见图3A),每个颜色代表一个模块,共得到32个表达模块。构建每个模块与样本特征的相关性热图和模块内基因数量的条形图,热图中横轴为样品特征,纵轴为32个模块,热图中颜色越深表示模块与特征值的相关性越高(见图3B)。最大的模块为Turquoise模块,包含2 577个基因,最小基因模块为Violet模块,有66个基因(见图3B)。
经筛选发现与品系(Cultivar)相关的模块有 6个(图3B中相关性系数绝对值高于0.7的模块): Yellow模块(cor=-0.75,P=0.005)、Magenta模块(cor=0.74,P=0.006)、Greenyellow模块(cor=0.87,P=3e-4)、Cyan模块(cor=-0.75,P=0.005)、Darkgreen模块(cor=-0.79,P=0.002)五个模块,以及一个备选模块 Purple模块(cor=0.68,P=0.02)。相关性为正值,表示基因在981品系中呈现较高表达水平(与鲁龙1号相比),相关性为负值,表示基因在981品系中呈现较低表达水平(与鲁龙1号相比);P<0.5显示相关性分析可靠。
另一方面,由于981品系表现出四分孢子放散时期的低育特性,因此选择四分孢子形成的时期即981.II期作为关注对象,发现与此特征相关的模块(以0.8为阈值)有:Magenta模块(cor=0.82,P=0.001)、Green模块(cor=0.97,P=1e-07)、Turquoise模块(cor=0.99,P=5e-10)3个模块。相关性的正负表示表达水平的高低。
2.3 品系相关模块的分析与富集
对与品系(Cultivar)相关的6个模块的基因及模块特征基因进行表达量分析,构建样品的表达热图(见图4)。热图和柱状图为相应颜色模块基因表达量热图,每一行基因都进行了相应的均一化,颜色越红表明基因相对表达量越高,颜色越绿则越低;柱状图为表达量均一化后的相对表达量,可以代表每一列,即每一个样品的表达情况。其中Yellow模块、Cyan模块和Darkgreen模块在981品系的I~IV期样品中均下调表达,而Magenta模块、Greenyellow模块和Purple模块的基因在981品系中均有不同程度的上调表达。
进一步地将6个模块的基因混合,通过FPKM数据处理分为981中高表达基因集与鲁龙1号品系中高表达基因集。显示在981品系中,在I~IV期样品中均保持高表达水平的基因为515个,设为Up组;相反,表达水平比鲁龙1号品系低的基因有817个,设为Down组。分别对Up与Down组使用binGO进行 GO富集分析。
图5中有颜色的圆圈表示有基因富集,颜色越深显著性越高,圆圈越大相应富集的基因越多。981中高表达的基因中(Up组),主要富集到信号转导(Signal transduction )(GO: 0007165, FDR= 1.39e-01)、转运(Transport) (GO: 0006810, FDR= 1.39e-01)、细胞分化(Cell differentiation)(GO: 0030154, FDR= 9.66e-02)等终端GO term上,表明981品系在四分孢子形成、成熟至放散过程中,信号调节、细胞转运、细胞分化等生物学过程比较活跃。
981品系低表达组(Down组)主要富集到翻译(Translation)(GO: 0006412, FDR=6.82e-09)、细胞稳态(Cellular homeostasis)(GO: 0019725, FDR=7.07e-03)、生物合成过程(Biosynthetic process)(GO: 0009058, FDR= 3.82e-02)等终端GO term上,表明981品系在四分孢子成熟至放散过程中生物合成、细胞稳态等受到抑制。同样,核糖体(Ribosome)GO: 0005840, FDR=3.22e-11)、细胞溶质(Cytosol)(GO: 0005829, FDR=2.50e-05)等细胞成分也有不同程度的减少(见图6)。以上结果表明,981品系在四分孢子形成、成熟至放散的过程里,在mRNA翻译的水平和生物活性大分子的合成上都没有鲁龙1号品系活跃;另外,鲁龙1号品系在细胞内调节维持细胞稳态的过程中表现较好。
2.4 与981品系II期相关的模块基因分析与富集
将与981品系II期相关的Magenta、Green和Turquoise 3个模块的基因表达量聚类构建热图(见图7),可以看出这3个模块在981品系的II期普遍上调表达,尤其Green和Turquoise两个模块基因是特异性上调表达。
将3个模块内的基因进行KEGG代谢通路富集(见图8)。3个模块中均有富集到Cellular Processes相关的代谢通路,如Magenta模块富集到溶酶体(Lysosome)(ko04142,P=0.006 899 873)和干细胞全能型调节信号通路(Signaling pathways regulating pluripotency of stem cells)通路(ko04550,P= 0.117 777 5);green模块富集到酵母细胞周期(Cell cycle-yeast)通路(ko04111,P=0.168 894 3)和肌动蛋白细胞骨架调节通路(Regulation of actin cytoskeleton)(ko04810,P=0.197 946 7);turquoise模块富集到卵母细胞减数分裂(Oocyte meiosis)(ko04114,P=0.018 493 97)、细胞周期(Cell cycle)(ko04110,P= 0.122 86)、酵母细胞周期(Cell cycle-yeast)(ko04111,P=0.142 086 7)、酵母减数分裂(Meiosis-yeast)(ko04113,P=0.143 526 6)和紧密连接(Tight junction)(ko04530,P=0.157 927 7)等代谢通路。

(B中热图内每个方框内上方数值表示相关性系数,下方数值表示显著性。箭头标识为品系相关,*标识为981.II期相关。红框所指为与品系相关的模块(相关系数>0.7),绿框所指为与981.II期相关的模块(相关系数>0.8)。The value above each cell in heatmap indicates the correlation coefficient, and the lower value indicates the significance. The arrow indicates the cultivar feature, and the * is marked as the 981.II period feature.The red box is marked as the module related to the strain (correlation coefficient>0.7), and the green box refers to the module related to the 981.II period (correlation coefficient>0.8).)

图4 与品系相关的6个模块的基因在龙须菜表达谱12个样品下的聚类分析

图5 Up组生物过程的GO富集网络图

(A:生物过程;B:细胞成分。A: Biological process; B:Cell component.)图6 Down组的GO富集网络图

图7 与981品系II期相关的3个模块的基因在表达谱12个样品下的聚类分析

(Rich factor 是该模块中位于该通路的基因数目与所有该通路的基因数的比值。箭头所指为属于细胞过程的代谢通路。A:Magenta模块;B:Green模块;C:Turquoise模块。Rich factor represent the ratio of the hub genes in this pathway and all the genes of this pathway. Arrows refer to metabolic pathways that are part of the cellular process. A: Magenta;B: Green;C: Turquoise.)
将3个模块内前30个富集到Cellular Processes相关代谢通路的基因整合汇总,共得到30个基因。将这30个基因的FPKM值调出,经过均一化处理后构建样品间的表达热图(见图9)。发现在981品系的II期样品内有10个基因表达量明显下调,另外20个基因则呈现表达量上升的特征。
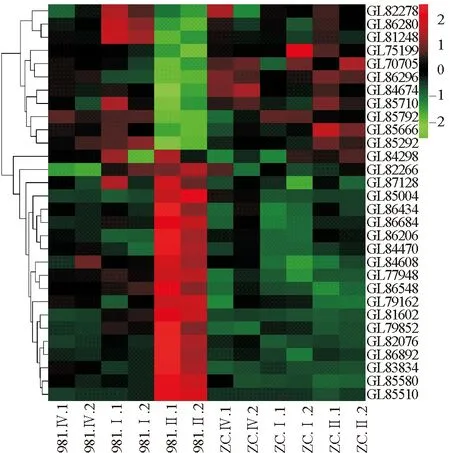
图9 30个备选基因在表达谱样品内的表达热图
统计这30个基因在KEGG通路注释中的结果发现有21个基因集中富集在卵母细胞减数细胞分裂(Oocyte meiosis, ko04114)、细胞周期(Cell cycle, ko04110)、酵母细胞周期(Cell cycle-yeast, ko04111)和酵母减数分裂(Meiosis-yeast, ko04113)通路中,将这21个基因分别进行KO(Keggorthology)注释和GO注释(见表2)。这些特异表达的基因主要集中在细胞周期的S期和M期。其中,处于S期的组蛋白去乙酰化酶HDAC1-2(GL84608、K06067)、复制起始点识别复合物ORC4(GL79162,K02606)和DNA复制许可因子MCM2(GL86684、K02540)上调表达,表明DNA复制可能异常进行;而细胞周期检验点蛋白RAD24(GL84298、K06662)主要参与S/G2期转换中的DNA损伤检验点,该基因上调表达间接表明S期DNA复制出现异常。另外在G1/S期中介导泛素化降解途径的SCF蛋白亚基CUL1(GL85292、K03347)下调表达,其参与G1期周期蛋白的降解,SCF改变可能影响到S期的正常调控。G2/M期的转化通常由CDK1起到关键性调控作用,而其活性依赖于细胞周期蛋白cyclin B的积累,但在是981.II期样品中CCNB1(GL81248,K05868)出现下调,直接影响CDK1活性,相应地,由CDK1介导的底物磷酸化水平降低,可能影响核纤层解聚、核仁解体等等,进而影响G2/M期的转化。
细胞分裂期M期又可分为前、前中、中、后和末期五个时期。前期中的黏连蛋白亚基SCC1(GL83834、K06670)、凝缩蛋白复合物亚基CAPH(GL85004、K06676)以及染色体结构维持蛋白SMC1(GL86206、K06636)参与姐妹染色单体黏着和染色体的凝缩,其在981品系的II期中过量表达可能导致染色体凝缩状态紧密。减数分裂重组蛋白DMC1(GL70705、K10872)在减数分裂前期I中同源染色体联会和交换重组中发挥作用,下调可能导致交换重组出现缺陷。细胞分裂从中期向后期转换主要依赖于后期促进复合体APC的作用,无活性的APC需要在CDC20的结合下有活性,在中期染色体排列正确后,一方面降解cyclin B使CDK1失活,解除对分离酶ESP的磷酸化作用,进而分解SCC1使姐妹染色单体分离,另一方面APC利用泛素化降解途径使ESP的抑制物降解,恢复ESP活性,进而使姐妹染色单体分离[32]。而在981.II期中,尽管APC3(GL86434、K03350)上调,但作为正调控因子的CDC20(GL84674、K03363)下调,因此对ESP活化作用减弱,即使ESP1(GL86892、K02365)的表达量上调,仍有可能是处于无活性状态,由此推测在姐妹染色单体分离阶段由于分离酶的作用降低,姐妹染色体分离出现异常。末期的细胞分裂控制蛋白CDC15(GL85666、K06683)参与细胞分裂的退出调控,在981.II期中同样下调。

表2 21个基因的KO和GO注释信息以及在细胞分裂中可能发挥的作用
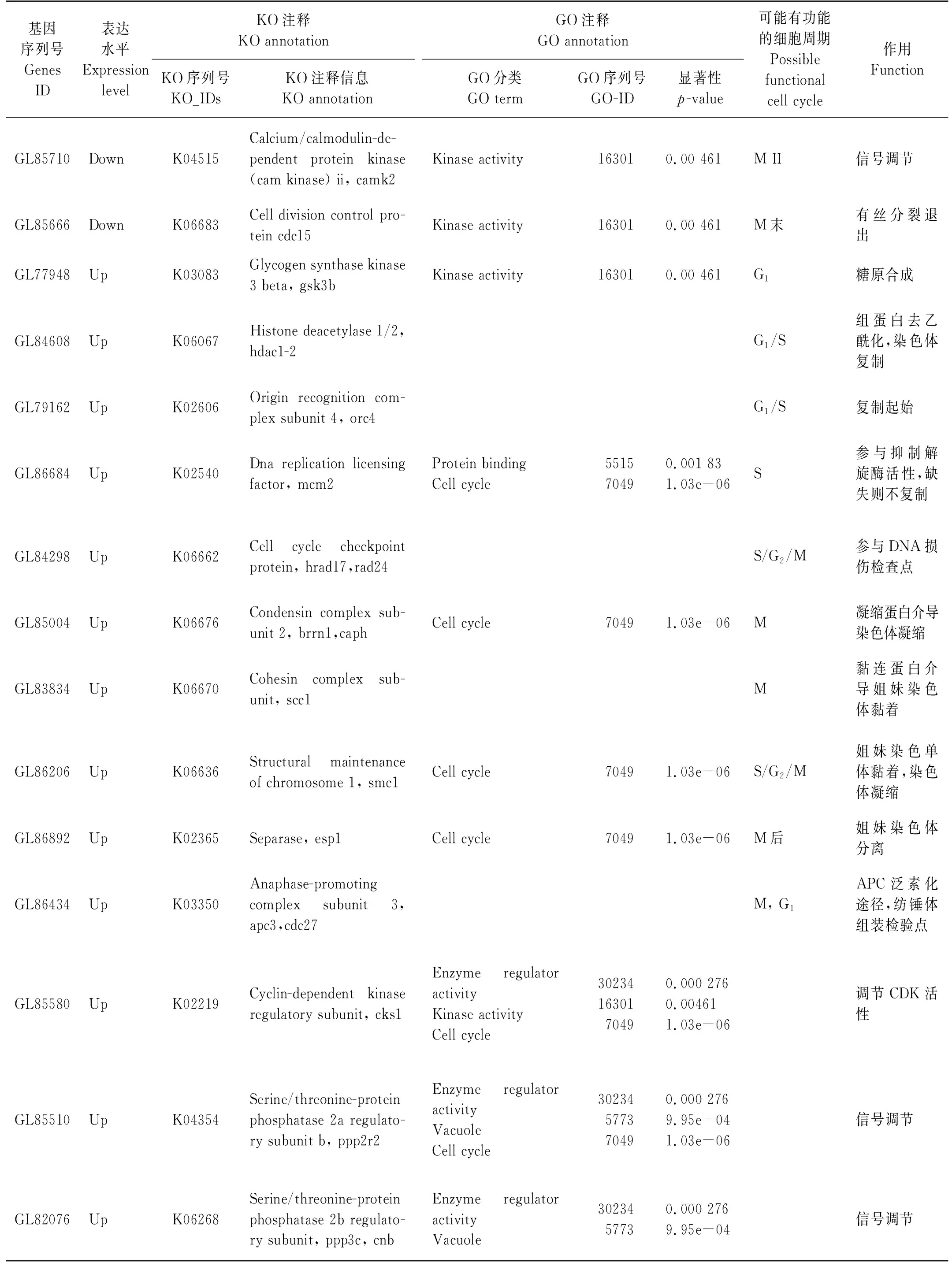
续表2
除此之外,还有一些参与信号转导调控途径的基因如丝氨酸/苏氨酸蛋白激酶PPP2家族(GL75199、K04382;GL85510、K04354;GL82076、K06268)表达水平也发生变化,这些都可能是981品系在孢子形成阶段出现异常的原因。
3 讨论
目前尚未有龙须菜的全基因组测序数据,现有龙须菜基因序列很多是不完整的,仅仅是基因全长的一部分。在进行数字基因表达谱分析中,测序所获得的序列信息都要以参考基因组序列为模板进行注释及后续定量,参考序列的质量高低直接影响到后续分析。而经过对数据的初步分析,发现龙须菜转录组分析可能存在污染以及龙须菜参考基因组数目太低的问题。龙须菜基因组survey数据不够完整,预测基因数目只有3 490条[21],严重低于正常藻类基因数目。采用该预测基因集会因信息缺失而影响准确性或丢失重要结果。因此在构建参考序列时采取二次mapping,以survey信息为参考根据比对到的RNA-seq的数据进行de novo拼接,最大化保留转录本信息。对转录本参考序列的质检也表明龙须菜转录组的完整性应该没有问题,无基因信息的损失。
通过基因的层次聚类,共划分出32个共表达模块,模块基因数从66个到2 577个不等。每个模块代表了具有相同表达模式的基因集,模块的数量由参数设置,模块数量多则可能使相似表达模式的基因分离,给后续研究带来工作量,模块数量少则可能使不同基因混合,掩盖基因表达信息。杨宇昕等[16]对玉米花期基因共表达模块筛选时划分出20个模块;鲜小华等[33]对甘蓝型油菜黄籽的研究中对1 461个低显著性关联基因进行划分得到8个共表达模块;鞠正等[19]对番茄果实成熟的研究中得到24个共表达模块。
根据模块与特征性状的相关性关联分析,找到与品系相关的6个模块,对模块内的差异表达基因进行GO富集结果表明981品系的信号调节、细胞转运、细胞分化等生物学过程比较活跃,而生物合成、细胞稳态、代谢过程等受到抑制,胞内物质有不同程度减少。以上结果表明,与鲁龙1号品系相比,981品系在四分孢子形成、成熟至放散的过程里,mRNA翻译水平和生物活性大分子合成都维持在较低水平,并且细胞内稳态紊乱,信号传递活跃,推测981品系的细胞活动受到干扰而出现异常。在此前对鲁龙1号品系的生理形状研究中也表明鲁龙1号品系具有比981品系更快的生长速度和更高的总蛋白含量[11]。
981品系的低育特性直观表现在成熟期III期即放散四分孢子时期,针对四分孢子形成时期即成熟前期II期的样品分析,筛选出3个高度相关的模块:magenta、green和turquoise模块。从模块内基因的KEGG代谢通路富集结果中找出30个参与细胞过程的基因。基因的表达热图表明有10个基因在981的II期下调而另外20个基因表现上调,这种表达水平上的异常变化符合要寻找的目的基因特征。对30个基因进一步筛选得到21个参与细胞周期的基因,分析各自的功能发现981品系在四分孢子发育阶段可能存在DNA复制、染色体凝缩状态、交换重组缺陷、姐妹染色单体分离、细胞分裂的退出调控等异常问题。对981品系四分孢子形成过程的形态学观察结果表明981品系形成和释放的四分孢子多表现出形态畸形和发育不完全[12],这些基因的调控异常表明981品系可能在孢子形成时期的细胞减数分裂出现异常而导致其形态变化,或可作为深入研究的备选基因。
4 结语
对前期获得的龙须菜不同育性品系(981和鲁龙1号)在不同发育阶段的表达谱数据,采用WGCNA构建基因共表达网络,共获得32个模块。其中,模块最大基因数量为2 577个,最小基因数为66个。经过筛选发现与品系相关的模块(以0.7为阈值)共5个,涉及基因共2 301个,对模块基因进行GO功能富集分析,结果表明主要富集以下功能上:信号转导、转运、细胞分化、翻译、细胞稳态、生物合成过程等。981品系与鲁龙1号品系的富集结果对比表明981品系在孢子放散过程中生物活性不高,生物合成水平较低,内环境稳态较差。筛选到21个与981品系II期四分孢子形成相关的备选基因,注释到细胞周期的不同阶段,主要集中在DNA复制和染色体形态变化上。对这些基因的进一步研究将有助于深入揭示龙须菜四分孢子发育过程中的分子调控机制。
