影像组学在肺肿瘤良恶性分类预测中的应用研究
2020-04-10周天绮朱超挺石峰
周天绮,朱超挺,石峰
浙江医药高等专科学校,宁波市,315100
0 引言
据国家癌症中心统计,我国肺癌发病人数和死亡人数已连续10年位居恶性肿瘤之首,每年新发肺癌约78.7万人,因肺癌死亡约63.1万人[1]。中国抗癌协会科普宣传部部长支修益表示,很多的肺癌患者都不是早期肺癌患者,所以才导致目前肺癌患者的高死亡率。
经过五十年的努力,肺癌的五年生存期并没有得到明显的提高。究其原因,一是常规影像PET、CT、MRI、超声无法诊断5 mm以下肿瘤,缺乏早期诊断手段;二是肿瘤异质性不确定性高,缺乏定量评估方法。
尽管已经得到大量肿瘤基因数据,但癌症仍然无法被治愈。肿瘤基因组的时空异质性限制了靶向治疗的效果,缺乏有效的手段去全面定量评估肿瘤异质性。影像组学(Radiomics)利用海量影像(结构影像、功能影像、分子影像)和基因病理信息来量化肿瘤微环境,早期定量评价肿瘤异质性,实现精准诊疗决策,延长病人生存期。
影像组学利用大数据挖掘技术从影像、基因、临床等信息中提取海量特征来量化肿瘤等重大疾病[2]。通过预测算法帮助临床医生制定科学的诊疗决策,如肿瘤分型、肿瘤分期、生存期预测及诊疗方案优化等,提高疾病早诊率和病人生存期。美国莫菲特癌症中心针对影像数据挖掘不足,肺癌患者预后难以定量评估的问题,在1 000余例病理、影像数据完整的肺癌患者数据中提取强度、形状纹理、小波等特征建立标签,进行智能分析,结果表明影像组学标签具有显著的预后价值,并与基因显著相关。中国科学院分子影像重点实验室基于4 000例信息完善肺癌数据研发的肺癌影像组学预测软件,可以进行肺癌的良恶性、生存期、TNM分期等预测,以及最优诊疗方案建议,可实现肿瘤的自动分割和592个特征的提取,已在国内20余家三甲医院进行试点检测,临床预测精度达80%以上。
可见,影像组学能无创地鉴别肺肿瘤的良恶性、疗效及预后评估等,实现早发现、早诊断、早治疗,帮助医生进行临床辅助诊断。本研究介绍影像组学的基本概念及其在肺肿瘤良恶性分类预测中的应用。
1 影像组学概述
1.1 定义
影像组学最早由荷兰学者Lambin等在2012年提出。其是指高通量地从MRI、PET、CT影像中提取大量高维的定量影像特征,实现肿瘤分割、特征提取与模型建立,凭借对海量影像数据信息进行更深层次的挖掘、预测和分析来辅助医生做出最准确的诊断[2]。医生需要医学有用的特征信息来诊断疾病处于哪一个层次,影像组学便可将影像转换为可挖掘的特征数据。
影像组学的数据范围在获取、存储、管理和分析等方面都大大超出了传统数据库软件工具能力范围。具有大数据的“4V”特征:①Volume:海量的数据规模;②Velocity:快速的数据流转;③Variety:多样的数据类型;④Value:价值大,价值密度低。影像组学利用大数据挖掘技术定量肿瘤异质性,实现精准诊疗决策,延长患者的生存期[3]。
1.2 处理流程
影像组学处理的一般流程包括:影像数据采集、图像分割、特征提取和特征降维、模型构建等步骤。
(1)影像数据采集
现代医院影像设备包括CT、MRI、PET等。影像组学的入组数据需要具有相同或相似的采集参数,保证数据不会受到机型、参数的影响[2]。如,NSCLC肿瘤中,影像组学特征的可变性与不同CT扫描的图像有关。应考虑影像扫描的差异性对肿瘤异质性分析所造成的影响。因此,为保证入组数据的一致性需制定好入组标准和规范。另外,在结构或功能影像以外还应采集必要的临床病历信息或基因、病理数据,以提高诊断的准确性[4]。
(2)图像分割
影像组学研究需先对病变肿瘤区域精准定位。通过将图像分割为感兴趣区域(Region of Interest,ROI),使肿瘤区域和其他组织分离,用以标定肿瘤区域。分割方法包括:人工手动分割、半自动分割和自动分割。手动分割被视为病灶分割的金标准,由专业的影像医生来勾画,精度最高,但临床肿瘤影像数据量庞大,手工勾画肿瘤边缘费时费力且主观性较强。针对影像组学的大数据集,可采用基于区域生长的半自动分割法或基于雪橇自动生长分割法(Toboggan Based Growing Automatic Segmentation Approach,TBGA)的自动图像分割。具体选用哪种方法进行分割应结合具体情况合理选择。
(3)特征提取和特征降维
高通量影像学特征用于定量描述分割完成后的ROI的属性。特征提取是通过变换的方法用低维空间表示高维度特征数据。在ROI分割完成后,就可以对其进行特征提取。目前常用的四大类高通量影像学特征包括:强度、形状、纹理、小波。为提高分类和预测的精确度,应将计算机定量特征、经验特征、文本信息、基因信息和病理信息相结合,全面量化肿瘤异质性,如表1所示。

表1 特征提取Tab.1 Features extraction
由表1可以看出,高维度特征包含海量信息且冗余性大,需特征降维以减少特征的数量,找到少数真正关键的特征。可以采用机器学习或者统计学方法来实现,如通过最大相关最小冗余(Maximum Relevance and Minimum Redundancy,mRMR)或主成分分析法(Principal Component Analysis,PCA)进行特征降维[5]。
(4)模型构建
针对具体临床问题,建立计算机定量影像特征与临床研究问题标签之间的分类模型。从影像大数据原始像素出发,提取高维手工设计特征并进行特征选择,或自主挖掘与临床问题相关的影像组学特征,构建影像特征与临床问题的分类模型。常用模型有支持向量机(Support Vector Machine,SVM)模型、卷积神经网络(Convolutional Neural Networks segmentation)模型等。
2 肺肿瘤良恶性诊断
肺肿瘤良恶性的辅助判断是影像组学目前应用较多的临床实践之一。本实验针对肺癌患者CT影像的样本数据,提取和优化特征空间,将优化后的特征空间数据作为SVM的输入,创建肺部肿瘤良恶性分类预测模型,预测患者患癌的可能性。并验证SVM分类器模型对不同结节大小良恶性预测的准确性。
2.1 数据获取及预处理
本次实验的数据样本选自公开数据集LIDCIDRI上的816例患者的CT影像数据。包括恶性肿瘤451例,良性肿瘤365例。切片厚度均小于3 mm,图像大小为512×512×400像素。
为使不同切片厚度的CT图像能够应用于同一模型中,需对数据集进行归一化处理。首先,对CT图像进行缩放,每一像素大小调整为1 mm3的体积大小;然后,将CT图像的像素强度转换为HU值,并将HU值(-1 000~400)线性变换为0~255;同时确保所有CT图像都具有相同的方向;最后,对CT图像进行粗略肺部分割,消除与肺部的不相交区域,如图1所示。
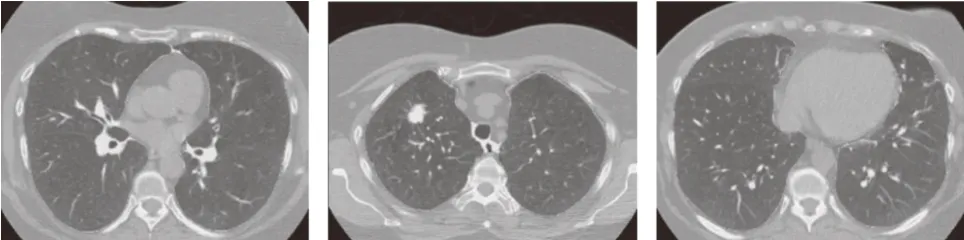
图1 肺部分割Fig.1 Partial pulmonary segmentation
2.2 图像分割
为满足基于大数据的影像组学分析,需采用高效的图像分割算法实现自动、可重复、精准的分割。先由经验丰富的放射科医生标记1 200个肺结节,用于神经网络的学习。从整体CT图像中的结节标记周围裁剪出小型3D图像块,将这些更小的3D图像块与结节标记直接对应,然后通过训练神经网络模型来检测肺结节。
本实验采用中科院田捷项目组提出的中心池化卷积神经网络分割方法。中心池化运算保留关键特征、双分支网络融合2D和3D信息、多尺度输入提取多尺度特征,对组织粘连和空腔等多种肺结节分割精度较高,如图2所示。
2.3 特征提取和特征降维
肺癌七大典型的影像学特征:分叶征、毛刺征、胸膜凹陷征、空泡征、细支管充气征、空洞、血管聚集征等都是临床诊断经验化的信息[6],并没有量化成为确切诊断的标准。本实验采用影像组学特征提取包Pyradiomics,从CT图像的结节中提取97维定量图像特征,包括灰度共生矩阵特征、一阶统计特征、灰度级区域矩阵特征等。
然后,采用FSelector 特征筛选模型进行特征降维。FSelector由以下五个函数构成:①identify_missing;②identify_collinear;③identify_zero_importance;④identify_low_importance;⑤identify_single_unique。这5个函数分别用于删除特征集中高missing-values百分比的特征、高相关性的特征、无贡献的特征、低贡献的特征和单值特征。
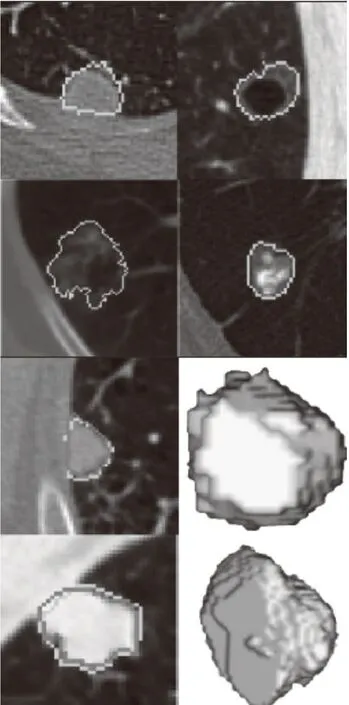
图2 组织粘连和空腔等肺结节分割Fig.2 Segmentation of pulmonary nodules such as tissue adhesion and cavity
采用统计学中的皮尔森(Pearson)相关系数计算特征的权重并排序。按权重降序选择多个特征构建优化特征空间。基于皮尔森相关系数计算得到的特征权重(前20个特征),如图3所示。权重排在前5的特征分别是:最大2D直径尺寸、延伸度、球度、平整度、表面体积比。
2.4 模型构建
Vapnik提出的支持向量机(SVM)是一种有坚实理论基础的新颖的小样本学习方法。它基本上不涉及概率测度及大数定律等,因此不同于现有的统计方法。从本质上看,它避开了从归纳到演绎的传统过程,实现了高效的从训练样本到预报样本的“转导推理”,大大简化了通常的分类和回归等问题[7]。近年来SVM方法已经在图像识别、信号处理和基因图谱识别等方面得到了成功的应用,显示了它的优势[8]。
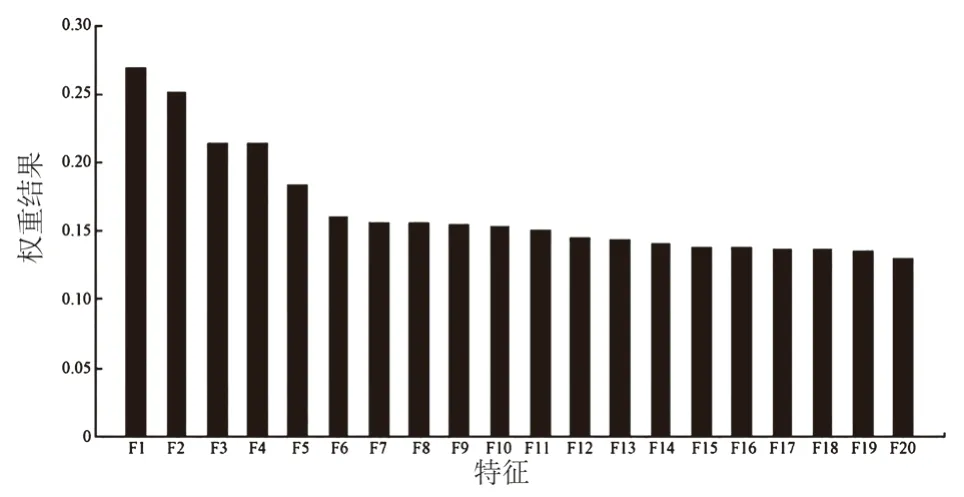
图3 基于皮尔森相关系数计算得到的特征权重Fig.3 Characteristic weight based on pearson coefficient
训练集含516例患者的CT影像数据,包括258例恶性肿瘤,258例良性肿瘤;测试集含300例CT影像数据,包括恶性肿瘤189例,良性肿瘤111例。为了确定优化特征空间的特征数量,计算SVM模型在不同特征数量下的可靠性,如图4所示。从图中可看出,当权重较大的前20个特征作为SVM模型的输入时,模型的可靠性最高。

图4 SVM模型的可靠性计算Fig.4 Reliability calculation of SVM model
由权重较大的20个特征组成优化特征空间,将训练集的优化特征空间作为SVM分类器的输入,训练肿瘤良恶分类预测模型,通过学习肺癌的这些特征,实现对肺肿瘤良恶性的有效判别。
2.5 模型验证
k折交叉验证一般用来验证机器学习中分类算法的准确性[9]。本实验采用十折交叉验证。将样本数据随机分成10份,轮流将其中9份作为训练集,剩余一份作为测试集,10次结果的均值作为分类算法的预测准确率。
肺结节的大小与其良恶性有一定的相关性,一般结节越大,恶性的可能性越高[10]。为验证SVM模型对不同结节大小良恶性分类预测的准确性,本实验按照肺部结节的大小分别将样本中的肺结节(直径小于30 mm)、小结节(直径小于20 mm)、微小结节(直径小于5 mm)的CT影像数据输入SVM分类器,对肺结节、小结节、微小结节的良恶性进行分类,并计算其预测准确率,如表2所示。

表2 不同结节大小的分类预测准确率(%)Tab.2 Classification and prediction accuracy of different nodule sizes
绘制基于肺结节和小结节良恶性分类预测的接收器工作特征曲线(Receiver Operating Characteristics,ROC),如图5所示。然后通过计算曲线下面积(Area under the Curve,AUC)以评价SVM分类器模型的好坏。
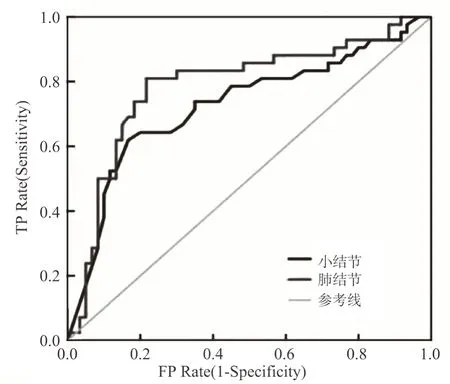
图5 结节良恶性分类预测的ROC 曲线Fig.5 ROC curve for classification and prediction of benign and malignant nodules
本实验中SVM分类器模型针对肺结节、小结节良恶性分类的预测准确率分别为83.7%、80.4%、AUC的值分别为0.824、0.792,表明SVM分类器可以准确地预测直径大于5 mm的结节的良恶性,可辅助临床医生进行诊断。
3 结束语
本实验构建的SVM分类器模型应用于肺肿瘤良恶性的定量预判,以辅助临床医生进行诊断。本实验中也存在一些不足,如样本数较少,应通过努力扩大样本数量;训练集中的ROI手动分割主观强,可结合多个医生手动分割的ROI进行分析。随着深度学习在影像学领域研究的不断深入,基于深度学习的预测分析方法将是未来的发展方向。
