3株鸡传染性贫血病毒的全基因组序列分析
2020-04-08邓显文张民秀谢芝勋张艳芳谢丽基谢志勤刘加波罗思思曾婷婷
邓显文,张民秀,谢芝勋,张艳芳,谢丽基,谢志勤,刘加波,罗思思,曾婷婷
(广西壮族自治区兽医研究所, 广西兽医生物技术重点实验室, 南宁 530001)
鸡传染性贫血病(Chicken infection anemia, CIA)是由鸡传染性贫血病毒(Chicken infection anemia virus, CIAV)引起的鸡免疫抑制病,易感雏鸡感染该病毒主要表现为再生性贫血障碍、骨髓萎缩和胸腺淋巴组织萎缩等症状[1]。我国于1992年首次在鸡群中分离鉴定CIAV[2],随后在黑龙江省、安徽省、广西省等地的鸡群均检测到CIAV的存在[3~5]。CIAV基因组全长约为2.3 kb,为单股负链DNA,含有3个开放阅读框(Open reading frames, ORFs),分别是ORF1,ORF2和ORF3,依次编码Viral protein 3(VP3)、Viral protein 1(VP1) 和Viral protein 2(VP2)[6]。VP1蛋白是CIAV的核衣壳蛋白,并且是CIAV的唯一结构蛋白和主要免疫原蛋白,可刺激机体产生较高的中和抗体。VP2蛋白是CIAV的支架蛋白,并且是VP1蛋白的辅助蛋白,帮助VP1蛋白形成正确的构像,从而使VP1蛋白暴露出中和表位[7]。VP3蛋白是CIAV的非结构蛋白,又称为凋亡素,具有诱导鸡胸腺淋巴母细胞和原始造血细胞凋亡的作用,从而导致雏鸡出现再生性贫血障碍症[8]。
根据CIAV全基因组的遗传进化规律,将CIAV主要分为4个基因群,分别是Group A、Group B、Group C和Group D[9-10]。目前研究显示Group A在世界范围内广泛流行,是优势的基因群[11-13]。我国主要流行的基因群为Group A,鸡群中散发存在Group B 和Group D基因群[9]。解慧梅[14]在2012-2104年间对江苏省鸡群CIA进行了血清学调查,发现血清抗体阳性率平均达到31.9%。储鸿蒙[15]对安徽省CIA血清学调查显示,血清阳性率达63.6%。这些数据再一次证实了我国CIAV感染鸡群的广泛性。本研究分别于2018年1月、4月和8月在广西南宁市规模化鸡场采集到3份病鸡肝脏组织样品,运用CIAV的检测引物鉴定为CIAV阳性后,分段扩增CIAV的基因组,并进行测序后将分段扩增的目的片段进行拼接,获得3株CIAV的全基因组,对这3株CIAV的基因组进行序列分析,以期获得广西南宁市CIAV流行毒株的分子变异趋势,为进一步丰富CIAV的分子流行病学提供依据。
1 材料与方法
1.1 病料来源及CIAV PCR鉴定 2018年1月、4月和8月份于广西南宁市规模化鸡场商品代肉鸡采集到3份肝脏组织。样品处理:磷酸缓冲溶液(PBS)按照(肝脏:PBS)1∶5稀释混成匀浆,将匀浆吸入到EP管中,反复冻融3次,3000 r/min离心20 min,取上清液并-70 ℃保存备用;运用参考文献[16]中的引物鉴定这3份病料均为CIAV阳性。
1.2 主要试剂 分子克隆所需试剂盒EasyPure Genomic DNA kit、DH5α感受态细胞和胶回收试剂盒购自北京全式金生物技术有限公司;pMD18-T、TaKaRa Ex Taq和DL2000 Maker购自宝日医生物技术(北京)有限公司。
1.3 CIAV全基因扩增引物 扩增CIAV全基因引物如表1,引物由华大基因生物科技(深圳)有限公司合成。

表1 引物序列信息
1.4 病料DNA的提取 3份阳性CIAV组织样品的DNA按照EasyPure Genomic DNA kit的操作说明书提取,提取好的DNA于-70 ℃保存备用。
1.5 CIAV全基因的扩增及序列分析 将提取好的DNA参照TaKaRa Ex Taq操作说明进行CIAV全基因的扩增。PCR产物进行电泳纯化后按照常规操作进行基因克隆,挑取阳性克隆菌液送华大基因生物科技(深圳)有限公司测序。应用LaserGene7.1对获得的序列进行拼接、拼接完成后运用LaserGene7.1和MEGA 4.0 软件对CIAV全基因序列的核苷酸、VP1、VP2和VP3蛋白氨基酸序列进行分析,并构建CIAV全基因、VP1、VP2和VP3基因的遗传进化树。从GenBank上获得28条CIAV全基因序列,其中包括13条国外参考毒株的全基因序列,15条国内参考毒株的全基因序列。
2 结 果
2.1 CIAV全基因扩增和序列测定结果 应用CIAV分段扩增引物对3份阳性CIAV的全基因进行分段扩增,扩增产物大小分别约为872、907和1254 bp,见图1。3株CIAV基因组经过基因测序鉴定后,运用LaserGene7.1对3段基因进行拼接后,3条全基因序列大小均为2298 bp,将3株CIAV进行命名,分别是GX1801、GX1804和GX1810,将序列上传至GenBank基因库,得到的相应序列号,分别是MK484614、MK484615和MK484616。
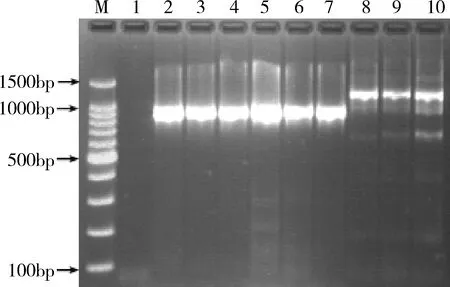
M: 100bp ladder; 1: 阴性对照; 2-4: 872bp片段;5-7: 907bp片段; 8-10: 1254bp片段M: 100bp ladder; 1: Negative control; 2-4: 872bp fragments;5-7: 907bp fragments; 8-10: 1254bp fragments
2.2 3株CIAV全基因序列同源性分析及遗传进化分析 GX1801、GX1804和GX1810全基因核苷酸序列之间的同源性为99.3%~99.7%,与28株参考毒株的核苷酸同源性为95.6%~99.8%;GX1801、GX1804和GX1810全基因序列与参考毒株的遗传进化分析表明(图2),CIAV遗传进化树可分为4个基因群,分别是Group A、Group B、Group C和Group D,所参考的中国毒株分别分布在Group A、Group B和Group D群;本研究的3株CIAV与12株中国参考毒株同属于Group A,并与GD-104和GD-103的亲缘关系最近,同源性在99.3%~99.9%;该结果说明了国内的CIAV流行毒株主要以Group A基因群为主(图2)。根据遗传进化树的基因分群,将本研究3株CIAV全基因序列分别与属于Group A、Group B、Group C和Group D的参考毒株进行核苷酸进行同源性分析,结果显示与Group A的参考序列间的核苷酸同源性为96.5%~99.8%;与Group B的参考序列间的核苷酸同源性为97.7%~98.2%;与Group C的参考序列间的核苷酸同源性为96.2%~96.9%;与Group D的参考序列间的核苷酸同源性为95.6%~96.5%。

图2 CIAV全基因遗传进化树
2.3 运用VP1基因、VP2基因和VP3基因构建进化树的结果比较 比较基于VP1、VP2和VP3基因构建的遗传进化树,发现基于VP1构建的遗传进化树可分为Group A、Group B、Group C和Group D 4个基因群,并且与基于CIAV的全基因进化树分群相一致,而VP2和VP3的遗传进化树仅能分为Group A、Group B、Group C或Group A、Group D、Group C(图3);以上数据说明CIAV的分群可依据VP1基因进行区分。
2.4 GX1801、GX1804和GX1810 VP1、VP2和VP3氨基酸序列分析 将本研究获得毒株和28株参考毒株的VP1蛋白的氨基酸序列进行比对,发现VP1蛋白的变异主要集中在第75、97、125、139、144、287、370、376、413和447位氨基酸位点;GX1801、GX1804和GX1810 VP1蛋白与28株参考毒株的氨基酸同源性为96.8%~100%,其中与GD-103 和GD-104的氨基酸同源性为99.8%~100%;分析GX1801、GX1804和GX1810 VP1蛋白高变区(139aa~151aa)发现,3个毒株在该区域没有出现氨基酸位点的增加和缺失,并且具有高度保守的特性;分析VP1蛋白上与毒力相关的氨基酸位点(第75、89、125、141、144、394位氨基酸位点),发现GX1801、GX1804和GX1810与国外强毒株Cuxhaven-1在第75、89、141、394位氨基酸位点上保持一致,与弱毒株C369仅在第394位氨基酸存在差异;与日本强毒株C368、国内强毒株GD-103和GD-104株在第75、89、125、141、144、394位氨基酸位点全部一致,推测GX1801、GX1804和GX1810与日本强毒株C368、国内强毒株GD-103和GD-104株可能具有相似的致病性(表2)。

图3 基于VP1、VP2和VP3基因构建的遗传进化树
本研究中VP2蛋白氨基酸序列比对结果显示VP2蛋白上氨基酸位点的变异较少,说明VP2蛋白氨基酸序列高度保守,GX1801、GX1804和GX1810 VP2蛋白与28株参考毒株的氨基酸同源性为98.6%~100%;其中与GD-103 和GD-104的氨基酸同源性为99.5%~100%;GX1801、GX1804和GX1810 VP2蛋白上与磷酸酶活性相关的基序(I94CNCGQFRKH103)均未发生变异。比较所有VP3蛋白的氨基酸序列,结果发现VP3蛋白的氨基酸序列高度保守,未见有明显的变异;GX1801、GX1804和GX1810 VP3蛋白与28株参考毒株的氨基酸同源性为96.7%~100%;其中与GD-103 和GD-104的氨基酸同源性均为100%;分析GX1801、GX1804和GX1810 VP3蛋白与诱导细胞凋亡相关的重要区域(VP3蛋白N端结构域的第1-69位氨基酸),发现这个区域高度保守。

表2 GX1801、GX1804、GX1810和Cuxhaven-1、C368、C369、GD-103、GD-104间的氨基酸位点变化
3 讨 论
蒋玲艳[5]于2002-2004年间对广西鸡群的CIAV的感染情况进行了流行病学调查,并证明CIAV在广西鸡群中普遍存在。邓显文等[17]于2006年对一株广西CIAV分离株进行遗传进化分析,发现该分离株与天津株TJBD40亲缘关系较近。近年来,未见有关于广西CIAV的流行情况的报道,本试验从临床组织样品中扩增并获得3株广西南宁CIAV的全基因序列,为广西CIAV的分子流行病学调查提供了重要材料。
本研究参考Eltahir等[9]对CIAV的分群,将CIAV遗传进化树分为4个基因群(Group A、Group B、Group C和Group D),国内所参考的15株中国毒株分别分布在Group A、Group B和Group D群,与Eltahir Y M的研究相一致,本研究的GX1801、GX1804和GX1810毒株与12株中国参考毒株同属于Group A,再次证实了国内流行毒株主要以Group A群为主,并且GX1801、GX1804和GX1810毒株与国内毒株GD-103、GD-104的亲缘关系较近,其与GD-103、GD-104具有较高的氨基酸同源性,因此,推测GX1801、GX1804和GX1810毒株可能与GD-103、GD-104来源于同一毒株。本研究比较了基于CIAV全基因、VP1基因、VP2基因和VP3基因构建的进化树,发现CIAV全基因和VP1基因所构建的进化树相一致,而基于VP2基因和VP3基因构建的进化树不能够更细致的分群,因此基于VP1基因对CIAV进行分群给CIAV在基因群上的划分上带来一定的指导意义。
CIAV的致病性和在细胞上的增殖、复制与VP1蛋白的氨基酸位点关系密切。Renshaw 等[18]发现VP1上第139和第144位氨基酸位点均突变为谷氨酰胺(Q)时,会降低病毒在MACC-MSB1细胞上的增殖和传播效率。GX1801、GX1804和GX1810 VP1蛋白上第139和144的氨基酸位点分别为赖氨酸(K)和谷氨酸(E),而与之比较的C368和C369株均能在MACC-MSB1细胞上增殖并具有较高的病毒效价[19],因此,推测GX1801、GX1804和GX1810可能在MACC-MSB1细胞上具有很好的适应性。Todd等[20]对Cuxhaven-1株VP1蛋白位点第75、89、125、141、144都进行了突变,但仍然保留394位氨基酸为谷氨酰胺(Q),发现该Cuxhaven-1突变株毒力减弱,并且不能引起贫血、骨髓和胸腺萎缩。本研究中参考的C368和C369株已经经过Yamaguchi等[19]的鉴定,分别是具有高致病性强毒株和低致病性的弱毒株,而国内毒株GD-103和GD-104毒株也已经经过蔺文成等[21]鉴定为强毒株,GX1801、GX1804和GX1810 VP1蛋白在毒力相关位点(第75、89、125、141、144和394位)上均与C368、GD-103和GD-104在同位点上保持一致,因此,推测本研究中的GX1801、GX1804和GX1810株可能具有高致病性,但需要进一步的人工感染试验才能确定。
CIAV 的VP2蛋白具有双重特异性磷酸酶活性,与磷酸酶活性相关的基序是位于VP2蛋白上第94~103位氨基酸(I94CNCGQFRKH103),基序内的第95位和第97位氨基酸的突变会减弱病毒在MDCC-MSB1细胞上的复制能力和减少细胞病变[22]。本研究中GX1801、GX1804和GX1810 VP2蛋白上的I94CNCGQFRKH103高度保守,这是否能够导致MDCC-MSB1细胞上产生明显的细胞病变还需要进一步研究。Astrid等[23]研究发现,VP3蛋白与诱导肿瘤细胞凋亡相关的重要区域是位于N端结构域的第1~69位的氨基酸位点,该区域的缺失不能诱导肿瘤细胞的凋亡。本研究中GX1801、GX1804和GX1810 VP3蛋白 N端结构域的第1~69位氨基酸均高度保守,结合VP1毒力相关位点的分析,推测GX1801、GX1804和GX1810在感染鸡体后可能能够引起鸡类淋巴细胞和骨髓细胞的凋亡。
