阪崎克罗诺菌特异性适配子筛选研究
2020-03-13张云怡梅玲玲陈鸿鹄张俊彦陈建才
张云怡,梅玲玲,占 利,陈鸿鹄,张俊彦,陈建才,张 政
克罗诺杆菌(Cronobacterspp.)是婴幼儿奶粉中常见的检出菌,患儿感染后多有发热、腹痛、腹泻、呕吐、粘血便等症状,严重者可致坏死性小肠结肠炎、败血症和脑膜炎,病死率达40%~80%[1-5]。国标GB10765-2010中规定婴幼儿配方食品中不得检出克罗诺菌。克罗诺菌检测目前主要采用生化检测法。该法特异性高,但耗时长。不利于对检测周期有高要求的工作。适配子是人工合成的单链DNA或RNA,可以和靶物质高特异性结合,具有抗体无法比拟的优势:靶物质范围广;更高的亲和力和特异性;制备中不需进行动物免疫,成本低;该技术出现以来已经在临床检验、食源性致病菌检测等方面表现出了很好的应用效果[6-9]。
在目前的细菌分类学中,克罗诺杆菌属包含7个种,分别为阪崎克罗诺菌(Cronobactersakazakii)、丙二酸盐克罗诺菌(Cronobactermalonaticus)、苏黎世克罗诺菌(Cronobacterturicensis)、莫金斯克罗诺菌(Cronobactermuytjensii)、Cronobactercondimenti、Cronobacteruniversalis和Cronobacterdublinensis。其中,阪崎克罗诺菌在环境、食品和病人样本中的分布最为广泛[10-11]。在本研究中,我们通过SELEX(指数富集配基系统进化技术)筛选出阪崎克罗诺菌特异性适配子,为建立基于适配子技术的阪崎克罗诺菌快速检测方法奠定基础,并对适配子的二级空间结构进行了分析。
1 材料与方法
1.1生物信息学分析 使用BLAST 2.2.25、ClustalX软件对40株阪崎克罗诺菌的基因组序列进行比较分析,菌株具体信息见表1。核心基因组参数设定:氨基酸序列覆盖度和相似度均为50%。使用pSORTb 软件分析蛋白亚细胞定位,使用SignalP 和TMHMM进行蛋白信号肽和跨膜区预测。
表1 40株阪崎克罗诺菌基因组信息
Tab.1 Genomic profiles of 40Cronobactersakazakiistrains
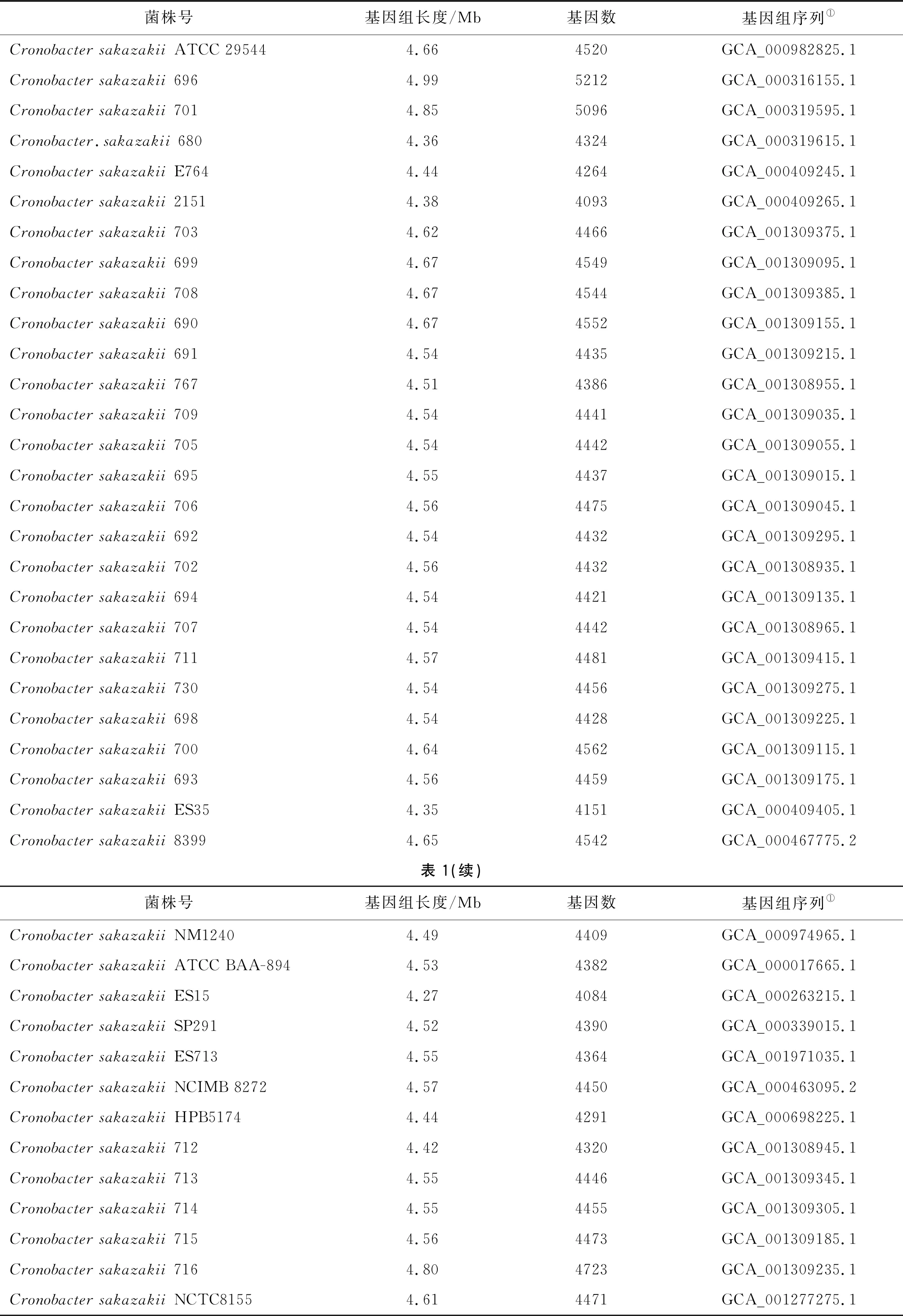
菌株号基因组长度/Mb基因数基因组序列①Cronobacter sakazakii ATCC 295444.664520GCA_000982825.1Cronobacter sakazakii 6964.995212GCA_000316155.1Cronobacter sakazakii 7014.855096GCA_000319595.1Cronobacter.sakazakii 6804.364324GCA_000319615.1Cronobacter sakazakii E7644.444264GCA_000409245.1Cronobacter sakazakii 21514.384093GCA_000409265.1 Cronobacter sakazakii 7034.624466GCA_001309375.1Cronobacter sakazakii 6994.674549GCA_001309095.1Cronobacter sakazakii 7084.674544GCA_001309385.1Cronobacter sakazakii 6904.674552GCA_001309155.1Cronobacter sakazakii 6914.544435GCA_001309215.1Cronobacter sakazakii 767 4.514386GCA_001308955.1Cronobacter sakazakii 7094.544441GCA_001309035.1Cronobacter sakazakii 705 4.544442GCA_001309055.1Cronobacter sakazakii 6954.554437GCA_001309015.1Cronobacter sakazakii 7064.564475GCA_001309045.1Cronobacter sakazakii 692 4.544432GCA_001309295.1Cronobacter sakazakii 7024.564432GCA_001308935.1Cronobacter sakazakii 6944.544421GCA_001309135.1Cronobacter sakazakii 7074.544442GCA_001308965.1Cronobacter sakazakii 7114.574481GCA_001309415.1Cronobacter sakazakii 7304.544456GCA_001309275.1Cronobacter sakazakii 6984.544428GCA_001309225.1Cronobacter sakazakii 700 4.644562GCA_001309115.1 Cronobacter sakazakii 6934.564459GCA_001309175.1Cronobacter sakazakii ES354.354151GCA_000409405.1Cronobacter sakazakii 83994.654542GCA_000467775.2表1(续)菌株号基因组长度/Mb基因数基因组序列①Cronobacter sakazakii NM12404.494409GCA_000974965.1Cronobacter sakazakii ATCC BAA-8944.534382GCA_000017665.1Cronobacter sakazakii ES15 4.274084GCA_000263215.1Cronobacter sakazakii SP2914.524390GCA_000339015.1 Cronobacter sakazakii ES7134.554364GCA_001971035.1Cronobacter sakazakii NCIMB 82724.574450GCA_000463095.2Cronobacter sakazakii HPB51744.444291GCA_000698225.1Cronobacter sakazakii 7124.424320GCA_001308945.1Cronobacter sakazakii 7134.554446GCA_001309345.1Cronobacter sakazakii 7144.554455GCA_001309305.1Cronobacter sakazakii 7154.564473GCA_001309185.1Cronobacter sakazakii 716 4.804723GCA_001309235.1Cronobacter sakazakii NCTC81554.614471GCA_001277275.1
①:基因组序列来自NCBI 数据库(https://www.ncbi.nlm.nih.gov/genome)。
1.2基因表达量测定 使用RNA 抽提试剂盒(TaKaRa, 中国)和反转录试剂盒(TaKaRa, 中国)进行C.sakazakiiATCC 29544菌株的RNA提取和反转录,使用SYBR green PCR试剂盒(Qiagen, 德国)检测表达量。
1.3候选靶蛋白的克隆、表达和纯化 基因克隆引物见表2,表达载体使用pet28a,表达诱导条件:0.1~0.5 mmol/L IPTG。Ni亲和层析柱纯化蛋白。聚丙烯酰胺凝胶电泳(SDS-PAGE)鉴定蛋白纯度。
表2 基因克隆引物
Tab.2 Primers for gene clone

基因引物(5'-3')酶切位点WP_071601484.1_2838F:GGAATTCCATATGGCCAGCCAGAGCGACGACTACGTGCNdeI, HindIIIR:CCCAAGCTTTTAGTGATGATGATGGTGATGAGGCACCGGTGCCAGCAGWP_029039202.1_2678F:GGAATTCCATATGCTGGAAGGCGACGACGCGCTGCCGCNdeI, HindIIIR:CCCAAGCTTTTAGTGATGATGATGGTGATGTGGCGTACCCTCGCGAWP_007892488.1_775F:GGAATTCCATATGATGAAAAACAGGTTTCGCCAGGGAGANdeI, XhoIR:CCCTCGAGTTAGTGATGATGATGGTGATGTCCGGCCGTGAGCGCCWP_007892506.1_762F:CTAGTCTAGAATGATAAAAAAAATCTCGCTGCTGAXbaI, HindIIIR:CCCAAGCTTTTAGTGATGATGATGGTGATGGAAGGTGTAGCTGCCTWP_007865575.1_4015F:GGAATTCCATATGTCGGCGCTGGAAGCGCCACGCACCGNdeI, HindIIIR:CCCAAGCTTTTAGTGATGATGATGGTGATGAAACTGCAAGCGCAGC
下划线序列为不同限制性内切酶酶切位点的核苷酸序列
1.4蛋白偶联 候选靶蛋白和BioMagPlus羧基磁珠(Polysciencss,美国)偶联,活化及偶联过程按BioMagPlus说明书进行。紫外分光光度计测定偶联前后280 nm吸光度,计算磁珠包被蛋白的效率。包被效率=(OD偶联前-OD偶联后)/OD偶联前×100%。
1.5ssDNA文库的构建和引物合成 用于WP_007892506.1_762蛋白适配子筛选的文库和引物序列见表3。
表3 用于WP_007892506.1_762蛋白适配子筛选的文库和引物序列
Tab.3 Library and primers for adaptor screening of protein WP_007892506.1_762
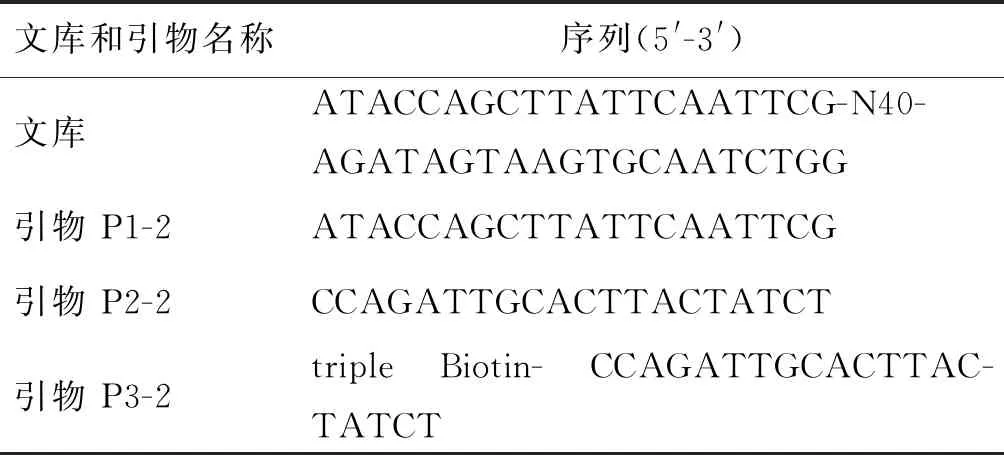
文库和引物名称序列(5'-3')文库ATACCAGCTTATTCAATTCG-N40-AGATAGTAAGTGCAATCTGG引物 P1-2ATACCAGCTTATTCAATTCG引物 P2-2CCAGATTGCACTTACTATCT引物 P3-2triple Biotin- CCAGATTGCACTTAC-TATCT
1.6SELEX 筛选 ssDNA文库变性处理后与候选靶蛋白包被的羧基磁珠混匀,加入酵母tRNA竞争结合,反应体系置磁力架,吸弃上清。缓冲液洗去未结合的 ssDNA,重复洗涤2 次。结合了 ssDNA 的羧基磁珠中加入去离子水,100 ℃加热 5 min,置磁力架 3~4 min,取上清为 PCR 模板。反应体系:模板 5 μL,P1-1或P1-2 (10 μmol/L)1 μL,P3-1或P3-2 (10 μmol/L) 1μL,dNTP(各2.5 mmol/L) 4 μL,10×Buffer 5 μL,Taq DNA聚合酶 0.25 μL,ddH2O 36.75 μL,共50 μL。PCR产物使用DNA纯化试剂盒 (Qiagen,德国)进行纯化。纯化后的一端标有生物素的dsDNA与链霉亲和素磁珠结合,碱分离获得不带生物素的另一条ssDNA,测定核酸浓度,作为下一轮筛选的富集库。重复以上步骤进行筛选。从第三轮每轮使用空磁珠反筛。每隔1轮使用OliGreen 荧光染料标记ssDNA 文库,TBS-380荧光仪测试筛选前后的荧光强度,计算ssDNA与靶蛋白结合百分率。
1.7适配子克隆和测序 WP_007892506.1_762第10轮筛选产物进行PCR扩增,体系:模板 2 μL,P1-1或P1-2(10 μmol/L)1 μL,P2-1或P2-2(10 μmol/L)1 μL,2×Taq PCR Master Mix 25 μL,加ddH2O 至50 μL。扩增产物2% 琼脂糖凝胶电泳检测,回收纯化。纯化产物与pMD19-T载体(Invitrogen, 美国)连接,连接产物转化E.coliDH5α感受态细胞。挑取阳性单克隆于LB(Amp 100 μg/mL)37 ℃过夜。送上海生工测序。
1.8适配子结构分析 使用RNA structure软件模拟适配子二级结构。
1.9单个适配子的获得 根据候选适配子的核酸序列,人工合成5’端标记地高辛的候选适配子。
1.10适配子与靶蛋白的亲和力测定 分别配制50 μg/mL候选靶蛋白、BSA溶液,取100 μL于微孔板, 4 ℃过夜。封闭液37 ℃封闭1 h。弃去孔内液体, 200 μL PBS洗涤4遍。地高辛适配子浓度500 nmol/L变性处理后每孔加入100 μL,37 ℃孵育1 h。弃去孔内液体,200 μL PBST洗涤4遍。每孔加入100 μL(1∶1 000)碱性磷酸酶标记的抗地高辛抗体,37 ℃孵育30 min,200 μL PBST洗涤3遍。加入100 μL底物,37 ℃显色10 min,加入50 μL终止液,酶标仪405 nm读数。
1.11生物素化适配子和链霉亲和素磁珠偶联试验
根据Dynabeads M-280 链霉亲和素磁珠(Thermo Fisher Scientific, 美国)单链寡核苷酸偶联剂量,每毫克磁珠偶联200 pmol/L的生物素化适配子,偶联步骤按说明书进行。
1.12适配子磁珠吸附性能比较 使用C.sakazakiiATCC29544的培养物对适配子磁珠的吸附能力进行比较。制备一定浓度的菌液,平板菌落计数。同时,各取1 mL菌液加入0.5 mg适配子磁珠中,37 ℃孵育1 h,磁力架分离磁珠,取上清进行平板菌落计数。吸附率=(吸附前菌落数-吸附后菌落数)/吸附前菌落数×100%。
1.13适配子磁珠捕获目标菌特异性实验 选取非目标菌:大肠埃希氏菌ATCC29522、肠炎沙门氏菌ATCC54001、福氏志贺氏菌ATCC12022、蜡样芽孢杆菌CMCC63033进行磁珠捕获特异性研究,菌株处理方法和吸附率计算同1.12。
2 结 果
2.1阪崎克罗诺菌核心基因组分析和编码蛋白亚细胞定位 阪崎克罗诺菌核心基因组分析显示共包括2 188个核心基因。使用pSORTb 3.0 软件对核心基因编码蛋白进行亚细胞定位预测,其中34个蛋白位于外膜(表4)。选择其中15个在阪崎克罗诺菌种内氨基酸序列相似度≥90%,与其它22种常见食源性细菌(Escherichiacoli、Salmonellaenteritidis、Salmonellatyphimurium.、Shigelladysenteriae、Shigellaflexneri、Shigellaboydii、Shigellasonnei、Bacilluscereus、Bacillusthuringiensis、Bacillusmycoides、Staphylococcusaureus、Yersiniapseudotuberculosis、Yersinia.enterocolitica、Vibrioparahaemolyticus、Vibriovulnificus、Vibriofluvialis、Vibrioalginolyticus、Campylobacterjejuni、Listeriamonocytogenes、Listeriainnocua、Clostridiumperfringens、Pseudomonasaeruginosa)氨基酸序列最高相似度≤70%的编码蛋白作为筛选适配子的候选靶蛋白。
表4 阪崎克罗诺菌核心基因组中的外膜蛋白编码基因
Tab.4 Outer membrane protein encoding genes in core genome ofCronobactersakazakii

序号基因编码蛋白序列ID1nlpENlpEWP_007896680.1_742bmaABamAWP_004386130.1_923traTTraTWP_004388372.1_1444-Hypothetical proteinWP_071601447.1_2405-chitinaseWP_007889641.1_2556fecAFecAWP_007898891.1_4257-Sucrose porinWP_007896602.1_5128-phospholipase AWP_004388410.1_7129-outer membrane vitamin B12 receptor①②WP_007892506.1_76210papCPilus assembly protein PapC①②WP_007892488.1_77511papCPilus assembly protein PapCWP_029039177.1_101412envCmurein hydrolase activator EnvCWP_032975803.1_105213hofQDNA transporter HofQ①②WP_071601442.1_125214-maltoporinWP_007888874.1_137915-hemolysin activator proteinWP_029039409.1_139716blcOuter membrane lipoprotein BlcWP_071844239.1_145217-fimbrial assembly proteinWP_029039348.1_151418-murein transglycosylase AWP_029039229.1_176519-host specificity protein①②WP_046623022.1_217120mlaAphospholipid-binding lipoprotein MlaAWP_004387036.1_220321-phage tail length tape measure proteinWP_029039590.1_235222-inverse autotransporter beta-barrel domain-containing protein①②WP_052367211.1_239223-fimbrial assembly protein①②WP_029039202.1_267824-hypothetical protein①②WP_007888065.1_277725-invasin①②WP_071601484.1_283826-TonB-dependent siderophore receptor①②WP_029039152.1_287327-transporter①②WP_029039384.1_298028-Autotransporter domain-containing esterase①WP_007900185.1_313629-Hypothetical proteinWP_029039106.1_319230-Hypothetical protein①WP_029038961.1_340331mipAMltA-interacting protein MipA①②WP_007893938.1_348432-host specificity protein①WP_046623084.1_352033-phospholipid: lipid A palmitoyltransferase①WP_007865575.1_401534-TonB-dependent siderophore receptorWP_029039173.1_4042
①:在阪崎克罗诺菌种内氨基酸序列相似度≥90%的蛋白, 与其它种细菌氨基酸序列最高相似度≤70%的蛋白。
②:无跨膜区蛋白。
2.2候选靶蛋白跨膜区预测 因为外膜蛋白的跨膜区位于外膜的内部,如果和适配子结合的部位位于这个部分,则会影响适配子和目标菌的结合效果。因此使用TMHMM v2.0软件对15个候选蛋白进行跨膜区预测,选择跨膜区少或没有的蛋白作为进一步的候选蛋白。预测结果显示:13个蛋白不具有跨膜区(表4),做为下步试验候选蛋白。
2.3候选蛋白表达量测定 以阪崎克罗诺菌ATCC 29544培养物为检测对象。检测上述13个候选蛋白的表达量,以16S rDNA为内参(表5),选择表达量最高的WP_071601484.1_2838,WP_029039202.1_2678,WP_029039152.1_2873,WP_007892506.1_762,WP_007865575.1_4015进行基因克隆和表达。
表5 候选蛋白编码基因荧光定量PCR检测引物及检测结果
Tab.5 Primers and detection results of fluorescence PCR

基因上游引物(5'-3')下游引物(5'-3')Ct①WP_007892506.1_762GCGTGGGTGGGCTTTCTGTGGCAGTCACAATTTCGACCA23.25WP_007892488.1_775TATCCTGGCGTGTTTGTGGCTACTTTCTGCGACGTGTTCT32.27WP_071601442.1_1252TCGCTGGTGGCGGATGAAAGACGACGCCCTCAACACCC35.68WP_046623022.1_2171AGGCGGTCAGCGGTCGTAATTCCAGGTGCCGTCGTTGTTGUNWP_052367211.1_2392TGATTTCCTGGCTCCGTTCTTACCGCCGAACATCCAGT29.17WP_029039202.1_2678GGCACGCATCACGCTTATTCCTGCTGTACCGTCACTTTGC21.20WP_007888065.1_2777ATGCCGCCACAGCCTATCCGGTTACTACGGTCTGAACCTG33.25WP_071601484.1_2838GACGCTGTCGGTATTCCTTGCCGGCGTCAGGCTCAGCGC26.17WP_029039152.1_2873CAGACGCCGACGATTACGACTGGCGTTGCTGTTGCTGTT27.26WP_029039384.1_2980 TCCCGCAGATTCAGCACCGCAACGACGACGGCAACT30.66WP_007893938.1_3484CGCAAAGCAACGCTGATGCCGTTGGTGTAGCGATAGAG33.49WP_046623084.1_3520AAGTGGAAGCGGTGATGGACGAAACTCACCATCCGGTC35.02WP_007865575.1_4015ATCCCGATTCCGCTGGTGCGCAGCCAGGCGAAGTAA25.1116SrDNAATGGACGCAAGTCTGATGGTGACTTTCTGGTTGATTACCG9.16
①Ct为两次实验的平均值。
2.4基因克隆、表达、蛋白纯化及羧基磁珠包被 5个基因通过克隆,蛋白表达和Ni柱纯化后,WP_007892506.1_762基因获得大量纯化蛋白,SDS-PAGE电泳检测未发现明显的非特异性条带(图1),选择WP_007892506.1_762编码蛋白进行适配子筛选。使用500 μg WP_007892506.1_762蛋白包被10 mg羧基磁珠,包被效率为93%。

图1 WP_007892506.1_762纯化蛋白SDS-PAGE电泳检测图Fig.1 SDS-PAGE of WP_007892506.1_762 purified protein
2.5WP_007892506.1_762蛋白适配子筛选 使用WP_007892506.1_762蛋白包被的羧基磁珠对人工合成的适配子文库进行筛选,经过10轮筛选,结合率不再上升。最高结合率为43.2%(表6)。
表6 WP_007892506.1_762蛋白适配子筛选参数
Tab.6 Screening parameters of adaptor for WP_007892506.1_762

Selex轮数ssDNA文库/pmol蛋白/μg结合率(%)1200010-21301012.139810-4120817.551108-6130823.17896-8120639.89966-10110643.2111206-1280436.8131404-1490433.4
2.6候选适配子一级结构 在WP_007892506.1_762的TA克隆中各挑选20个克隆,测序,20个克隆中,1个为空载体,有3个不同的核酸序列(表7)。
表7 WP_007892506.1_762 蛋白候选适配子序列
Tab.7 Sequences of candidate adaptors for WP_007892506.1_762
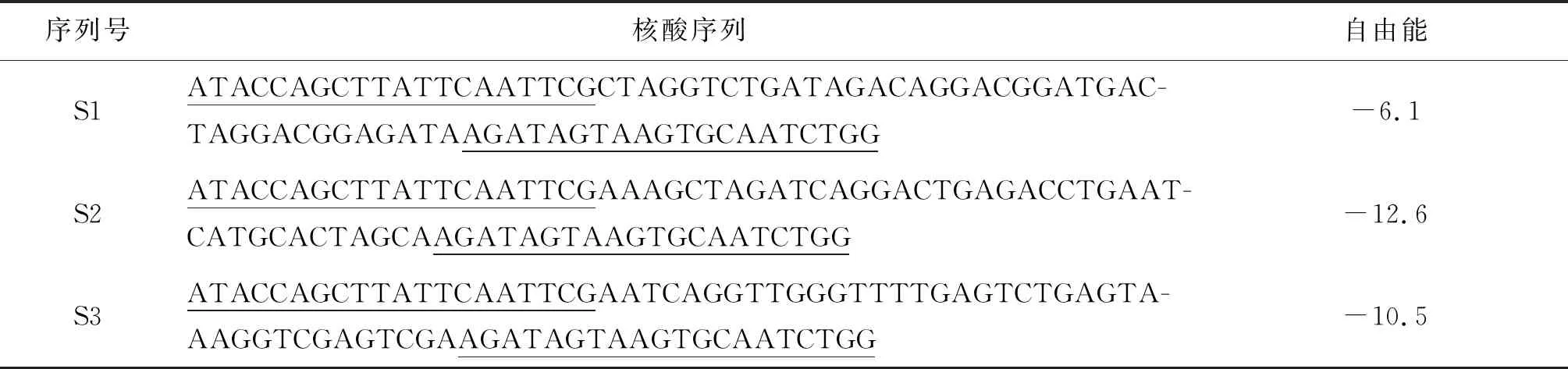
序列号核酸序列自由能S1ATACCAGCTTATTCAATTCGCTAGGTCTGATAGACAGGACGGATGAC-TAGGACGGAGATAAGATAGTAAGTGCAATCTGG-6.1S2ATACCAGCTTATTCAATTCGAAAGCTAGATCAGGACTGAGACCTGAAT-CATGCACTAGCAAGATAGTAAGTGCAATCTGG-12.6S3ATACCAGCTTATTCAATTCGAATCAGGTTGGGTTTTGAGTCTGAGTA-AAGGTCGAGTCGAAGATAGTAAGTGCAATCTGG-10.5
下划线序列为ssDNA文库的保守序列区。
2.7靶蛋白和候选适配子亲和力检测 使用酶联适配子直接检测法鉴定靶蛋白与候选适配子的结合力,结果显示:WP_007892506.1_762蛋白与适配子 S1结合后的吸光度值明显高于 BSA 和空白对照组(P=0.0021;P=0.0020), 见表8。
表8 靶蛋白和候选适配子的结合亲和力
Tab.8 Binding affinity between target protein and candidate adaptors
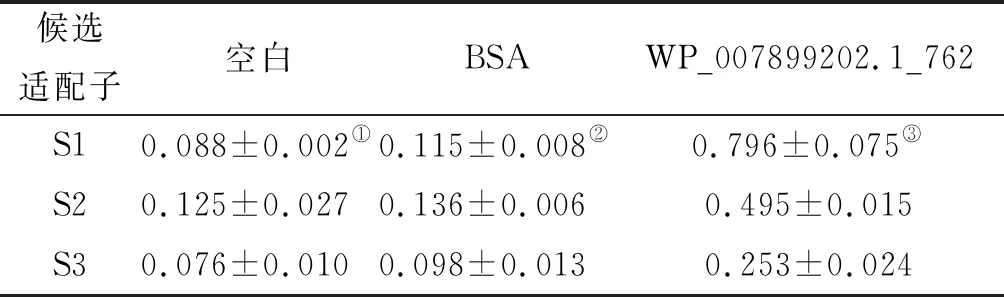
候选适配子空白BSAWP_007899202.1_762S10.088±0.002①0.115±0.008②0.796±0.075③S20.125±0.0270.136±0.0060.495±0.015S30.076±0.0100.098±0.0130.253±0.024
以上数值为3次实验的平均值
③和①、② 相比具有显著性差异
2.8适配子磁珠吸附性能比较 阪崎克罗诺菌ATCC29544的培养物和S1、S2、S3适配子磁珠的吸附效率见表9, 结果表明:S3适配子磁珠不具有目标菌吸附能力, S1适配子磁珠吸附效率较高,最高为23.1%。
表9 适配子磁珠吸附性能比较(%)
Tab.9 Adsorptivity of adaptors magnetic bead

原始菌量(CFU)S1适配子磁珠吸附率S2适配子磁珠吸附率S3适配子磁珠吸附率6.5×105---6.5×10420.015.5-6.5×10323.112.4-6.5×10215.4--
以上数值为两次实验的平均值
2.9S1、S2 适配子磁珠菌体吸附特异性研究 选取非目标菌:大肠埃希氏菌ATCC29522、肠炎沙门氏菌ATCC54001、福氏志贺氏菌ATCC12022、蜡样芽孢杆菌CMCC63033进行磁珠吸附特异性研究。原始菌量分别为2.1×105~2.1×102CFU, 8.5×105~8.5×102CFU, 5.2×105~5.2×102CFU和6.8×105~6.8×102CFU。结果表明:S1、S2 适配子磁珠对非目标菌无吸附能力。
2.10适配子二级结构 根据自由能最低原则,使用RNAstructure软件对适配子S1和S2的二级结构进行模拟,结果显示:适配子的二级结构以茎环结构和发夹结构为主(图2)。
3 讨 论
传统的微生物学检测方法步骤多,时间长,较难满足快速检测的需要。分子生物学和免疫学检测方法虽然快速、方便,但当样品中的目标菌浓度较低时,仍需要较长时间的前增菌过程。针对此种情况,免疫磁珠特异性富集样品中目标菌的方法得到快速发展,可以有效富集样品中的目标菌,缩短样品的前处理过程[12-13]。免疫磁珠是利用超顺磁性磁珠表面的功能基团,主要包括氨基、羧基和巯基等结合相应的抗体,再利用抗体和目标菌的特异性结合来实现菌体的富集。但免疫磁珠的制备过程相对复杂,需要通过动物免疫获得相应的特异性抗体,需要考虑动物是否会对靶标物质产生免疫耐受,而且成本较高。适配子是体外人工合成的单链DNA或RNA分子,长度一般为25-90nt, 具有一定的空间二级结构。通过SELEX技术,研究人员可以从人工合成的随机寡核苷酸文库中筛选针对靶物质(蛋白质、代谢产物、金属离子、药物、细菌、病毒等)的适配子[14-16]。适配子可以和靶物质高亲和力、高特异性结合形成靶物质-适配子复合物,具有传统单克隆抗体无法比拟的优势如:靶物质范围更加广泛;具有更高的亲和力和特异性;随机序列折叠形成的空间结构具有高度的可变性和多样性;在制备过程中不需进行动物免疫,成本较低。近年来,SELEX技术呈现跨越式的发展,磁珠分选-SELEX、细胞-SELEX、定量PCR-SELEX、流式细胞-SELEX、毛细管电泳-SELEX 、生物传感器-SELEX等技术相继出现[17-21]。目前,上述筛选方法的单一或联合使用已经使适配子的筛选效率极大提高。在新型SELEX技术应用前提下,已有包括朊病毒、溶藻弧菌、铜绿假单胞菌、金黄色葡萄球菌毒素B(SEB)和金黄色葡萄球菌毒素A(SEA)等病原微生物和毒素蛋白的适配子得到筛选[8-9,22-23],并应用于相应病原体和毒素的检测。适配子技术在微生物检测领域展现出广阔的应用空间。但是,目前对病原菌特异性适配子的筛选主要以病原菌菌体作为筛选的靶物质。以菌体作为筛选靶标,操作简单,但存在目标位点过多,适配子特异性不强的问题。在本研究中,我们首次采用比较基因组学,生物信息学预测的方法选择阪崎克罗诺菌适配子筛选的靶蛋白,以阪崎克罗诺菌中保守且较特异性存在的外膜蛋白为靶标,成功筛选到阪崎克罗诺菌特异性适配子,为病原菌核酸适配子的筛选提供了新的思路。但筛选到的适配子捕获目标菌的效率与已广泛应用的免疫磁珠相比[12-13]存在较大差距。阪崎克罗诺菌特异性适配子SELEX筛选优化工作需要进一步进行。

图2 适配子S1、S2的二级结构Fig.2 Secondary structures of adaptors S1 and S2
利益冲突:无
引用本文格式:张云怡,梅玲玲,占 利,等.阪崎克罗诺菌特异性适配子筛选研究[J].中国人兽共患病学报,2020,36(1):12-19,24. DOI: 10.3969/j.issn.1002-2694.2019.00. 192
