多重PCR技术研究进展
2020-03-11钟泽澄王进张师音
钟泽澄,王进,张师音
·综 述·
多重PCR技术研究进展
钟泽澄1,2,王进2,张师音1,2
1 厦门大学 公共卫生学院,福建 厦门 361100 2 厦门大学 国家传染病诊断试剂与疫苗工程技术研究中心,福建 厦门 361100
多重PCR (Multiplex polymerase chain reaction,MPCR) 是指通过一次PCR反应同时对多个靶标进行扩增,结合一定的检测手段对扩增产物进行检测从而实现对多个靶标进行诊断的技术。MPCR具有高效率、高通量、低成本的特性而被深入研究。目前MPCR技术已被广泛应用于科学研究和疾病诊断等领域,文中从MPCR扩增与检测两方面概述了MPCR技术发展及其应用,讨论了MPCR技术的优点及存在的问题,并且提出将反应体系分隔成小液滴或结合管式对流PCR (Capillary convective PCR,CCPCR) 技术的方式有望提高固相载体表面的扩增效率,从而开发出扩增效率高、一致性好、体系稳定、检测重数高的多重PCR技术。
多重PCR,熔解曲线,微流控,膜层析,Luminex液相芯片
传统的多重PCR (Multiplex polymerase chain reaction,MPCR)是在普通PCR的基础上,在同一个反应体系中加入不同的引物对,针对不同的模板或同一模板的不同区域进行特异性的扩增,从而得到多个目的片段,结合一定的检测手段进而实现同时对多个靶标进行诊断的技术。自Chamberlain[1]于1988年首次提出这个概念以来,MPCR已经应用于许多领域,包括基因突变与缺失、基因分型与定量、遗传检测、伴药诊断等[2-6]。MPCR所涵盖的范围广,能够同时对多个靶标进行检测,大幅提升检测效率的同时,还降低了检测成本。
随着科学技术的发展,MPCR技术在扩增和检测方面已经取得了许多新的突破。在扩增方面,不再局限于在同一反应管中进行扩增,而是将不同的引物对和模板分散于相对独立的空间中进行扩增;在检测方面,不断有新的超多重检测技术的出现。文中从MPCR扩增与检测两方面概述了MPCR技术的发展及其应用,对影响MPCR扩增与检测效果的因素进行了总结,并对提高固相载体表面的扩增效率提出针对性策略。
1 多重荧光定量PCR技术
1.1 基于荧光探针的多重PCR技术
多重荧光定量PCR (Multiple fluorescence quantitative PCR)技术是在荧光定量PCR技术的基础上,利用几种不同荧光基团的组合,结合仪器对不同通道荧光的检测能力实现对多个靶标的实时定量检测。TaqMan水解探针 (Hydrolysis probes) 是多重荧光PCR体系中常用的一种探针,探针一端标记荧光基团,另一端标记淬灭基团,通过在不同序列末端标记不同荧光基团及相应的淬灭基团,即可形成不同的TaqMan水解探针,将上述探针及相应的扩增引物加至同一反应体系,即可实现对多个靶标的共同检测。Weller等[7]以TaqMan探针为基础,成功构建了两重PCR扩增和检测体系,Zhang等[8]通过单管逆转录反应成功地实现了对肠病毒71型 (EV71)、柯萨奇病毒A16 (CA16) 以及总肠道病毒 (EVs) 的三重检测。分子信标 (Molecular beacon) 是多重PCR体系中另一种常用的探针,基于荧光共振能量转移 (Fluorescence resonance energy transfer,FRET) 的原理,当体系中不存在特异性的靶标时,分子信标会自发形成茎环结构,淬灭基团与荧光基团相互接近从而发生FRET,不会产生荧光。如果体系中存在特异性的靶标,在一定的条件下,茎环结构会打开并且与靶标进行复性,从而产生荧光信号。那么在同一个反应体系中加入几种靶标特异性的分子信标就能够实现对多个靶标的同时检测。Vet等[9]针对4种不同的靶标设计了4种不同的分子信标并且首次在四色荧光PCR仪上实现了四重的检测,El-Hajj等[10]利用五色分子信标探针实现了对结核杆菌利福平耐药突变的检测。
1.2 基于探针编码的多重PCR技术
基于水解探针和分子信标探针的多重荧光定量PCR技术的优点是简便快捷,然而受仪器荧光检测通道的限制,往往最多只能达到四重或五重检测。组合探针编码 (Combination probe coding) 技术的开发,使得目前的多重实时核酸扩增可检测的靶标数目多于仪器可检测的数目。通过对4种不同的荧光基团进行相互组合可以形成15种指示探针 (图1),在多重反应体系中加入多种靶序列特异的置换探针 (Displacing probes),那么在检测通道有限的情况下仍能够实现对多种不同的靶标同时进行扩增和检测。Huang等利用多色组合探针编码技术(MCPC) 实现了对8种食源性病原菌的鉴定以及通过4色组合探针编码技术实现了对15种不同型别的HPV病毒进行分型[11-12]。
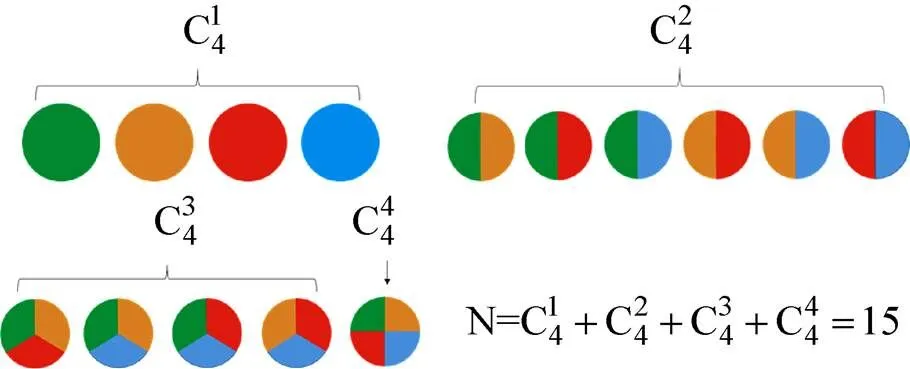
图1 荧光基本元素之间的相互组合
1.3 基于荧光染料的多重PCR技术
非特异性的荧光染料嵌入到DNA双链小沟中后,在特定的激发光激发后将会产生一定波长的荧光。以此为基础发展了熔解曲线 (Melting curve) 分析法,利用不同DNA序列具有不同m(Melting temperature) 值的特征,在PCR反应结束后,通过程序升温使双链逐渐解链成单链,当达到双链特异的m值所对应的温度时,荧光强度大幅度降低,利用这样的原理可以对PCR中不同长度的双链产物或相同长度不同GC含量的双链进行分析 (图2)。Singh等[13]利用多重实时PCR熔解曲线分析的方法,成功地实现了同时对氨苄西林、链霉素、磺胺、四环素、氯霉素耐药的沙门氏菌的检测与辨别,Mendes等[14]建立了一种多重熔解曲线分析方法并且实现了对不同基因编码的金属-β-内酰胺酶型别的检测和鉴别。针对不同的型别设计了特异性的引物对,从而对模板进行扩增生成不同的扩增子,扩增子之间m值各有差异,最后通过熔解曲线峰的位置进行鉴别。在熔解曲线分析技术的基础上发展而来的高分辨率熔解曲线技术是一种利用饱和染料,借助高分辨率的仪器,实现对单个核苷酸熔解温度不同从而形成不同形态熔解曲线,进而对基因进行分析的新技术,它具有极高的灵敏度,能够检测出单个碱基之间的差别,目前主要应用于单核苷酸多态性分析、甲基化分析、基因突变、基因分型等领域[15-18]。

图2 多重熔解曲线
1.4 基于荧光探针熔解曲线的多重PCR技术
由于荧光基团本身发射的不是单一波长的荧光,而且PCR仪对不同荧光基团发射的荧光信号的区分度有限,所以现有的PCR仪只能检测4–5种不同的荧光信号。而基于荧光染料的多重PCR技术由于染料不具备特异性的识别能力,所以同一反应体系中通常只能使用一种荧光染料。为了弥补这两种方法的不足而开发出来的荧光探针熔解曲线技术 (Fluorescence probe melting curve technique) 具有成本低、多重检测能力更强的优点。通过在靶序列中选取相对保守的区域设计通用扩增引物,然后在引物对之间选取差异度较大的区域设计能够识别各种目标核酸序列的特异性探针,探针在扩增的过程中不会被完全水解,并且根据检测通道的不同将探针进行分组,每个通道中包含多个特异性探针,通过调整探针的长度或GC含量对每条探针的m值进行人为的调控,使得同一检测通道内针对不同靶标的检测探针m值都间隔合适的差值,每个荧光通道均可定性检测多个靶标,最后通过熔解曲线进行分析。Elenitoba-Johnson等[19]利用荧光探针熔解曲线技术成功地区分了27种可能发生碱基替换的序列。Liao等[20]利用荧光探针熔解曲线技术成功地实现了对15种高危型HPV的分型以及实现了对48种人类单核苷酸多态性的分型和分析。
2 基于微流控的多重PCR技术
多重荧光定量PCR技术需将多个引物对、探针以及模板加入到同一个反应管中进行反应,伴随检测重数的增加,同管内核苷酸链数目也会增加,各核苷酸链之间互搭形成二聚体甚至多聚体的可能性大幅上升,轻则导致非特异性扩增的出现,靶标扩增效率下降,检测灵敏度和特异性降低,重则导致非特异性扩增占据主导,靶标扩增失败,造成检测结果假阳性和/或假阴性。基于微流控芯片 (Microfluidic chip) 的多重PCR技术通过在芯片微孔中预装填不同的引物对的方法将各重扩增限制在各自的独立空间内,再通过液流控制系统将模板引流到不同的微孔中,进而在微孔中进行特异的PCR扩增反应,最后再进行终点定性检测 (图3)[21],从而实现对多个靶标的区分和鉴别[21-24]。Poritz等结合微流控技术研发了一种可以同时检测多种病原体的诊断平台,称为“FilmArray”,该平台成功地实现了在1 h内对21种常见的病毒或细菌的检测和鉴别[25-27]。Cai等[28]设计的一种微流控芯片利用双向电泳技术和多重阵列PCR终点荧光检测技术实现了在全血中对不同病原菌的即时检测。多重液滴式数字PCR的作用原理与基于微流控芯片的多重PCR的原理相似,通过芯片装置和液流控制系统产生成千上万个液滴,每个液滴中只包含单个目的DNA分子,这样就可以使不同的靶标处于相对独立的空间进行各自特异性的扩增反应,最后通过计算各靶标发生有效扩增的液滴数,从而实现绝对定量。Jackson等[29]利用液滴式数字PCR成功地实现了对血液游离DNA (Free DNA) 中罕见癌症突变的多重检测,利用液滴式数字PCR对珍贵的临床标本进行多靶标的检测,极大地提高了标本的利用率并降低了成本[30-31]。基于微流控芯片的多重PCR技术和多重液滴式数字PCR的优点是在一定程度上避免了非特异性扩增产物的生成以及在荧光定量PCR的基础上增加了检测重数。但是当模板浓度较低时,基于微流控的方法将模板分流至含特异性引物微孔中的概率降低,易产生假阴性结果;而多重液滴式数字PCR的方法使每个液滴中仅包含单重引物和模板的概率相对较低,往往一个液滴中包含多对引物或者液滴中的引物与模板为非特异性配对,另外它对模板的要求较高,浓度过高的模板不能保证生成的每个液滴中仅包含单个目的DNA分子。此外在检测方面,上述两种技术都需要配合扩增的特殊性而使用到高精密度的检测仪器,其技术的复杂性使其实现较为困难。

图3 基于微流控的多重PCR[21]
3 基于固相载体的多重PCR技术
3.1 基于膜层析的多重检测技术
膜层析技术 (Membrane chromatography) 是在柱层析技术上发展而来的一项技术,以膜介质为基质,偶联相应的离子交换、亲和、疏水等化学基团,制作而成的一种新型的层析介质。膜层析技术具有简单快速并且能够自动分离液体混合物的优点,而多重PCR技术具有有效富集目的片段的优点,将这两种技术有机地结合在一起能够形成一种灵敏度高、特异性好、成本低、简便快速的多重核酸检测方法。该方法提前将靶标特异性的捕获探针固定在层析膜的不同位置上,然后使变性后的多重PCR扩增产物通过毛细作用经过层析膜,固定在特定位置的捕获探针通过碱基互补配对的作用将末端带有胶体金或荧光素的产物捕获,而非特异的扩增产物及引物被洗脱,最后通过肉眼或荧光检测仪对产生的荧光进行检测和识别 (图4)[32]。Mao等[33]基于胶体金标记的核酸探针和侧流试纸条建立了一种现场检测目的基因的方法,利用这种核酸层析金标试纸条能够在15 min内检测出核酸样本。Carter等[34]发明的侧流微阵列方法能够快速、灵敏地对多种核酸进行检测,并且不需要专门的仪器设备,这种方法以微型侧流设备为基础,利用核酸杂交介导靶标的捕获。Gomez-Martinez等[32]开发的多重核酸检测试纸条在现场实现了对108例献血者血型相关基因型的准确鉴定。基于膜层析的多重检测技术以其操作简单、携带便捷等优点拓宽了可检测的范围,使检测不再局限于实验室,对于现场检测也能够实现[35-37],但是膜层析技术中样品的阻滞、非特异性杂交等问题仍然是今后需要解决的问题。
3.2 基于Luminex液相芯片的多重检测技术
液相芯片技术 (Liquid chip technology) 被称为后基因组时代的芯片技术,也被称为xMAP技术。它可以同时对1份标本中的100种不同目的分子进行检测,并能在30 min内检测96个不同的样本。其液相微球编码原理为,将两种荧光染料分别用10种不同的浓度两两混合形成10×10的荧光配比阵列,并对微球进行染色,得到100种不同荧光编码的微球。将荧光编码且共价结合了不同捕获探针的微球悬浮于一个液相体系中,先加入待检测分子与其反应,再加入带有荧光标记的报告分子,从而形成“微球-捕获探针-靶标-报告分子”复合物。检测时,该复合物在鞘液的作用下将逐个通过检测通道,接受两束不同波长的激发光照射并对相应的发射光进行检测,其中一束激光用于识别微球种类,即判定该种微球是对哪种特定的靶标进行检测,另一束激光用于检测微球上报告分子的荧光强度,从而实现对待测物质的定性和定量分析 (图5)[38]。Sherry等[39]利用Luminex液相芯片技术实现了对沙门氏菌和其他病原菌的检测和鉴定。Rajeswari等[40]利用Luminex液相芯片技术和液滴生成技术成功地实现了对禽流感病毒、传染性猴气管炎病毒和空肠弯曲菌的检测和鉴别。Song等[41]利用Luminex液相芯片技术同时对11种不同的突变型进行检测和分型。虽然基于Luminex液相芯片技术能够实现同时对多种不同病原体的检测和鉴别,但它仅在检测方面发挥了其多通量、高效率的特性,其实质上并没有解决多重扩增中众多引物、探针、模板间相互错配的问题。因此,未来开发出微球表面多重扩增以及Luminex液相芯片检测一体化的技术是核酸多重检测的重要研究方向。

图4 基于膜层析的多重检测[32]

图5 基于Luminex液相芯片的多重检测技术[38]
4 总结与展望
MPCR技术的优势在于它的高效性,通过一次扩增反应即可实现对多种病原体进行检测和鉴别,相较多个单重检测而言成本显著降低。MPCR有着广泛的应用前景,但是仍然存在许多影响MPCR扩增与检测效果的因素 (表1),其中扩增效率的一致性是最大的难题,检测重数越多,效率一致性往往越低。针对MPCR扩增效率一致性的问题,传统单管多靶标扩增的方式通过不断优化反应体系中缓冲液、酶、引物、探针等的用量,确保每一重的扩增效率保持一致,然而随着检测重数的增加,形成二聚体甚至多聚体的可能性也大幅上升,轻则导致非特异性扩增的出现,靶标扩增效率下降,检测灵敏度和特异性降低,重则导致非特异性扩增占据主导,靶标扩增失败,造成假阳性和/或假阴性,影响检测准确性。而基于微流控的MPCR则将每一重的扩增分布在各自相对独立的空间内进行,在一定程度上降低了引物二聚体的生成,保证了扩增效率,但是微流控的实现相对较为困难,且当模板浓度较低时,易产生假阴性结果。相比于上述方法,基于固相载体表面扩增的MPCR则将不同的引物对分配至不同的固相载体上,使得每个靶标的扩增集中于以特定固相颗粒为载体的表面,从根本上解决MPCR引物间相互干扰的问题,此外将固相载体表面扩增与Luminex液相芯片检测技术相结合能够同时对各固相颗粒及颗粒表面的扩增信号进行特异性识别,并且理论上能够实现100重的核酸扩增与检测,极大地提高了检测重数的同时又能保证各靶标的扩增互不干扰,但是由于需要将引物束缚至固相载体,这在一定程度上限制了引物与模板的碰撞概率并且导致其扩增效率降低,那么如何提高固相载体表面的扩增效率值得深入思考。针对上述问题,可通过将反应体系分隔成微小液滴,或结合管式对流PCR技术使反应体系在扩增的过程中循环流动,将有望提高引物与模板之间碰撞的概率,同时提高固相载体表面的扩增效率,从而开发出扩增效率高、一致性好、体系稳定、检测重数高的多重PCR技术。

表1 影响MPCR扩增与检测效果的主要因素
[1] Chamberlain JS, Gibbs RA, Rainer JE, et al. Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification. Nucleic Acids Res, 1988, 16(23): 11141–11156.
[2] Zakrzewski F, Gieldon L, Rump A, et al. Targeted capture-based NGS is superior to multiplex PCR-based NGS for hereditary BRCA1 and BRCA2 gene analysis in FFPE tumor samples. BMC Cancer, 2019, 19: 396.
[3] Ward DG, Baxter L, Gordon NS, et al. Multiplex PCR and next generation sequencing for the non-invasive detection of bladder cancer. PLoS ONE, 2016, 11(2): e0149756.
[4] Liu ZD, Zhang PH, He XY, et al. New multiplex real-time PCR approach to detect gene mutations for spinal muscular atrophy. BMC Neurol, 2016, 16: 141.
[5] Gautam R, Mijatovic-Rustempasic S, Esona MD, et al. One-step multiplex real-time RT-PCR assay for detecting and genotyping wild-type group A rotavirus strains and vaccine strains (Rotarix®and RotaTeq®) in stool samples. PeerJ, 2016, 4: e1560.
[6] Saito T, Kikuchi A, Kaneko A, et al. Rapid and sensitive multiplex single-tube nested PCR for the identification of five humanspecies. Parasitol Int, 2018, 67(3): 277–283.
[7] Weller SA, Elphinstone JG, Smith NC, et al. Detection ofstrains with a quantitative, multiplex, real-time, fluorogenic PCR (TaqMan) assay. Appl Environ Microbiol, 2000, 66(7): 2853–2858.
[8] Zhang SY, Wang J, Yan Q, et al. A one-step, triplex, real-time RT-PCR assay for the simultaneous detection of enterovirus 71, coxsackie A16 and pan-enterovirus in a single tube. PLoS ONE, 2014, 9(7): e102724.
[9] Vet JAM, Majithia AR, Marras SAE, et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proc Natl Acad Sci USA, 1999, 96(11): 6394–6399.
[10] El-Hajj HH, Marras SAE, Tyagi S, et al. Detection of rifampin resistance inin a single tube with molecular beacons. J Clin Microbiol, 2001, 39(11): 4131–4137.
[11] Huang QY, Hu QH, Li QG. Identification of 8 foodborne pathogens by multicolor combinational probe coding technology in a single real-time PCR. Clin Chem, 2007, 53(10): 1741–1748.
[12] Huang QY, Zheng LL, Zhu YM, et al. Multicolor combinatorial probe coding for real-time PCR. PLoS ONE, 2011, 6(1): e16033.
[13] Singh P, Mustapha A. Development of a real-time PCR melt curve assay for simultaneous detection of virulent and antibiotic resistant. Food Microbiol, 2014, 44: 6–14.
[14] Mendes RE, Kiyota KA, Monteiro J, et al. Rapid detection and identification of metallo-β-lactamase- encoding genes by multiplex real-time PCR assay and melt curve analysis. J Clin Microbiol, 2007, 45(2): 544–547.
[15] Vossen RHAM, Aten E, Roos A, et al. High-resolution melting analysis (HRMA)—more than just sequence variant screening. Human Mutat, 2009, 30(6): 860–866.
[16] Lopez GH, McBean BS, Wilson B, et al. Molecular typing for the Indian blood group associated 252G>C single nucleotide polymorphism in a selected cohort of Australian blood donors. Blood Transfus, 2015, 13(1): 78–85.
[17] Qiu C, Zhi YF, Shen Y, et al. High-resolution melting analysis of HPV-16 L1 gene methylation: A promising method for prognosing cervical cancer. Clin Biochem, 2015, 48(13/14): 855–859.
[18] Nemcova E, Cernochova M, Ruzicka F, et al. Rapid identification of medically importantisolates using high resolution melting analysis. PLoS ONE, 2015, 10(2): e0116940.
[19] Elenitoba-Johnson KSJ, Bohling SD, Wittwer CT, et al. Multiplex PCR by multicolor fluorimetry and fluorescence melting curve analysis. Nat Med, 2001, 7(2): 249–253.
[20] Liao YQ, Wang XB, Sha C, et al. Combination of fluorescence color and melting temperature as a two-dimensional label for homogeneous multiplex PCR detection. Nucleic Acids Res, 2013, 41(7): e76.
[21] Lu Y, Chen S, Wei L, et al. A microfluidic-based SNP genotyping method for hereditary hearing-loss detection. Anal Chem, 2019, 91(9): 6111–6117.
[22] Oshiki M, Miura T, Kazama S, et al. Microfluidic PCR amplification and MiSeq amplicon sequencing techniques for high-throughput detection and genotyping of human pathogenic RNA viruses in human feces, sewage, and oysters. Front Microbiol, 2018, 9: 830.
[23] Moltzahn F, Olshen AB, Baehner L, et al. Microfluidic-based multiplex qRT-PCR identifies diagnostic and prognostic microRNA signatures in the sera of prostate cancer patients. Cancer Res, 2011, 71(2): 550–560.
[24] Pholwat S, Liu J, Stroup S, et al. Integrated microfluidic card with TaqMan probes and high-resolution melt analysis to detect tuberculosis drug resistance mutations across 10 genes. mBio, 2015, 6(2): e02273–14.
[25] Poritz MA, Blaschke AJ, Byington CL, et al. FilmArray, an automated nested multiplex PCR system for multi-pathogen detection: development and application to respiratory tract infection. PLoS ONE, 2011, 6(10): e26047.
[26] Buss SN, Leber A, Chapin K, et al. Multicenter evaluation of the BioFire FilmArray gastrointestinal panel for etiologic diagnosis of infectious gastroenteritis. J Clin Microbiol, 2015, 53(3): 915–925.
[27] Altun O, Almuhayawi M, Ullberg M, et al. Clinical evaluation of the FilmArray blood culture identification panel in identification of bacteria and yeasts from positive blood culture bottles. J Clin Microbiol, 2013, 51(12): 4130–4136.
[28] Cai DY, Xiao M, Xu P, et al. An integrated microfluidic device utilizing dielectrophoresis and multiplex array PCR for point-of-care detection of pathogens. Lab Chip, 2014, 14(20): 3917–3924.
[29] Jackson JB, Choi DS, Luketich JD, et al. Multiplex preamplification of serum DNA to facilitate reliable detection of extremely rare cancer mutations in circulating DNA by digital PCR. J Mol Diagn, 2016, 18(2): 235–243.
[30] Zhong Q, Bhattacharya S, Kotsopoulos S, et al. Multiplex digital PCR: breaking the one target per color barrier of quantitative PCR. Lab Chip, 2011, 11(13): 2167–2174.
[31] Didelot A, Kotsopoulos SK, Lupo A, et al. Multiplex picoliter-droplet digital PCR for quantitative assessment of DNA integrity in clinical samples. Clin Chem, 2013, 59(5): 815–823.
[32] Gomez-Martinez J, Silvy M, Chiaroni J, et al. Multiplex lateral flow assay for rapid visual blood group genotyping. Anal Chem, 2018, 90(12): 7502–7509.
[33] Mao X, Ma YQ, Zhang AG, et al. Disposable nucleic acid biosensors based on gold nanoparticle probes and lateral flow strip. Anal Chem, 2009, 81(4): 1660–1668.
[34] Carter DJ, Cary RB. Lateral flow microarrays: a novel platform for rapid nucleic acid detection based on miniaturized lateral flow chromatography.Nucleic Acids Res, 2007, 35(10): e74.
[35] Zhuang LL, Ji YX, Tian PL, et al. Polymerase chain reaction combined with fluorescent lateral flow immunoassay based on magnetic purification for rapid detection of canine parvovirus 2. BMC Vet Res, 2019, 15: 30.
[36] Roth JM, De Bes L, Sawa P, et al.detection and differentiation by direct-on-blood PCR nucleic acid lateral flow immunoassay: development, validation, and evaluation. J Mol Diagn, 2018, 20(1): 78–86.
[37] Jauset-Rubio M, Tomaso H, El-Shahawi MS, et al. Duplex lateral flow assay for the simultaneous detection ofand. Anal Chemistry, 2018, 90(21): 12745–12751.
[38] Schmitt M. Pawlita High-throughput detection and multiplex identification of cell contaminations. Nucleic Acids Res, 2009, 37(18): e119.
[39] Dunbar SA, Jacobson JM. Quantitative, multiplexed detection ofand other pathogens by Luminex®xMAP™ suspension array//Schatten H, Eisenstark A, Eds. Totowa: Salmonella. Humana Press, 2007, 394: 1–19.
[40] Rajeswari PKP, Soderberg LM, Yacoub A, et al. Multiple pathogen biomarker detection using an encoded bead array in droplet PCR. J Microbiol Methods, 2017, 139: 22–28.
[41] Song YJ, Roumagnac P, Weill FX, et al. A multiplex single nucleotide polymorphism typing assay for detecting mutations that result in decreased fluoroquinolone susceptibility inserovars Typhi and Paratyphi A. J Antimicrob Chemoth, 2010, 65(8): 1631–1641.
Advances in multiple PCR technology studies
Zecheng Zhong1,2, Jin Wang2, and Shiyin Zhang1,2
1 School of Public Health, Xiamen University, Xiamen 361100, Fujian, China 2 National Institute of Diagnostics and Vaccine Development in Infectious Diseases, Xiamen University, Xiamen 361100, Fujian, China
Multiple PCR (Multiplex polymerase chain reaction, MPCR) is a technology to simultaneously amplify multiple targets through a single reaction and to detect the amplification products by reliable detection means so as to realize the diagnosis of multiple targets. MPCR has been well studied for its high efficiency, high throughput and low cost. At present, MPCR has been widely used in scientific research, disease diagnosis and other fields. Here, we summarize the development and application of MPCR from amplification and detection, and discuss the advantages and existing problems of MPCR. We propose that separate the reaction mixture into droplets or combined MPCR with the capillary convective PCR is expect to further improve the amplification efficiency of the surface of the solid phase carrier, so as to provide reference for the development of multiple PCR with high amplification efficiency, good consistency, good stability and multiplex detection.
multiple PCR, melting curve, microfluidic, membrane chromatography, Luminex liquid chip
钟泽澄, 王进, 张师音. 多重PCR技术研究进展. 生物工程学报, 2020, 36(2): 171–179.
Zhong ZC, Wang J, Zhang SY. Advances in multiple PCR technology studies. Chin J Biotech, 2020, 36(2): 171–179.
May 20, 2019;
July 18, 2019
Supported by:National Natural Science Foundation of China (Nos. 81501835, 81871505), Science and Technology Development Project of Fujian Province (No. 2017J05136).
Shiyin Zhang. Tel: +86-592-2183111; E-mail: zhangshiyin@xmu.edu.cn
10.13345/j.cjb.190206
国家自然科学基金 (Nos. 81501835, 81871505),福建省自然科学基金 (No. 2017J05136) 资助。
2019-07-30
http://kns.cnki.net/kcms/detail/11.1998.Q.20190729.1224.002.html
(本文责编 郝丽芳)
