分子生物学技术在诺如病毒检测中的应用*
2020-03-02徐泽炎吴莺
徐泽炎,吴莺,2
(1.江苏大学医学院,江苏镇江212000;2. 江苏大学附属医院消化科,江苏镇江 212001)
诺如病毒(norovirus, NOV)是引起人类非细菌性胃肠炎的主要病原, 全球约有50%以上的病毒性胃肠炎是由NOV引起[1]。该病毒引起的急性肠胃炎的发病率在我国呈上升趋势。该病毒感染剂量可低至10~100个病毒粒子,具有很强的外界抵抗力,传播速度快,极易造成大规模群体感染事件,尤其易在学校、幼儿园、养老院等的抵抗力弱的人群聚集地引起暴发[2]。根据NOV的传播途径,粪便、食品和水可作为检测该病毒的主要样本。为了适应NOV疫情暴发的处置,检测技术必须快速和灵敏。传统检测NOV的方法有电镜法[3]和免疫学方法[4],前者设备昂贵,操作要求高,敏感性低;后者缺乏可用于捕获高变异NOV保守性片断的特异性抗体。近年来,随着分子生物学检测技术的快速发展,测定与分析NOV全基因组及部分序列已被广泛应用于临床快速诊断和流行病学研究[5-6]。
1 基于核酸扩增法的NOV检测技术
核酸扩增法是目前检测该病毒的主要手段,主要包括实时荧光定量RT-PCR (real-time quantitative RT-PCR,qRT-PCR)、逆转录PCR(reverse transcription PCR, RT-PCR)和逆转录-环介导等温扩增法(reverse transcription-loop-mediated isothermal amplification, RT-LAMP)。由于NOV为单股正链RNA病毒[7],须提取NOV的RNA,逆转录成cDNA, 再进行PCR。
1.1RT-PCR RT-PCR先将RNA逆转录成cDNA,再用PCR扩增和琼脂糖凝胶电泳进行判别。Jiang等[8]采用RT-PCR首次在粪便中发现NOV。Kojima等[9]针对NOV衣壳蛋白保守区的基因序列设计的引物GI-SKF/GI-SKR和GⅡ-SKF/GⅡ-SKR是检测NOV的理想引物,其引物具有良好的稳定性与保守度。该方法比较灵敏,但由于模板和试剂等影响,对样品中的模板只能定性,而且因置于开放体系中操作,污染风险高。
1.2qRT-PCR 其原理为非特异性荧光染料偶联到目标特异性探针上,通过连续监测荧光信号的变化,并与加入已知量的标准品比较,可以实时检测核酸生成和目的基因的初始量[10]。Kageyama等[11]选择NOV最保守的基因区ORF1-ORF2连接区N/S结构域,设计引物COG1F/COG1R和COG2F/COG2R,可用于qRT-PCR检测NOV不同基因型。qRT-PCR的敏感性和特异性要高于常规RT-PCR,能定量快速分析NOV,这对于致病剂量和风险评估有着重要意义[12],但qRT-PCR无法同时检测多种病毒。
1.3RT-LAMP LAMP全程恒温,其反应液中含有大量的甜菜碱,在60~65 ℃时,DNA双链处于动态平衡之中,有利于引物与靶序列的结合,在DNA聚合酶的作用下,通过自我循环合成大量目的DNA,并产生白色沉淀,结果的判读采用直观观察浑浊度或浊度阅读器[13]。张昭华等[14]建立RT-LAMP检测病毒性腹泻样本的Ⅱ型诺如病毒方法:利用一对内引物、一对外引物和一条加速引物在62 ℃、70 min内充分扩增模板RNA。该法和qRT-PCR法比较,两种方法敏感性一致,但前者检测速度快、只需简单的加热器或水浴,观察结果简单,更适合基层使用。
2 高通量测序技术检测NOV
高通量测序(high-throughput sequencing technology)也称为第二代基因测序技术(next generation sequencing, NGS),具有通量高、速度快、成本低等特点,广泛应用于生物包括病毒的基因组学、转录组学等领域。由于NOV基因组全长只有7 500 bp,高通量测序可从临床样本中鉴定NOV[15],并可追踪该病毒的基因进化,发现新突变株,有助于了解病毒的结构、中和表位、抗原变异和宿主受体的关系[16]。
3 基因芯片技术检测NOV
基因芯片技术,是将寡核苷酸等DNA探针按一定顺序固定在载体上,形成分子点阵,当样品中的核酸与探针发生杂交后,检测和分析杂交信号[17]。根据杂交体系分为固相基因芯片技术和液相基因芯片技术。该技术可同时检测样品中的多个基因,或成百上千种样本中相同的基因,因此,该技术具有高通量的优势,并且可以自动化和微型化。
3.1液相基因芯片技术 也称为悬浮芯片(suspension array)技术或液态芯片(liquid chip)技术,是美国Luminex公司开发一种具有多指标同步分析(unknown x multi-analyte profiling,xMAP)功能的芯片技术[18-19]。Luminex公司利用微球为载体,微球内含有3色荧光,每种微球可以偶联特异核酸探针,每个微球能结合约1×106个探针分子,杂交时荧光信号强,再通过流式细胞仪检测,结合激光检测装置识别和测量单个微球荧光强度,所得到的数据经数字信号处理器处理,即可判断结果。何雅青等[20]将多重RT-PCR结合液相基因芯片技术检测粪便样本中A型轮状病毒和NOV,将PCR产物与偶联这些病毒核酸探针的微球混合物进行杂交, 利用Luminex 200液相芯片检测仪,可以快速、准确地检测到这些病毒。Chung等[21]基于液相基因芯片和静电吸附技术,快速、敏感地检测到牡蛎中的鼠诺如病毒(murine norovirus, MNV)。
3.2基于磁珠的可视化基因芯片技术 荧光素标记芯片技术需要昂贵的荧光扫描仪判断结果,这限制了大规模地推广应用芯片技术。基于磁珠的可视化基因芯片(magnetic bead-based visual gene chip)技术主要利用生物素与链霉亲合素特异性结合的特点,采用生物素标记样品核酸,链霉亲合素包被磁珠或纳米金等标记物,通过链霉亲合素与生物素的亲和作用,产生不同颜色的斑点,并且肉眼可以识别,因此,检测结果可以可视化[22]。史蕾等[23]以生物素标记GⅠ型和GⅡ型NOV的引物扩增临床粪便样本,带有生物素标记的PCR产物与芯片上GⅠ型和GⅡ型NOV的互补探针结合形成稳定的双链,再杂交链霉亲合素包被的磁珠后,以肉眼或通过普通显微镜观察。田甜等[24]建立RT-PCR-反向斑点杂交检测NOV的方法,也属于可视化基因芯片技术,生物素标记引物的NOV扩增产物经热变性后,与固定在硝酸纤维素膜上的NOV探针进行杂交反应,经显色后判定结果。
4 生物传感器技术
生物传感器由生物感应元件和信号处理元件组成,将生物感应元件(如核酸、受体、病毒、肽等)固定在换能器上,待测物与感应元件作用后,产生响应信号,该信号被处理元件接受、加工、转换、输出和定量分析[25]。根据响应信号分为光学信号、化学信号和压电信号等,生物传感器技术也可分为光学生物传感器、电化学生物传感器和压电晶体传感器等。
4.1F0F1-ATP合酶(ATPase)生物传感器 F0F1-ATPase是一个生物分子马达,可以转化生物体内的能量。由F0(ab2cn)和F1(α3β3γδε)组成,F0、F1分别位于细菌囊泡的载色体膜内和突出膜外(图1)。F0利用质子跨膜的能量形成旋转矩力;而F1用ATP水解的动力来产生相反的机械矩力。有研究表明,若不同的负荷连接在F0F1-ATPase上,可改变F1的旋转速度[26-27]。张捷等[28]根据NOV的ORF1和ORF2 连接处的高度保守区设计探针,利用ε亚基抗体-生物素-链霉亲和素-生物素-NOV探针,将该病毒探针连接在F0F1-ATPase的ε亚基上构建生物传感器(图1)。当探针识别NOV时,F0F1-ATPase活性发生改变,合成不同的ATP,检测ATP试剂luciferase/luciferin 判断结果。该方法所需仪器简单,无需提取核酸,具有快速、灵敏、经济等优点的特点。
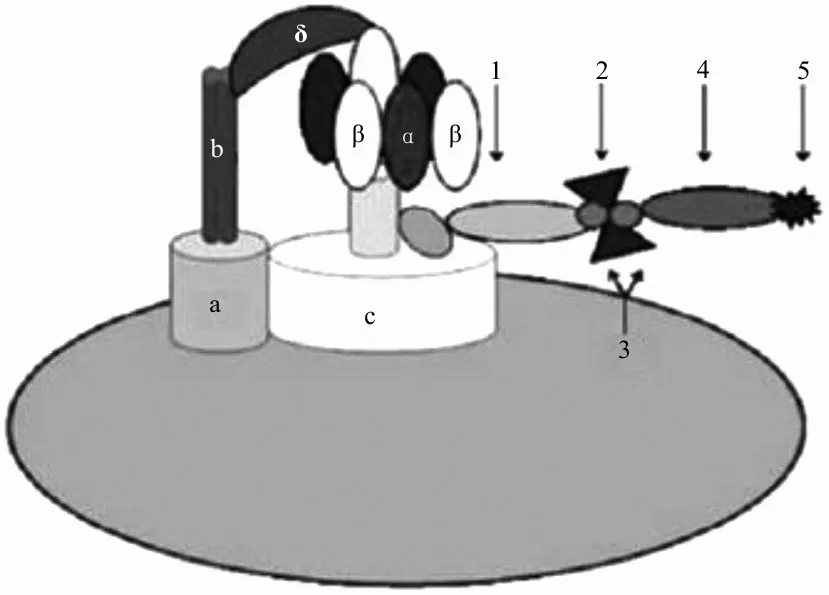
注:1,ε亚基单克隆抗体; 2,链霉亲合素;3,生物素;4,NOV探针;5,NOV病毒。
图1检测NOV的F0F1-ATPase生物传感器的模式[28]
4.2基于适体(aptamer)-表面等离子共振传感器技术 表面等离子共振(surface plasmon resonance,SPR)传感器是一种化学生物传感器,其原理是利用光线穿透金属导电层,引发其表面的自由电子出现等离子体化。其实时监测生物分子相互反应的动力学过程,包括键的结合或解离。适体是利用指数富集的配基系统进化技术 (systematic evolution of ligands by ex-ponential enrichment,SELEX), 从人工合成的文库中随机筛选出短链寡核苷酸(DNA或RNA)。适体通过氢键、疏水作用力、范德华力等作用力,高亲和力及高特异性结合靶标分子。首先将适体修饰至SPR基底上,然后适体特异性地识别目标物,导致共振条件发生变化,并改变SPR的输出信号,从而对目标物进行分析检测[29]。Giamberardino等[30]首先通过SELEX筛选与MNV特异性结合并带有荧光的单链寡核苷酸DNA,作为适体(AG3),再将5′末端标记有巯基的AG3在金硫键成键驱动作用力下,有序吸附在SPR炭电极的纳米金膜(gold nanoparticles-modified screen-printed carbon electrode,GNPs-SPCE)表面,MNV的标本流经SPR时,MNV和AG3特异性地结合,导致SPR的输出信号改变(图2)。该方法敏感,特异性高,对待测样品的纯度要求低,不需要对样品进行标记,适体具有分子量小、结构简单、易进行修饰、靶标范围广泛、可反复使用和长期保存的优点。
4.3基于纳米粒子结合石英晶体微天平传感器技术 石英晶体微天平(quartz crystal microbalance,QCM)传感器,又称电晶体传感器,其原理是利用石英晶体芯片作为换能器,将芯片表面吸附物质的质量转换为频率信号。QCM作为一种质量敏感型压电传感器,相对分子质量较小,通过密度大的金纳米颗粒(AuNPs)修饰肽,放大信号[31]。Hwang等[32]利用噬菌体展示技术筛选出来和NOV衣壳蛋白(rP2)高亲和力结合的肽,与巯基(thiol)修饰的金纳米颗粒形成的自组装单层(self-assembly monolayer, SAM)结合,改善电子的传递效率,提高QCM传感器的检测效率(图3),具有检测时间短和检测限低的优势。

注:1,吸附在SPR炭电极的纳米金膜(GNPs-SPCE); 2,有巯基的适体(AG3)单链寡核苷酸; 3,鼠诺如病毒(MNV)。
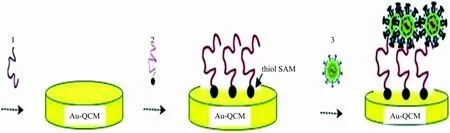
注:1,利用噬菌体展示技术筛选出来肽;2,与thiol修饰金纳米的SMA结合的肽;3,吸附NOV。
5 展望
由于NOV具有感染性强、发病急、传播快等特点,易造成大规模群体感染,因此,能够快速和准确地鉴定NOV,显得非常重要。目前核酸扩增法、高通量测序、基因芯片以及生物传感器等分子生物学技术检测NOV已逐渐成熟,可有效缩短检验NOV的时间,提高检验精度和敏感性。这些技术在NOV实际检测中具有不同的适用范围和优缺点。qRT-PCR是目前检测NOV的主要手段,由于不可预测NOV单核苷酸的多态性,基于保守区设计的引物和探针无法确保检测新突变株的敏感性。RT-LAMP检测速度快,设备简单,适合基层使用,但还不成熟稳定。高通量测序分析NOV全部基因信息,追踪NOV的基因进化和发现新突变株,但费用相对昂贵。基因芯片检测法是一种高通量、快速的、自动化检测技术,用于出入境等需要时快速筛查NOV感染,但该病毒变异较大,收集与设计探针是一个难题。随着多学科的不断融合渗透,生物传感器已将生物学、生物电子学和微电子学等学科结合起来,依据电化学、光效应等来设计探测装置,以提高精确度。生物传感元件的稳定性还是一个难题,此外,如何选择活性强、敏感性高的生物传感元件也是需要考虑的问题。
