基于RF_AdaBoost模型的血液种属鉴别算法
2020-01-16魏曼曼路皓翔杨辉华
魏曼曼, 路皓翔, 杨辉华,3
(1. 桂林电子科技大学计算机与信息安全学院, 2. 电子工程与自动化学院, 桂林 541004;3. 北京邮电大学自动化学院, 北京 100876)
拉曼光谱分析技术属于光谱分析技术中的一种, 具有丰富的分子结构信息, 且对检测样本无污染、 检测速度快[1], 因此在高聚物、 珠宝鉴别、 药物鉴别、 食品检测以及石油化工等领域中的应用极为广泛[2,3]. 近年来, 血液种属鉴别在珍稀动物保护、 海关以及刑侦等方面起到重要作用[4], 然而, 当前尚无快速、 准确、 可靠性强的血液种属鉴别方法, 导致在实际应用中极为不便.
拉曼光谱反映了物质内部分子的极化度[5], 不同物质的拉曼光谱信息不同, 如水分的拉曼光谱比较弱而血液中其它成分的拉曼光谱则较强, 基于拉曼光谱的这一特点, 研究人员越来越重视拉曼光谱分析技术在血液种属鉴别方面的应用研究. 如Kelly等[6]将统计学分析方法如慢特征分析、 主成分分析和交叉验证法应用于人、 犬和猫3个物种的分类研究, 通过对采集到的拉曼光谱进行分析并提取主成分, 实现了这3个物种的有效分离. McLaughlin等[7]将二元偏最小二乘判别模型应用于人、 猫和狗等12个种属血液样本的鉴别. Mistek等[8]采集了人、 猫和犬血迹的傅里叶变换红外光谱样本数据, 将其用于建立偏最小二乘判别模型并实现3个物种的分类. Fujihara等[9]建立主成分分析模型, 将便携式拉曼光谱仪采集的人类和非人类血液样本的拉曼光谱进行有效区分, 该模型在血液于室温下保存90 d的情况下仍然适用. 陈秀丽等[10]利用激光镊子拉曼光谱技术采集正常细胞和地贫红细胞的光谱数据, 并结合主成分分析和反向传播算法实现了细胞鉴别. 潘建基等[11]以血清的显微共聚焦拉曼光谱数据为研究对象, 结合主成分分析和判别分析法实现了鼻咽癌的早期诊断, 但对鼻咽癌组血清的检测灵敏度仅为89.7%. 白鹏利等[12]获取了人与动物血液拉曼光谱数据, 利用杠杆值和残差值剔除异常数据, 并结合主成分分析法进行检测研究. 郑祥权等[13]以小波去噪和基线校正对人血和比格犬血的拉曼光谱进行预处理, 并结合主成分分析法构建了线性判别模型. 文献[6~9]均采用干燥后的血液样品进行实验, 无法适应海关进出口血液鉴定等场景的需求; 文献[10,11]分别采用俘获的单个红细胞和晾干后的血清样品进行血液分析, 会对样本造成破坏; 文献[12,13]虽然在样本制备时采用了无损的方式, 且对人类和非人类血液种属识别准确度分别达到95%和90%, 但检测性能仍有待提高.
近年来, 随着人工智能的兴起, 机器学习技术在石油化工、 药品鉴别等行业备受关注[14,15], 作为机器学习的一个分支, 集成学习更是凭借分类准确度高、 可靠性强等优点逐渐应用到众多领域并占据越来越重要的地位[16,17], 其中随机森林(RF)及Adaptive Boosting Algorithm(AdaBoost)算法是集成学习领域的杰出代表. 本文采用泛化性能强、 分类准确度高的RF算法作为AdaBoost的弱分类器, 通过弱分类器判别准确度确定权重从而组合为强分类器. 为了验证该方法的性能, 以人和动物血液的拉曼光谱数据为实例, 并与支持向量机(SVM)、 极限学习机(ELM)、 核极限学习机(KELM)、 堆栈自编码网络(SAE)、 反向传播网络(BP)、 主成分分析-线性判别法(PCA-LDA)、 偏最小二乘判别分析(PLS-DA)和RF算法进行对比, 从分类准确度、 模型运行时间和稳定性3个方面验证了该方法的有效性.
1 RF_AdaBoost模型
RF算法由Leo Breiman等[18]于2001年提出, 具有稳定性强、 分类准确度高的优点, 其训练集在Bootstrap重采样的过程下随机产生, 且内部决策树选取属性时也是随机的. Freund等[19]对Boosting算法进行改良, 使算法能够自适应调整样本权重和弱分类器级联权重, 形成性能优良的AdaBoost算法. 该算法采用加权样本训练下一个弱分类器, 同时根据每次迭代中的预测误差调整弱分类器权重, 从而将弱分类器按照权重整合成符合实际需求的强分类器. 本文结合RF较强的分类预测准确度及AdaBoost算法自适应调整数据分布的优点, 将RF作为AdaBoost算法的弱分类器, 旨在提高算法分类准确率及抗噪声能力, 模型具体框架如图1所示.

Fig.1 Framework of RF_AdaBoost model
该模型进行血液种属鉴别的过程主要分为2个阶段: RF弱分类器训练阶段和强分类器样本类别决策阶段. 模型对样本进行分类预测的详细流程如下:
初始化. 对RF_AdaBoost模型中RF弱分类器的数目及单个RF中决策树的数目进行初始化, 并初始化样本权重u1,i:

(1)
式中:m为训练集包含的样本数目.
弱分类器训练. 根据RF投票决策策略, 第t个分类器的分类结果Gt(x)为

(2)

预测误差计算. 为使预测结果更准确, 应根据RF弱分类器的预测误差率对样本权重进行调整, 使上一轮误分类样本在下一轮迭代中所占比重更大, 因此, 计算第t个RF弱分类器对于序列Gt(x)的预测误差率et:

(3)
式中:ut,i表示第t个RF弱分类器的样本权重;I为单位向量;y为期望分类结果. 预测误差率越小表示弱分类器对样本的预测结果越准确.
弱分类器组合. 根据在模型中作用越大的弱分类器预测误差应越小的原则, 计算第t个RF弱分类器的权重λt:

(4)
式中:et为第t个RF弱分类器的预测误差率.
样本权重调整. 为使下一轮迭代中RF弱分类器能够正确区分误分类样本, 应对样本权重进行更新, 放大误分类样本权重并缩小正确分类样本权重. 假设前t-1轮迭代所产生的分类器ft-1(x)已知, 即
ft-1(x)=ft-2(x)+λt-1Gt-1(x)=λ1G1(x)+…+λt-1Gt-1(x)
(5)
则模型的损失函数在当前样本权重下应最小:
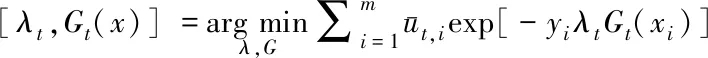
(6)

(7)
强分类函数集成. 训练T轮后得到T组弱分类函数Gt(x), 由Gt(x)组合得到强分类函数f(x):

(8)
式中:λt为第t轮训练时的弱分类器权重. 强分类函数f(x)经二值化后得到最终用于血液种属鉴别的强分类器h(x):

(9)
2 实验部分
2.1 材 料
实验用血液样本共计1033例, 其中535例人类血液样本由广西桂林市某医院提供, 498例动物血液样本由广西桂林市某研究中心提供.
2.2 样品的制备
所有血液样本不进行任何前处理, 均置于EDTA抗凝管中. 用移液枪移取少量抗凝管中的血液样本, 滴至清洗并干燥后的镀铝载玻片上, 进行拉曼光谱测量. 载玻片采用体积分数为75%的乙醇清洗, 以防止干扰拉曼信号并避免样品间的交叉污染.
2.3 光谱采集及预处理

Fig.2 Raman spectroscopy of blood
2.3.1 光谱采集 采用Finder Vista激光共聚焦显微拉曼光谱仪(北京卓立汉光仪器有限公司)对血液样本进行测量, 激发波长设置为785 nm, 积分时间为10 s. 将200~2000 cm-1的拉曼位移范围等间隔划分为1778个特征波长点, 测量每点对应的拉曼强度值, 进而得到其拉曼光谱曲线. 每例样本采集5条光谱, 取其平均光谱作为该样本的光谱曲线. 实验时将人类血液光谱标记为正类样本, 非人类血液光谱标记为负类样本, 样品的光谱信息如图2所示. 由图2可见, 血液拉曼光谱在417, 754, 1003, 1226, 1547和1620 cm-1处均有明显出峰. 血液样本中核酸和蛋白质组成成分的多样性以及含量差异导致不同拉曼位移处的光谱相对强度不同, 位于754 cm-1处谱峰的形成是由于核酸结构的差异性所致, 而位于417, 1003, 1226, 1547和1620 cm-1处的拉曼谱峰是由蛋白质中物质含量的多样性造成的. 这证明血液拉曼光谱中含有丰富的遗传信息, 并且对血液中分子组成、 结构、 含量等信息分析具有重要的参考价值, 可用于提高血液种属判别的准确度.
采用如图2所示的血液拉曼光谱数据集, 对RF_AdaBoost模型从分类准确度、 模型稳定性及算法运行时间3个方面进行性能评估. 在实验所用数据集中, 正、 负类样本数目之比约为1∶1, 为使数据分布保持一致, 避免引入额外的偏差影响实验结果, 采用1∶1的比例随机选取正、 负类样本构造训练集. 如训练集所含样本数目为100时, 应随机选取正样本、 负样本各50例, 其中正样本占比为50/535≈0.093, 负样本占比为50/498≈0.100, 因此随机选取正样本总数的9.3%以及负样本总数的10%构成训练集, 剩余样本构成测试集. 具体划分情况如表1所示, 按此方式构建出9种不同规模的训练集进行实验, 以验证RF_AdaBoost模型在不同规模训练集下的性能.

Table 1 Distribution of positive and negative samples in the training sets

Fig.3 Raman spectroscopy of blood after pretreatment
2.3.2 光谱预处理 在样品拉曼光谱获取过程中, 由于外界环境变化、 激光功率波动等原因, 导致光谱数据中包含大量无关信息和噪声. 为减少无关信息对鉴别模型的影响, 提高模型的预测能力, 需要对实验所用相关数据进行预处理. 本实验采用Savitzky-Golay(S-G)平滑法和求导数2种方法对实验样品的拉曼光谱数据进行预处理. 首先, 采用S-G 5点平滑法消去样品光谱数据中噪声对分析模型建立的影响, 较好地保留光谱的原始信息; 然后, 对平滑处理后的样品光谱数据求一阶导数, 移除背景成分使拉曼特征峰更加显著. 预处理之后的样品拉曼光谱如图3所示.
2.4 相关参数设置
2.4.1 决策树的数目 RF_AdaBoost血液种属鉴别模型采用RF作为弱分类器, 而RF本身就是多棵决策树集成在一起的, 决策树的数目会对RF的性能产生一定影响. 为保证模型整体性能达到最优, 首先需要确定单个弱分类器中决策树的数目. 按照表1所示方式随机选取训练集样本, 在不同样本规模下验证了决策树的数目与血液种属鉴别准确度的关系, 具体关系如图4所示. 可见, 当训练集规模不同时, 使RF分类准确度达到峰值的决策树数目不尽相同, 但都分布在100~200之间. 根据实际情况为不同规模的训练集选择不同的决策树数目, 当训练集规模为200, 300, 600, 700和800时, 单个RF弱分类器的决策树数目设为100; 当训练集数目为500和900时, 决策树数目设为150; 当训练集规模为100和400时, 决策树数目设为200.
Fig.4 Relationship between the number of decision trees and the classification accuracy of RF

Fig.5 Relationship between the number of weak classifiers and the classification accuracy of model
2.4.2 弱分类器的数目 RF_AdaBoost模型对人类和非人类血液种属鉴别过程中, 弱分类器数目对模型预测能力及泛化性能等也会产生一定影响, 过多的弱分类器数目会增加模型的时间复杂度, 甚至会降低模型的预测准确度, 故RF_AdaBoost模型在进行血液种属鉴别时需要选取合适的弱分类器数目. 按照表1所示数据集的划分情况进行鉴别实验, 结果如图5所示. 可见, 弱分类器数目在8~18之间时模型分类准确度达到最高, 在不同训练集规模下使分类准确度达到峰值的弱分类器数目不同, 综合考虑模型的运行时间, 建立RF_AdaBoost血液种属鉴别模型时对各个样本规模下的弱分类器数目分层次设置. 当训练集数目为100时, 模型中弱分类器的数目设为18; 当训练集数目为200~400时, 弱分类器数目设为12; 当训练集数目为500~900时, 弱分类器数目设为8.
2.5 鉴别模型的建立
实验中RF算法选用Randomforest-matlab工具箱(https://code.google.com/p/randomforst-matlab/), 采用MATLAB R2014a为编程软件, 运行在Intel(R) Core(TM) i5-6500 CPU@3.20GHz 3.19 GHz环境下. 实验所用RF_AdaBoost模型主要分为实验数据预处理、 弱分类训练及级联、 强分类器预测输出3个阶段.
2.5.1 光谱数据预处理 为除去拉曼光谱数据中夹杂的噪声, 提高数据集的信噪比, 首先对实验数据进行预处理. 依次采用S-G 5点平滑法和一阶导数法处理样品的光谱数据, 使拉曼峰值更显著, 同时增强模型抗干扰能力并提高预测性能.
2.5.2 弱分类器训练 弱分类器初始化, 根据2.4节相关参数设置中的讨论结果对单个RF弱分类器中的决策树数目以及模型中的弱分类器个数进行设置, 样本初始权重1/m, 其中m为训练集样本个数, 并在迭代过程中不断自动调整样本权重和各弱分类器权重.
2.5.3 强分类器预测输出 将各训练集规模下的全部弱分类器根据权重线性整合为强分类器, 输入测试集得到RF_AdaBoost模型预测结果.
2.5.4 对比实验 采用RF, SVM, ELM, KELM, SAE, BP, PCA-LDA以及PLS-DA模型进行对比实验, 其中每种训练集规模下RF模型所含决策树的数目与RF_AdaBoost模型中单个弱分类器所含的决策树数目相同; SVM选用线性核函数, 参数c=1, gamma=0.3; ELM和KELM的网络结构均设置为1778-train*0.4-2(train为训练集样本个数), KELM选用RBF核函数; SAE和BP网络均设置为1778-400-200-2, 迭代次数为100, 学习率为0.01, 激活函数选用sigmoid.
3 结果与讨论
按照表1所示随机抽取血液拉曼光谱数据组成训练集, 其余样本组成测试集, 同时选取RF, ELM, KELM, SAE, BP, PCA-LDA以及PLS-DA进行对比实验. 每个比例的对比实验分别进行10次, 每次实验时均需按照表1中数据集的构成情况重新将数据随机分配为训练集和测试集, 取10次测试的平均值作为最终结果, 并根据分类准确度、 模型稳定性以及运行时间3个指标对模型的鉴别能力进行评估.
3.1 血液光谱特征分析

Fig.6 Raman representative spectroscopy of human and non-human blood
人类和非人类血液的代表性拉曼光谱如图6所示. 可见, 人类和非人类的血液拉曼光谱谱峰大致相同, 但谱峰的相对强度有明显差异. 在2条代表性拉曼光谱中, 拉曼位移在240~1220 cm-1范围内时, 人类血液光谱强度小于非人类血液; 而在1220~1670 cm-1范围内, 人类血液拉曼光谱强度明显超过非人类血液. 此外, 在330~450 cm-1和1220~1670 cm-1范围内, 人类血液拉曼谱峰更明显且更易于识别. 这表明人类和非人类遗传物质不同, 因此所包含的生物化学信息具有特异性, 不同核酸碱基和蛋白质中氨基酸的构成具有多样性, 表现在拉曼光谱中即为谱峰强度的差异, 这为血液种属鉴别提供了条件.
3.2 分类准确度
分类准确度是检验模型性能的关键指标. 采用表1所示的样本集合构建方式选取训练集和测试集, 得到各模型在10次实验下对于测试集的平均分类准确度如表2所示. 可见, 在每种规模的训练集下, RF_AdaBoost模型的分类准确度均保持在98%以上, 与其它模型相比有显著提高; 模型分类准确度随着样本规模的增大而提高, 当训练集包含600~900个训练样本时, 分类准确度达到100%, 表明该模型在此训练集规模下可以准确实现血液种属鉴别. 这是由于该模型较好地结合了RF的随机性和AdaBoost的自适应性, 抗噪声能力得到有效增强, 使得非线性建模能力更优.

Table 2 Test sets classification accuracy of each model under different training set scales
随着训练集中样本数量的增加, RF, SVM, ELM, KELM和PLS-DA的分类准确度均呈上升趋势, SVM的准确度仅次于RF_AdaBoost模型, 表明其解决了高维空间的分类问题, 可用于鉴别血液种属, 但与RF_AdaBoost模型相比鉴别能力较弱. RF的分类准确度次之, 这是因为重采样技术和决策树集成方式中的随机特性提高了模型性能. PLS-DA的分类准确度较高, 表明观测变量和预测变量投影到新空间后, 建立的模型能够较好地预测血液种属, 模型受训练集规模的影响较小, 准确度约为94%. 此外, 在血液种属鉴别实验中KELM和ELM也保持了较高的分类准确度, 但KELM稍差于ELM, 表明核函数的加入并未起到改善模型性能的作用. PCA-LDA首先提取出了6个主成分, 进而通过线性判别法对数据进行分类, 但得到的预测结果较差, 表明此时的PCA未能提高建模质量, 反而损失了部分数据信息. SAE和BP在不同规模训练集下得到的分类准确度均较低, 表明其非线性建模能力弱, 预测效果较差, 在血液种属鉴别场景下这2种模型预测结果的可靠性较差.
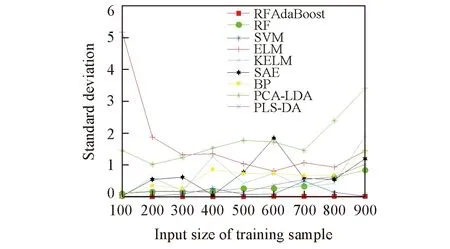
Fig.7 Standard deviation of prediction accuracy of each model
3.3 模型的稳定性
模型的稳定性决定其实际应用的可靠性与泛化性能. 对于建立的RF_AdaBoost血液种属鉴别模型, 采用10次重复实验的预测标准偏差作为模型稳定性的衡量标准, 各模型在不同规模训练集下的预测标准偏差如图7所示. 可见, RF_AdaBoost模型稳定性最优, 在任意规模训练集下均表现出最低的预测标准偏差, 表明与其它模型相比RF_AdaBoost模型用于血液种属鉴别时效果是最稳定. RF, SVM和PLS-DA的稳定性优于ELM, KELM, SAE以及BP模型, 表明这3种模型鲁棒性较好, 但与RF_AdaBoost相比效果较差. SVM的表现仅次于RF_AdaBoost模型, 而PCA-LDA, SAE, ELM, KELM及BP模型的稳定性最差.
3.4 模型运行时间
模型运行时间可用于衡量模型的预测效率, 实验所得结果如表3所示. 可见, RF_AdaBoost模型运行时间较长, 这是由于除了受到训练集样本数目的影响外, 其构建时集成了多个RF弱分类器, 且受不同规模的训练集下单个RF弱分类器中决策树棵数的影响较大.

Table 3 Training time of each model under different training set scales
随着训练集样本的增加, SVM, ELM, KELM, SAE, BP以及PLS-DA模型的单次运行时间均逐渐延长. SVM和以决策树为基础的RF模型运行速度较快, 因为其包含的可调参数较少且模型简洁. 而ELM和KELM运行速度较快, 是因为其模型结构由输入层、 隐含层和输出层组成, 分别采用随机设定和解方程组即可确定前两者的连接权值以及隐含层的阈值、 后两者的连接权值等重要参数, 解决方式简便易行, 无需大量时间即可完成运算. PLS-DA模型向新空间投影后用于解释样本的观测数目少, 因此能较为快速的得出实验结果. 同样的, PCA-LDA对数据降维后再进行分类, 降低了算法的计算开销, 因此运行时间较短. SAE和BP的运行时间与其它模型相比较长, 因为这两种模型在实验中均为包含两层隐含层的神经网络结构, 且需要对各节点参数层层传播及训练.
4 结 论
以RF作为AdaBoost的弱分类器, 提出了一种同时具备RF的随机性和AdaBoost的自适应性的血液种属鉴别方法, 旨在提高模型预测能力及抗噪声性能. 为扩充实验内容, 在一定程度上反映所建立模型的科学性和普适性, 采用构造不同规模训练集进行实验的形式对模型性能进行评估. 实验结果表明, 在训练集中样本数目为600~900时, 准确度达到100%, 且不论训练集规模如何, 预测标准偏差一直保持趋近于0. RF_AdaBoost模型具有分类准确度高、 稳定性好的优点, 可用于人类和非人类的血液种属鉴别.
