小麦主要过敏原CM16线性B细胞表位的预测及初步鉴定
2020-01-07李雪娇李欣芮范卓妍张亚妮车会莲
李雪娇,李欣芮,范卓妍,张亚妮,车会莲*
(北京食品营养与人类健康高精尖创新中心,中国农业大学食品科学与营养工程学院,北京 100083)
小麦蛋白在小麦籽粒中占10%~15%,是小麦籽粒中的重要营养物质,各蛋白质组分按其在不同溶剂中的溶解度不同分为4 类:清蛋白、球蛋白、醇溶蛋白、麦谷蛋白[1-2]。小麦致敏机制比较复杂,不同种类的小麦蛋白都有可能引起过敏反应,有不同的致敏性及发病机制,并呈现不同的临床症状[3]。流行病学的调查结果显示,近年来过敏性疾病的发病率逐年升高,世界0.4%人口受小麦过敏症困扰[4]。北京协和医院发布的中国过敏性休克诱因研究表明,超七成致敏源来自食物,其中以小麦居多[5]。小麦过敏会影响内脏、呼吸道与皮肤的健康,引起运动激发过敏症、乳糜泻、哮喘、鼻炎、接触性荨麻疹等[6]。
有研究表明,CM16是引起日本面包师哮喘的主要过敏原[7],并且可以引发刚摄入辅食的婴幼儿发生口服性的过敏反应[8]。CM16分子质量17 kDa,由143 个氨基酸组成,属于醇溶蛋白超家族中的大型谷物胰蛋白酶或α-淀粉酶抑制剂家族[9]。该家族蛋白质广泛存在于谷类、豆类以及其他较高等植物中,对胰淀粉酶具有较强的抑制作用[10]。
B细胞表位是抗原中可被B细胞抗原受体或抗体特异性识别并结合的线性片段或空间构象性结构,其刺激机体产生B细胞介导的体液免疫应答,并产生效应分子(抗体)和效应细胞[11]。在免疫学中,表位才是抗原刺激机体免疫系统产生特异性免疫应答的真正部位[12]。B细胞表位预测是表位预测的一个重要组成部分,大多数研究是针对线性B细胞表位预测,通过组合抗原蛋白物理化学性质、结构性质、统计显著性度量等特征属性进行表位预测[13-14]。
通过生物信息学软件分析抗原表位是目前常用的表位研究方法之一。其原理是对已知的线性B细胞表位特点进行归纳后,发现蛋白质序列中亲水性和表面可及性高的区域更容易分布在分子表面,而可塑性高的区域容易发生折叠和扭曲,易与抗体接触,可能产生抗原表位[15-17]。但该方法主要基于蛋白质的一级氨基酸序列,忽略了氨基酸之间的分子作用力,具有一定的局限性,需要结合抗消化实验以及质谱分析加以验证。
因此,本研究拟利用生物信息学法预测CM16线性B细胞表位,与质谱得到的抗消化肽段信息进行比对,以期证明抗消化性结合生物信息学分析鉴定过敏原线性B细胞表位的方法的可行性和应用性。
1 材料与方法
1.1 材料与试剂
实验用小麦粉为中粮香雪美味富强粉。
BCA蛋白定量试剂盒 上海碧云天生物技术有限公司;标准蛋白分子质量Marker 美国Thermo Fisher公司;牛血清白蛋白、胃蛋白酶(P7000)、胰酶(P3292) 美国Sigma公司;0.22 μm硝酸纤维素膜美国Whatman公司。
1.2 仪器与设备
JB-3磁力搅拌器 上海富磁新径仪器有限公司;TGL-16M台式高速冷冻离心机 广州广一科学仪器有限公司;多功能酶标仪 美国Thermo Scientific公司;DYY-7C型电泳仪 北京市六一仪器厂;GenoSens 1850凝胶成像分析系统 上海勤翔科学仪器有限公司;CC-K6加热制冷型恒温水浴锅 德国Huber公司。
1.3 方法
1.3.1 小麦蛋白粗提物的制备
参考冼静雯等[18]的方法提取小麦蛋白粗提物。称取约100 g小麦粉,1∶5(g/mL)加入预冷丙酮于4 ℃去脂,至上清液澄清,通风橱下干燥,称质量,以1∶10(g/mL)的比例加入0.01 mol/L磷酸盐缓冲液,提取12~24 h,10 000×g离心10 min,上清液即为蛋白粗提物溶液。
1.3.2 生物信息学方法预测小麦过敏原CM16线性B细胞表位序列
从Uniprot数据库获取小麦过敏原α-淀粉酶/胰蛋白酶抑制剂CM16的氨基酸序列。使用生物信息学软件DNAStar Protean中Hoop-woods的氨基酸亲水性分析方案、Emini-Surface Probability的表面可及性算法、Kparlus-Schuzl的可塑性分析和Jameson-Wolf的抗原指数分析方案,对蛋白质的一级序列性质进行分析,综合分析这些参数预测结果的重叠部分后,得出过敏原的线性B细胞表位序列[19]。
1.3.3 生物信息学方法分析小麦过敏原CM16二级结构
使用DNAStar Protean载入NCBI中下载的FASTA格式的α-淀粉酶/胰蛋白酶抑制剂CM16氨基酸序列,使用Chou-Fasman和Garnier-Robson方案对蛋白质的二级结构进行预测分析。
1.3.4 基于生物信息学方法的小麦过敏原CM16同源建模
在PDB数据库中使用BLAST搜索与过敏原α-淀粉酶/胰蛋白酶抑制剂CM16同源性较高,且已由实验分析出高级结构数据的蛋白质序列。以相似度最高的蛋白质为模板,使用SWISS-MODEL在线网站进行相同结构区域的同源建模,根据分子动力学模拟和能量最小化原理优化得到最佳蛋白高级结构。使用Spdbv软件对模拟结构的拉氏构象图进行合理性分析,使用Pymol软件对预测线性B细胞表位及抗消化肽段区域进行空间定位。
1.3.5 小麦蛋白粗提物抗消化肽段的质谱分析
分别取经过胃蛋白酶或胰酶消化60 min后的小麦粗蛋白消化产物,进行高效液相色谱-质谱联用检测,鉴定氨基酸长度多于4 个的抗消化肽段,并确定多肽所属的蛋白质序列。
1.4 数据处理
数据统计与图表绘制工具主要为DNAStar软件、Spdbv软件version 4.1(http - //spdbv.vital-it.ch/)、Pymol软件version 2.2(http-//www.pymol.org/)等。
2 结果与分析
2.1 小麦粗蛋白提取物主要成分分析
小麦蛋白粗提物电泳结果显示,小麦总蛋白具有5 条明显的电泳条带,其中分子质量在17 kDa的蛋白质含量最高,经灰度值分析,约占总蛋白含量的40%,其分子质量对应小麦过敏原α-淀粉酶/胰蛋白酶抑制剂CM16。由图1可知,小麦蛋白粗提物主要是由17 kDa处的α-淀粉酶/胰蛋白酶抑制剂CM16和大于180 kDa的高分子质量谷蛋白低聚物组成,这两种蛋白质具有热稳定性,经37 ℃温育60 min后,含量未发生明显变化。在胃蛋白酶作用下,消化第30秒时,高分子质量谷蛋白低聚物迅速被降解,产生分子质量低于10 kDa的片段,而17 kDa处的α-淀粉酶/胰蛋白酶抑制剂CM16在第30秒时保持完整,第5分钟时才被完全降解。随着消化进行,低于10 kDa的抗消化片段直至消化结束时仍可被检测。结果表明,高分子质量谷蛋白低聚物极易被胃蛋白酶消化,α-淀粉酶/胰蛋白酶抑制剂CM16易被消化,但小麦过敏原在消化过程中会产生消化抗性片段,具有潜在致敏活性。因此后续选取α-淀粉酶/胰蛋白酶抑制剂CM16进行线性B细胞表位与抗消化肽段之间关系的研究。

图1 胃蛋白酶消化对小麦蛋白质粗提物的影响Fig. 1 Effect of pepsin digestion on WPE
2.2 小麦过敏原CM16可能线性B细胞表位的预测结果
一般认为,亲水性和表面可及性高的区域易分布在蛋白质分子的表面,且柔性区域易于发生折叠和扭曲,容易产生表位与抗体结合[20]。如图2所示,在氨基酸亲水性分析中,亲水性指数大于0表明亲水性好,CM16的亲水性区域分布较为均匀,其中亲水性较高的区域分别为AA 88~100和AA 137~143。在可塑性分析中,发现AA 26~32、AA 39~51、AA 54~62、AA 64~67、AA 75~78、AA 89~98、AA 103~108、AA 130~132和AA 138~140区域具有一定的柔性,容易发生折叠、弯曲,易与抗体结合。在抗原指数分析中,以抗原指数大于0为筛选条件,结果显示抗原指数较高的区域为AA 26~34、AA 42~63、AA 66~70、AA 71~84、AA 88~99、AA 100~111和AA 137~143,可能含有潜在的优势抗原表位。最后对于CM16的表面可及性进行了分析,表面可及性指数大于1为筛选条件,结果显示AA 47~50、AA 58~60、AA 62~65、AA 90~96和AA 138~143区域具有较好的表面可及性。综合以上各参数,将同时满足4 个参数筛选条件的表位预测为CM16可能的线性B细胞表位,如表1所示。
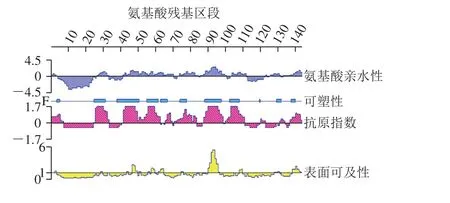
图2 DNAStar对CM16一级氨基酸序列的分析Fig. 2 DNAStar analysis of the amino acid sequence of CM16

表1 DNAStar预测得到的CM16的线性B细胞表位Table 1 Predicted linear B-cell epitopes of CM16 by DNAStar
2.3 过敏原CM16线性B细胞表位的二级结构预测结果
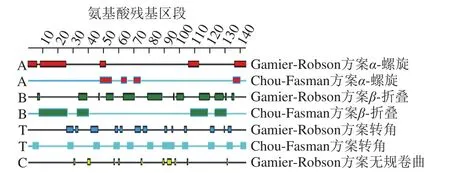
图3 CM16的二级结构预测Fig. 3 Prediction of CM16 secondary structure
图3 为使用DNAStar软件中的Chou-Fasman和Garnier-Robson方案对CM16二级结构进行预测的结果[21]。分析可知,两种方案在预测CM16的转角区域时具有一定的相似性,但对于α-螺旋和β-折叠的预测具有基本相反的结果,还需要结合三级结构进行分析。总体来看,CM16蛋白质二级结构中有序的螺旋、转角结构占据了主要优势,反映了蛋白质具有良好的紧密结构。
2.4 过敏原CM16预测线性B细胞表位的空间定位
使用PDB蛋白质结构数据库,对小麦α-淀粉酶/胰蛋白酶抑制剂CM16的氨基酸序列进行BLAST检索,共检索到16 种已有空间结构数据的蛋白质序列,其中PDB检索号为1bfa.1.A的重组玉米双功能蛋白因子/淀粉酶抑制剂与CM16的序列相似性可达41.88%,具有较高的序列覆盖率,模拟结构评分最高,因而被选为CM16建模的最佳模板。拉氏构象图是α-碳与酰胺平面的交角图,可以评价蛋白质结构中转角的易变程度。通过了解氨基酸残基的Φ和Ψ角信息,获得拉氏构象图的允许构象和不允许构象区域,如图4所示。在空间上允许和不允许出现的区域用不同颜色的等高线表示,黄色封闭区域为允许区,构象最为稳定,在该区域内的任何成对二面角(φ,ψ)所规定的构象都是立体化学所允许的[22]。蓝色封闭区域为临界区,该区域内任何成对二面角所规定的构象虽然是立体化学所允许的,但不够稳定,除此之外的区域为不允许区[23]。图中每个点代表一个氨基酸残基,红色点为组成α-螺旋的氨基酸,黄色为β-折叠。由图4可知,α-螺旋主要集中于第3象限的允许区域内,而β-折叠主要集中于第2象限的允许区域内。由此可证,CM16的二级结构主要由α-螺旋组成。只有6 个点位于不允许区域,8 个点位于蓝色的临界区域,其他多位于黄色的允许区域内,允许区域的氨基酸覆盖率达88%,此结果表明,构建的CM16的三维结构在一定程度上具有合理性和稳定性。
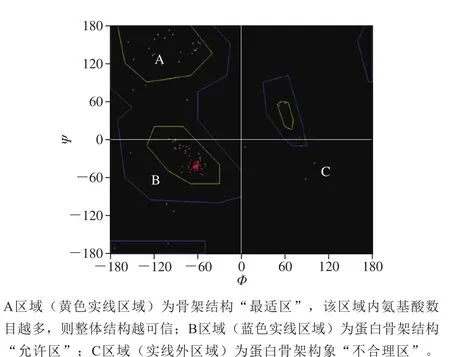
图4 拉氏构象图Fig. 4 Ramachandran plot of three-dimensional model of CM16
利用Pymol软件对生物信息学模拟的CM16线性B细胞表位在其空间结构上进行定位[24],将生物信息学模拟的4 条线性B细胞表位按不同颜色标注在三维结构图上(图5)。图5A为卡通拓扑图,彩色区域是不同抗消化肽段在其中的定位。CM16的空间结构是由4 个分子内的α-螺旋以及其他无序结构组成的,具有醇溶蛋白超家族典型的结构特性。由二硫键形成α-螺旋在维持蛋白质空间结构稳定性方面起到非常重要的作用,使CM16蛋白形成致密的球状分子,为其致敏性提供了结构基础。除标注为红色的表位位于α-螺旋上以外,其他的3 个表位均位于无规卷曲处,构象上具有可塑性,容易与抗体接触形成表位。图5B显示CM16表面分子暴露情况,4 个预测的线性表位均位于球状结构表面,具有与抗体结合的表面可及性,可以形成线性B细胞表位。综上可知,CM16的线性B细胞表位多位于无规卷曲处或是α-螺旋与无规卷曲的连接处,具有可塑性及表面可及性。
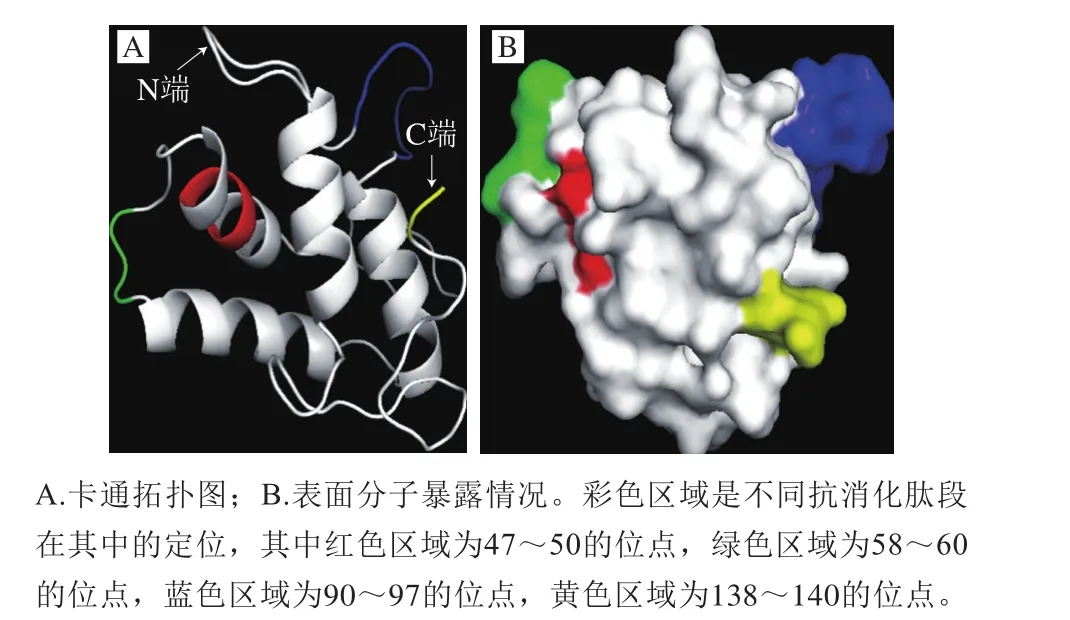
图5 预测线性B细胞表位在CM16三维结构中的定位Fig. 5 Location of the predicted linear B-cell epitopes in CM16 3D structure
2.5 过敏原CM16可能线性B细胞表位与抗消化肽段的比较
将小麦蛋白粗提物经胃蛋白酶消化60 min后的产物,通过高效液相色谱-质谱联用进行鉴定。经过60 min的胃蛋白酶消化,共产生6 753 个二级质谱图。根据Uniprot数据库提供的小麦过敏原氨基酸序列构建检索数据库,使用pFind软件搜索,共鉴定出23 条属于过敏原蛋白的片段,且大部分来源于α-淀粉酶/胰蛋白酶抑制剂亚型和高分子质量谷蛋白亚基,α-淀粉酶/胰蛋白酶抑制剂亚型CM16的主要过敏原肽段如表2所示。

表2 胰酶消化质谱与小麦主要过敏原匹配的肽段Table 2 Matching of trypsin-digested peptides with wheat major allergens
将预测表位与抗消化肽段在过敏原CM16的一级氨基酸序列上分布进行标注,如图6所示,预测表位和抗消化肽段主要分布于中部和C末端,而1、2号抗消化肽段与2、3号预测表位有部分序列重合,证明了预测表位的抗消化性,也体现了表位与抗消化肽段之间的联系。

图6 抗胰酶消化肽段及预测表位在CM16氨基酸序列的定位Fig. 6 Location of anti-trypsin-digested peptides and predicted epitopes in amino acid sequence of CM16
3 讨 论
本研究首先对小麦蛋白质粗提物进行电泳分析,发现分子质量在17 kDa的小麦过敏原α-淀粉酶/胰蛋白酶抑制剂CM16量最高,约占总蛋白含量的40%。进一步进行体外模拟胃肠道分析,结果显示小麦过敏原α-淀粉酶/胰蛋白酶抑制剂CM16在模拟消化过程中会产生消化抗性片段,提示其可能具有潜在的致敏活性。Posch等[20]使用双方电泳法分离出小麦过敏原,经过氨基酸序列检测,确定其主要过敏原为14~18、27 kDa和37 kDa处的蛋白,这与本研究结果一致。
为研究小麦中的醇溶蛋白超家族过敏原α-淀粉酶/胰蛋白酶抑制剂CM16的线性B细胞表位的结构特征,通过生物信息学方法对小麦过敏原CM16的线性B细胞表位进行探究。首先通过DNAStar对CM16一级氨基酸序列进行分析,通过筛选,最终确定了4 个CM16的线性B细胞表位。尽管近年来B细胞表位预测的方法得到了一定的发展和应用,但这些研究方法还存在一定的问题[25-26]。首先,所有预测表位的方法都缺乏评价标准,这使得各种预测方法的结果难以比较与评估[27]。其次,90%以上的表位为构象表位[28],然而大多数预测线性表位的方法都具有一定的局限性,它们仅仅是根据少数的几个表位的特征(氨基酸的性质、残基的表面可及性、空间分布、分子间接触)预测表位[29-30]。有研究结果表明[31],仅根据氨基酸的性质来预测线性表位的方法并不可靠,要提高预测的准确性,需与非表位特征结合起来预测。
因此,本研究进一步通过生物信息学预测CM16的二级结构,并通过同源建模确定CM16的三级结构。在进一步完善线性B细胞表位预测研究的基础上,深入研究了线性表位的构象分布。结果表明,CM16是由4 个α-螺旋以及无规卷曲组成的球状结构,预测的线性B细胞表位则位于无规卷曲处或是α-螺旋与无规卷曲的连接处,且暴露于球状结构的表面,具有表面可及性及结构可塑性。
为探究生物信息学方法预测线性B细胞表位的抗消化肽段的可行性,本研究利用生物信息学方法预测出CM16的4 条线性B细胞表位后,结合质谱得到的抗消化肽段信息进行比对分析,发现其中2 条肽段存在部分重合,含有线性B细胞表位的可能性更高,也证明了抗消化性结合生物信息学分析鉴定过敏原线性B细胞表位的方法的可行性和应用性。
4 结 论
小麦中的醇溶蛋白超家族过敏原α-淀粉酶/胰蛋白酶抑制剂CM16是由4 个α-螺旋以及无规卷曲组成的球状结构,其线性B细胞表位则位于无规卷曲处或α-螺旋与无规卷曲的连接处,且暴露于球状结构的表面,具有表面可及性及结构可塑性。探究小麦过敏原CM16线性B细胞表位有助于进一步认识小麦过敏原,对小麦过敏原的识别和检测具有重要的参考和借鉴作用。
