基于提高CRISPR/Cas基因编辑效率的研究进展
2019-12-14张玉苗李蓉鲁瑶林玉玲赖钟雄徐涵
张玉苗 李蓉 鲁瑶 林玉玲 赖钟雄 徐涵


摘 要 CRISPR基因编辑技术发展迅速,为功能基因的研究及遗传改良提供了技术支持。提高基因组编辑效率成为使用基因编辑技术的基本点,目前已取得了重要进展。本文结合CRISPR/Cas系统的作用原理、晶体结构,着重介绍目前提高编辑效率的最新研究进展,对CRISPR/Cas基因编辑效率的提高进行分析并提出改进策略。
关键词 CRISPR/Cas;基因组编辑;基因编辑效率中图分类号 Q943.2 文献标识码 A
Research Progress on Improving CRISPR/CasGenome Editing Efficiency
ZHANG Yumiao1, LI Rong2, LU Yao2, LIN Yuling2, LAI Zhongxiong2*, XUHAN Xu2,3*
1. College of Biological and Environmental Engineering / Shandong Key Laboratory of Eco-Environmental Science for the Yellow River Delta, Binzhou University, Binzhou, Shandong 256600, China; 2. Institute of Horticultural Biotechnology, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 3. Institut de la Recherche Interdisciplinaire de Toulouse, IRIT-ARI, Toulouse 31300, France
Abstract CRISPR genome editing technology has developed rapidly and provided technical support for the functional genes research and genetic improvement. High efficiency of genome editing is critical for the application of the technology, and important progresses have been made recently. The present review summarized historic events in the improvement of the technology especially of the editing efficiency in relation to the functional mechanism and structure of the CRISPR/Cas system, and proposed several technological hints to the improvement of the editing efficiency.
Keywords CRISPR/Cas; genome editing; genome editing efficiency
DOI10.3969/j.issn.1000-2561.2019.10.013
基因工程,特別是转基因和基因编辑技术,对人类的福祉和安全带来机遇和挑战,坚守生物安全底线早已经是国际社会的共识[1],而生物安全的前提是基因工程技术的精准、高效和可控。目前基因测序技术的迅猛发展使很多物种的全基因组序列得到展现。为揭示各基因的功能,对基因序列进行精准而有效的编辑是实验分子生物学直接和有效的方法。基因编辑(gene editing/ genome editing),不仅有利于基因功能的研究,而且对植物的遗传育种具有重大意义。本文主要通过对CRISPR/Cas系统的技术原理、晶体结构等进行分析,同时综合前人对编辑效率提高的研究,以期为提高基因编辑效率提供参考。
1 CRISPR/Cas技术的发现
现有的基因编辑系统主要包括锌指核酸酶(zinc finger nucleases, ZFNs)系统、类转录激活因子效应物核酸酶(transcription activator-like effector nucleases, TALENs)系统以及CRISPR/Cas(clustered regularly interspaced short palindromic repeat-associated protein)系统[2-3]。ZFN和TALEN技术均依赖蛋白质对DNA序列的特异性识别,载体组装的复杂性是它们在基因编辑中应用的主要障碍。2013年,CRISPR/Cas[成簇的、规律间隔的短回文重复序列及相关蛋白(clustered regularly interspaced short palindromic repeats)/associated protein]技术作为第三代基因组编辑技术迅速发展起来[4-5]。CRIPSR/Cas体系普遍存在于细菌中,是细菌的一种适应性免疫体系,可使细菌高效辨认和切割入侵的外源核酸。CRISPR/Cas体系可将外源DNA片断捕获并将其插入到细菌基因组CRISPR-array中,转录产生向导CRISPR/RNA(crRNA)后,进而引导Cas核酸内切酶切割入侵外源核酸。CRISPR/Cas系统包含3种类型:I型,II型和III型;I型和III型系统较为复杂,需要多个Cas蛋白形成复合物开展切割;而II型系统仅需要一个Cas蛋白即可对外源核酸进行更高效、特异的切割,成为基因编辑工具的最佳选择[6]。
随着研究的深入,CRISPR/Cas技术编辑效率(脱靶效应、蛋白切割效率以及递送方法)的问题亟待解决。
2 CRISPR/Cas系统的技术原理
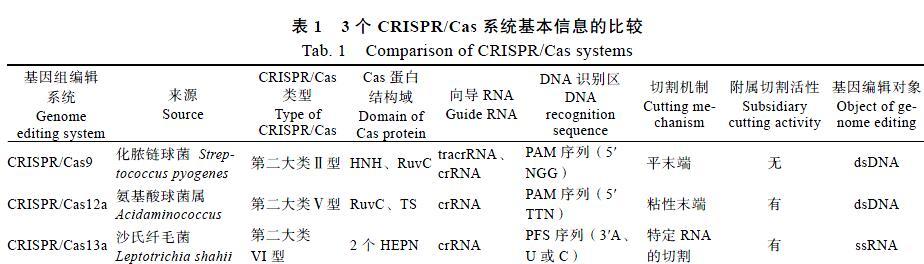
目前广泛应用的Cas9蛋白属于基因编辑的第二类系统[7],Cas12a(Cpf1),Cas13a(C2c2)等其他类型的Cas蛋白也相继被发现[8-9],进一步丰富了CRISPR/Cas系统,其中几个基因编辑系统的基础信息见表1。在多样性自然进化系统中这种固有的可编程性的存在,使CRISPR/Cas系统的应用扩展到了精确的基因组编辑领域[10-11]。
CRISPR/Cas9系统中Cas9核酸酶发挥作用,引导RNA(sgRNA)需要crRNA和tracrRNA形成复合体才能引导Cas9蛋白识别靶位点,然后蛋白的2个核酸内切酶结构域分别对DNA的2条单链进行特异性切割[12];Cas9核酸内切酶对靶位点的识别切割依赖靶序列3′端的PAM序列。不同的Cas9核酸内切酶对应的PAM序列亦不同,例如:spCas9核酸内切酶PAM序列为“NGG” [12],而saCas9核酸内切酶PAM序列为(NNGRRT)[13]。
CRISPR/Cas12a系统发挥切割作用的核酸内切酶Cas12a(Cpf1)与Cas9蛋白相比,具备以下特点:(1)CRISPR/Cas12a系统的引导RNA不需要tracrRNA,只需要crRNA即可發挥作用,Cas12a-crRNA复合体较小,进入细胞或组织更加容易;(2)核酸内切酶切割位置距离DNA靶点较远,在编辑位点上为基因组DNA编辑提供了更多选择;(3)与Cas9切割双链DNA产生的缺口是平末端,Cas12a切割产生的缺口是黏性末端,此末端更易于DNA的插入[6]。
CRISPR/Cas13a系统发挥切割作用的核酸内切酶为Cas13a(C2c2)蛋白,包含2个高等真核生物和原核生物核苷酸结合结构域,具有RNA介导的RNA酶切割活性,可以切割单链RNA(single-stranded RNA,ssRNA)[14]。相比Cas9和Cas12a,Cas13a核酸内切酶使CRISPR系统在基因编辑领域的应用领域更为广阔。
3 CRISPR/Cas系统的晶体结构
蛋白质的一级结构决定高级结构,高级结构决定生物功能,因此要想充分研究CRISPR/Cas系统的功能以及存在的问题,就需要对其结构有更清楚的了解,解析CRISPR/Cas系统的晶体结构以期找到限制CRISPR/Cas系统发展的关键因素。
3.1Cas9蛋白的晶体结构
科研工作者目前已对SpCas9蛋白的晶体结构进行解析,研究发现SpCas9蛋白由1368个氨基酸残基构成,包括REC-lobe和NUC-lobe 2个叶片状(lobe)结构(图1A)分别负责靶DNA的识别以及剪切工作;REC-lobe包括REC1、REC2以及1个长的α螺旋区域3个结构域,NUC-lobe包括RuvC、HNH以及PI(PAM interaction,PI)3个结构域;另外,RuvC结构域可与PI结构域相互作用,形成一个可与sgRNA发生相互作用的带正电荷的表面[15-16]。REC-lobe和NUC-lobe间可形成带正电荷的凹槽,在此凹槽内,SpCas9、sgRNA和靶DNA3者可以相互作用,完成对双链DNA的切割;在相互作用的过程中,首先REC-lobe与sgRNA结合,2者协同作用在靶DNA上寻找PAM序列;然后,sgRNA与靶DNA上的碱基互补配对,REC-lobe完成了对靶DNA的识别;接着,NUC-lobe中的RuvC和HNH结构域分别对靶DNA的2条链进行切割,完成NUC-lobe对靶DNA的剪切功能(图1B)。SpCas9蛋白单独存在是无活性的,当其与sgRNA结合后,SpCas9蛋白的构象会发生巨大的变化,处于活性状态,完成对靶DNA的切割[15-16]。
3.2 Cas12a(Cpf1)蛋白的晶体结构
黄志伟团队最先解析了LbCpf1系统的晶体结构[17]。LbCpf1蛋白结构呈三角形结构,中间是一个带有正电荷的凹槽。在未与crRNA结合时LbCpf1蛋白处于松散的构象,结合后crRNA发卡结构与LbCpf1蛋白紧密结合,LbCpf1-crRNA的复合体由N端螺旋结构域、C端RuvC结构域及中央的寡核苷酸结合结构域(oligonucleo tide- binding domain,OBD)3部分组成;OBD中的一个凸起的环状螺旋结构对LbCpf1蛋白与底物的结合起决定性作用;crRNA的3?末端与靶DNA的配对同样位于三角结构的凹槽内,仅crRNA的重复序列部分就可以引起LbCpf1蛋白构象的巨大改变,不需要tracrRNA的参与(图2)[17]。
3.3 Cas13a(C2c2)蛋白的晶体结构
Cas13a(C2c2)蛋白属于第二类VI型CRISPR/Cas系统的核酸内切酶,对LshC2c2晶体结构的解析显示,该蛋白同样包括REC-lobe和NUC-lobe 2个叶片状(lobe)结构分别负责靶DNA的识别以及靶序列的剪切工作;REC-lobe由N端结构域(N-terminal domain,NTD)和Helical-1结构域构成,NUC-lobe由Helical-2结构域及2个HEPN结构域构成;与SpCas9和LbCpf1蛋白不同的是REC-lobe中的NTD结构域高度保守,NTD结构域与Helical-1结构域间包含有催化pre-crRNA成熟的位点;crRNA与LshC2c2蛋白结合会使LshC2c2蛋白构象发生巨大改变(图3)[18-19]。
4 基因编辑效率存在问题及研究进展
尽管以CRISPR/Cas为工具的基因编辑技术发展迅速,与其相关的技术还有很多问题亟待解决,如脱靶效应、基因编辑效率不够高等问题。针对这些问题,人们一直在寻找新的解决方法,例如开发更精确的脱靶效应检测方法、修饰Cas蛋白或sgRNA从而提高复合物的稳定性,以及尝试新的递送方法等。
4.1脱靶与基因编辑
在CRISPR/Cas系统进行定点基因编辑过程中,间隔区序列与目标基因序列進行碱基互补配对时,对目标DNA序列的匹配具有一定程度的容忍度,允许个别碱基的错配,这一特点导致基因组中与目标DNA序列有较少碱基差别的非目
标DNA也会被误切,基因编辑中这种现象称为脱靶现象,脱靶现象的存在很大程度上阻碍了CRISPR/Cas系统在生产实践中的应用[20]。
CRISPR/Cas9脱靶现象首先在人类细胞中被验证[21]。Keith团队发现CRISPR/Cas9系统在人类细胞中的脱靶切割频率较高,甚至存在5个碱基差别的非目标基因仍可被切割,并因此导致突变[21]。在整个目标基因的识别过程中,sgRNA和PAM发挥非常重要的作用,脱靶的产生也很大程度上源自对目标基因识别准确性的降低;除sgRNA和PAM的影响之外[22],Cas9蛋白的构象以及切割方式也影响基因编辑系统的精确度[23]。表2列举了目前在sgRNA的设计、PAM序列设计、CRISPR系统的优化(Cas9的构象、Cas9-sgRNA的用量等)等3个方面在降低脱靶效应方面的研究。
4.2 Cas蛋白切割效率
基因组编辑效率(efficiency of genome editing)指转化事件中目的基因被成功编辑(发生插入、缺失或替换等)的百分数[47]。基因编辑效率的提高对于CRISPR/Cas9基因编辑技术的开发应用具有重要作用。目前科研工作者主要通过CRISPR/Cas9基因编辑系统的内部序列优化、基因编辑递送系统的改善以及基因编辑修复策略的改变等方面的来研究提高编辑效率。
4.2.1 优化CRISPR/Cas9基因编辑序列
(1)密码子优化。根据研究对象进行密码子优化可以有效提高基因编辑效率。Xing等[48]研究发现经过密码子优化后的Cas9基因对玉米基因编辑效率有重要影响,经过玉米自身密码子优化后的Cas9比经过人类密码子优化的Cas9获得的基因编辑效率更高。
(2)启动子选择。研究表明使用不同的启动子启动sgRNA转录对基因编辑效率有影响。Xing等[48]构建了分别含AtU6-26启动子、OsU3启动子和TaU3启动子的CRISPR/Cas9载体启动sgRNA转录,对目标基因ZmHKT1的基因编辑效率分别是14.5%、26.3%和33.8%;因此在玉米基因编辑系统构建时TaU3启动子启动sgRNA转录是比较高效的选择[48]。不同启动子启动Cas9核酸内切酶的表达也对编辑效率有影响;2015年,Svitashev等构建了组成型启动子玉米泛素启动子(maize ubiquitin promoter,UBI启动子)、温度调控型玉米甘露醇脱氢酶基因启动子(maize mannitol dehydrogenase gene promotor,MDH启动子)2种不同启动子控制经过玉米密码子优化后的Cas9蛋白表达的CRISPR/Cas编辑系统,研究不同载体系统对玉米无叶舌基因LIG1(Liguleless1)的基因编辑效率,结果表明,组成型UBI启动子的基因编辑效率比诱导型MDH启动子的基因编辑效率更高[49]。而Feng等利用玉米减数分裂特异基因dmc1的启动子启动Cas9蛋白,其转基因阳性愈伤中的突变效率高达100%,并且无脱靶效应,实现了CRISPR/Cas系统高效的基因组编辑[50]。
(3)靶序列及sgRNA序列优化。研究表明同一目标基因内不同位点的基因编辑效率也是不同的。研究人员对人EMX1基因设计不同位点进行CRISPR/Cas9系统基因编辑脱靶效率的研究结果表明,PAM远端(5′端)序列的单碱基差异的脱靶率要高于PAM近端(3′端)的8~12个核心序列的单碱基差异;尤其是核心序列中若存在2~3个碱基的差异,脱靶效率大大降低,若存在5个碱基及以上的差异则几乎无脱靶现象产生[46]。研究还发现3′端靠近PAM位点第20位、16位、7位、1位均存在碱基偏好性[51-52]。Farboud等发现sgRNA的3′端若为2个G切割效率最高[53]。刘小乐团队分析发现sgRNA的3′端的3个序列为CGG时切割效率较高,而靠近PAM的3′端的4位序列若是T,则切割效率会降低[52]。
4.2.2 改善递送系统 CRISPR/Cas基因编辑系统需要一定的递送系统才能进入生物体内发挥编辑功能,如何高效地将编辑系统送入生物体内直接影响到基因编辑的效率。目前递送系统主要包括传统的递送系统如生物学介导的递送(最常用的为细菌/病毒介导的传递系统)、非生物介质递送、粒子轰击递送、电脉冲及电磁辐射递送等方法,以及近来使用的纳米技术递送系统[54]。传统的细菌/病毒介导的传递系统是植物遗传转化最常用的工具,但由于宿主范围的限制,仅适用于部分植物物种或组织,同时还涉及将外源基因整合到宿主基因组中;粒子轰击递送方法可以通过基因枪或外力将生物分子递送到多种植物种或组织的细胞中,但是此种递送系统应用局限性强,且经常由于外力操作而导致组织损伤[54]。
纳米材料目前已经广泛用于医学领域中,作为靶向给药、癌症治疗和遗传性疾病治疗的递送载体。在植物中,一些研究已经使用纳米材料将质粒DNA、dsRNA、siRNA、蛋白质和植物激素递送到原生质体或细胞中[54]。目前很多研究已经开发了基于不同材料的纳米传输载体,主要包括MSN、CNT、LDH、DNA纳米结构和磁性纳米颗粒[55-63]。在对纳米传输载体递送外源材料进入植物细胞内的研究方面取得了显著性进展,如功能化的CNT将质粒DNA或siRNA形式的外源性功能基因传递到细胞中,从而导致外源基因的强烈表达或高效的基因沉默,并且CNT还可以在一段时间内保护多核苷酸免于核酸酶降解[58-59];另外CNT在无外部轰击或化学助剂的情况下可以选择性地将质粒DNA递送到叶绿体中[57]。LDH纳米片可以促进dsRNA向模式植物烟草细胞中传送[61]。磁性纳米颗粒可在有存在磁场的情况下通过花粉孔将外源DNA递送进花粉中[62]。对于难以使用常规方法进行遗传转化的植物来说,纳米技术递送系统无疑是有效的。这些开创性研究在开发基于纳米材料的运输系统方面取得了重要进展,为植物生物技术的未来应用铺平了道路。
与以往的转基因递送方法相比,基于纳米材料的递送系统为植物基因编辑提供了多种有利条件。首先,它同其他物化方法一样克服了宿主范围的限制,能适用于广泛的植物物种和不同的组织,特别是那些难以进行组织培养获得再生植株的植物可以通过原位递送进行转导;其次,在根据纳米材料的特性进行选择和实现高效递送的同时,还可将不同的生物分子同时运送到目标位置进行递送。该方法应用到基因组编辑领域,可通过进一步研究避免外源载体片段进入植物细胞,增加公众对于基因编辑的接受度[54]。图4展示了纳米材料介导的植物基因工程的过程。
4.2.3 改善DSB修复策略 Cas9核酸内切酶切割目标DNA序列可引起DNA双链断裂(double strand break,DSB)。DSB可由2种内源性的修复机制修复,非同源末端连接(non-homologous end joining,NHEJ)和同源重组介导的修复(homology-directed repair,HDR),从而实现基因的定向敲除、敲入等编辑过程[64]。因NHEJ修复易于出错,目前大部分基因编辑研究倾向于利用HDR修复DSB的策略。由于NHEJ修复途径
也属于细胞内源性修复机制,会与HDR修复进行竞争,因此若能抑制NHEJ的效率从而提高HD的修复效率,则会提高基因编辑的效率。目前已有一些方法经研究证明对NHEJ修复过程有抑制作用,如使用Scr7这种DNA连接酶IV抑制剂抑制NHEJ活性[65],适当改造基因编辑中的供体质粒[66],设计非同源依赖的靶向整合(homology-in- dependent targeted integration,HITI)方法[67]等,這些方法均可有效提升基因编辑的效率。
5 提高基因编辑效率的其他几个方面
随着人们操控基因组技术的进步,有3项基因组技术在生物学领域的研究中起着举足轻重的作用:高通量基因组测序技术、CRISPR基因编辑技术和单细胞基因组学(单细胞测序)。
基因组学是阐明整个基因组的结构、结构与功能的关系以及基因之间相互作用的科学。从全基因组的整体水平而不是单个基因水平,研究生命这个具有自身组织和自装配特性的复杂系统,认识生命活动的规律,更接近生物的本质和全貌。高通量测序技术(high-throughput sequencing)又称下一代测序技术(next-generation sequencing technology),以能一次并行对几十万到几百万条DNA分子进行序列测定和一般读长较短等为标志。
对编辑效果,包括打靶效率(targeting efficiency)和脱靶效应的检测是评估基因组编辑实验成败的关键。随着高通量测序的普及,全基因组测序将越来越普遍(花更少的时间和金钱),因此整合高通量测序与相关计算分析,利用CRISPR/ Cas介导的基因组编辑实现全基因组水平基因的功能筛选是基因编辑效果检测评估的趋势。
前人对于提高基因编辑效率做了大量的研究,尚未从根本上解决基因编辑过程中精准有效切割的问题。要想解决这一问题需内因与外因相结合进行全面考量。从目前研究较多的CRISPR/ Cas系统序列特征(GC含量、碱基频率特征、sgRNA与靶向DNA的错配特征、切割位点对碱基序列的偏好性等)延伸至物理学、化学、生物学各方面的多种影响因素。
单细胞测序以单个细胞为单位,通过全基因组或转录组扩增,进行高通量测序,能够揭示单个细胞的基因结构和基因表达状态,反映细胞间的异质性,在肿瘤学、发育生物学、微生物学、神经科学等领域发挥重要作用,正成为生命科学研究的焦点单细胞测序技术[68],特别是单个细胞分离并逐个测序技术。单细胞测序技术发展下去一个很可能的结果,就是做到基因可控性。可控基因技术在单细胞测序基础上进行数据总结,对基因行为的结果达到可预测水平,从基因层面全面获知细胞乃至植物个体的生理变化,这一信息的反馈,成为基因编辑结果的实时评价工具,将对提高基因编辑效率发生根本性的智能指导作用。
对于植物基因编辑而言,提高CRISPR/Cas系统进入细胞的效率,影响转导递送的生物物理学层面上的障碍主要是细胞壁、细胞膜和核膜的通透性,也会涉及染色体形态结构以及核小体的结构等的问题[69]。在植物胚性细胞一般细胞壁比较薄,细胞壁结构相对松散,转导不仅因其发育潜能高而提高效率,而且因为细胞壁的松散而容易侵染递送和理化方式的递送,从而提高最终的基因编辑效率。核膜对DNA、RNA和蛋白质都有屏障作用[70-71]。充分利用细胞周期中M期核膜解聚的窗口期(分裂前期至分裂后期,prophase- telophase/M)进行递送是对核膜造成的障碍进行针对性改善的有效措施。这种方法可以与同步化的诱导相结合,使更多的细胞在转导中处于M期状态。一些胁迫因子也可以短时间改变细胞壁、细胞膜的通透性,有助于细胞周期和递送的调节[72-73]。借助纳米技术进行CRISPR/Cas系统递送方法的开发也是比较理想的一种方式,有待进一步开发研究。目前在核小体不易展开的物理死角处如何进行基因编辑尚无确切研究数据[74]。
提高CRISPR/Cas系统的效率,在生化层面上,主要以CRISPR/Cas系统作用的原理以及晶体结构为基础,研究晶体结构的热力学特征,通过对关键碱基进行改变以及利用特定的化学修饰方法,改变蛋白的构象,提高引导RNA以及Cas蛋白的活性及稳定性,进而增强引导RNA的引导特异性以及Cas蛋白对于目标序列的切割效率。在生物学层面上,以细胞生长状态和发育潜能为基础,建立高效同步的繁殖体系[75],对基因编辑效率的提高具有明确的效益。在染色质状态特征等基础上,研究DNA甲基化、组蛋白乙酰化、核小体占位、以及编辑位点是否处于超敏感位点等对编辑效率的影响[24, 76-77],使得CRISPR/Cas系统进行基因编辑时所处的细胞多数为可以行使功能的活细胞,同时Cas蛋白进行切割时处于染色质疏松状态,DNA与组蛋白解聚,以及细胞分裂周期S期到G2期[78]。
本文在对CRISPR/Cas系统的技术原理、晶体结构等进行分析的基础上,着重分析CRISPR/ Cas系统序列特征,并讨论了物理学、化学、生物学等多种因素的影响,认为基因编辑效率的提高可以通过内外因结合的方式,在基因编辑系统本身改造、基因编辑系统递送时期和递送方式选择以及影响酶活和蛋白稳定性等方面进行优化。
参考文献
[1]于 辉, 徐 涵. 基因工程与国际生物安全规范[J]. 中国国际法年刊, 1997: 71-105.
[2]Gaj T, Gersbach C A, Barbas C F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7): 397-405.
[3]李希陶, 刘耀光. 基因组编辑技术在水稻功能基因组和遗传改良中的应用[J]. 生命科学, 2016, 28(10): 1243-1249.
[4]Jinek M, East A, Cheng A,et al.RNA-programmed genome editing in human cells[J]. eLife, 2013, 2: e00471.
[5]Marraffini L A. CRISPR-Cas immunity in prokaryotes[J]. Nature, 2015, 526(7571): 55-61.
[6]單琳琳, 夏海滨. 成簇的规律间隔的短回文重复序列(CRISPR)介导的基因组编辑技术研究进展[J]. 细胞与分子免疫学杂志, 2018, 34(9): 856-862.
[7]Hsu P D, Lander E S, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278.
[8]Abudayyeh O O, Gootenberg J S, Konermann S,et al. C2c2 is a single-component programmable RNA-guided RNA-tar geting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573.
[9]Zetsche B, Gootenberg J S, Abudayyeh O O,et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771.
[10]Knott G J, Doudna J A. CRISPR-Cas guides the future of genetic engineering[J]. Science, 2018, 361(6405): 866-869.
[11]Murugan K, Babu K, Sundaresan R,et al. The revolution continues: newly discovered systems expand the CRISPR-Cas toolkit[J]. Molecular Cell, 2017, 68(1): 15-25.
[12]Nishimasu H, Shi X, Ishiguro S,et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space[J]. Science, 2018, 361(6408): 1259-1262.
[13]Nishimasu H, Cong L, Yan W X,et al. Crystal structure ofStaphylococcus aureusCas9[J]. Cell, 2015, 162(5): 1113-1126.
[14]Tambe A, East-Seletsky A, Knott G J,et al. RNA binding and HEPN-nuclease activation are decoupled in CRISPR-Cas13a[J]. Cell Reports, 2018, 24(4): 1025-1036.
[15]Nishimasu H, Ran F A, Hsu P D,et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2014, 156(5): 935-949.
[16]Jinek M, Jiang F, Taylor D W,et al. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation[J]. Science, 2014, 343 (6176): 1247997.
[17]Dong D, Ren K, Qiu X L,et al. The crystal structure of Cpf1 in complex with CRISPR RNA[J]. Nature, 2016, 532: 522-526.
[18]Liu L, Li X Y, Wang J Y,et al. Two distant catalytic sites are responsible for C2c2 RNase activities[J]. Cell, 2017, 168(1-2): 121-134.
[19]Liu L, Li X Y, Ma J,et al. The molecular architecture for RNA-guided RNA cleavage by Cas13a[J]. Cell, 2017, 170(4): 714-726.
[20]王 影, 李相敢, 邱麗娟. CRISPR/Cas9基因组定点编辑中脱靶现象的研究进展[J]. 植物学报, 2018, 53(4): 528-541.
[21] Fu Y F, Foden J A, Khayter C, et al. High-frequencyoff-target mutagenesis induced by CRISPR-Cas nucleases inhuman cells[J]. Nature Biotechnology, 2013, 31(9): 822-826.
[22] Pattanayak V, Lin S, Guilinger J P, et al. High-throughputprofiling of off-target DNA cleavage reveals RNA-programmedCas9 nuclease specificity[J]. Nature Biotechnology,2013, 31(9): 839-843.
[23] Cho S W, Kim S, Kim Y, et al. Analysis of off-target effectsof CRISPR/Cas derived RNA-guided endonucleases andnickases[J]. Genome Research, 2014, 24(1): 132-141.
[24] Wu X, Scott D A, Kriz A J, et al. Genome-wide binding ofthe CRISPR endonuclease Cas9 in mammalian cells[J]. NatureBiotechnology, 2014, 32(7): 670-676.
[25] Ren X, Yang Z, Xu J, et al. Enhanced specificity and efficiencyof the CRISPR/Cas9 system with optimized sg RNAparameters in Drosophila[J]. Cell Reports, 2014, 9(3):1151-1162.
[26] Wang T, Wei J J, Sabatini D M, et al. Genetic Screens inHuman Cells Using the CRISPR-Cas9 System[J]. Science,2014, 343(6166): 80-84.
[27] Labuhn M, Adams F F, Ng M, et al. Refined sgRNA efficacyprediction improves large- and small-scale CRISPR-Cas9applications[J]. Nucleic Acids Research, 2018, 46(3):1375-1385.
[28] Cencic R, Miura H, Malina A, et al. Protospacer adjacentmotif (PAM)-distal sequences engage CRISPR Cas9 DNAtarget cleavage[J]. PLoS One, 2014, 9(10): e109213.
[29] Fu Y, Sander J D, Reyon D, et al. Improving CRISPR-Casnuclease specificity using truncated guide RNAs[J]. NatureBiotechnology, 2014, 32(3): 279-284.
[30] Singh K, Evens H, Nair N, et al. Efficient in vivo liver-directed gene editing using CRISPR/Cas9[J]. MolecularTherapy, 2018, 26(5): 1241-1254.
[31] Kim D, Kim S, Kim S, et al. Genome-wide target specificitiesof CRISPR-Cas9 nucleases revealed by multiplex Digenome-seq[J]. Genome Research, 2016, 26(3): 406-415.
[32] Rahdar M, Mcmahon M A, Prakash T P, et al. SyntheticCRISPR RNA-Cas9-guided genome editing in humancells[J]. Proceedings of the National Academy of Sciences ofthe United States of America, 2015, 112(51): E7110-E7117.
[33] Hendel A, Bak R O, Clark J T, et al. Chemically modifiedguide RNAs enhance CRISPR-Cas genome editing in humanprimary cells[J]. Nature Biotechnology, 2015, 33(9):985-989.
[34] Cromwell C R, Sung K, Park J, et al. Incorporation ofbridged nucleic acids into CRISPR RNAs improves Cas9endonuclease specificity[J]. Nature Communications, 2018,9(1): e1448.
[35] Yin H, Song C Q, Suresh S, et al. Partial DNA-guided Cas9enables genome editing with reduced off-target activity[J].Nature Chemical Biology, 2018, 14(3): 311-316.
[36] Doench J G, Fusi N, Sullender M, et al. Optimized sgRNAdesign to maximize activity and minimize off-target effectsof CRISPR-Cas9[J]. Nature Biotechnology, 2016, 34,184-191.
[37] Meng X B, Hu X X, Liu Q, et al. Robust genome editing ofCRISPR-Cas9 at NAG PAMs in rice[J]. Science China: LifeSciences, 2018, 61(1): 122-125.
[38] Müller M, Lee C M, Gasiunas G, et al. Streptococcus thermophilesCRISPR-Cas9 systems enable specific editing ofthe human genome[J]. Molecular Therapy, 2016, 24(3):636-644.
[39] Jinek M, Chylinski K, Fonfara I, et al. A programmabledual-RNA-guided DNA endonuclease in adaptive bacterialimmunity[J]. Science, 2012, 337(6096): 816-821.
[40] Dianov G L, Hubscher U. Mammalian base excision repair:the forgotten archangel[J]. Nucleic Acids Research, 2013,41(6): 3483-3490.
[41] Wyvekens N, Topkar V V, Khayter C, et al. DimericCRISPR RNA-guided FokI-dCas9 nucleases directed bytruncated gRNAs for highly specific genome editing[J]. HumanGene Therapy, 2015, 26(7): 425-431.
[42] Slaymaker I M, Gao L, Zetsche B, et al. Rationally engineeredCas9 nucleases with improved specificity[J]. Science,2016, 351(6268): 84-88.
[43] Kleinstiver B P, Pattanayak V, Prew M S, et al. High-fidelityCRISPR-Cas9 nucleases with no detectable genome-wideoff-target effects[J]. Nature, 2016, 529(7587): 490-495.
[44] Chen J S, Dagdas Y S, Kleinstiver B P, et al. Enhancedproofreading governs CRISPR-Cas9 targeting accuracy[J].Nature, 2017, 550(7676): 407-410.
[45] Shalem O, Sanjana N E, Hartenian E, et al. Genome-scaleCRISPR-Cas9 knockout screening in human cells[J]. Science,2014, 343(6166): 84-87
[46] Hsu P D, Scott D A, Weinstein J A, et al. DNA targetingspecificity of RNA-guided Cas9 nucleases[J]. Nature Biotechnology,2013, 31(9): 827-832.
[47] Zhu C, Bortesi L, Baysal C, et al. Characteristics of genomeediting mutations in cereal crops[J]. Trends in Plant Science,2017, 22(1): 38-52.
[48] Xing H L, Dong L, Wang Z P, et al. A CRISPR/Cas9 toolkitfor multiplex genome editing in plants[J]. BMC Plant Biology,2014, 14: 327.
[49] Svitashev S, Young J K, Schwartz C, et al. Targeted mutagenesis,precise gene editing, and site-specific sene- insertionin maize using Cas9 and guide RNA[J]. Plant Physiology,2015, 169(2): 931-945.
[50] Feng C, Su H, Bai H, et al. High-efficiency genome editingusing a dmc1 promoter-controlled CRISPR/Cas9 system inmaize[J]. Plant Biotechnology Journal, 2018, 16(11):1848-1857.
[51] Doench J G, Hartenian E, Graham D B, et al. Rational designof highly active sgRNAs for CRISPR-Cas9–mediatedgene inactivation[J]. Nature Biotechnology, 2014, 32(12):1262-1267.
[52] Xu H, Xiao T F, Chen C H, et al. Sequence determinants ofimproved CRISPR sgRNA design[J]. Genome Research,2015, 25(8): 1147-1157.
[53] Farboud B, Meyer B J. Dramatic enhancement of genomeediting by CRISPR/Cas9 through improved guide RNA design[J]. Genetics, 2015, 199(4): 959-971.
[54] Wang P, Zhao F J, Kopittke P M. Engineering Crops withoutGenome Integration Using Nanotechnology[J]. Trends inPlant Science, 2019, 24(7): 574-577.
[55] Martin-Ortigosa S, Peterson D J, Valenstein J S, et al. Mesoporoussilica nanoparticle-mediated intracellular Cre proteindelivery for maize genome editing via loxP site excision[J]. Plant Physiology, 2014, 164(2): 537-547.
[56] Torney F, Trewyn B G, Lin V S Y, et al. Mesoporous silicananoparticles deliver DNA and chemicals into plants[J]. NatureNanotechnology, 2007, 2(5): 295-300.
[57] Kwak S-Y, Lew T T S, Sweeney C J, et al. Chloroplast-selective gene delivery and expression in planta usingchitosan-complexed single-walled carbon nanotube carriers[J]. Nature Nanotechnology, 2019, 14(5): 447-455.
[58] Demirer G S, Zhang H, Matos J L, et al. High aspect rationanomaterials enable delivery of functional genetic materialwithout DNA integration in mature plants[J]. Nature Nanotechnology,2019, 14(5): 456-464.
[59] Yang C Y, Chen S H, Wang J H, et al. A facile electrospinningmethod to fabricate polylactide/graphene/MWCNTsnanofiber membrane for tissues scaffold[J]. Applied SurfaceScience, 2016, 362: 163-168
[60] Zhang H, Demirer G S, Zhang H L, et al. DNA nanostructurescoordinate gene silencing in mature plants[J]. Proceedingsof the National Academy of Sciences of the UnitedStates of America, 2019, 116(15): 7543-7548.
[61] Mitter N, Worrall E A, Robinson K E, et al. Clay nanosheetsfor topical delivery of RNAi for sustained protection againstplant viruses[J]. Nature Plants, 2017, 3(2): 16207.
[62] Zhao X, Meng Z G, Wang Y, et al. Pollen magnetofectionfor genetic modification with magnetic nanoparticles as genecarriers[J]. Nature Plants, 2017, 3(12): 956-964.
[63] Bao W L, Wan Y L, Balu?ka F. Nanosheets for delivery ofbiomolecules into plant cells[J]. Trends in Plant Science,2017, 22(6): 445-447.
[64] Ding Y, Li H, Chen L L, et al. Recent advances in genomeediting using CRISPR/Cas9[J]. Frontiers of Plant Science,2016, 7: 703.
[65] Ma Y, Zhang J, Yin W, et al. Targeted AID-mediated mutagenesis(TAM) enables efficient genomic diversification inmammalian cells[J]. Nature Methods, 2016, 13(12):1029-1035.
[66] He X, Tan C, Wang F, et al. Knock-in of large reporter genesin human cells via CRISPR/Cas9-induced homologydependentand independent DNA repair[J]. Nucleic AcidsResearch, 2016, 44(9): e85.
[67] Suzuki K, Tsunekawa Y, Hernandez-Benitez R, et al. In vivogenome editing via CRISPR/Cas9 mediated homology independenttargeted integration[J]. Nature, 2016, 540(7631):144-149.
[68] 朱忠旭, 陳 新. 单细胞测序技术及应用进展[J]. 基因组学与应用生物学, 2015, 34(5): 902-908.
[69] Ou H D, Sébastien P, Deerinck T J, et al. ChromEMT:Visualizing 3D chromatin structure and compaction in interphaseand mitotic cells[J]. Science, 2017, 357(6349):eaag0025.
[70] Foisner R. Cell Cycle Dynamics of the Nuclear Envelope[J].The Scientific World journal, 2003, 3: 1-20.
[71] Evans D E, Shvedunova M, Graumann K. The nuclearenvelope in the plant cell cycle: structure, function and regulation[J]. Annals of Botany, 2011, 107(7): 1111-1118.
[72] Xu K D, Chang Y X, Liu K, et al. Regeneration of Solanumnigrum by somatic embryogenesis, involving frog egg-likebody, a novel structure[J]. PLoS One, 2014, 9(6): e98672.
[73] Xu K D, Chang Y X, Zhang Y, et al. Rorippa indica regenerationvia somatic embryogenesis involving frog egg-likebodies efficiently induced by the synergy of salt and droughtstresses[J]. Scientific Reports, 2016, 6: 19811.
[74] Chen Y, Zeng S, Hu R, et al. Using local chromatin structureto improve CRISPR/Cas9 efficiency in zebrafish[J]. PLoSOne , 2017, 12(8): e0182528.
[75] XuHan X, Jing H C, Cheng X F, et al. Polyploidization inembryogenic microspore cultures of Brassica napus L. cv.Topas enables the generation of doubled haploid clones bysomatic embryogenesis[J]. Protoplasma, 1999, 208: 240-247.
[76] 李 干, 王国栋, 怀 聪, 等. SpCas9 结构域相互作用关键氨基酸的动态网络分析[J]. 计算生物学, 2016, 6(3):50-61.
[77] Kim H K, Min S, Song M, et al. Deep learning improvesprediction of CRISPR-Cpf1 guide RNA activity[J]. NatureBiotechnology, 2018, 36(3): 239-241.
[78] Heyer W D, Ehmsen K T, Liu J. Regulation of homologousrecombination in eukaryotes[J]. Annual Review of Genetics,2010, 44: 113-139.
