菠萝R2R3-MYB基因家族鉴定与表达分析
2019-12-14陈哲胡福初阮城城范鸿雁郭利军张治礼
陈哲 胡福初 阮城城 范鸿雁 郭利军 张治礼

摘 要 R2R3-MYB轉录因子参与植物众多生命活动的调控,在植物生长发育中至关重要。本研究采用生物信息学分析方法,从菠萝基因组中筛选鉴定R2R3-AcMYB转录因子,并对其基因结构、编码蛋白和系统进化进行了分析;基于转录组数据和荧光定量PCR分析,研究了乙烯利处理后菠萝顶端分生组织MYB基因的表达模式。研究结果显示,菠萝基因组含有103个R2R3-AcMYB转录因子,其中67个的基因结构组成为3(外显子)+2(内含子),17个为2(外显子)+1(内含子)。103个R2R3-AcMYB蛋白中,有31个偏碱性,55个显酸性和17个呈中性。亚细胞定位预测显示,有67个编码蛋白定位于细胞质,20个定位于细胞核。保守基序分析发现,有91个R2R3-AcMYB的序列包含SANT结构的motif 1和motif 2。系统进化树分析表明,81个R2R3-AcMYB转录因子可以分别被归入18个亚组,其余22个R2R3-AcMYB转录因子则未能进行明确归类。基于转录组数据和荧光定量PCR分析结果,发现菠萝顶端分生组织中多个MYB基因的表达受到乙烯利的诱导或抑制,暗示这些基因可能参与了乙烯利诱导条件下菠萝生长发育(包括开花诱导等)调控。这些研究结果为进一步挖掘菠萝激素响应、生长发育和开花调控等基因,揭示菠萝生长发育调控机制提供了数据支持。
关键词 菠萝;R2R3-MYB;生物信息学;乙烯利;基因表达中图分类号 S668.3 文献标识码 A
Bioinformatics and Gene Expression Analysis of Pineapple R2R3-MYB Gene Family
CHEN Zhe, HU Fuchu, RUAN Chengcheng, FAN Hongyan, GUO Lijun, ZHANG Zhili*
Institute of Tropical Fruit Trees, Hainan Academy of Agricultural Sciences / Key Laboratory of Tropical Fruit Tree Biology of Hainan Province / Haikou Investigation Station of Tropical Fruit Trees, Ministry of Agriculture and Rural Affairs, Haikou, Hainan 571100, China
Abstract R2R3-MYB transcription factors are very important in plant growth and development, ich are involved in the regulation of many life activities of plants. In this study, the R2R3-AcMYB transcription factors were screened and identified from the pineapple genome by bioinformatics analysis. Based on the transcriptome data and fluorescence-quantitative PCR analysis, the expression patterns of MYB genes in the apical meristem of pineapple treated with ethylene were studied. The results showed that there were 103 R2R3-AcMYB transcription factors in the pineapple genome, of which 67 gene were composed of 3 (exons) +2 (introns) and 17 were 2 (exons) +1 (introns). Of the 103 R2R3-AcMYB proteins, 31 were alkaline, 55 were acidic and 17 were neutral, and the main secondary structure in 83 proteins was random curl. Subcellular localization predictions showed that 67 proteins were located in the cytoplasm and 20 in the nucleus. Conservative motif analysis found that 91 of the R2R3-AcMYB sequences contained motif 1 and motif 2 of the SANT structure. Phylogenetic analysis showed that 81 R2R3-AcMYB transcription factors could be classified into 18 subgroups, and the remaining 22 R2R3-AcMYB transcription factors could not be clearly classified. Based on transcriptome data and fluorescence-quantitative PCR analysis, it was found that the expression of several MYB genes in the apical meristem of pineapple was induced or inhibited by ethrel, suggesting that the genes may be involved in the responses of pineapple growth and development including the induction of flowering to ethrel. The results would provide data supports for further excavation of genes involved in hormone response, growth and development, and flowering regulation, which would contribute to revealing the regulation mechanism of pineapple growth and development.
Keywords pineapple; R2R3-MYB; bioinformatics; ethrel; gene expression
DOI10.3969/j.issn.1000-2561.2019.10.008
MYB类转录因子家族是指含有MYB结构域的一类转录因子,是真核生物中存在的最大的一类转录因子家族。典型的MYB类转录因子一般由DNA结合结构域、1个转录激活结构域和1个不完全界定的负调节区3个保守功能域组成[1-2]。其中,DNA结合结构域最为保守,一般由1~4个不完全重复序列(R)组成,每个重复片段R则由50~53个保守的氨基酸残基组成。根据MYB转录因子所含MYB结构域的个数将其分为1R-MYB、2R-MYB(R2R3)、3R-MYB(R1R2R3)和非典型MYB(4R-MYB和5R-MYB)[3-4]。其中,动物中以3R-MYB为主要群体,植物中则是2R-MYB居多[3, 5-6]。
MYB转录因子家族庞大的数目预示着其对植物生长发育过程调控的重要性。目前的研究表明,R1/R2-MYB主要参与植物生物钟调节[7-8]和DNA结合蛋白调控[9],R2R3-MYB转录因子则参与植物苯丙烷代谢途径[10]、生物和非生物胁迫响应[11]、细胞形态建成与分化[12]、激素响应[13]和植物防御机制反应[14]等生命过程,说明数目较为庞大的R2R3-MYB转录因子在植物生长发育过程中扮演者着更为重要的角色。随着基因组测序和转录组测序等技术在植物研究中的应用和推广,科研人员陆续报道了大量植物的R2R3-MYB转录因子,包括拟南芥(Arabidopsis thaliana)(125个)[8]、葡萄(Vitis)(117个)[15]、杨树(Populus)(192个)[16]和柑橘(Citrus)(103个)[17]等。Dubos等[3]还将拟南芥R2R3-MYB转录因子细分为23个亚族。然而,菠萝作为我国最重要的热带水果之一,关于MYB转录因子家族的研究尚未见报道,关于菠萝R2R3-MYB转录因子的研究也未见报道。本研究以福建农林大学公布的菠萝基因组数据为资源平台[18],利用生物信息学相关手段,在菠萝基因组范围内筛选、鉴定菠萝R2R3-MYB转录因子,分析其編码蛋白特性、编码序列特征和分类信息,以期为菠萝R2R3-MYB转录因子的克隆表达和功能验证等研究奠定基础。
1 材料与方法
1.1材料
菠萝植株样品采自海南省澄迈县海南厚德农业开发有限公司海南缘聚兴中阳专业合作社,常规栽培管理。2017年1月进行40%浓度乙烯利灌心处理,处理后1 h、3 h、6 h、1 d、6 d、31 d、34 d、40 d、45 d、50 d、63 d等分别采样,包括根、茎、茎尖分生组织、成熟叶、果实等5个组织部位,所有的样品迅速用液氮冷冻,放入–80 ℃超低温冰箱保真备用。
1.2 方法
1.2.1 菠萝MYB转录因子获取 从Phytoz ome11.0数据库获取被注释为MYB转录因子的基因组DNA序列、CDS序列和蛋白序列,利用NCBI的Conserved Domain Search分析所有MYB Unigene蛋白序列的结构域,去除不具有MYB保守结构域或者不具有完整MYB保守结构域的序列,根据MYB转录因子所含MYB结构域个数筛选获得R2R3-MYB转录因子,并对其进行进一步分析。
1.2.2 蛋白信息分析 菠萝R2R3-MYB蛋序列的分子量、等电点和氨基酸数目等参数均通过Prot Param[19]在线分析工具获取。通过在线分析程序https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl/page=npsa_sopma.html对菠萝R2R3-MYB蛋白进行二级结构分析[20],利用PSORT Prediction在线分析软件预测亚细胞定位。
1.2.3 基因结构分析 通过GSDS在线工具分析菠萝R2R3-MYB转录因子家族的内含子和外显子组成情况[21]。
1.2.4 保守基序分析 利用在线软件MEME(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)获取菠萝R2R3-MYB蛋白的保守基序,查找的保守基序最大数目为5,查找片段大小为10~70个氨基酸之间[22]。通过SMART(http://smart.embl- heidelberg.de/)在线分析软件对查找获得的保守基序进行功能注释。
1.2.5 系统进化分析与分类 参照Dubos等[3]的分类方式,利用125个拟南芥R2R3-MYB蛋白序列和筛选鉴定出的103个菠萝R2R3-MYB蛋白序列构建的系统进化树获取分类信息。系统进化树构建在MEGA6.0上进行,采用NJ法则(neighbor-joining method)的P-距离(P-distance)模型,选择部分删除(pairwise deletion)空位的选项,Bootstrap method取值1000。
1.2.6 转录组测序 使用Invitrogen公司的TRIzol Reagent RNA提取试剂盒提取总RNA,通过核酸蛋白仪检测RNA纯度(OD260/280比值)和浓度后,用Agilent2100(Stratagene, La Jolla, CA, USA) 精确检测RNA的完整性。选取从对照、乙烯利处理1 h、3 h、1 d菠萝茎尖样品提取的RNA送北京诺禾致源科技股份有限公司进行转录组测序,后续分析基于此测序结果数据。
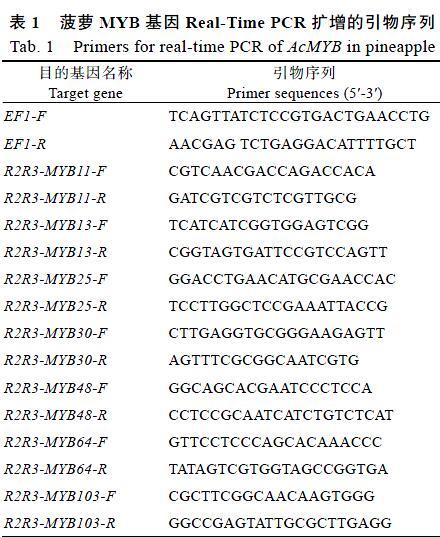
1.2.7 基于菠萝转录组MYB类转录因子的表达模式分析与验证 转录组数据采用FPKM(Fragments Per Kilobase of exon model per Million mapped reads)来衡量基因的表达水平。采用Tbtools软件对103个MYB 转录因子在乙烯灌心处理后3个时期的表达数据进行分层聚类分析。以总RNA反转录后的cDNA为模板,EF1为内参基因,选取7个MYB转录因子进行实时荧光定量PCR分析(表1)。该反应在LightCycler480实时荧光定量PCR仪上进行。实时定量PCR的反应体系为10 μL,包括1 μL cDNA,5 μL SYBR Green I Master,10 μmol/L的正反向引物各1 μL,2 μL ddH2O。数据采用2–ΔΔCt法进行相对定量分析,利用Excel、GraphPad Prism 5.0等软件进行统计学分析。
2 结果与分析
2.1菠蘿R2R3-MYB转录因子家族的鉴定和分类
Phytozome11.0数据库中菠萝基因组信息的检索结果发现,共有297个菠萝MYB转录因子的Unigene序列,保守结构域分析发现有63个Unigene序列不具有MYB转录因子的保守结构域、或不具有完整的保守结构域的序列,另外
Aco013173和Aco025712则是2个完全重复的序列。去除不具有MYB保守结构域序列、不具有完整MYB保守结构域的序列和重复序列,得到126个1R-MYB转录因子、103个2R-MYB转录因子、3个3R-MYB转录因子和1个4R-MYB转录因子。本研究以103个2R-MYB转录因子作为研究对象,将其编号为R2R3-MYB1~R2R3- YB103(表2)。
2.2 菠萝R2R3-MYB转录因子蛋白基本信息分析
通过各种在线分析软件得到的菠萝R2R3- MYB转录因子蛋白基本信息分析见表2。由表2可以看出,菠萝R2R3-MYB转录因子编码蛋白的大小和氨基酸数目均差异很大,编码氨基酸的大小在200~400之间的菠萝R2R3-MYB转录因子占大多数,共85个,占比82.52%,其中最小的R2R3- MYB96由180个氨基酸组成,最大的R2R3- MYB9由1591个氨基酸组成,他们对应的菠萝R2R3-MYB蛋白分子量大小为20 411.55~ 172 545.82 ku之间。菠萝R2R3-MYB转录因子蛋白中分别有55个酸性蛋白(pH<6.5)、17个中性蛋白(6.5
菠萝R2R3-MYB蛋白质二级结构在线分析结果表明,83个菠萝R2R3-MYB蛋白的主要二级机构为无规则卷曲(random coil),占比80.58%,其他20个蛋白预测的二级结构均以α-螺旋(Alpha helix)为主。93个编码蛋白预测数量最少的二级结构为β-转角(Beta turn),占比90.29%;8个编码蛋白预测数量最少的二级结构为扩展链(extend strand)。此外,R2R3-MYB15和R2R3-MYB64编码蛋白预测数量最小的二级结构是β-转角和扩展链。
定位预测结果显示,67个菠萝R2R3-MYB蛋白预测定位于细胞质,占比65.04%。其次,20个预测定位于细胞核,占比19.42%;预测定位于微体的有R2R3-MYB13、R2R3-MYB17、R2R3-MYB58、R2R3-MYB69、R2R3-MYB70和R2R3-MYB96等7个蛋白。预测定位于叶绿体基质的有R2R3-MYB67、R2R3-MYB100和R2R3-MYB103等3个蛋白;预测定位于线绿体基质的有R2R3-MYB18和R2R3-MYB40等2个蛋白;预测定位于内质网膜的有R2R3-MYB2和R2R3-MYB71等2个蛋白。此外,R2R3-MYB1和R2R3-MYB87分别预测定位于细胞外和线绿体内膜。
2.3 菠萝R2R3-MYB蛋白保守基序分析
MEME在线软件分析得到R2R3-MYB转录因子的5个保守基序,SMART分析表明Motif 1、Motif 2和Motif 4均为SANT结构,Motif 3和Motif 5均未得到其功能结构信息。具有Motif 1的转录因子有91个,占比88.50%;具有Motif 2的转录因子有103个,占比100%;说明Motif 1和Motif 2均为菠萝R2R3-MYB转录因子的保守基序。此外,仅有28个转录因子具有Motif 3,具有Motif 4和Motif 5的转录因子均为7个(表3)。
2.4 菠萝R2R3-MYB基因结构及家族系统进化分析
GSDS分析结果表明,除了R2R3-MYB18、R2R3-MYB25、R2R3-MYB48和R2R3-MYB103 4个转录因子不具备内含子,其余99个转录因子的基因结构组成均包含外显子和内含子(图1)。其中,有67个转录因子的基因结构组成为3+2(外显子+内含子),所占比率最高,达65.04%;其次是17个转录因子的基因结构组成为2+1内含子,占比16.35%;此外还有8个转录因子的基因结构组成为4+3,3个转录因子的基因结构组成为5+4,2个转录因子的基因结构组成为12+11。而基因结构组成为9+8和14+13的转录因子均为1个,分别是R2R3-MYB20和R2R3-MYB87。外显子和内含子数量最多的转录因子都是R2R3-MYB87。
参照Dubos等[3]对拟南芥R2R3-MYB转录因子家族的分类,采用NJ法构建拟南芥和菠萝R2R3-MYB蛋白的进化树如图2。由图2可以看出,22个菠萝R2R3-MYB转录因子未归入分类小组,占比21.36%;此外,除了S3、S6、S12、S15和S19等5个亚组没有菠萝R2R3-MYB转录因子成员,其他81个菠萝R2R3-MYB转录因子分别被归入18个亚组。根据拟南芥R2R3-MYB的基因信息对其进行功能预测,其中S1、S13和S24亚组R2R3-MYB基因功能与木质素生物合成、气孔关闭以及粘液沉积有关;S2和S23亚组R2R3-MYB基因功能与非生物胁迫响应相关;S3、S4以及S11亚组R2R3-MYB基因功能与非生物胁迫响应以及苯丙醇类途径相关;S5、S9和S10亚组R2R3-MYB基因功能與原花青素类生物合成相关;S6和S18亚组R2R3-MYB基因功能与花发育、花青素、ABA敏感相关;S7和S12亚组R2R3-MYB基因功能与黄酮醇及芥子油苷生物合成有关;S22和S20亚组R2R3-MYB基因功能与逆境响应有关;S15和S25亚组R2R3-MYB基因功能与胚胎形成和根形成有关;S21亚组R2R3-MYB基因功能与腋生分生组织发育有关。
2.5基于转录组菠萝MYB转录因子基因表达模式分析
基因的表达模式与基因的功能关系密切,为了了解MYB转录因子在响应乙烯刺激诱导菠萝成花过程中可能起到调控作用,据此从菠萝茎尖转录组测序结果的基因表达数据库中提取103个R2R3-MYB家族基因在乙烯利处理后4个响应期(CK、乙烯利处理后1 h、乙烯利处理后3 h、乙烯利处理后1 d)的表达数据,采用Heml软件对这些基因的差异表达进行分层聚类分析(图3)。
根据转录组数据,我们发现103个R2R3- MYB家族基因在乙烯利处理后4个响应期表达差异显著,其中有78个R2R3-MYB家族基因在4个样品中均有表达(FPKM>0),其中有8个R2R3-MYB家族基因在4个样品中FPKM均大于100(Aco007902,Aco016984,Aco001378,Aco013641,Aco002989,Aco009589,Aco000514,Aco006386)。大部分R2R3-MYB家族基因表现出组成型表达,也有少数基因仅在乙烯利处理后的时期有较高表达,其中Aco001378在乙烯利处理3 h后有较高表达,而Aco001748、Aco002582、Aco005389以及Aco006402在乙烯利处理1 h后有较高表达。而Aco001378、Aco001748、Aco002582、Aco005389、Aco006402、Aco011071以及Aco031816属于R2R3-MYB基因S20和S22亚组,基因功能可能与逆境响应相关。同时,对以上基因进行Q-PCR验证分析发现,AcMYB11(Aco001748)、AcMYB13(Aco002582)、AcMYB25(Aco006402)、AcMYB30(Aco007733)及AcMYB48(Aco011071)均在乙烯利处理6 h后开始有明显的上调表达,部分基因在乙烯利处理1 d后到达峰值后又下降;而AcMYB11(Aco031816)则在乙烯利处理1 d后到达峰值(图4)。Q-PCR的验证结果与转录组数据相符,基于转录组数据的MYB差异表达模式和基于荧光定量PCR分析表明,部分MYB基因可能在响应乙烯刺激诱导菠萝成花过程中可能起到调控作用。
3 讨论
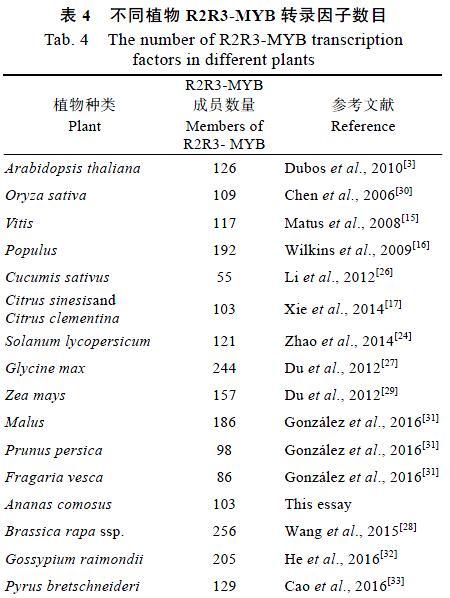
MYB转录因子作为最大的一类转录因子家族,因其对植物生长发育过程重要的调控作用,受到国内外研究学者的普遍关注,关于植物MYB转录因子的研究也屡见报道[23]。本研究利用生物信息学方法筛选鉴定出103个菠萝R2R3-MYB转录因子,这与柑橘(Citrus reticulataBlanco)(103个)和桃(Prunus persica)(98个)相当,但与前人报道的杨树(192个)、大豆(Glycine max)(244个)、拟南芥(126个)和黄瓜(Cucumis sativus)(55个)等植物中R2R3-MYB转录因子的数目相差较大。不同植物R2R3-MYB转录因子数目及其文献来源见表4。植物基因组经历的基因序列变化包括小片段的易位、插入、倒位、缺失和重复有关,还有全基因组复制事件之后的染色体重排与融合密切相关[24],这些序列变化的进程是物种多样化形成的根本原因,也是不同物种R2R3-MYB转录因子家族数目差异的根本原因。
擬南芥、水稻(Oryza sativa)和番茄(Solanum lycopersicum)等植物具有较大的R2R3-MYB转录因子家族可能是它们都经历了基因复制事件[24-25],而黄瓜没有经历基因复制事件,所以具有目前发现数目最小的R2R3-MYB转录因子家族[26]。大豆(Glycine max)和菜心(Brassica rapassp.)具有庞大的R2R3-MYB转录因子家族则可能是因为它们分别为古四倍体和三倍体有关[27-28]。
MYB转录因子可根据MYB结构域的个数将其分为1R-MYB、2R-MYB(R2R3)、3R-MYB(R1R2R3)和非典型MYB(4R-MYB和5R- MYB)[3-4]。目前针对R2R3-MYB转录因子亚组的研究较为常见,本研究参照拟南芥R2R3-MYB转录因子的分组方式,将81个菠萝R2R3-MYB转录因子和97个拟南芥R2R3-MYB转录因子分别被归入23个亚组(22个菠萝R2R3-MYB转录因子和28个拟南芥R2R3-MYB转录因子未被归于任何一个亚组)。相对于拟南芥在23个亚组均有转录因子分布,菠萝R2R3-MYB转录因子未分布于S3、S6、S12、S15和S19五个亚组。
MYB蛋白参与植物的多个生命活动。同样,S3亚组的AtMYB58和AtMYB63都参与木质素生物合成的正调控[34],S6亚组中的AtMYB75、AtMYB113和AtMYB114参与控制花色苷的生物合成[35],S12亚组成员中的AtMYB28、AtMYB76
和AtMYB29则主要参与硫代葡萄糖苷的生物合成[36-37],S15亚组中的AtMYB0、AtMYB23和AtMYB66共同决定拟南芥表皮细胞的类型[3],S16亚组中的AtMYB21、AtMYB24和AtMYB57参与花药发育的调控[38]。本研究通过转录组数据分析发现103个R2R3-MYB家族基因在乙烯利处理后4个响应期表达差异显著,部分基因仅在特殊的响应时期有高表达,另外,通过对7个候选基因Q-PCR验证分析发现,AcMYB11(Aco001748)、AcMYB13(Aco002582)、AcMYB25(Aco006402)、AcMYB30(Aco007733)及AcMYB48(Aco011071)均在乙烯利处理6 h后开始有明显的上调表达,部分基因在乙烯利处理1 d后到达峰值后又下降;而AcMYB11(Aco031816)则在乙烯利处理1 d后到达峰值。基于转录组数据的MYB差异表达模式和基于荧光定量PCR分析表明,部分MYB基因可能在响应乙烯刺激诱导菠萝成花过程中可能起到调控作用。
4 结论
本研究以公布的菠萝基因组数据为基础信息,利用生物信息学方法筛选鉴定出103个R2R3-MYB转录因子,分析掌握了103个R2R3-MYB转录因子的基因结构组成,编码蛋白的理化性质、二级结构组成、保守基序和亚细胞定位预测等信息,通过构建进化树获取了菠萝R2R3-MYB转录因子的分类情况;基于转录组数据的MYB差异表达模式和基于荧光定量PCR分析发现,部分MYB基因可能参与了乙烯利诱导菠萝成花的调控。本研究结果将为进一步挖掘菠萝激素响应、生长发育和开花调控等基因,揭示菠萝生长发育调控机制提供了数据支持。
参考文献
[1] Frampton J. Myb transcription factors: their role in growth, differentiation and disease[M]. Dordrecht: Springer Netherlands, 2004: 6-8.
[2] Thompson M A, Ramsay R G. MYB: an old oncoprotein with new roles[J]. Bioessays, 1995, 17(4): 341-350.
[3] Dubos C, Stracke R, Grotewold E,et al. Myb transcription factors inArabidopsis[J]. Trends in Plant Science, 2010, 15(10): 573-581.
[4] Katiyar A, Smita S, Lenka S K,et al. Genome-wide classification and expression analysis of MYB transcription factor families in rice andArabidopsis[J]. BMC Genomics, 2012, 13: 544.
[5] Lipsick J S. One billion years of Myb[J]. Oncogene, 1996, 13(2): 223-235.
[6] Martin C, Paz-Ares J. MYB transcription factors in plant[J].Trends Genet, 1997, 13(2): 67-73.
[7] Schaffer R, Ramsay N, Samach A, et al. The late elongatedhypocotyl mutation of Arabidopsis disrupts circadianrhythms and the photoperiodic control of flowering[J]. Cell,1998, 93(7): 1219-1229.
[8] Schaffer R, Wisman E. Microarray analysis of diurnal andcircadian-regulated genes in Arabidopsis[J]. The Plant Cell,2001, 13(1): 113-123.
[9] Yu E Y, Kim S E, Kim J H, et al. Sequence-specific DNArecognition by the MYB-like domain of plant telomeric proteinRTBP1[J]. Journal of Biological Chemistry, 2000,275(31): 24208-24214.
[10] Hichri I, Barrieu F, Bogs J, et al. Recent advances in thetranscriptional regulation of the flavonoid biosyntheticpathway[J]. Journal of Experimental Botany, 2011, 62(8):2465-2483.
[11] Segarra G, Van der E S, Trillas I, et al. MYB72, a node ofconvergence in induced systemic resistance triggered by afungal and a bacterial beneficial microbe[J]. Plant Biology,2009, 11(1): 90-96.
[12] Noda K I, Glover B J, Linstead P, et al. Flower colour intensitydepends specialized cell shape controlled by a MYB relatedtranscription factor[J]. Nature, 1994, 369(6482):661-664.
[13] Iturriaga G, Leyns L, Villeges A, et al. A family ofnovelMYB-related genes from the resurrection plant Craterostigmaplantagineum are specifically expressed in callusand roots in response to ABA or dessication[J]. Plant MolecularBiology, 1996, 32(4): 707-716.
[14] 杜 海, 唐曉凤, 刘 蕾, 等. MYB 转录因子在植物抗逆胁迫中的作用及其分子机理[J]. 遗传, 2008, 30(10):1265-1271.
[15] Matus J T, Aquea F, Arce-Johnson P. Analysis of the grapeMYB R2R3 subfamily reveals expanded wine quality-relatedclades and conserved gene structure organization across Vitisand Arabidopsis genomes[J]. BMC Plant Biology, 2008, 8:83.
[16] Wilkins O, Nahal H, Foong J, et al. Expansion and diversificationof the populus R2R3-MYB family of transcriptionfactors[J]. Plant Physiology, 2008, 149(2): 981-993.
[17] Xie R, Li Y, He S, et al. Genome-wide analysis of citrusR2R3MYB genes and their spatiotemporal expression understresses and hormone treatments[J]. PLoS One, 2014, 9(12):e113971.
[18] Ming R, Buren R V, Wai C M, et al. The pineapple genomeand the evolution of cam photosynthesis[J]. Nature Genetics,2015, 47: 1435-42.
[19] Wilkins M R, Gasteiger E, Bairoch A, et al. Protein identification and analysis tools in the ExPASy server[M]// Link A J. 2-D Proteome analysis protocols: Methods in molecular biology, vol 112, Humana Press, 1999: 531-552.
[20] Levin J, Garnier J. Improvements in a secondary structure prediction method basedon a search for local sequence homologies and its use as a model building tool[J]. Biochimica et Biophysica Acta (BBA)-Protein Structure and Molecular Enzymology. Acta, 1988, 955(3): 283-295.
[21] Hu B, Jin J, Guo A Y, et al. Gsds 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2014, 31: 1296.
[22] Bailey T L, Bodén M, Buske F A, et al. "MEME Suite: tools for motif discovery and searching"[J]. Nucleic Acids Research, 2009, 37: 202-208.
[23] Ambawat S, Sharma P, Yadav N,et al. Myb transcription factor genes as regulators for plant responses: an overview[J]. Physiology and Molecular Biology of Plants, 2013, 19: 307-321.
[24] Zhao P, Li Q, Li J, et al. Genome-wide identification and characterization of R2R3MYB family in Solanum lycopersicum[J]. Molecular Genetics and Genomics, 2014, 289: 1183-1207.
[25] Cannon SB, Mitra A, Baumgarten A,et al. The roles of segmental and tandem gene duplication in the evolution of large gene families inArabidopsis thaliana[J]. BMC Plant Biology, 2004, 4: 10.
[26] Li Q, Zhang C, Li J, et al. Genome-Wide Identification and Characterization of R2R3MYB Family inCucumis sativus[J]. PLoS One, 2012, 7(10): e47576.
[27] Du H, Yang S S, Liang Z, et al. Genome-wide analysis of the MYB transcription factor superfamily in soybean[J]. BMC Plant Biololgy, 2012, 12: 106.
[28] Wang Z, Tang J, Hu R, et al. Genome-wideanalysis of the R2R3-MYB transcription factor genes in Chinese cabbage (Brassica rapassp.pekinensis) reveals their stress and hormone responsive patterns[J]. BMC Genomics, 2015, 16: 17.
[29] Du H, Feng B R, Yang S S, et al. The R2R3-MYB transcription factor gene family in maize[J]. PLoS One, 2012, 7(6): e37463.
[30] Chen Y H, Yang X Y, He K, et al. The MYB transcriptionfactor superfamily of Arabidopsis: expression analysis andphylogenetic comparison with the rice MYB family[J]. PlantMolecular Biology, 2006, 60: 107-124.
[31] González M, Carrasco B, Salazar E. Genome-wide identificationand characterization of R2R3MYB family inRosaceae[J]. Genomics Data, 2016, 9: 50-57.
[32] He Q, Jones D C, Li W, et al. Genome-Wide Identificationof R2R3-MYB Genes and Expression Analyses DuringAbiotic Stress in Gossypium raimondii[J]. Scientific Reports,2016, 6: 22980.
[33] Cao Y, Han Y, Li D, et al. MYB Transcription Factors inChinese Pear (Pyrus bretschneideri Rehd.): Genome-WideIdentification, Classification, and Expression Profiling duringFruit Development[J]. Frontiers in Plant Science, 2016, 7:10.
[34] Zhou J , Lee C , Zhong, et al. MYB58 and MYB63 are transcriptionalactivators of the lignin biosynthetic pathway duringsecondary cell wall formation in Arabidopsis[J]. PlantCell, 2009, 21: 248-266.
[35] Gonzalez A, Zhao M, Leavitt J M, et al. Regulation of theanthocyanin biosynthetic pathway by the TTG1/bHLH/Mybtranscriptional complex in seedlings[J]. The Plant Journal,2008, 53(5): 814-827.
[36] Gigolashvili T, Yatusevich R, Berger B, et al. TheR2R3-MYB transcription factor HAG1/MYB28 is a regulatorof methionine-derived glucosinolate biosynthesis inArabidopsis thaliana[J]. Plant Journal, 2007, 51(2): 247-261.
[37] Gigolashvili T, Yatusevich R, Berger B, et al.HAG2/MYB76 and HAG3/MYB29 exert a specific and coordinatedcontrol on the regulation of aliphatic glucosinolatebiosynthesis in Arabidopsis thaliana[J]. The New Phyologist,2008, 117: 627.
[38] Cheng H, Song S, Xiao L, et al. Gibberellin acts throughjasmonate to control the expression of MYB21, MYB24, andMYB57 to promote stamen filament growth in Arabidopsis[J]. PLoS Genetics, 2009, 5: e1000440.
